Genetic analysis for six wild and selection populations of common carp (Cyprinus carpio) using microsatellites
-
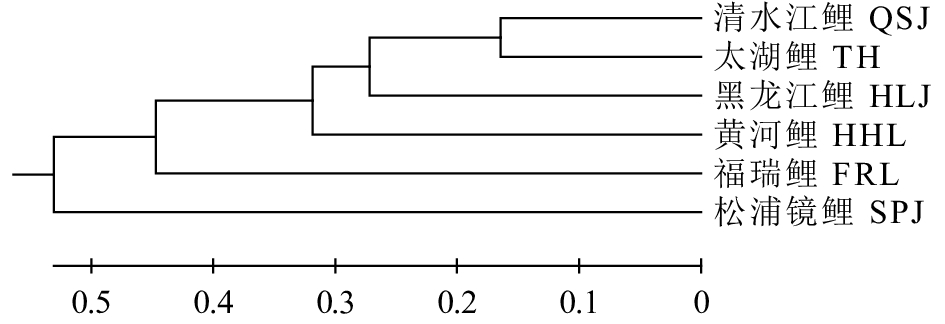
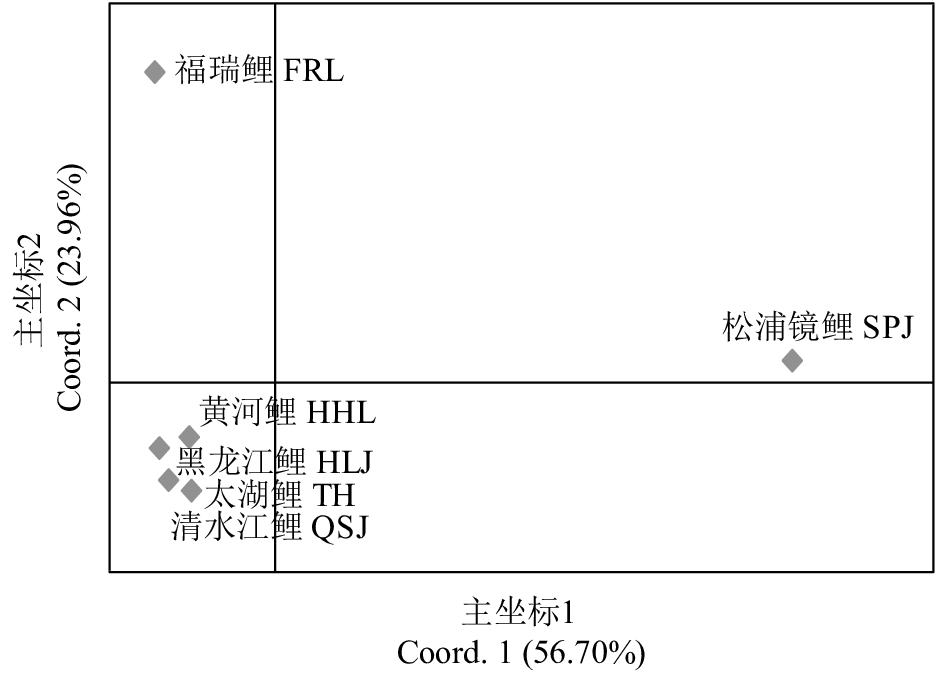
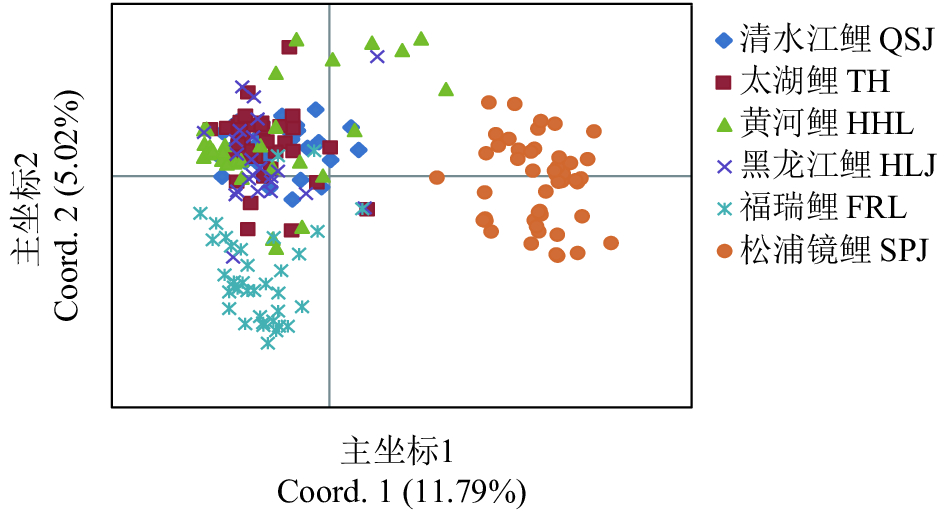
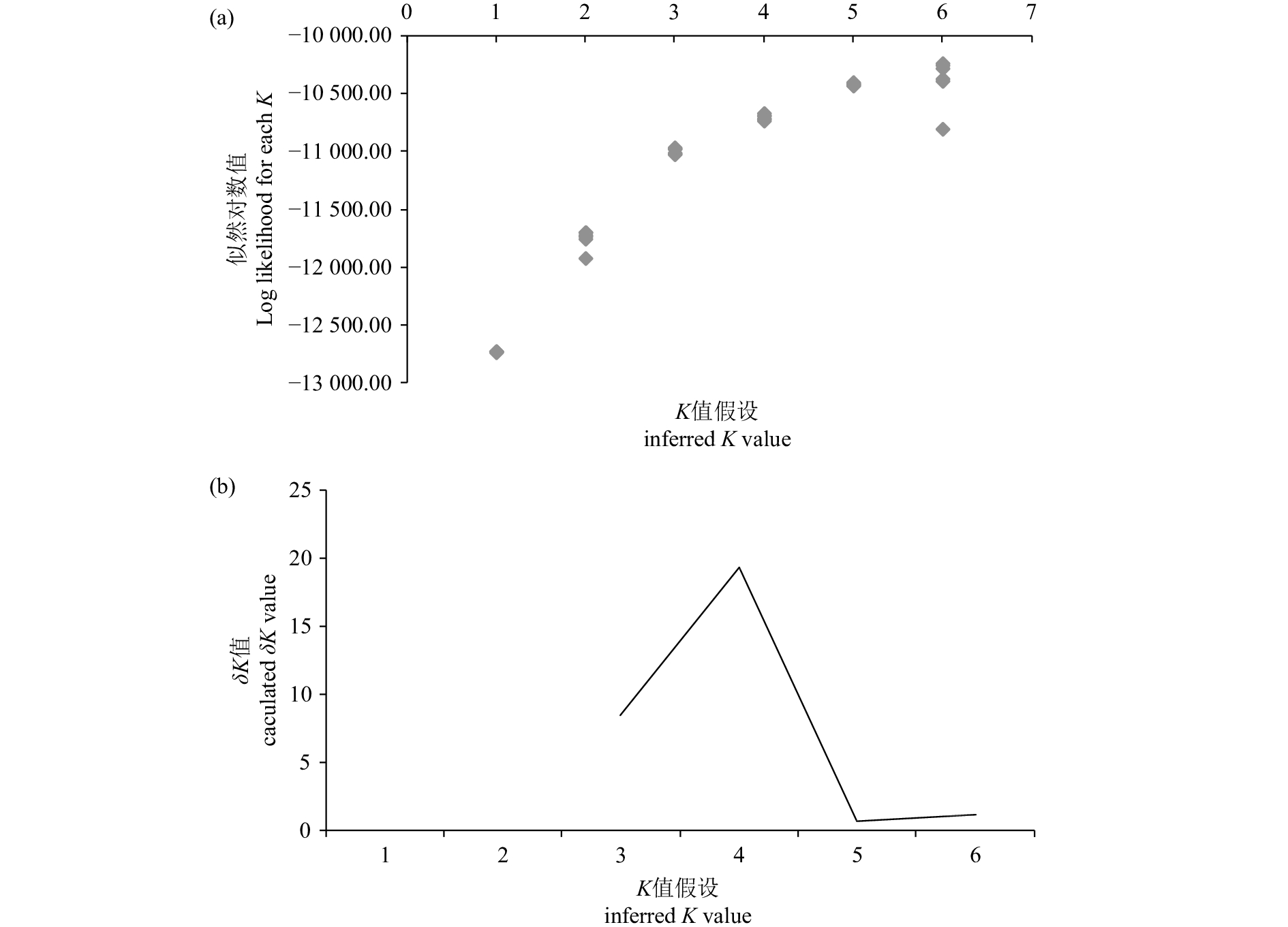
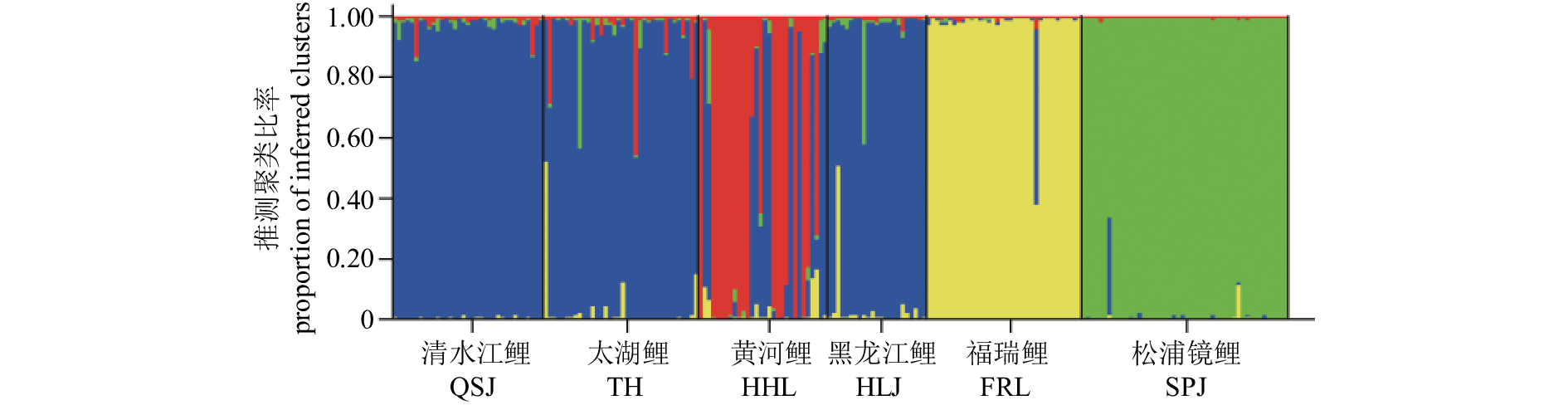
摘要: 利用12个微卫星标记对鲤(Cyprinus carpio)的4个野生群体 [清水江鲤、太湖鲤、黄河鲤(C.carpio haematopterus)和黑龙江鲤(C.carpio amurensis)] 和2个选育群体 [福瑞鲤(C.carpio var. FFRC)和松浦镜鲤(C. carpio var. specularis 'Song-pu')] 共208尾个体进行遗传分析。结果显示,12个位点共检测到341个等位基因,平均等位基因数为28.67,其中1个位点(HLJ1127)检测到正向选择压力;选育群体的遗传多样性参数普遍低于野生群体,其中松浦镜鲤群体的各项参数均值最低(Na=6.82,Ho=0.54,PIC=0.50),清水江鲤群体的各项参数均值最高(Na=21.25,Ho=0.80,PIC=0.91);分子方差分析显示,整体遗传变异主要来自群体内,但群体间呈极显著遗传分化(P<0.01);基于群体Nei's遗传距离的UPGMA聚类树和PCoA分析表明,鲤4个野生群体间遗传距离较近,而与2个人工选育群体间遗传距离较远;基于个体遗传结构及PCoA分析显示部分野生个体遗传结构比较混杂,而选育个体的遗传结构则相对单一。研究表明,中国鲤野生资源具有较高的遗传多态性,而人工选育群体维持着较纯的遗传种质。Abstract: We genotyped a total of 208 individuals of common carp (Cyprinus carpio) from four wild populations [Qingshuijiang (QSJ), Taihu (TH), C.carpio haematopterus (Huangheli, HHL) and C.carpio amurensis (Heilongjiang, HLJ)] and two selected strains [C.carpio var. FFRC (FRL) and C.carpio var. specularis 'Song-pu' (SPJ)] by using 12 microsatellite markers. Altogether 341 alleles were detected in 12 loci, with an average of 28.67 alleles per locus, and one locus (HLJ1127) was under strong selection pressure. Two selection strains showed lower genetic diversity parameters values than four wild populations. Strain SPJ showed the lowest genetic diversity parameters values (Na=6.82, Ho=0.54 and PIC=0.50), whereas Strain QSJ presented the highest parameters values (Na=21.25, Ho=0.80 and PIC=0.91). The analysis of molecular variance indicates that most of the genetic variance was within populations; however, significant genetic divergence among populations was detected (P<0.01). Similar results had been found by UPGMA dendrogram and PCoA plots using Nei's genetic distances among six populations. The wild populations had closer genetic distance and clustered together earlier, but were clearly separated from two selection strains. The PCoA plots and genetic structure analysis reveal the distinct genetic structure of individuals from selection individuals and partial complexity genetic structure of wild individuals. In brief, wild C.carpio populations in China have high genetic diversity and good genetic purity in their selection strains.
-
Keywords:
- Cyprinus carpio /
- population /
- selection /
- microsatellite /
- genetic diversity
-
细胞色素P450(cytochrome P450,CYPs)是一类广泛分布于生物体内的超基因家族,其酶系能够催化内源物质的合成、降解以及外源性物质的代谢[1],在解毒代谢阶段I起到重要作用[2]。CYPs基因具有结构多样性,这决定了细胞色素P450酶系可能是自然界最具催化多样性的酶系之一[3]。NELSON等[2]基于481个细胞色素P450基因和22个假基因的趋异进化关系以及氨基酸序列相似度将CYPs归为不同的“家族(family)”、“亚家族(subfamily)”,同一家族序列同源性大于40%,同一亚家族序列同源性大于55%[2, 4]。目前无脊椎动物中已发现的CYPs家族数已超过了70多个[5],而软体动物中CYPs基因的相关研究基础十分薄弱,仅在栉孔扇贝(Chlamys farreri)[6]、菲律宾蛤仔(Ruditapes philippinarum)[7]、翡翠贻贝(Perna viridis)[8]以及长牡蛎(Crassostrea gigas)[9]等少数物种有相关基因的研究报道,且缺乏系统的命名。
福寿螺(Pomacea canaliculata)具有适应性强、繁殖力高等特点,严重危害水域生态、农作物及人类健康,是中国恶性外来入侵种之一[10]。目前普遍采用四聚乙醛(methaldehyde)为有效成分的药物对其进行杀灭防治,然而四聚乙醛的持续重复施用诱导了福寿螺对其产生微弱抗药性[11-12]。细胞色素P450作为生物体内三大解毒酶系之一,已经被证明是害虫对杀虫剂产生抗性和交互抗性的主要原因之一[13-14],研究认为海洋贝类CYPs酶活力与水体有毒有机物代谢密切相关[7],而福寿螺耐药机理还不明了,相关研究十分薄弱。基于此,文章克隆分析了福寿螺CYP3192A1,采用荧光定量PCR(real time quantitative PCR,RT-qPCR)检测了CYP3192A1基因表达水平的组织及性别差异性,进而研究浓度及时间维度上四聚乙醛处理与CYP3192A1表达水平的相关性,初步探索CYP3192A1在四聚乙醛代谢中的作用地位,以为研究福寿螺CYPs基因结构与解毒功能,细胞色素P450功能及底物特异性奠定理论基础。
1. 材料与方法
1.1 材料处理
福寿螺采集于中国水产科学研究院珠江水产研究所外来水生生物入侵风险评估中心,螺体质量为9.32~15.28 g,壳高3.5~4.5 cm,取自相同世代群体。分别取雌、雄螺的肝、心、中肠、鳃、胃及腹足等6个组织,每个组织设3个样重复,采用RT-qPCR测定CYP3192A1基因表达水平,每样3个重复。
分别以0.6 mg·L-1、2.0 mg·L-1的亚致死浓度四聚乙醛处理福寿螺雌性群体,在用药后第0、第2、第6、第12、第24小时采集样品,选取鳃、肝、肠、胃4个组织,每个组织设3个样重复,RT-qPCR测定CYP3192A1表达水平,每样3个重复。
1.2 总RNA的提取
应用Mollusc RNA Kit R6875-01(OMEGA Bio-TEK)试剂盒提取福寿螺组织中总RNA,以电泳仪(BG-submidi)和核酸蛋白分析仪(东南D30)检测RNA质量与浓度后,置于-80 ℃保存备用。
1.3 细胞色素P450核心区cDNA克隆
应用PrimeScriptTMⅡ1st Strand cDNA synthesis Kit(TaKaRa)试剂盒进行反转录获得cDNA,-20 ℃保存。Primer Premier 5.0软件设计目的片段扩增引物P3-F和P3-R(表 1),以TaKaRa LA Taq®(RR02MA)进行PCR,测序;纯化目的片段、连接至pMD19-T载体,转染感受态细胞E.coli DH5α,菌液涂布于SOC培养基上12 h后挑菌落,经克隆、测序、鉴定后与已知序列进行比对。
1.4 cDNA末端快速扩增
以核心区序列为模板设计5′RACE-PCR套式引物P3-5′-1、P3-5′-2(表 1),应用Clontech SMAR-Ter® RACE cDNA Amplification Kit试剂盒进行反转录和Outer-PCR,通用引物为UPM;利用LA Taq®和Ex Taq®(TaKaRa)进行Inner-PCR,通用引物为NUP,依次进行PCR测序和克隆测序。设计引物P3-3′-1、P3-3′-2、P3-3′-3、P3-3′-4进行3′ RACE-PCR,利用试剂盒3′-Full RACE core Set with Prime ScriptTM RTase、LA Taq®(TaKaRa)、HiFi酶(Tran-sgene bio-tech)获得3′末端序列,通用引物为3′ Outer Primer、3′ Inner Primer,克隆测序。PCR主程序为94 ℃,30 s;退火30 s;72 ℃,30~60 s;30个循环;退火温度值见表 1。
表 1 细胞色素CYP3192A1扩增、RACE-PCR引物及荧光定量PCR引物Table 1. Primers used for PCR, RACE-PCR and quatitative real-time PCR引物名称
primer引物序列(5′→3′)
primer sequence扩增片段/bp
amplified fragment退火温度/℃
annealing temperatureP3-F
P3-R
P3-5′-1
P3-5′-2
P3-3′-1
P3-3′-2
P3-3′-3
P3-3′-4
UPM
NUP
3′ Outer Primer
3′ Inner Primer
18S-f
18S-r
P3-QT-F
P3-QT-R5′-ATCATCCACACTCTCACTGC-3′
5′-GAATCGTTACGCCGTTGA-3′
5′-ATCCTCTGCCTTGGCGTTTTGTTCC-3′
5′-TAACCGAATGACCTTCCACACAGCC-3′
5′-ATCCACCCGACAGACTCTCACAGGC-3′
5′-CTCAGCAAGAGGTTGTCTCCCAGTG-3′
5′-GCTACCCACGACCCCACCAACTACC-3′
5′-CTTCCCTTCGGTCTGGGTCCTCGTC-3′
5′-CTAATACGACTCACTATAGGGCAAGCAGTGGTATCAACGCAGAGT-3′
5′-CTAATACGACTCACTATAGGGC-3′
5′-AAGCAGTGGTATCAACGCAGAGT-3′Control Reagents
5′-TACCGTCGTTCCACTAGTGATTT-3′
5′-CGCGGATCCTCCACTAGTGATTTCACTATAGG-3′
5′-AATACATGCAAACCAGCTCC-3′
5′-ATTTTTCGTCACTACCTCCC-3′
5′-TTCGGTCTGGGTCCTCGTCA-3′
5′-TACAGCCCTGGTGGAAGTGG-3′1 247
1 247
758
672
1 225
1 094
1 010
922
5′ universal Primer mix
nested universal primer
universal
universal
329
329
322
32256
56
60
62
60
60
62
62
57
57
57
571.5 实时荧光定量PCR
预实验基础上以18S rDNA为荧光定量表达内参基因。参考福寿螺18S(EU520453.1)设计引物18S-f、18S-r;设计福寿螺CYP3A序列克隆片段引物P3-QT-F、P3-QT-R(表 1),分别克隆、连接载体、转染感受态细胞制备18S rDNA和CYP3192A1质粒。分别将质粒以10-1数量级稀释,共稀释6个浓度梯度;分别与SYBR® Select Master Mix体系混合,置于LightCycler480 Software Setup(Roche罗氏)进行荧光定量PCR,获得荧光信号强度与浓度的标准曲线,结果显示18S内参基因表达的标准曲线为y=-3.404x+38,R2=0.993 3,E=98.4%;CYP3192A1基因的为y=-3.404 262x+34.09,R2=0.990 7,E=101.3%。说明实验数据可靠,可继续进行实验数据的检测。同实验组每个组织3个样品重复,然后每个cDNA样品分别进行荧光定量PCR,测定每个样CYP3192A1绝对定量表达,每个样品3次检测重复。
1.6 序列分析与数据统计
序列拼接与组装,获得CYP3192A1基因cDNA全序列;ORF finder在线工具预测开放阅读框(open reading frame,ORF)序列;NCBI数据库对蛋白序列进行Blastp和SmartBLAST搜索相似序列,MEGA 5.0进行ClustalW比对并构建Neighbor-Joining树;用ProtParam在线工具预测蛋白质的理论等电点pI、分子质量和亲水性等氨基酸特性;Motif Scan工具预测序列的模体结构;TMHMM server.V.2.0预测跨膜结构区;Interpro预测蛋白结构域并归类;SWISS-MODEL Workspace预测蛋白三维结构并分析底物识别位点(substrate recognition sites,SRS);相似序列比对分析CYP3192A1序列保守位点。
用2-ΔΔCT法处理分析荧光定量PCR数据[15],采用软件Excel 2010作图,SPSS 19.0统计分析性别、组织表达差异性及药物处理与福寿螺CYP3192A1表达水平的相关性。
2. 结果
2.1 CYP3192A1的克隆与鉴定分析
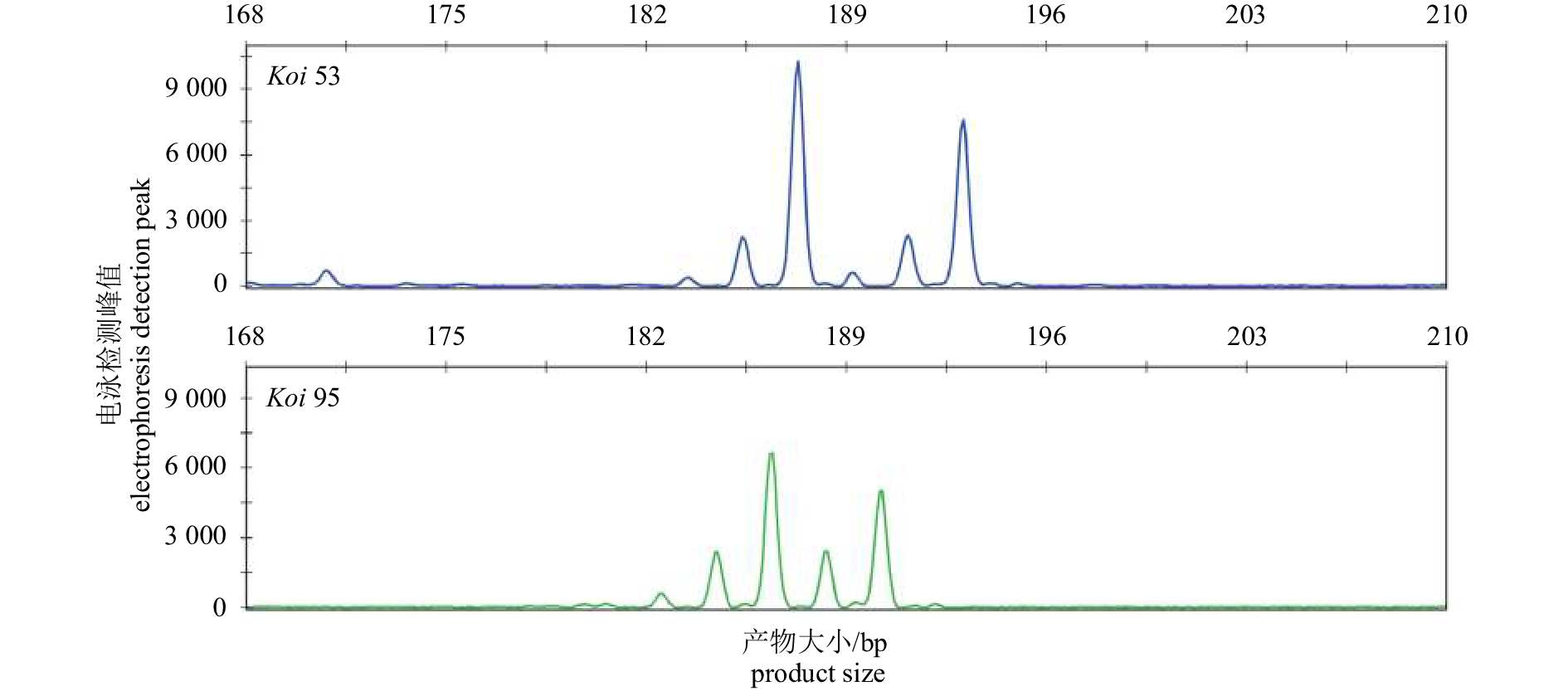
通过PCR和RACE-PCR从福寿螺鳃组织中克隆获得一条长度为2 523 bp的CYPs基因cDNA全序列(GenBank号KU923379);包含了260 bp的5′非编码区(UTR)和712 bp的3′UTR序列,以及1 551 bp的开放阅读框序列,预测显示其编码517个氨基酸(图 1),将氨基酸序列提交至细胞色素P450命名委员会,定名为CYP3192A1。预测蛋白质分子量约为58.0 kD,理论等电点为8.61,亲水性较弱,亮氨酸(Leu)含量高达12.6%。
![]() 图 1 福寿螺CYP3192A1克隆及氨基酸序列分析CYP3192A1的核酸及编码氨基酸序列,不同背景颜色氨基酸序列为P450超家族E class的标签序列;蓝色字体标注的序列为跨膜螺旋;红色字体的序列依次为C螺旋(W***R)、I螺旋(AG*ET)、K螺旋(E**R)和血红素结合区F**G*** C*G。Figure 1. Cloning and amino acid sequence analysis of P.canaliculata CYP3192A1The CYP3A-like sequences of nucleic acids and amino acids encoding; the sequence amino acids were marked by different background colors for P450 superfamily E class groupⅠsignature. Blue font tags sequence represent transmembrane helix. Red fonts represent helix C (W***R), I helix (AG*ET), helix K (E**R) and heme combination zone F**G***C*G.
图 1 福寿螺CYP3192A1克隆及氨基酸序列分析CYP3192A1的核酸及编码氨基酸序列,不同背景颜色氨基酸序列为P450超家族E class的标签序列;蓝色字体标注的序列为跨膜螺旋;红色字体的序列依次为C螺旋(W***R)、I螺旋(AG*ET)、K螺旋(E**R)和血红素结合区F**G*** C*G。Figure 1. Cloning and amino acid sequence analysis of P.canaliculata CYP3192A1The CYP3A-like sequences of nucleic acids and amino acids encoding; the sequence amino acids were marked by different background colors for P450 superfamily E class groupⅠsignature. Blue font tags sequence represent transmembrane helix. Red fonts represent helix C (W***R), I helix (AG*ET), helix K (E**R) and heme combination zone F**G***C*G.跨膜结构域预测显示1~11号氨基酸位于生物膜以内,35~517氨基酸位于细胞质内,而12~34氨基酸为一个跨膜螺旋(图 1),其中12~31氨基酸镶嵌于生物膜内部;蛋白质结构分析显示其归类于细胞色素P450酶,具有P450超家族E类(E class)的标签(signature)序列(PR00463):69~88氨基酸(amino acid,AA)、93~114 AA、380~398 AA、420~444 AA、452~462 AA以及462~485 AA(图 1);还具有CYP450高度保守序列血红素结合区F** G***C*G、K螺旋(E**R)、I螺旋(AG*ET)、C螺旋(W***R)(图 1)等,其中亚铁血红素结合区视为CYPs的特征基序,与I螺旋、K螺旋等参与稳定蛋白核心结构[16],空间结构如图 2。
2.2 CYP3192A1的序列比对与系统发育分析
以福寿螺CYP3192A1氨基酸进行Blastp同源比对搜索,获得不同物种同源性序列,均为CYP3A亚家族序列;基于不同物种的CYP3A亚家族氨基酸序列同源性构建了系统发育树(图 3),聚类分析显示福寿螺CYP3192A1与长牡蛎的CYP3A24归为一支,鱼类的CYP3A亚家族序列归为一支,人(Homo sapiens)和印度野牛(Bos taurus)等哺乳动物CYP3A亚家族归为一支,各种鸟的CYP3A亚家族序列归为一支,物种亲缘关系的亲疏符合传统的动物分类学中分类法则。与福寿螺CYP3192A1基因氨基酸序列同源性最高的为长牡蛎的CYP3A24序列,同源性仅为34%;推测福寿螺该CYPs属于细胞色素P450新家族的一个成员;将其序列提交至细胞色素P450命名委员会,正式定名为CYP3192A1。
表 2 图 3中基因、物种名及对应的登录号Table 2. Gene, Latin name, Chinese name of species and their Accession No. in Fig. 3基因gene 物种species 登录号Accession no. CY3A9 白鹭Egretta garzetta KFP21783.1 CYP3A21 红蜂虎Merops nubicus KFQ25408.1 CYP3A24 红冠蕉鹃Tauraco erythrolophus KFV20621.1 CYP3A24 鹃鴗Leptosomus discolor KFQ05822.1 CYP3A9 马来犀鸟Buceros rhinoceros silvestris KFO86946.1 CYP3A9 金领娇鹟Manacus vitellinus KFW84447.1 CYP3A9 短嘴鸦Corvus brachyrhynchos KFO59023.1 CYP3A9 刺鹩Acanthisitta chloris KFP82045.1 CYP3A24 双领鸻Charadrius vociferous KGL97267.1 CYP3A37 鸵鸟Struthio camelus BAN33784.1 CYP3A24 南非鸵鸟Struthio camelus australis KFV75188.1 CYP3A13 小家鼠Mus musculus NP 031845.1 CYP3A43 智人Homo sapiens NP 476436.1 CYP3A4 普通牛Bos taurus NP 001092837.1 CYP3A56 底鳉Fundulus heteroclitus NP 001296866.1 CYP3A27 安大略鲑Salmo salar ACI33861.1 CYP3A45 虹鳟鱼Oncorhynchus mykiss AAK58569.1 CYP3A24 长牡蛎Crassostrea gigas EKC24008.1 2.3 CYP3192A1基因表达的性别及组织差异性
福寿螺CYP3192A1表达具有性别差异性,性别对福寿螺肝、肠、鳃和心组织中CYP3192A1表达水平具有极显著影响(P < 0.01),而对胃及腹足中CYP3192A1表达水平无显著影响(P>0.05)。肠组织CYP3192A1的表达量表现为雄性高于雌性,而在其他组织中CYP3192A1的表达水平均为雄性低于雌性(图 4)。
![]() 图 4 福寿螺CYP3192A1基因在雌雄各组织中的表达情况A、B表示福寿螺组织CYP3192A1基因表达水平受性别的影响;a、b、c表示福寿螺不同组织CYP3192A1基因表达差异。Figure 4. Sex-dependent and tissue-dependent differences of P.canaliculata CYP3192A1 gene expressionA and B represent CYP3192A1 expression level affected by gender in different tissues; a, b and c represent tissue-dependent differences of CYP3192A1 expression in male and female individuals.
图 4 福寿螺CYP3192A1基因在雌雄各组织中的表达情况A、B表示福寿螺组织CYP3192A1基因表达水平受性别的影响;a、b、c表示福寿螺不同组织CYP3192A1基因表达差异。Figure 4. Sex-dependent and tissue-dependent differences of P.canaliculata CYP3192A1 gene expressionA and B represent CYP3192A1 expression level affected by gender in different tissues; a, b and c represent tissue-dependent differences of CYP3192A1 expression in male and female individuals.福寿螺CYP3192A1表达具有组织差异性,且在雌、雄个体中的表达有差异。雄螺CYP3192A1在肠组织表达量最高,显著高于其他组织,而其他组织间无显著性差异;雌螺CYP3192A1在肠和鳃组织表达量最丰富,其次是肝和胃组织,心和腹足组织内表达量最低。
2.4 四聚乙醛处理对CYP3192A1基因表达的影响
分别以0.6 mg·L-1、2.0 mg·L-1的四聚乙醛处理雌性福寿螺24 h,RT-qPCR检测不同时刻肝、鳃、肠和胃组织的表达情况。结果如图 5所示,四聚乙醛的处理质量浓度为0.6 mg·L-1时,肝和胃组织CYP3192A1表达量在0~6 h呈逐渐上升趋势;鳃和肠组织CYP3192A1表达量在2~6 h呈逐渐上升趋势。四聚乙醛处理质量浓度为2.0 mg·L-1时,肝和胃组织CYP3192A1表达量与0.6 mg·L-1时变化趋势类似,但是表达量上升时间缩短至2 h,表达量峰值降低;鳃和肠组织CYP3192A1表达量实验期间呈持续下降趋势,未检测到CYP3192A1的诱导表达。四聚乙醛诱导福寿螺CYP3192A1表达的持续时间随浓度变化在时间维度上表现出不同的趋势:低浓度诱导期更长,高浓度诱导期缩短甚至在实验时间内未检测到诱导作用。
3. 讨论
该研究利用在线工具对克隆基因的编码蛋白序列进行分析鉴定,在氨基酸序列中未发现明显的跨膜信号肽,表明该蛋白质不属于分泌蛋白;跨膜结构区分析结果显示CYP3192A1具有由12~34号氨基酸组成的跨膜螺旋,镶嵌于生物膜内,35~517号氨基酸位于细胞质基质内,据此推测CYP3192A1可能依赖于自身的跨膜螺旋锚定于内质网上[1]。
细胞色素P450是一类含有亚铁血红素的超家族单加氧酶,参与多类底物的不同催化反应类型,但是不同形态的细胞色素P450具有位点特异性、立体结构特异性和底物特异性[17-18],引起这种特异性的结构基础归结于氨基酸和核酸序列的差异性。研究认为这种差异性是细胞色素P450超家族通过基因扩增和适应多样化而形成的,另外细胞色素P450也具有特定的保守序列[19]。细胞色素P450结构的一般特征为C端富集螺旋状物且高度保守,N端富集β折叠物;CYPs标志性的区域为F**G***C*G基序,最保守的部分是围绕血红素的蛋白质中心,该保守中心空间上接近螺旋体I及螺旋K[20];CYPs的可变区通常与N端锚定、底物结合和识别有关,具有底物识别位点(SRSs)[21]。该研究中的蛋白质序列具有Cyt_P450_ E_ grp-I(Cytochorome P450 E class group I)(IPR002401)的特征结构域,由于大多数微粒体细胞色素P450均属于此分类[2]研究基础,推测CYP3192A1蛋白序列属于微粒体细胞色素P450酶。另外CYP3192A1蛋白序列具有细胞色素P450酶的高度保守区域,空间结构呈球形,具有血红素结合区及蛋白中心区域;N端结构变异性较大,空间上没有结构域包围,易于三维结构的变化,符合细胞色素的一般结构特征。系统发育分析显示,与福寿螺CYP3192A1同源性最高的序列是长牡蛎的CYP3A24,同源性仅为34%,并未达到同家族序列同源性所规定的40%;但GenBank数据库中与此同源性较高的其他物种CYPs序列均为CYP3A亚家族的成员,且空间结构高度保守,而研究认为细胞色素P450超家族的多样性是基因扩增进化而来的[4],推测福寿螺CYP3192A1基因可能是由CYP3A进化而来,其在蛋白结构上呈现高度相似性,因此CYP3192A1在结构及功能研究上可以参考CYP3A亚族基因的研究成果。研究认为CYP3A亚家族酶系是药物代谢中最重要且唯一具多个底物识别位点的CYP亚家族同工酶,其代谢底物种类繁多[13];目前,在人、老鼠、鱼类等多种生物体内已发现与药物代谢相关的CYP3A亚家族成员[22-24]。因此福寿螺CYP3192A1序列结构特征的克隆与鉴定,与CYP3A亚家族结构与功能研究相互促进,为福寿螺细胞色素P450酶学特性及药物代谢研究提供重要理论基础。
细胞色素P450广泛存在于不同的生物个体中,且同种基因在不同生物、不同组织中分布具有差异性[25];水生动物研究显示鳃是异源物质吸收、生物转化及代谢物排泄的重要器官[26],消化器官因其细胞色素P450的高水平表达而作为主要的解毒器官研究[27],因此该实验以鳃和消化系统作为实验研究对象组织。研究认为细胞色素P450基因的表达是在体内的信号或外界异生物质的复杂而精确地调控下进行的,其表达水平除了与物种和组织有关,还应考虑其性别及其所处环境条件等情况[19]。该研究中,性别和组织特异性对福寿螺CYP3192A1的表达水平有显著影响。整体表现为雌性表达量高于雄性,这可能与体内激素分泌相关;不同性别个体的CYPs的表达量不同,并且特定CYPs在体内的表达及其活性也存在性别差异性[28-29];而在组织间,表现为表达量最丰富的组织是肠和鳃,肝和胃次之,在心和腹足中表达最少。这与常见水生动物组织特异性研究有相似之处,但也有诸多差异性;如异育银鲫(Carassius auratus gibelio)CYP3A亚家族基因在肝和肠组织中转录水平最高,在肾和鳃中次之,在其余组织较低[30]。
检测代谢酶系的cDNA表达水平是研究化合物对CYPs的抑制或诱导效应的常用方法之一[31]。为了解福寿螺CYPs表达水平与四聚乙醛处理的相关性,文章在四聚乙醛对福寿螺急性攻毒实验的基础上,参考韩文素等[32]的实验方法选取不同倍数LC50的亚致死浓度的四聚乙醛处理福寿螺,检测了四聚乙醛处理后CYP3192A1基因表达的变化。研究认为胁迫环境下福寿螺几乎不活动,新陈代谢维持在很低水平[33],而该实验结果显示在福寿螺受到四聚乙醛胁迫后活力下降的同时,不同组织均出现了CYP3192A1表达量一定时期内升高的现象,说明四聚乙醛能增强福寿螺组织CYP3192A1的表达,总体表现为“低浓度诱导,高浓度缩短诱导期”;另外,随着四聚乙醛浓度的升高,福寿螺CYP3192A1表达水平也发生显著的变化,证明了四聚乙醛处理与福寿螺CYP3192A1表达量变化具有密切相关性。可能由于四聚乙醛在福寿螺体内存在药物的“首过效应”[34],最新接触到药物的组织对药物存在吸收和稀释作用,鳃和肠诱导期更早出现;最后所有组织CYP3192A1表达量均表现为持续下降的趋势,可能是由于福寿螺自身解毒系统并不能完全消除四聚乙醛的毒害作用,机体生命活动状态趋向衰竭,高浓度四聚乙醛能够增强四聚乙醛作用效果。
总体来说四聚乙醛能够诱导福寿螺组织内CYP3192A1表达水平的增加,研究认为由表达增加或活力增强等引起的代谢酶酶活性增加一般会促使药物代谢增加[35],因此CYP3192A1在药物胁迫下的诱导表达可能在增强四聚乙醛代谢中起到作用,然而这需要对四聚乙醛在福寿螺体内代谢途径及CYP3192A1底物特异性进行深入研究验证。
-
表 1 鲤6个群体样本采集信息
Table 1 Sample information of six populations of C.carpio
群体
population群体来源
source经纬度
longitude, latitude样本数
number of sample清水江鲤 QSJ 贵州凯里 107.97°E,26.58°N 35 太湖鲤 TH 江苏无锡 120.29°E,31.59°N 36 黄河鲤 HHL 河南郑州 114.08°E,32.13°N 30 黑龙江鲤 HLJ 黑龙江抚远 134.28°E,48.37°N 23 福瑞鲤 FRL 淡水渔业研究中心 119.82°E,31.36°N 36 松浦镜鲤 SPJ 黑龙江水产研究所 126.63°E,45.75°N 48 表 2 鲤群体遗传研究所用12个微卫星位点的基本信息
Table 2 Basic information of 12 microsatellite loci used in genetic analysis for C.carpio populations
位点
locus重复单元
repeat unit荧光修饰
fluorescent dye引物序列 (5'–3')
primer sequence产物大小/bp
size range退火温度/℃
annealing temperatureKoi53 CA 6-FAM AGGTCACTGAAACAATACA 167~209 55 CTGATGCTTCTGGATAAA Koi87 GT HEX ATGCTTTTTTTCTCGGATTC 284~330 55 GCAAATCAGTCAAACATCAC Koi95 CA HEX TGTATTGCTTATTGTTAGTT 157~223 46 AGTCTGTCTGTTTCCGTCTT Koi113 CA 6-FAM CACTTTGCCATTTTCCTATC 119~201 55 TTCATTCGCTCTTTTTTTAT Koi115 GT HEX GAGGAAATGATGGAATAAAT 232~292 55 TAAGAGGGTTTTGTAGTGTA MFW18 CA 6-FAM GTCCCTGGTAGTGAGTGAGT 131~219 55 GCGTTGACTTGTTTTATACTAG MFW20 CA HEX CAGTGAGACGATTACCTTGG 162~264 55 GTGAGCAGCCCACATTGAAC HLJ307 AC 6-FAM ATCATTTGTATTCGTGCTTG 180~246 55 GATCCACTGGGTCCTTTT HLJ308 GT 6-FAM TGACAGGAAGAGCAGGAC 107~195 55 TCTCGAAGAACAGACACCC HLJ316 AC HEX TGCTAATCGGTGTTTCAT 173~249 55 TTCTGCTTCACAGCCATA HLJ322 AC 6-FAM GGGAGATGGGATGGATGA 183~261 55 GAGGGAGTATTTGTGAGTGTTG HLJ1127 AC HEX GTTACGTCTTTGCCCTGAGC 205~289 55 TGCCCTTCAATAAACGCTTC 表 3 12个微卫星对6个鲤群体遗传多样性的检测结果
Table 3 Statistics for genetic variation at 12 microsatellite loci in six populations of C.carpio
位点
locus等位基因数
Na有效等位基因数
Ne观测杂合度
Ho期望杂合度
He多态信息含量
PIC哈-温平衡 HWE 野生群体
wild population选育群体
selected populationKoi53 18 5.86 0.48 0.83 0.81 NS ** Koi87 23 12.70 0.68 0.92 0.92 NS ** Koi95 28 13.85 0.69 0.93 0.92 NS ** Koi113 35 18.89 0.66 0.95 0.94 NS ** Koi115 23 7.68 0.48 0.87 0.86 NS ** MFW18 28 8.50 0.79 0.88 0.87 NS ** MFW20 35 18.89 0.75 0.95 0.95 NS ** HLJ307 30 14.99 0.85 0.94 0.93 NS ** HLJ308 30 8.83 0.83 0.89 0.88 NS NS HLJ316 28 7.08 0.73 0.86 0.85 NS ** HLJ322 32 9.42 0.87 0.89 0.89 NS ** HLJ1127 31 16.18 0.95 0.94 0.94 NS ** 平均 mean 28.67 11.88 0.73 0.91 0.90 – – 注:NS. 不显著(P>0.05);**. 极显著偏离哈-温平衡(P<0.01) Note: NS. insignificant difference (P>0.05); **. very significant deviation from HWE (P<0.01) 表 4 鲤6个群体遗传多样性参数
Table 4 Genetic diversity parameters of six populations of C.carpio
参数
index清水江鲤
QSJ太湖鲤
TH黄河鲤
HHL黑龙江鲤
HLJ福瑞鲤
FRL松浦镜鲤
SPJ等位基因数 Na 21.25 19.42 12.50 12.83 7.41 6.82 有效等位基因数 Ne 13.06 11.76 7.83 7.68 4.77 3.41 观测杂合度 Ho 0.80 0.78 0.79 0.83 0.73 0.54 期望杂合度 He 0.93 0.92 0.88 0.87 0.78 0.60 多态信息含量 PIC 0.91 0.90 0.85 0.84 0.73 0.50 表 5 鲤群体基于微卫星标记的分子方差分析
Table 5 Analysis results of molecular variance of microsatellites in C.carpio populations
变异来源
source of variation平方和
sum of squares方差组分
variance component百分率/%
percentage of variation分化指数
fixation index不分组 ungrouped 群体间 among populations 218.48 0.62 12.19 FST=0.12** 群体内 within populations 1 691.64 4.45 87.81 总体 total 1 910.12 5.06 区分组 (选育和野生) grouped (selection and wild) 不同组间 among groups 78.86 0.21 4.04 FCT=0.04** 组内不同群体间 among populations within groups 139.62 0.50 9.63 FSC=0.10** 群体内 within populations 1 691.64 4.45 86.33 FST=0.14** 总体 total 1 910.12 5.15 注:**. 差异极显著(P<0.01);后表同此 Note: **. very significant difference (P<0.01);the same case in the following table. 表 6 鲤群体间的遗传分化指数 (FST,对角线以上) 和Nei's遗传距离 (DA,对角线以下)
Table 6 Pair-wise FST (above diagonal) and Nei's genetic distance (below diagonal) among C.carpio populations
清水江鲤
QSJ太湖鲤
TH黄河鲤
HHL黑龙江鲤
HLJ福瑞鲤
FRL松浦镜鲤
SPJ清水江鲤 QSJ 0.018** 0.023** 0.032** 0.094** 0.185** 太湖鲤 TH 0.328 0.011** 0.027** 0.074** 0.182** 黄河鲤 HHL 0.595 0.533 0.035** 0.076** 0.211** 黑龙江鲤 HLJ 0.650 0.437 0.783 0.087** 0.193** 福瑞鲤 FRL 1.054 0.895 0.807 0.821 0.212** 松浦镜鲤 SPJ 0.901 0.966 1.183 1.105 1.152 -
[1] HULAK M, KASPAR V, KOHLMANN K, et al. Microsatellite-based genetic diversity and differentiation of foreign common carp (Cyprinus carpio) strains farmed in the Czech Republic[J]. Aquaculture, 2010, 298(3/4): 194-201.
[2] REN W, HU L, GUO L, et al. Preservation of the genetic diversity of a local common carp in the agricultural heritage rice-fish system[J]. Proc Natl Acad Sci USA, 2018, 115(3): E546-E554.
[3] 董在杰, 刘念, 朱文彬, 等. 清水江鲤基于微卫星标记和形态指标的遗传分析[J]. 南方水产科学, 2018, 14(1): 1-8. [4] XIAO T, LU C, XU Y, et al. Screening of SSR markers associated with scale cover pattern and mapped to a genetic linkage map of common carp (Cyprinus carpio L.)[J]. J Appl Genet, 2015, 56(2): 261-269.
[5] 刘伟, 苏胜彦, 董在杰, 等. 3个鲤群体的微卫星标记与生长性状相关性分析[J]. 南方水产科学, 2012, 8(3): 17-24. [6] ZHENG X, KUANG Y, LV W, et al. Quantitative trait loci for morphometric traits in multiple families of common carp (Cyprinus carpio)[J]. Sci China Life Sci, 2017, 60(3): 287-297.
[7] 常玉梅, 孙效文, 梁利群. 中国鲤几个代表种群基因组DNA遗传多样性分析[J]. 水产学报, 2004, 28(5): 481-486. [8] 全迎春, 孙效文, 梁利群. 应用微卫星多态分析四个鲤鱼群体的遗传多样性[J]. 动物学研究, 2005, 26(6): 595-602. [9] XU L H, WANG C H, WANG J, et al. Selection pressures have driven population differentiation of domesticated and wild common carp (Cyprinus carpio L.)[J]. Genet Mol Res, 2012, 11(3): 3222-3235.
[10] LIAO X L, YU X M, TONG J O. Genetic diversity of common carp from two largest Chinese lakes and the Yangtze River revealed by microsatellite markers[J]. Hydrobiologia, 2006, 568(1): 445-453.
[11] LI D, KANG D, YIN Q, et al. Microsatellite DNA marker analysis of genetic diversity in wild common carp (Cyprinus carpio L.) populations[J]. J Genet Genom, 2007, 34(11): 984-993.
[12] 鲁翠云, 张晓丽, 顾颖, 等. 福瑞鲤与豫选黄河鲤选育群体的遗传结构及亲本间遗传距离分布[J]. 中国水产科学, 2016, 23(5): 1091-1098. [13] 桑滨, 鲁翠云, 李超, 等. 微卫星标记分析3个耐寒鲤品种的遗传多样性[J]. 生物学杂志, 2017, 34(3): 24-32,56. [14] 董在杰. 福瑞鲤选育技术和养殖对比试验[J]. 科学养鱼, 2011(6): 41-42. [15] 石连玉, 李池陶, 葛彦龙, 等. 黑龙江水产研究所鲤育种概要[J]. 水产学杂志, 2016, 29(3): 1-8. [16] 单云晶, 鲁翠云, 李超, 等. 基于线粒体COI基因序列的5种鲤养殖品种遗传多样性研究[J]. 中国水产科学, 2013, 20(5): 931-938. [17] 刘念, 傅建军, 董在杰, 等. 中国6个鲤群体的mtDNA D-loop序列遗传变异分析[J]. 水生态学杂志, 2017, 38(3): 75-82. [18] DAVID L, RAJASEKARAN P, FANG J, et al. Polymorphism in ornamental and common carp strains (Cyprinus carpio L.) as revealed by AFLP analysis and a new set of microsatellite markers[J]. Mol Genet Genom, 2001, 266(3): 353-362.
[19] CROOIJMANS R P M A, van der POEL J J, GROENEN M, et al. Microsatellite markers in common carp (Cyprinus carpio L.)[J]. Anim Genet, 1997, 28(2): 129-134.
[20] 孙效文, 贾智英, 魏东旺, 等. 磁珠富集法与小片段克隆法筛选鲤微卫星的比较研究[J]. 中国水产科学, 2005, 12(2): 126-132. [21] NEI M. Estimation of average heterozygosity and genetic distance from a small number of individuals[J]. Genetics, 1978, 89(3): 583-590.
[22] KALINOWSKI S T, TAPER M L, MARSHALL T C. Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment[J]. Mol Ecol, 2007, 16(5): 1099-1106.
[23] FOLL M, GAGGIOTTI O. A genome-scan method to identify selected loci appropriate for both dominant and codominant markers: a Bayesian perspective[J]. Genetics, 2008, 180(2): 977-993.
[24] JEFFREY H. Some tests of significance, treated by the theory of probability[J]. Math Proc Cambridge Philos Soc, 1935, 31(2): 203-222.
[25] EXCOFFIER L, LISCHER H E. Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows[J]. Mol Ecol Resour, 2010, 10(3): 564-567.
[26] TAMURA K, PETERSON D, PETERSON N, et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Mol Biol Evol, 2011, 28(10): 2731-2739.
[27] PEAKALL R, SMOUSE P E. GENALEX 6: genetic analysis in Excel. Population genetic software for teaching and research[J]. Mol Ecol Notes, 2006, 6(1): 288-295.
[28] PRITCHARD J K, STEPHENS M, DONNELLY P. Inference of population structure using multilocus genotype data[J]. Genetics, 2000, 155(2): 945-959.
[29] EVANNO G, REGNAUT S, GOUDET J. Detecting the number of clusters of individuals using the software STRUCTURE: a simulation study[J]. Mol Ecol, 2005, 14(8): 2611-2620.
[30] BALLOUX F, LUGON-MOULIN N. The estimation of population differentiation with microsatellite markers[J]. Mol Ecol, 2002, 11(2): 155-165.
[31] BOTSTEIN D, WHITE R L, SKOLNICK M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. Am J Hum Genet, 1980, 32(3): 314-331.
[32] 高雅, 李生斌. STR遗传多态性研究中样本数量对等位基因检出数量的影响[J]. 遗传, 2008, 30(3): 313-320. [33] 傅建军, 李家乐, 沈玉帮, 等. 草鱼野生群体遗传变异的微卫星分析[J]. 遗传, 2013, 35(2): 192-201. [34] LIU F, XIA J H, BAI Z Y, et al. High genetic diversity and substantial population differentiation in grass carp (Ctenopharyngodon idella) revealed by microsatellite analysis[J]. Aquaculture, 2009, 297(1/2/3/4): 51-56.
[35] 朱冰, 樊佳佳, 白俊杰, 等. 金草鱼与中国4个草鱼群体的微卫星多态性比较分析[J]. 南方水产科学, 2017, 13(2): 51-58. [36] 刘青, 刘皓, 吴旭干, 等. 长江、黄河和辽河水系中华绒螯蟹野生和养殖群体遗传变异的微卫星分析[J]. 海洋与湖沼, 2015, 46(4): 958-968. [37] DANISH M, SINGH I J. Genetic diversity analysis of common carp (Cyprinus carpio var. communis) and Labeo rohita (Hamilton, 1822) collected from hatchery by using microsatellite markers[J]. Int J Chem Stud, 2017, 5(3): 513-518.
[38] SINGH E, SHARMA O P, JAIN H K, et al. Microsatellite based genetic diversity and differentiation of common carp, Cyprinus carpio in Rajasthan (India)[J]. Nat Acad Sci Lett, 2015, 38(3): 193-196.
[39] 古珍珍, 杨新鑫, 胡建尊, 等. 瓯江彩鲤酪氨酸酶(TYR)基因的选择压力分析[J]. 上海海洋大学学报, 2016, 25(3): 329-336. [40] LEHOCZKY I, MAGYARY I, HANCZ C, et al. Preliminary studies on the genetic variability of six Hungarian common carp strains using microsatellite DNA markers[J]. Hydrobiologia, 2005, 533(1/2/3): 223-228.
[41] 曲疆奇, 毕滢佳, 董在杰, 等. 应用SRAP标记分析福瑞鲤及其原始亲本的遗传结构[J]. 动物学杂志, 2011, 46(5): 120-125. [42] KOHLMANN K, KERSTEN P, FLAJSHANS M. Microsatellite-based genetic variability and differentiation of domesticated, wild and feral common carp (Cyprinus carpio L.) populations[J]. Aquaculture, 2005, 247(1/2/3/4): 253-266.
[43] 岳兴建, 邹远超, 王永明, 等. 元江鲤种群遗传多样性[J]. 生态学报, 2013, 33(13): 4068-4077. [44] 张倩倩, 陈杰, 蒋霞云, 等. 不同鳊鲂鱼类群体微卫星DNA指纹图谱的构建和遗传结构分析[J]. 水产学报, 2014, 38(1): 15-22. [45] 杨慧荣, 欧阳徘徊, 李桂峰, 等. 用微卫星技术研究珠江流域三个野生大眼鳜群体的遗传多样性[J]. 中山大学学报(自然科学版), 2015, 54(1): 103-109. [46] 唐首杰, 毕详, 王成辉, 等. 团头鲂3个选育群体遗传潜力的微卫星分析[J]. 南方水产科学, 2017, 13(2): 59-68. [47] 唐首杰, 杨洁, 王成辉, 等. 驯养、选育条件下尼罗罗非鱼群体的选择压力分析[J]. 中国水产科学, 2016, 23(4): 900-913. -
期刊类型引用(26)
1. 黄晓兰,王苗苗,刘海静,杨奇慧,张会兰,陈厚宏,黎杰,徐静,陈岗富,李华涛. 当归副产物对颗粒饲料的保护作用及其对鲫生长和缺氧应激的影响. 动物营养学报. 2025(02): 1173-1187 .  百度学术
百度学术
2. 朱文婷,李文嘉,宣雄智,赵娟. 渔用中草药种类、作用机理及应用效果研究进展. 中国饲料. 2024(05): 94-100 .  百度学术
百度学术
3. 杨鹏,李锐,查红刚,叶志祥,史庆超,张志勇. 复合植物多糖对澳洲淡水龙虾血清生化指标的影响. 现代畜牧兽医. 2023(06): 36-39 .  百度学术
百度学术
4. 李蕾,蒋昕彧,朱雷,孔祥会. 多糖类免疫增强剂在鱼类养殖中的应用研究进展. 水产学杂志. 2023(06): 127-135+145 .  百度学术
百度学术
5. 刘辉,鹿瑶,辛运腾,董乐,董福霖,林基亮,于朝磊,张黎黎. 饲料中添加植物乳杆菌对花鲈生长性能、血清生化指标及抗哈维弧菌感染的影响. 饲料研究. 2023(23): 49-55 .  百度学术
百度学术
6. 曹雪,孙佳,杨质楠,梁爽,李月红. 中草药在水产动物养殖中的研究进展. 饲料研究. 2023(24): 133-137 .  百度学术
百度学术
7. 刘永,闫世雄,杜彦丽,何洋,施红梅,王坤,豆腾飞,刘丽仙,申志超,贾俊静,葛长荣. 植物多糖在鱼类养殖中的应用研究进展. 山西农业科学. 2022(02): 272-280 .  百度学术
百度学术
8. 肖芳,陈涛,伍振煌,王俊龙. 植物多糖的提取工艺、生物学功能及其在动物生产中的研究进展. 饲料研究. 2022(14): 125-128 .  百度学术
百度学术
9. 韦宏杰,李雪鹤,吴远彩,易远名,朱东文君,杨奇慧,谭北平. 月桂酸单甘油酯对凡纳滨对虾生长、肌肉氨基酸、非特异性免疫及肠道菌群的影响. 水产学报. 2022(10): 1912-1926 .  百度学术
百度学术
10. 卫明亮,张志伟,张志勇,林志杰,祝斐,贾超峰,孟乾,徐大凤,张曹进. 冷应激对黑鲷组织损伤及细胞凋亡基因表达的影响. 南方水产科学. 2022(05): 110-117 .  本站查看
本站查看
11. 杨欣仪,类延菊,阳佩蓉,蔡佳玲,杨品红,杨春英. 植物多糖的生物学功能及其对鱼类健康的影响. 饲料研究. 2022(20): 130-136 .  百度学术
百度学术
12. 贾慧凝,侍苗苗,卞永乐,侍崇敬,刘恒蔚,宋学宏,秦粉菊. 纳米硒对低氧胁迫下中华绒螯蟹免疫保护和抗氧化能力的影响. 南方水产科学. 2022(06): 100-109 .  本站查看
本站查看
13. 袁仲瑾,岑剑伟,李来好,杨贤庆,黄卉,魏涯,郝淑贤,赵永强,王悦齐,林织. 低温暂养对珍珠龙胆石斑鱼存活、非特异性免疫及抗氧化指标的影响. 南方水产科学. 2022(06): 118-126 .  本站查看
本站查看
14. 李忠琴,张新艳,杨求华,林茂,江兴龙,翟少伟. 五种中草药复方体外激活花鳗鲡(Anguilla marmorata)外周血白细胞活性的评价. 海洋与湖沼. 2022(06): 1487-1493 .  百度学术
百度学术
15. 孙彩云,董宏标,王文豪,李勇,古群红,段亚飞,张家松,许晓东. 月桂酸单甘油酯对花鲈脂质代谢的影响. 南方水产科学. 2021(01): 67-75 .  本站查看
本站查看
16. 虞为,杨育凯,林黑着,黄小林,黄忠,李涛,周传朋,马振华,荀鹏伟,杨长平. 牛磺酸对花鲈生长性能、消化酶活性、抗氧化能力及免疫指标的影响. 南方水产科学. 2021(02): 78-86 .  本站查看
本站查看
17. 杨蕊,周胜杰,方伟,马振华. 营养强化对卵形鲳鲹仔、稚鱼骨骼发育基因表达的影响. 水产科技情报. 2021(03): 126-131 .  百度学术
百度学术
18. 曾祥兵,董宏标,韦政坤,段亚飞,陈健,张慧,孙彩云,许晓东,张家松. 鸡内金多糖对尖吻鲈幼鱼生长、消化、肠道抗氧化能力和血清生化指标的影响. 南方水产科学. 2021(04): 49-57 .  本站查看
本站查看
19. 韩梦瑶,王晓梅,王占旗,叶金云,张忠山. 天然活性多糖在水产动物养殖中的应用. 水产学杂志. 2021(04): 85-92 .  百度学术
百度学术
20. 李正花,周建虹,陈土艳. 八珍汤加味治疗气滞血瘀证月经不调患者的疗效及作用机制探析. 世界中西医结合杂志. 2021(09): 1719-1723 .  百度学术
百度学术
21. 杨大俏,王锦旭,李来好,杨贤庆,马海霞,胡晓. 近江牡蛎膜分离联产制备多糖多肽及其功能特性研究. 大连海洋大学学报. 2020(01): 126-133 .  百度学术
百度学术
22. 杨大俏,王锦旭,李来好,杨贤庆,马海霞,岑剑伟,王悦齐. 近江牡蛎多糖的结构鉴定及免疫调节能力分析. 食品科学. 2020(10): 38-46 .  百度学术
百度学术
23. 李琛琛,何建,纪鹏,魏彦明,刘胜利,袁子文,张晓松,文艳巧,张亚辉,华永丽,姚万玲. 当归多糖对头孢噻呋钠联合LPS致鸡肝损伤的防治效果. 动物医学进展. 2020(06): 74-80 .  百度学术
百度学术
24. 杨玲,胡睿智,夏嗣廷,贺建华. 植物多糖的功能性研究进展及其在动物生产中的应用. 动物营养学报. 2019(06): 2534-2543 .  百度学术
百度学术
25. 虞为,杨育凯,陈智彬,林黑着,黄小林,周传朋,杨铿,曹煜成,黄忠,马振华,李涛,王珺,王芸,荀鹏伟,黄倩倩,于万峰. 饲料中添加螺旋藻对花鲈生长性能、消化酶活性、血液学指标及抗氧化能力的影响. 南方水产科学. 2019(03): 57-67 .  本站查看
本站查看
26. 赵香菊,刘秀玲,王中华. 大蒜多糖对肉鸡血清生化指标及抗氧化能力的影响. 中国家禽. 2019(20): 52-54 .  百度学术
百度学术
其他类型引用(9)



 下载:
下载:





 粤公网安备 44010502001741号
粤公网安备 44010502001741号
