Stability comparison of reference genes of Pinctada fucata by real-time qPCR
-
摘要:
以合浦珠母贝(Pinctada fucata)为研究对象, 应用实时荧光定量PCR技术检测了甘油醛-3-磷酸脱氢酶(GAPDH)、β-肌动蛋白(β-actin)和18S核糖体rRNA(18S rRNA)3个管家基因在合浦珠母贝不同组织、性腺发育时期、胚胎发育时期mRNA水平的表达情况。同时比较了BestKeeper、geNorm和NormFinde软件对试验数据处理的差异, 从中选出合适的内参基因。结果表明β-actin在不同组织和胚胎发育不同阶段表达最稳定, 18S rRNA在性腺发育不同时期表达最稳定。
Abstract:We analyze the expression stability of the housekeeping genes in Pinctada fucata, including Glyceraldehyde-3-Phosphate Dehydrogenase (GAPDH), beta-actin (β-actin) and 18S rRNA (18S), by quantitative real-time polymerase chain reaction (qRT-PCR) in different tissues, at different stages of gonadal development and embryonic development.Besides, we select the suitable reference genes by using softwares of BestKeeper, geNorm and NormFinder algorithms.The results indicate that β-actin is the most stable reference gene for different tissues and different embryonic developmental stages, while 18S is the most stable reference gene for different gonadal developmental stages.
-
Keywords:
- Pinctada fucata /
- qPCR /
- reference genes /
- stability analysis
-
随着生物技术的不断发展,实时荧光定量RT-PCR(real-time quantitative PCR,qPCR)已成为研究基因表达水平的常规检测手段。其原理是在PCR反应体系中添加特定荧光基团,通过实时监测整个PCR进程中产物的变化,即荧光信号的累积,对DNA、RNA样品或者目标基因的表达进行定性或者定量分析[1],可以实时监测不同细胞、组织、个体、发育不同阶段或是特殊处理条件下某个基因的表达水平。该技术以其定量准确、高重现性、高灵敏度、高通量等特点当前已广泛用于单个核酸多态性分析、RNA干扰后基因表达差异的验证、基因芯片数据验证以及疾病诊断等各领域[2-5]。然而定量PCR的数据处理和分析结果往往会受到起始材料的质量、RNA质量、反转录效率、内参基因的选择、扩增效率等因素的影响,从而与目标基因特异性表达的真实值存在一定的差异,因此,为了减少样本间的误差[6],获得真实可靠的试验结果,需要采用适当的内参基因对试验数据进行校正和标准化。理想的内参基因是在不同组织、不同发育阶段以及不同处理因素下表达水平均恒定的基因[7]。然而稳定表达的基因几乎不存在,因此仅利用单一的管家基因标准化,可能造成与基因的实际表达水平有很大差异。传统的管家基因如18S核糖体RNA(18S rRNA),28S核糖体RNA(28S rRNA),转录延伸因子基因(EF),多聚泛素酶基因(UBQ),甘油醛-3-磷酸脱氢酶基因(GAPDH),肌动蛋白基因(ACT)等常常作为内参基因[8]。但是研究表明管家基因的表达水平在不同的细胞类型及试验体系中可能变化很大,许多经典的管家基因并不适合广泛的应用。所以针对不同的试验体系,有必要筛选出最合适的内参基因。目前对于qPCR内参基因的选择过程发展出不同的算法,常用的有geNorm[9]、NormFinder[10]和BestKeeper[11],用于从若干候选内参基因中筛选出表达最为稳定的一个或一组基因作为内参系统。近年来筛选内参基因的研究已经有了大量的报道,多见于植物和动物中,如人(Homo sapiens)、亚麻(Linum usitatissimum)、西红柿(Solanum lycopersicon)、大豆(Giycine max)等[12-15]。水产动物方面,青鳉(Oryzias latipes)[16]、大西洋鲑(Salmo salar)[17]、虾夷扇贝(Patinopecten yessoensis)[18]等也展开内参稳定性的相关研究。杨桂梅等[19]利用抑制差减技术检测出GAPDH和β-actin基因在牙鲆(Paralichthys olivaceus)发育过程中表达水平上可能存在差异。
合浦珠母贝(Pinctada fucata)是中国人工培育海水珍珠的最主要珍珠贝,其所产珍珠量占整个海水珍珠产量的95%以上,具有重要的生产价值。当前国内尚未见关于合浦珠母贝基因表达定量PCR分析内参基因筛选的相关报道。为此,笔者研究选择合浦珠母贝18S rRNA、GAPDH、β-actin等3个常见的管家基因,对其作为内参基因的效应进行检测和比较,为后续qPCR最适内参基因的选择提供指导。
1. 材料与方法
1.1 样品采集及处理
合浦珠母贝样品采集于深圳市大亚湾海区养殖群体,由所在课题组进行常规养殖。试验共分为3个组:1)组织分布组(包括精巢、卵巢、外套膜、闭壳肌、鳃、消化腺、心脏);2)性腺发育组(包括休止期、增殖期、生长期、成熟期、排放期;部分样品固定于Bouin′s液,4 ℃保存用于常规组织学切片鉴定性腺所属时期);3)胚胎发育组(包括受精卵、2~4细胞期、32细胞期、囊胚期、担轮幼虫期、D型幼虫期、壳顶幼虫期)。用于RNA提取的样品经液氮速冻并转移至-80 ℃保存。此过程所有用具均由DEPC处理或者180 ℃烘箱烘烤6 h。
1.2 主要试验试剂及仪器
RNA提取试剂盒E.Z.N.A Mollusc RNA Kit购自OMEGA公司,DNase I和DNase I buffer均购自Fermentas公司,反转录试剂盒ReverTra Ace qPCR RT Kit SYBR Green Realtime PCR Master Mix购自TOYOBO公司。荧光定量PCR仪(Roche LightCycler 480),高速冷冻离心机(Eppendorf 5810R),分光光度计(Quawell Q5000)。
1.3 方法
1.3.1 引物设计与合成
根据GenBank中合浦珠母贝的18S rRNA、GAPDH、β-actin、EF-1α基因的mRNA序列设计引物,其中GAPDH合成2对引物。为确保荧光染料同PCR产物的结合,所选序列均为线性mRNA序列,引物的扩增长度在93~152 bp之间。所有引物均由华大基因生物工程有限公司合成,Nuclease-free Water溶解引物,置于-20 ℃保存备用。
1.3.2 总RNA提取
每个样品取约100 mg,按照OMEGA Mollusc RNA Kit说明书分别提取总RNA,Nuclease-Free Water溶解总RNA,通过1.2%的琼脂糖凝胶电泳和分光光度计检测总RNA的质量和浓度。
1.3.3 cDNA合成
每个样品的总RNA分别取1 μg,DNA酶Ⅰ(RNase-free DNase Ⅰ,美国Fermentas出品)37 ℃处理30 min,以去除基因组DNA的污染,各加入1 μL 10 mmol · L-1 EDTA,振荡混匀后,65 ℃ 10 min以灭活DNA酶Ⅰ;参照ReverTra Ace反转录试剂盒(日本TOYOBO出品)说明书合成第一链cDNA。反应体系为10 μL,反应程序为37 ℃ 15 min,98 ℃ 5 min,4 ℃保存,置-20 ℃备用。
1.3.4 PCR片段序列分析
将上述引物扩增得到的PCR产物进行亚克隆,送华大基因公司测序,并用DNAstar 5.0进行序列分析。
1.3.5 荧光定量PCR标准曲线的绘制和扩增效率分析
利用cDNA第一链为模板,依次进行梯度稀释,共设5个试验梯度,每个梯度稀释10倍,即cDNA初始模板浓度分别为100、10-1、10-2、10-3和10-4。分别进行荧光定量PCR,根据定量结果以cDNA初始模板的log值为横坐标,测得的Ct值为纵坐标,绘制标准曲线,计算其扩增效率。
1.3.6 荧光定量PCR
荧光定量PCR反应在Roche LightCycler 480 real-time PCR system进行。参照SYBRⓇ Green Real time PCR Master Mix(日本TOYOBO出品)试剂盒说明书准备反应体系为SYBR Green mix5 μL、上下游引物各0.2 μL(10 pmol · μL-1)、反应模板0.5 μL和Nuclease-free Water 4.1 μL,经试验摸索得到优化的PCR反应条件为94 ℃ 1 min;40个循环,94 ℃ 15 s,55 ℃ 15 s,72 ℃ 1 min;80~85 ℃(收集荧光,不同基因有所不同),反应完成后进行溶解曲线分析,以确定PCR产物的质量和结果的可信度。组织样品和性腺发育样品各设3个生物学重复,每个qPCR反应设3个上样重复。
1.3.7 数据分析和处理
数据由罗氏lightcycler-480-Software 1.5导出至Excel 2007和SPSS 19.0,利用BestKeeper、geNorm和NormFinder软件对各候选内参基因的表达稳定性进行分析。BestKeeper软件分析是基于Ct值,并通过标准变异系数(SD)和变异相关系数(CV)来判断内参基因的稳定性,SD、CV值越小,则该内参基因表达越稳定[11, 20];VANDESOMPELE等[9]编写的geNorm程序是根据M值(某一个内参基因与其他所选内参基因表达水平的两两比值经对数变换,计算其平均标准差,作为基因表达稳定度的平均值[21])对选择的内参基因稳定性进行排序,从而找出合适的内参基因。M值越小则说明该内参基因越稳定;NormFinder是ANDERSEN等[10]研发的另一种用于选择合适内参基因的程序,原理是通过程序运行生成基因表达的稳定值,稳定值越低则表明内参基因表达越稳定。
2. 结果
2.1 内参基因的选择
根据文献记载及GenBank上已知的序列信息,笔者研究筛选了4个功能不同的常用管家基因作为候选基因。利用Primer Premier 5.0和Oligo 6.0软件,设计荧光定量PCR引物并进行检测,其中GAPDH设计了2对引物。PCR扩增产物经测序均与目标序列100% 一致。普通RT-PCR电泳检测均为单一条带(图 1)。采用10倍梯度稀释后构建4个基因的标准曲线,PCR扩增效率和R2值见表 1。PCR扩增效率显示,4个看家基因的扩增效率在0.905~0.960,R2值均在0.99以上,说明标准曲线的线性关系很好。而由EF-1α和GAPDH2的溶解曲线图(图 2)看到2对引物得到的溶解曲线不是单峰,说明这2对引物的反应特异性不高,或者是引物和模板的匹配程度不高,舍弃EF-1α和GAPDH2,由于马氏珠母贝中目前得到的EF-1α序列不完整(AB205403.1),对引物设计有所限制,因此对其他3个基因进行后续试验。
表 1 侯选基因的扩增参数Table 1 Amplification parameters for candidate reference genes基因
geneGenBank
登录号
GenBank accession No.引物序列(5′→3′)
primer sequence扩增片段/bp
amplification size斜率
slope扩增效率
amplification efficiency回归系数
R2β-肌动蛋白β-actin AB252571.1 F: TGGTATGGGACAGAAGGAC
R: GACAATGCCGTGCTCAAT94 -3.434 0.950 0.998 延伸因子-1α EF-1α AB205403.1 F: CCTGGCCACAGAGATTTCAT
R: AATTCCCCAACACCAGCAG93 -3.356 0.926 0.999 甘油醛-3-磷酸脱氢酶GAPDH1 AB205404.1 F : TGGCATTGAGGAAGGTTTG
R: GTGGAGGATGGTATGATGTTAGA129 -3.352 0.960 0.998 甘油醛-3-磷酸脱氢酶GAPDH2 AB205404.1 F: TATTTCTGCACCGTCTGCTG
R: ATCTTGGCGAGTGGAGCTAA129 -3.400 0.938 0.999 18S核糖体RNA 18S rRNA AY877529.1 F: CGTTTCAACAAGACGCCAGTAG
R: ACGAAAAAAAGGTTTGAGAGACG152 -3.463 0.905 0.998 2.2 内参基因表达的稳定性分析
2.2.1 Ct值比较
一个基因的表达丰度越低,Ct值越大,反之则越小。3个候选内参基因在各组别的Ct值如图 3,表明在不同组织及不同发育阶段,每个内参基因的表达水平都有一定的变化。其中GAPDH在3个组别中平均值分别为30.92±3.85、23.06±2.17和24.88±2.84。Ct值所在范围均较大,基因表达丰度低。18S在性腺发育组和胚胎发育组中的表达丰度最高,均值为16.55±1.64和19.01±1.87;β-actin在组织分布组表达丰度最高,均值分别为23.65±1.42。
2.2.2 BestKeeper软件分析
从表 2数据可以看出,在合浦珠母贝的不同组织中β-actin最稳定,其次为18S rRNA和GAPDH;在性腺发育的不同时期,18S rRNA表达最稳定,其次为β-actin和GAPDH;在胚胎发育的不同时期,β-actin表达最稳定,其次为18S rRNA和GAPDH。
表 2 3个候选内参基因在各组织、性腺发育不同时期及胚胎发育不同时期中的表达稳定性(BestKeeper软件)Table 2 Expression stability of 3 candidate reference genes in different tissues, different developmental stages of gonads and embryos calculated by BestKeeper参数
factor各组织
tissue性腺发育不同时期
different gonadal developmental stage胚胎发育不同时期
different embryonic developmental stage18S核糖体RNA
18S rRNAβ-肌动蛋白
β-actin甘油醛-3-磷酸脱氢酶
GAPDH18S核糖体RNA
18S rRNAβ-肌动蛋白
β-actin甘油醛-3-磷酸脱氢酶
GAPDH18S核糖体RNA
18S rRNAβ-肌动蛋白
β-actin甘油醛-3-磷酸脱氢酶
GAPDHCt值的算术平均数(AM [Ct]) arithmetic mean of Ct 25.00 23.65 30.92 16.55 20.30 23.06 19.39 20.00 24.88 Ct值的最小值(Min [Ct]) Min values of Ct 21.18 21.54 23.51 13.87 16.75 17.02 14.42 16.03 20.59 Ct值的最大值(Max [Ct]) Max values of Ct 27.97 27.23 39.29 27.10 26.50 28.67 26.15 24.47 30.32 Ct值的标准差(SD [±Ct]) standard deviation of Ct 1.75 1.11 2.88 1.16 1.51 1.63 1.88 1.60 2.42 Ct值的变异系数(CV [%Ct]) coefficient of variance expressed as a percentage on Ct level 6.98 4.70 9.31 6.99 7.46 7.08 9.68 7.99 9.71 2.2.3 geNorm软件分析
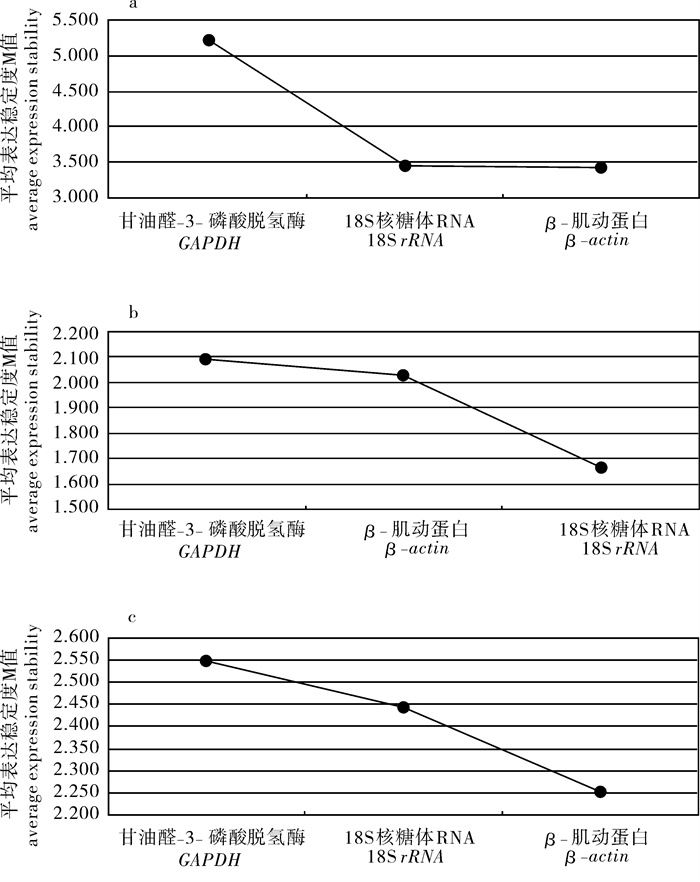
将样本的相对定量值分别输入geNorm软件,根据软件分析结果,18S rRNA、β-actin和GAPDH在合浦珠母贝外套膜、闭壳肌、性腺、鳃等组织中,平均表达稳定度值M分别是3.449、3.425和5.236,由高到低依次排序为β-actin>18S rRNA>GAPDH(图 4-a);在合浦珠母贝雌、雄性腺发育的增殖期、生长期、成熟期和排放期,18S rRNA、β-actin和GAPDH平均表达稳定度M分别是1.665、2.024和2.092,表达稳定度由高到低依次排序为18S rRNA>β-actin>GAPDH(图 4-b);18S rRNA、β-actin和GAPDH在合浦珠母贝胚胎发育的不同时期如2~4细胞期、囊胚期、D型幼虫期等,平均表达稳定度M分别是2.443、2.253和2.547,表达稳定度由高到低依次排序为β-actin>18S rRNA>GAPDH(图 4-c)。
2.2.4 NormFinder软件分析
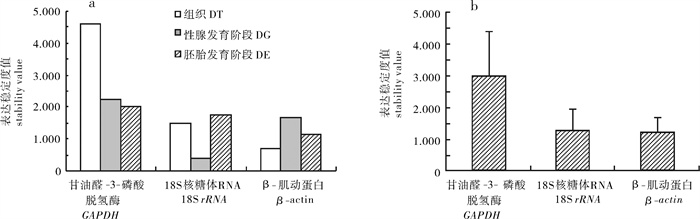
软件分析结果与BestKeeper和GeNorm软件分析结果基本一致。在不同组织中表达稳定性从高到低依次为β-actin>18S>GAPDH,性腺发育不同阶段3个基因的表达稳定性从高到低依次为18S>β-actin>GAPDH,胚胎发育的不同阶段3个基因的表达稳定性从高到低依次为β-actin>18S>GAPDH(图 5-a)。3个组别3个基因表达稳定值的均值分别为2.954±1.421, 1.219±0.728和1.176±0.486(图 5-b)。
![]() 图 5 3个候选内参基因在合浦珠母贝不同组织(DT)、性腺发育阶段(DG)、胚胎发育阶段(DE)的表达稳定性(a),3个内参基因表达稳定性的均值(b)(NormFinder软件)Fig. 5 Average expression stability of 3 candidate reference genes in different tissues (DT), at different developmental stages of gonads (DG) and embryos (DE) from P.fucata (a); mean value of expression stability of 3 reference genes calculated (b) by NormFinder
图 5 3个候选内参基因在合浦珠母贝不同组织(DT)、性腺发育阶段(DG)、胚胎发育阶段(DE)的表达稳定性(a),3个内参基因表达稳定性的均值(b)(NormFinder软件)Fig. 5 Average expression stability of 3 candidate reference genes in different tissues (DT), at different developmental stages of gonads (DG) and embryos (DE) from P.fucata (a); mean value of expression stability of 3 reference genes calculated (b) by NormFinder3. 讨论
在实时荧光定量PCR中,内参基因被用来作为数据标准化的对照,以校正作为模板的cDNA所存在的数量差异[22]。近年来,关于不同物种内参基因的选择已有很多报道[23-24]。当前,在合浦珠母贝中使用较多的内参基因有GAPDH、18S rRNA、β-actin和EF-1α[25-27]等。笔者研究根据文献选择了这4个功能不同的管家基因(其中EF-1α由于引物设计的原因没有后续研究),对其在合浦珠母贝
不同组织中、性腺发育不同时期、胚胎发育不同阶段等样品中的表达差异进行探讨,并分别采用BestKeeper,geNorm、NormFinder 3种算法对基因稳定性进行计算,以筛选表达相对稳定的基因作为内参基因。为了尽可能减少试验误差,样品制备的整个过程都严格按照试验要求进行,总RNA制备、逆转录、荧光定量PCR均采用相同方法在同一试验仪器进行。荧光定量PCR使用SYBR Green DNA结合染料,与其他荧光染料如Taqman、Beacon等相比,具有试验设计简单,不需合成特异探针,通用型好,成本低等优点,并且能够通过溶解曲线分析检验扩增反应的特异性。geNorm、NormFinder和BestKeeper是目前内参基因筛选常用的几种工具。geNorm通过基因配对的形式筛选最不稳定的基因,将其余基因重新配对并筛选出适当数目合适的内参基因。geNorm可以对任意数量管家基因进行筛选,选出2个以上作为内参基因,更有助于校正系统偏差。NormFinder则是基于方差分析对内参基因的稳定性进行评价,产生基因表达稳定值,然后按照稳定值大小排序,表达稳定值最小的基因最稳定,缺点是只能选择一个合适的内参基因作为参照。BestKeeper是直接根据Ct值进行计算,不但可以分析内参基因的稳定性,还可以用于比较目标基因的表达水平。缺点是只能计算一次配对相关系数,当排除某个基因后该程序不能重新计算[28]。因此笔者研究使用这3种软件进行分析,有助于相互验证以确定试验结果的准确性。由数据可知,3种内参基因筛选工具分析得出的结果一致,即表达较为稳定的2个内参基因是18S rRNA和β-actin。其中在不同组织及胚胎发育不同时期β-actin表达最稳定,而在性腺发育不同时期18S rRNA表达最稳定。YOKO等[27]对于合浦珠母贝几个内参基因的研究中18S为最稳定表达的基因,与笔者的结论略有不同。YOKO等仅选择胚胎发育不同时间点及稚贝样品,用3个内参基因的相对表达量分析其稳定性,研究方法较单一且不够深入。而笔者研究中不仅比较了Ct值大小,还利用几种专业的内参基因稳定性分析软件。另外所取样品时间点与之不同,可能造成数据结论不同。虽然所选内参基因相同,但是设计引物序列不同,扩增所得片段位置不同,因此扩增效率及Ct值变化均与笔者所用引物不同,也会造成结论不同。
GAPDH在3个试验组均表现为稳定性最差,在3个组别中GAPDH的表达水平差别最大(表 2)。Ct值分别在23.51~39.29,17.02~28.67和20.59~30.32变化。可见GAPDH并不适合广泛作为合浦珠母贝定量表达的内参基因。GAPDH是糖酵解和糖异生过程的关键酶,参与糖酵解过程中第一个ATP形成,在不同功能的细胞类型中表达差异较大。PEREZ等[29]和BUSTIN[30]发现GAPDH mRNA在不同癌组织(包括肺癌、乳腺癌、肾细胞癌等)中的表达比正常组织高,在低氧、胰岛素、表皮生长因子、激素等因子的刺激下,培养细胞的GAPDH mRNA表达水平也各不相同。另外笔者研究中GAPDH基因设计了2对引物,第二对引物的溶解曲线不是单峰,不符合实时定量PCR的要求,推测该引物扩增的片段内重复序列较多或者引物与模板容易产生错配,导致产物不单一。说明在实时定量PCR中引物的设计和验证至关重要。
GAPDH、β-actin、18S rRNA被超过90%的定量PCR研究单独用作参照基因[31]。然而有大量研究认为管家基因在不同细胞类型和不同生理状态下的表达并不是恒定不变的[32-33]。RADONIC等[8]研究表明内参基因β-actin和GAPDH均受有丝分裂原刺激的调节。这使传统的管家基因作为qRT-PCR内参基因的功能受到挑战。研究人员尝试通过基因表达芯片数据和EST数据库等方法筛选新的可以稳定表达的基因作为内参基因[34-35]。如在小麦(Triticum aestivum)和大麦(Hordeum vulgare)中,一些新的内参基因的筛选都利用Unigene和TIGR数据库,并且发现这些新的内参基因在表达水平上比传统的内参基因具有更好的稳定性[36]。有研究人员通过基因芯片技术扫描拟南芥(Arabidopsis thaliana)和水稻(Oryza sativa)的整个基因组来发掘新的内参基因[37-38]。另外,在追踪基因表达的微小变化时,与使用2个以上的内参基因相比,单独使用一个内参基因往往不能得到准确的定量结果,在相对表达的分析中可能会导致更大的误差。选择2个以上内参基因有助于得到更为可靠的结果。由于文章所分析基因为合浦珠母贝几种常见的管家基因,数目较少,无法进行最适内参基因数目的分析。后续试验将筛选更多稳定表达的基因进行分析。
-
图 5 3个候选内参基因在合浦珠母贝不同组织(DT)、性腺发育阶段(DG)、胚胎发育阶段(DE)的表达稳定性(a),3个内参基因表达稳定性的均值(b)(NormFinder软件)
Figure 5. Average expression stability of 3 candidate reference genes in different tissues (DT), at different developmental stages of gonads (DG) and embryos (DE) from P.fucata (a); mean value of expression stability of 3 reference genes calculated (b) by NormFinder
表 1 侯选基因的扩增参数
Table 1 Amplification parameters for candidate reference genes
基因
geneGenBank
登录号
GenBank accession No.引物序列(5′→3′)
primer sequence扩增片段/bp
amplification size斜率
slope扩增效率
amplification efficiency回归系数
R2β-肌动蛋白β-actin AB252571.1 F: TGGTATGGGACAGAAGGAC
R: GACAATGCCGTGCTCAAT94 -3.434 0.950 0.998 延伸因子-1α EF-1α AB205403.1 F: CCTGGCCACAGAGATTTCAT
R: AATTCCCCAACACCAGCAG93 -3.356 0.926 0.999 甘油醛-3-磷酸脱氢酶GAPDH1 AB205404.1 F : TGGCATTGAGGAAGGTTTG
R: GTGGAGGATGGTATGATGTTAGA129 -3.352 0.960 0.998 甘油醛-3-磷酸脱氢酶GAPDH2 AB205404.1 F: TATTTCTGCACCGTCTGCTG
R: ATCTTGGCGAGTGGAGCTAA129 -3.400 0.938 0.999 18S核糖体RNA 18S rRNA AY877529.1 F: CGTTTCAACAAGACGCCAGTAG
R: ACGAAAAAAAGGTTTGAGAGACG152 -3.463 0.905 0.998 表 2 3个候选内参基因在各组织、性腺发育不同时期及胚胎发育不同时期中的表达稳定性(BestKeeper软件)
Table 2 Expression stability of 3 candidate reference genes in different tissues, different developmental stages of gonads and embryos calculated by BestKeeper
参数
factor各组织
tissue性腺发育不同时期
different gonadal developmental stage胚胎发育不同时期
different embryonic developmental stage18S核糖体RNA
18S rRNAβ-肌动蛋白
β-actin甘油醛-3-磷酸脱氢酶
GAPDH18S核糖体RNA
18S rRNAβ-肌动蛋白
β-actin甘油醛-3-磷酸脱氢酶
GAPDH18S核糖体RNA
18S rRNAβ-肌动蛋白
β-actin甘油醛-3-磷酸脱氢酶
GAPDHCt值的算术平均数(AM [Ct]) arithmetic mean of Ct 25.00 23.65 30.92 16.55 20.30 23.06 19.39 20.00 24.88 Ct值的最小值(Min [Ct]) Min values of Ct 21.18 21.54 23.51 13.87 16.75 17.02 14.42 16.03 20.59 Ct值的最大值(Max [Ct]) Max values of Ct 27.97 27.23 39.29 27.10 26.50 28.67 26.15 24.47 30.32 Ct值的标准差(SD [±Ct]) standard deviation of Ct 1.75 1.11 2.88 1.16 1.51 1.63 1.88 1.60 2.42 Ct值的变异系数(CV [%Ct]) coefficient of variance expressed as a percentage on Ct level 6.98 4.70 9.31 6.99 7.46 7.08 9.68 7.99 9.71 -
[1] GIULIETTI A, OVERBERGH L, VALCKX D, et al. An overview of real-time quantitative PCR: applications to quantify cytokine gene expression[J]. Methods, 2001, 25(4): 386-401. doi: 10.1006/meth.2001.1261
[2] KUBISTA M, ANDRADE J M, BENGTSSON M, et al. The real-time polymerase chain reaction[J]. Mol Aspects Med, 2006, 27(2/3): 95-125.
[3] VANGUILDER H D, VRANA K E, FREEMAN W M. Twenty-five years of quantitative PCR for gene expression analysis[J]. Biotechniques, 2008, 44(5): 619-626. doi: 10.2144/000112776
[4] WONG M L, MEDRANO J F. Real-time PCR for mRNA quantitation[J]. Biotechniques, 2005, 39(1): 75-85. doi: 10.2144/05391RV01
[5] BUSTIN S A. Quantification of mRNA using real-time reverse transcription PCR (RT-PCR): trends and problems[J]. Mol Endocrinol, 2002, 29(1): 23-39. doi: 10.1677/jme.0.0290023
[6] TUNBRIDGE E M, EASTWOOD S L, HARRISON P J. Changed relative to what?Housekeeping genes and normalization strategies in human brain gene expression studies[J]. Biol Psychiat, 2010, 69(2): 173-179.
[7] SUZUKI T, HIGGINS P J, CRAWFORD D R. Control selection for RNA quantitation[J]. Biotechniques, 2000, 29(2): 332-337. doi: 10.2144/00292rv02
[8] RADONIC A, THULKE S, MACKAY I M, et al. Guideline to reference gene selection for quantitative real-time PCR[J]. Biochem Bioph Res Co, 2004, 313(4): 856-862. doi: 10.1016/j.bbrc.2003.11.177
[9] VANDESOMPELE J, DE PRETER K, PATTYN F, et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes[J]. Genome Biol, 2002, 3(7): 1-11. https://xueshu.baidu.com/usercenter/paper/show?paperid=106j0mf0wy7h0rj0mp5506j0a4321095&site=xueshu_se&hitarticle=1
[10] ANDERSEN C L, JENSEN J L, RNTOFT T F. Normalization of real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets[J]. Cancer Res, 2004, 64(15): 5245-5250. doi: 10.1158/0008-5472.CAN-04-0496
[11] PFAFFL M W, ALES T, CHRISTIAN P, et al. Determination of stable housekeeping genes, differentially regulated target genes, and sample integrity: BestKeeper-Excel-based tool using pair-wise correlations[J]. Biotechnol Lett, 2004, 26(6): 509-515. doi: 10.1023/B:BILE.0000019559.84305.47
[12] CICINNATI V R, SHEN Q L, SOTIROPOULOS G C, et al. Validation of putative reference genes for gene expression studies in human hepatocellular carcinoma using real-time quantitative RT-PCR[J]. BMC Cancer, 2008, 8(11): 350. DOI: 10.1186/1471-2407-8-350.
[13] HUIS R, HAWKINS S, NEUTELINGS G. Selection of reference genes for quantitative gene expression normalization in flax (Linum usitatissimum)[J]. BMC Plant Biol, 2010, 10(4): 71. DOI: 10.1186/1471-2229-10-71.
[14] EXPOSITO-RODRIGUEZ M, BORGES A A, BORGES-PEREZ A, et al. Selection of internal control genes for quantitative real-time RT-PCR studies during tomato development process[J]. BMC Plant Biol, 2008, 8(12): 131. DOI: 10.1186/1471-2229-8-131.
[15] LIBAULT M, THIBIVILLIERS S, BILGIN D D, et al. Identification of four soybean reference genes for gene expression normalization[J]. The Plant Genome, 2008, 1(1): 44-54. https://xueshu.baidu.com/usercenter/paper/show?paperid=033ae767139c586f34a44d3c26de60fa&site=xueshu_se&hitarticle=1
[16] ZHANG Z B, HU J Y. Development and validation of endogenous reference genes for expression profiling of Medaka (Oryzias latipes) exposed to endocrine disrupting chemicals by quantitative real-time PCR[J]. Toxicol Sci, 2007, 95(2): 356-368. doi: 10.1093/toxsci/kfl161
[17] INGERSLEV H C, PETTERSEN E F, JAKOBSEN R A, et al. Expression profling and validation of reference gene candidates in immune relevant tissues and cells from Atlantic salmon (Salmo salar L. )[J]. Mol Immunol, 2006, 43(8): 1194-1201. doi: 10.1016/j.molimm.2005.07.009
[18] 鲍相渤, 刘卫东, 姜冰, 等. 内参基因在虾夷扇贝定量PCR中表达稳定性的比较[J]. 水产科学, 2011, 30(11): 603-608. https://xueshu.baidu.com/usercenter/paper/show?paperid=018744c2d22bbf8f839dd93525c3754a&site=xueshu_se [19] 杨桂梅, 鲍宝龙, 任大明. 3-磷酸甘油醛脱氢酶、β-肌动蛋白和18S rRNA作为相对定量的内标在牙鲆发育阶段的稳定性比较[J]. 上海水产大学学报, 2005, 14(1): 84-88. https://xueshu.baidu.com/usercenter/paper/show?paperid=4dc56e17a5bd9ba07ea174f7bc668d5c&site=xueshu_se&hitarticle=1 [20] 侯维海, 孙鹏, 陈全家, 等. 地黄实时定量PCR内参基因的筛选[J]. 中国农学通报, 2011, 27(1): 76-82. https://xueshu.baidu.com/usercenter/paper/show?paperid=1957bc902b8b9daa4e4b29f0a0b43c59&site=xueshu_se [21] 王忠伟, 郭景茹, 王建发, 等. 新生籽鹅组织RT-PCR分析中内参基因的选择[J]. 中国兽医学报, 2012, 32(3): 427-431. https://xueshu.baidu.com/usercenter/paper/show?paperid=2e0279cb02680d407dabb8a534ef57c4&site=xueshu_se&hitarticle=1 [22] GUTIERREZ L, MAURIAT M, GU NIN S, et al. The lack of a systemic validation of reference genes: a serious pitfall undervalued in reverse transcription-polymerase chain reaction (RT-PCR) analysis in plants[J]. Plant Biotech J, 2008, 6(6): 609-618. doi: 10.1111/j.1467-7652.2008.00346.x
[23] Van HIEL M B, Van WIELENDAELE P, TEMMERMAN L, et al. Identification and validation of housekeeping genes in brains of the desert locust Schistocerca gregaria under different developmental conditions[J]. BMC Mol Biol, 2009, 10(6): 56. DOI: 10.1186/1471-2199-10-56.
[24] SCHARLAKEN B, De GRAARF D C, GOOSSENS K, et al. Reference gene selection for insect expression studies using quantitative real time PCR: the head of the honeybee, Apismel liferia, after a bacterial challenge[J]. J Insect Sci, 2008, 8(4): 33. DOI: 10.1673/031.008.3301.
[25] WANG Z, WU Z, JIAN J, et al. Cloning and expression of heat shock protein 70 gene in the haemocytes of pearl oyster (Pinctada fucata, Gould 1850) responding to bacterial challenge[J]. Fish Shellfish Immunol, 2009, 26(4): 639-645. doi: 10.1016/j.fsi.2008.10.011
[26] TAKESHI T, KAZUYOSHI E. Biphasic and dually coordinated expression of the genes encoding major shell matrix proteins in the pearl oyster Pinctada fucata[J]. Mar Biotechnol, 2006, 8(1): 52-61. doi: 10.1007/s10126-005-5037-x
[27] YOKO M, TAKUMA N, HIDEO A, et al. Expression of genes responsible for biomineralization of Pinctada fucata during development[J]. Comp Biochem Phys, 2010, 155(3): 241-248. doi: 10.1016/j.cbpb.2009.11.009
[28] WOOD S H, CLEMENTS D N, MCEWAN N A, et al. Reference genes for canine skin when using quantitative real-time PCR[J]. Vet Immunol Immunopathol, 2008, 126(3/4): 392-395. https://xueshu.baidu.com/usercenter/paper/show?paperid=4b2b292e62ef1968abd38ac7045acd82&site=xueshu_se&hitarticle=1
[29] PEREZ R, TUPAC-YUPANQUI I, DUNNER S. Evaluation of suitable reference genes for gene expression studies in bovine muscular tissue[J]. BMC Mol Biol, 2008, 9(11): 79. DOI: 10.1186/1471-2199-9-79.
[30] BUSTIN S A. Quantification of mRNA using real-time reverse transcription PCR (RT-PCR): trends and problems[J]. J Mol Endocrinol, 2002, 29(1): 23-39. doi: 10.1677/jme.0.0290023
[31] 陈凤花, 王琳. 实时荧光定量RT-PCR内参基因的选择[J]. 临床检验杂志, 2005, 23(5): 393-395. https://xueshu.baidu.com/usercenter/paper/show?paperid=bb750fc3c3e61c0e55920d47303866bb&site=xueshu_se&hitarticle=1 [32] BARBER R D, HARMER D W, COLEMAN R A, et al. GAPDH as a housekeeping gene: analysis of GAPDH mRNA expression in a panel of 72 human tissues[J]. Physiol Genomics, 2005, 21(3): 389-395. doi: 10.1152/physiolgenomics.00025.2005
[33] ROMANOWSKI T, MARKIEWICZ A, BEDNARZ N, et al. House-keeping genes as a reference in quantitative real-time RT-PCR[J]. Postepy Hig Med Dosw, 2007, 28(61): 500-510.
[34] FACCIOLI P, PECCHIONI N, CATTIVELLI L, et al. Expression sequence tags from cold-acclimatized barley can identify novel plant genes[J]. Plant Breeding, 2001, 120(6): 497-502. doi: 10.1046/j.1439-0523.2001.00652.x
[35] FACCIOLI P, PROVERO P, HERRMANN C, et al. From single genes to co-expression networks: extracting knowledge from barley functional genomics[J]. Plant Mol Biol, 2005, 58(5): 739-50. doi: 10.1007/s11103-005-8159-7
[36] PAOLACCI A R, TANZARELLA O A, PORCEDDU E, et al. Identification and validation of reference genes for quantitative RT-PCR normalization in wheat[J]. BMC Mol Biol, 2009, 10(2): 11. DOI: 10.1186/1471-2199-10-11.
[37] CZECHOWSKI T, STITT M, ALTMAN T, et al. Genome-wide identification and testing of superior reference genes for transcript normalization in Arabidopsis[J]. Plant Physiol, 2005, 139(1): 5-17. doi: 10.1104/pp.105.063743
[38] MUKECH J. Genome-wide identification of novel internal control genes for normalization of gene expression during various stages of development in rice[J]. Plant Sci, 2009, 176(5): 702-706. doi: 10.1016/j.plantsci.2009.02.001
-
期刊类型引用(14)
1. 费越越,南星羽,余路,罗扬,高钟元,许丹. 异育银鲫内参基因的筛选. 水产科学. 2020(03): 306-315 .  百度学术
百度学术
2. 张家炜,邬婧,周丹,郑哲,王庆恒. 光裸星虫(Sipunculus nudus)不同发育时期卵细胞内参基因的筛选. 基因组学与应用生物学. 2020(02): 575-583 .  百度学术
百度学术
3. 王爱云,曹滕飞,吕家森,丛明. 亚硝态氮胁迫下菲律宾蛤仔实时定量PCR内参基因的筛选. 生态毒理学报. 2019(01): 153-160 .  百度学术
百度学术
4. 车志群,孙仁杰,刘文爱,廖思明,阎冰. 六溴环十二烷胁迫下红树蚬荧光定量PCR内参基因稳定性分析. 广西科学院学报. 2018(03): 242-249 .  百度学术
百度学术
5. 李耀国,孙彤,武文丽,陈永志,蒋丹尼. 马氏珠母贝GAPDH基因克隆及其在去甲基化受精卵中的表达分析. 江苏农业科学. 2018(10): 38-42 .  百度学术
百度学术
6. 牟政强,闫路路,王化敏,霍忠明,闫喜武. 菲律宾蛤仔不同发育时期及成体不同组织中内参基因筛选. 水产学报. 2018(05): 663-672 .  百度学术
百度学术
7. 张磊,王锦旭,杨贤庆,魏涯,杨少玲. 海洋动物多糖的研究进展. 食品工业. 2018(01): 211-215 .  百度学术
百度学术
8. 张磊,王锦旭,杨贤庆,魏涯,岑剑伟. 响应面法优化合浦珠母贝糖胺聚糖咀嚼片的制备工艺. 食品工业科技. 2018(17): 159-166 .  百度学术
百度学术
9. 张磊,王锦旭,杨贤庆,魏涯,杨少玲. 合浦珠母贝糖胺聚糖泡腾片制备工艺优化及其质量分析. 大连海洋大学学报. 2018(05): 644-650 .  百度学术
百度学术
10. 王珍珍,黄桂菊,范嗣刚,刘宝锁,张博,苏家齐,喻达辉. 合浦珠母贝matrilin-1基因的克隆和表达分析. 南方水产科学. 2017(01): 76-84 .  本站查看
本站查看
11. 曹滕飞,丛明,李兆艳,赵建民,吕家森,李成华. 氨态氮对菲律宾蛤仔毒理机制研究的内参基因筛选. 生态毒理学报. 2017(02): 182-190 .  百度学术
百度学术
12. 莫祖英,车志群,廖思明,杨明柳,阎冰. 多氯联苯胁迫下红树蚬荧光定量PCR内参基因的筛选. 广西科学. 2015(03): 350-356 .  百度学术
百度学术
13. 朱文杰,黄桂菊,张东玲,范嗣刚,刘宝锁,毕晓敏,喻达辉. 合浦珠母贝表皮生长因子样(EGF-like)基因的克隆与表达分析. 水产学报. 2015(05): 648-657 .  百度学术
百度学术
14. 闫芳,罗少杰,田群莉,焦钰,邓岳文. 马氏珠母贝外套膜中央膜与边缘膜荧光定量PCR分析中内参基因的筛选. 广东海洋大学学报. 2014(06): 6-11 .  百度学术
百度学术
其他类型引用(9)




 下载:
下载:


 粤公网安备 44010502001741号
粤公网安备 44010502001741号
