Screening of antagonistic bacteria against visceral white-spots disease of Larimichthys crocea and preliminary study on its biological characteristics
-
摘要: 为实现大黄鱼 (Larimichthys crocea) 内脏白点病的生物防控,推进水产养殖用药减量,从健康大黄鱼肠道中筛选出对其内脏白点病病原菌——杀香鱼假单胞菌 (Pseudomonas plecoglossicid) 有拮抗作用的益生菌。采用琼脂扩散法筛选菌株,通过生理生化特征以及分子生物学分析,对菌株进行鉴定,并评价其溶血性、药敏性、安全性、产酶能力及抗菌广谱性。从健康大黄鱼肠道中初步分离出37株潜在益生菌,通过拮抗试验筛选出3株具有拮抗效果的菌株,分别命名为P1-17、P2-33和P3-11。通过生理生化特性及16S rRNA 测序分析,菌株P1-17和P2-33鉴定为贝莱斯芽孢杆菌 (Bacillus velezensis),菌株P3-11鉴定为粪肠球菌 (Enterococcus faecalis);溶血性试验和药敏试验结果表明3株菌均无显著溶血圈,且所含耐药因子较少,不具备潜在致病性;人工回感试验证实,3株拮抗菌对健康大黄鱼无致病性;抗菌谱测定结果表明,2株芽孢杆菌对溶藻弧菌 (Vibrio alginolyticus)、哈维氏弧菌 (V. harveyi)、美人鱼发光杆菌 (Photobacterium damselae) 等多种水产病原菌具有拮抗效果,同时2株芽孢杆菌具有产淀粉酶和蛋白酶的能力;而该株粪肠球菌只对杀香鱼假单胞菌有拮抗作用。研究结果可为后续大黄鱼肠道益生菌的筛选和应用提供科学依据。Abstract: In order to achieve the biological prevention and control of visceral white-spots disease of Larimichthys crocea, and promote the reduction of drug use in aquaculture, we isolated and screened the probiotics with an antagonistic effect on Pseudomonas plecoglossicid, which is a pathogen of visceral white spot disease in L. crocea, from the intestine of healthy L. crocea. The strains were screened by agar diffusion method, identified by physiological and biochemical characteristics and molecular biology analysis, and evaluated for hemolysis, drug sensitivity, safety, enzyme production ability and broad-spectrum antibacterial activity. Thirty-seven strains of potential probiotics were isolated and the three most potent strains were further characterized for their probiotic potential, named as P1-17, P2-33 and P3-11. The three most promising isolates were identified by sequencing the 16S rRNA gene and physiological and biochemical characteristics. Strains P1-17 and P2-33 were identified as Bacillus velezensis and P3-11 was identified as Enterococcus faecalis. According to the hemolytic test and disk diffusion method, none of the three strains had sigificant hemolytic rings, containing few drug resistance factors, so they had no potential pathogenicity. The result from the artificial infection safety test confirmed that these three strains of antagonistic bacteria had no pathogenicity to healthy L. crocea. The results of the antimicrobial spectrum show that two strains of Bacillus had an antagonistic effect on common aquatic pathogens such as Vibrio alginolyticus, V. harveyi and Photobacterium damselae. Moreover, two strains of Bacillus could produce amylase and protease, while the strain of E. faecalis only had an antagonistic effect on P. plecoglossicida. The study provides a scientific basis for the subsequent screening and application of intestinal probiotics in L. crocea.
-
大口黑鲈 (Micropterus salmoides),俗称加州鲈,隶属鲈形目、鲈亚目、太阳鱼科、黑鲈属。原自然分布于北美洲,于1983年从中国台湾引入广东省[1]。大口黑鲈适温范围较广,生长快且易起捕,同时其肉质鲜美、无肌间刺,因而深受养殖者和消费者欢迎,在我国多个省市与地区得以迅速推广养殖。近几年大口黑鲈人工配合饲料的突破与应用,使大口黑鲈养殖区域与规模迅速扩大。2017年,我国养殖大口黑鲈年产量已达45.7万吨[2]。但近两三年来养殖大口黑鲈抗病性能下降,病害问题频发。大口黑鲈引进中国已三十多年,虽然这期间也从原产地进行过为数不多的小规模引进,但相对于养殖规模而言,亲本的种质更新极为有限。因此抗病性能下降是否与种质质量与遗传多样性下降有关,进一步开展选择育种的种质遗传基础如何,都亟需对中国现有大口黑鲈养殖群体的遗传多样性进行评估。
微卫星 (microsatellite) 标记,又称简单重复序列 (simple sequence repeats, SSR) 或短串联重复序列 (simple tandem repeats, STR),是均匀分布于真核生物基因组中的简单重复序列,由2~6个核苷酸的串联重复片段构成,具有分布广泛、共显性遗传、多态性高等特点,广泛应用于多个生命学科领域[3-4]。20世纪90年代初,微卫星标记开始应用于鱼类遗传及育种研究中[5]。利用微卫星研究大口黑鲈的遗传多样性在近年有所报道。梁素娴等[6]用21对微卫星引物对广东的3个大口黑鲈养殖群体进行遗传多样性分析发现,3个不同群体平均杂合度处于中等偏低水平 (期望杂合度He分别为0.375、0.403和0.368),遗传多样性不高。李镕等[7]利用11对微卫星引物对大口黑鲈第2~第4代选育群体的遗传结构进行分析发现,与F2相比,F4的遗传多样性减少8.76%,表明选育群体的遗传多样性在逐步下降。樊佳佳等[8]用43个微卫星标记对目前国内养殖的大口黑鲈群体、2009年引进的佛罗里达亚种、2010年引进的佛罗里达亚种和北方亚种这4个群体进行遗传多样性检测,发现国内养殖的大口黑鲈群体遗传多样性水平明显低于国外新引进的大口黑鲈群体。以上研究表明,我国大口黑鲈养殖群体出现遗传多样性显著下降、种质退化及种群结构单一等问题。
广东省是中国大口黑鲈成鱼的主养区和主要苗种培育基地。2016年广东省大口黑鲈养殖面积约5 333.4 hm2,产量为23.5万吨,占全国总产量的62.8%。广东省大口黑鲈成鱼养殖主要集中在珠江三角洲的佛山市,其养殖产量近20万吨。佛山市的顺德区和南海区有大口黑鲈鱼苗培育专业村,仅南海区九江镇的大口黑鲈鱼苗生产量就接近40亿尾,约占广东全省生产量的75%,占全国生产量的60%[9]。因此佛山地区的大口黑鲈养殖群体极具代表性,研究其养殖群体的遗传多样性和遗传结构,可以反映出中国大口黑鲈养殖群体的遗传多样性现状。本研究选取广东省佛山市3个不同地方的大口黑鲈养殖群体作为研究对象,利用微卫星标记技术对其进行遗传多样性分析,旨在为当前大口黑鲈养殖群体的种质资源调查和进一步的选育种工作提供支撑。
1. 材料与方法
1.1 实验材料
实验所需大口黑鲈样品分别为佛山市南海区西樵镇的西樵群体 (编号为XJ)、九江镇沙头片区的沙头群体 (编号为ST)、九江镇九江片区烟南村的烟南群体 (编号为YN),这3个养殖群体的苗种来自不同苗种场。从3个养殖群体中各随机选取32尾个体,分别剪取尾鳍,–20 ℃保存于95%的乙醇中,用于DNA的提取。
1.2 基因组DNA的提取与检测
利用天根生化科技 (北京)有限公司的海洋动物组织基因组DNA提取试剂盒,提取大口黑鲈尾鳍的基因组DNA。用1%的琼脂糖凝胶进行电泳,并用酶标仪检测其质量和浓度,−20 ℃冰箱保存。
1.3 微卫星引物的筛选与合成
根据GenBank及文献上登录的大口黑鲈微卫星引物序列[10-14],选择并设计合成51对引物,进行预扩增,并用Qsep100全自动核酸蛋白分析系统 (中国,台湾) 检测引物的特异性和多态性,最终确定其中的12对重复性好、特异性强和多态性高的微卫星引物 (表1) 用于研究。引物委托生工生物工程 (上海) 股份有限公司进行合成。各对引物分别带有TAMRA、HEX和FAM的特异性荧光标记,用于扩增和测算目的条带的大小。
表 1 大口黑鲈微卫星引物序列及退火温度Table 1. Primer sequences and annealing temperatures of microsatellite marker位点
loci引物序列 (5'−3')
primer sequence片段长度/bp
size重复序列
repeat sequence退火温度/℃
annealingtemperature参考文献
Referencemdo3 AGGTGCTTTGCGCTACAAGT
CTGCATGGCTGTTATGTTGG119~143 (CA)20 46.2 [11] mdo4 TCTGAACAACTGCATTTAGACTG
CTAATCCCAGGGCAAGACTG151~155 (CA)11 48.6 [11] mdo7 TCAAACGCACCTTCACTGAC
GTCACTCCCATCATGCTCCT179~205 (CA)12 53 [11] lma120 TGTCCACCCAAACTTAAGCC
TAAGCCCATTCCCAATTCTCC204~224 (GT)28 60 [10, 13] MiSaTPW011 CAACATGGACGCTACTAT
CAACCATCACATGCTTCT170~194 (AGAT)13 60 [14] MiSaTPW038 AGTCAACATTCAAACCACTTTCCCAC
TTGTATGTTAGGATCGGCTGATTGTG227~312 (AGAT)6+5+9+5 60 [14] MiSaTPW025 CCAAGGTCAGGTTTAAC
ACCTTTGTGCTGTTCTGTC277~298 (AGAT)11 55 [14] MiSaTPW060 TATAGTTTGGTCCAGCAGGTGGCGT
TGTGGAATGACATTTAGCCGAGGCC294~561 (AGAT)14+10+3+2+4+9+10 60 [14] MiSaTPW116 CCAAACAGCCTACAGAATGTGCCT
AGTCCCTCGACTGAATGTGTGCAA191~219 (AC)21 55 [14] MiSaTPW001 AGTAAAGGACCACCCTTGTCCA
GCCTGGTCATTAGGTTTCGGAG293~299 (AC)16 60 [14] MiSaTPW058 CATTCCTGAGAGACGTGGTTGCTG
TGTGACTTGCTCTGTGAAAGGTGC143~169 (AAGT)6 60 [14] MiSaTPW154 TGCGGGCTAATAGGCCTGTCTG
GCACCACACAATCACACCTGGTAT182~228 (AC)18 55 [14] 1.4 PCR的反应体系与程序
PCR反应总体积20 μL,含有模板DNA 1 μL、上下游引物 (10 μmol·L–1) 各1 μL、ddH2O 7 μL、Premix rTaq (TaKaRa,中国大连) 10 μL,在96孔板中进行PCR反应。扩增程序为94 ℃预变性3 min;94 ℃变性30 s,退火 (各引物退火温度参照表1中的退火温度) 30 s,72 ℃延伸30 s,35个循环;72 ℃延伸10 min,4 ℃保存。
1.5 扩增产物的检测
引物筛选的预扩增产物通过Qsep100全自动核酸蛋白分析系统进行毛细管电泳检测。将PCR扩增产物室温放置2~3 min,开盖后按要求放在样品托上进行毛细管电泳。电泳参数为6 kV下进样2 s再电泳300 s。电泳完毕后,用Q-ANALYZER软件对结果进行矫正与分析,对扩增产物分子量的差异大小进行个体各微卫星基因座的分型。
荧光引物的扩增产物通过STR基因分型检测,由生工生物工程 (上海) 股份有限公司完成。使用毛细管电泳分离STR片段,激光诱导的荧光检测,由设备检测并数据化存储,自动数据采集并分型。荧光颜色分离,计算片段大小,确定各个等位座的基因分型。整个过程通过ABI 310遗传分析仪 (美国) 完成。
1.6 数据统计与分析
通过STR分型确定标记在个体中的基因型,运用POPGENE 32软件进行统计分析,计算各个群体微卫星位点的等位基因数 (Na)、有效等位基因数 (Ne)、观测杂合度 (Ho)、He、Shannon指数 (I)、遗传相似度 (S) 及群体间遗传距离 (Da)。运用FSTAT软件算出遗传分化指数 (FST)。根据Nei氏遗传距离的大小,运用MEGA 5软件构建系统树[15]。运用CERVUS 3.0软件统计出多态信息含量 (polymorphism information content, PIC)[16]。运用ARLEQUIN 3.1软件进行群体间FST计算、群体分子方差分析 (AMOVA)[17]。利用STRUCTURE 2.3软件分析群体的遗传结构[18]。
2. 结果
2.1 微卫星位点筛选
从不同地区采集到的大口黑鲈养殖群体中,随机选取12个大口黑鲈样本,利用51对微卫星引物进行PCR扩增,通过Qsep100全自动核酸蛋白分析系统进行毛细管电泳检测产物(图1),筛选出12对多态性好、特异性强的微卫星引物用于大群体的扩增分析 (表1)。
![]() 图 1 毛细管电泳检测引物MiSaTPW011部分扩增产物绿色箭头指示20 bp align Marker和1 000 bp align Marker;M. size Marker;1~8. 大口黑鲈8个个体的毛细管电泳结果Figure 1. Detection for partial PCR products of MiSaTPW011 with capillary electrophoresisGreen arrows indicate 20 bp align Marker and 1 000 bp align Marker, respectively; M. size Marker; 1−8. output of eight M. salmoides capillary electrophoresis
图 1 毛细管电泳检测引物MiSaTPW011部分扩增产物绿色箭头指示20 bp align Marker和1 000 bp align Marker;M. size Marker;1~8. 大口黑鲈8个个体的毛细管电泳结果Figure 1. Detection for partial PCR products of MiSaTPW011 with capillary electrophoresisGreen arrows indicate 20 bp align Marker and 1 000 bp align Marker, respectively; M. size Marker; 1−8. output of eight M. salmoides capillary electrophoresis2.2 微卫星位点多态性与群体遗传参数
用筛选出的12对微卫星引物扩增3个大口黑鲈群体共96个个体的DNA,将产物进行STR分型 (图2)。结果表明,各对引物在3个群体中均能扩增到目的条带,并体现了程度不一的多态性(表2)。其中,Na为1~6,Ne为1~3.52,I为0~1.427,Ho为0~0.854,He为0~0.720,PIC为0.02~0.693。
表 2 大口黑鲈12个微卫星位点的等位基因数、杂合度及多态信息含量Table 2. Number of alleles, heterozygosity and polymorphic information content of 12 microsatellite loci of M. salmoides位点
locus等位基因数
Na有效等位基因数
NeShannon指数
I观测杂合度
Ho期望杂合度
He多态信息含量
PICmdo3 1 1.00 0.000 0.000 0.000 0.040 mdo4 4 1.89 0.797 0.086 0.473 0.439 mdo7 2 1.13 0.225 0.118 0.112 0.158 lma120 4 2.89 1.211 0.552 0.658 0.604 MISATPW011 4 2.14 0.838 0.854 0.536 0.425 MISATPW038 4 1.72 0.658 0.523 0.420 0.442 MISATPW025 3 2.49 0.980 0.484 0.601 0.526 MISATPW060 6 3.52 1.427 0.667 0.720 0.693 MISATPW116 1 1.00 0.000 0.000 0.000 0.020 MISATPW001 2 1.12 0.219 0.115 0.109 0.102 MISATPW058 2 1.04 0.101 0.000 0.041 0.040 MISATPW154 4 1.65 0.749 0.208 0.397 0.363 平均 mean 3.08 1.80 0.600 0.301 0.339 0.321 3个大口黑鲈群体的遗传多样性见表3。其中3个群体的Na为2.583 3~2.666 7,Ne为1.659 2~1.911 0,I为0.533 7~0.620 6,Ho为0.294 7~0.304 9,He为0.312 5~0.360 6,PIC为0.294 2~0.335 6。沙头群体的6个遗传多样性参数略高于西樵和烟南群体,西樵和烟南群体的比较接近,且西樵群体的遗传多样性参数最低。
表 3 3个大口黑鲈养殖群体遗传多样性Table 3. Genetic diversity of three populations of M. salmoides群体
population等位基因数
Na有效等位基因数
NeShannon指数
I观测杂合度
Ho期望杂合度
He多态信息含量
PIC西樵 XJ 2.583 3 1.659 2 0.533 7 0.294 7 0.312 5 0.294 2 沙头 ST 2.666 7 1.911 0 0.620 6 0.304 9 0.360 6 0.335 6 烟南 YN 2.583 3 1.736 1 0.554 1 0.301 5 0.328 4 0.296 4 2.3 群体间的遗传分化及遗传距离分析
3个大口黑鲈养殖群体间的遗传分化指数 (FST)为0.012 1~0.028 9 (表4),属低水平遗传分化 (FST<0.05)。其中西樵和烟南群体间的遗传分化水平最低 (FST=0.012 1),沙头群体和烟南群体间的遗传分化水平最高 (FST=0.028 9,表4)。
表 4 3个大口黑鲈养殖群体的遗传分化指数Table 4. Fixation index (FST) of three populations of M. salmoides西樵 XJ 沙头 ST 烟南 YN 西樵 XJ − 沙头 ST 0.017 9 − 烟南 YN 0.012 1 0.028 9 − AMOVA分析显示,14.17%的遗传变异来自群体间,85.83%的遗传变异来自群体内 (P<0.01)。结果表明,遗传变异主要存在个体间,个体间的遗传变异程度远大于群体间 (表5)。
表 5 3个大口黑鲈养殖群体AMOVA分析Table 5. AMOVA analysis among three populations of M. salmoides变异来源
source of variation自由度
df平方和
sum of squares方差组分
variance component% 群体间 among populations 95 192.859 0.296 76 14.17 群体内 within populations 96 146.500 1.526 04 85.83** 总变异 total variation 191 339.359 1.778 07 注:**. 1 023次模拟检验后为极显著 (P<0.01) Note: **. very significant difference after 1 023 permutation tests (P<0.01) 3个群体间Nei's遗传距离 (Da)介于0.013 7~0.024 9,遗传相似度 (S)介于0.975 4~0.986 4 (表6),其中西樵群体和沙头群体的遗传距离最近 (Da=0.013 7)、遗传相似度最高 (S=0.986 4),而烟南群体和沙头群体的遗传距离最远 (Da=0.024 9)、遗传相似度最低 (S=0.975 4)。由遗传距离大小构建了UPGMA聚类树,聚类分析结果显示,3个大口黑鲈群体归为两大类,西樵与烟南群体为一类,沙头群体单独为一类 (图3)。
表 6 3个大口黑鲈养殖群体的遗传相似度S (对角线上) 和遗传距离Da (对角线下)Table 6. Genetic similarity (above diagonal) and genetic distance (below diagonal) of three populations of M. salmoides西樵 XJ 沙头 ST 烟南 YN 西樵 XJ − 0.982 5 0.986 4 沙头 ST 0.017 7 − 0.975 4 烟南 YN 0.013 7 0.024 9 − 2.4 群体遗传结构的分析
运用STRUCTURE软件对3个大口黑鲈群体的遗传结构进行分析,执行K=2~7的假设,设定10次重复,结果显示对数似然函数值lnP(D)随亚群数K的增加而降低,K=1时,lnP(D)最大,从而确定出最适宜的亚群数为1 (图4)。K=2时,3个群体的遗传结构图几乎没有分别 (图5),也说明3个养殖群体都来自一个亚群。
3. 讨论
本研究采用Qsep100全自动核酸蛋白分析系统,通过毛细管电泳筛选微卫星引物,在51对微卫星引物中挑选出12对多态性相对较好的引物进行大群体遗传多样性的检测。在检测阶段则是采用ABI310遗传分析仪通过荧光标记的引物进行PCR扩增,扩增产物经毛细管电泳后进行STR分析。Qsep100全自动核酸蛋白分析系统和ABI310遗传分析仪均采用毛细管电泳系统,2个系统都具有操作简单、速度快、准确性高的优点。不同之处在于,Qsep100系统在电泳介质中加入能结合核酸的荧光物质,利用激光诱导其发光来检测核酸[19],ABI310系统则是先对引物进行荧光标记,使得PCR产物带有荧光,毛细管电泳时直接检测产物荧光来确定产物大小[20]。Qsep100系统成本较高的部分是带有荧光的电泳介质 (即卡夹),ABI310系统成本较高的部分是荧光引物的合成,Qsep100系统的总成本略低。但ABI310系统毛细管的峰图则更清晰,更容易排除杂带的干扰,检测准确性更高。因此综合2套系统的优势,本研究在引物筛选阶段采用Qsep100系统,在检测分型阶段则采用ABI310系统,可以在保证准确性的前提下提高检测效率,节省成本。
对本研究筛选的12对微卫星引物的遗传参数分析发现,12对引物中有5对引物属于低度多态 (PIC<0.25),4对引物属于中度多态 (0.25<PIC<0.5),只有3对引物属于高度多态 (PIC>0.5)。其中有2对引物 (mdo3和MISATPW116)只有1个等位基因 (Na=1),其He均为0,说明这2对引物没有多态性。Malloy等[11]和Lutz-Carrillo等[13-14]通过磁珠富集法筛选到大量的大口黑鲈微卫星引物,本研究选择其中的51对引物用于扩增,并从中筛选出12对多态性高且特异性好的微卫星引物。在Malloy等[11]和Lutz-Carrillo等[13-14]的研究中,这12对引物大都属于高度多态性,其中有10对引物He为0.55~0.94,其遗传多样性参数均显著高于本研究所获得的参数值。另外,在他们的研究中报道有高度多态性的引物,大部分在本研究的3个群体中已不再具有多态性[10-14]。因此,从本研究12对引物在大口黑鲈3个养殖群体扩增所获得的多样性参数来看,本研究中的3个养殖群体的遗传多样性较Malloy等报道过的群体已显著下降。推测当前养殖的大口黑鲈群体大都是由最初引进的大口黑鲈群体繁育的后代,经过多年的近交繁育,遗传多样性已显著下降。
对本研究选取的广东佛山3个大口黑鲈养殖群体的遗传多样性分析发现,3个群体的He为0.312 5~0.360 6。而樊佳佳等[8]用微卫星标记对国内养殖群体和3个引进群体进行遗传多样性检测,其He为0.454 9~0.613 8,高于本研究的分析结果。本研究所选用的MISATPW001和mdo72位点,PIC值已下降为0.102和0.158,而在樊佳佳等[8]的结果中,国内养殖群体中这2个位点的PIC分别为0.141 1和0.239 2,这均说明近几年我国大口黑鲈养殖群体遗传多样性下降明显。另外,3个群体之间,沙头群体 (ST)的遗传多样性最高 (He=0.360 6),各遗传参数均高于另外2个群体,可能与苗种来源有关。
本研究的3个大口黑鲈养殖群体遗传分化指数 (FST)为0.012 1~0.028 9,遗传分化水平较低,说明3个群体遗传差异不明显。3个群体Da介于0.013 7~0.024 9,非常接近,另外,运用STRUCTURE软件进行遗传结构分析也表明3个养殖群体应来自于一个亚群。这佐证了上述观点,即当前大口黑鲈养殖群体极可能均由最初引进的大口黑鲈群体繁育而来。当前大口黑鲈养殖群体结构单一,遗传多样性低,抗病性能下降的问题除了与养殖模式及管理有关外,也极可能与种质退化、遗传多样性下降有关。由于遗传多样性下降,导致其对环境的适应能力变差,进而更容易被病原等侵袭。因此应高度重视提高与维持大口黑鲈种群遗传多样性的问题,并通过分子选育种技术进行优良品种的培育,以促进大口黑鲈养殖产业的健康可持续发展。
-
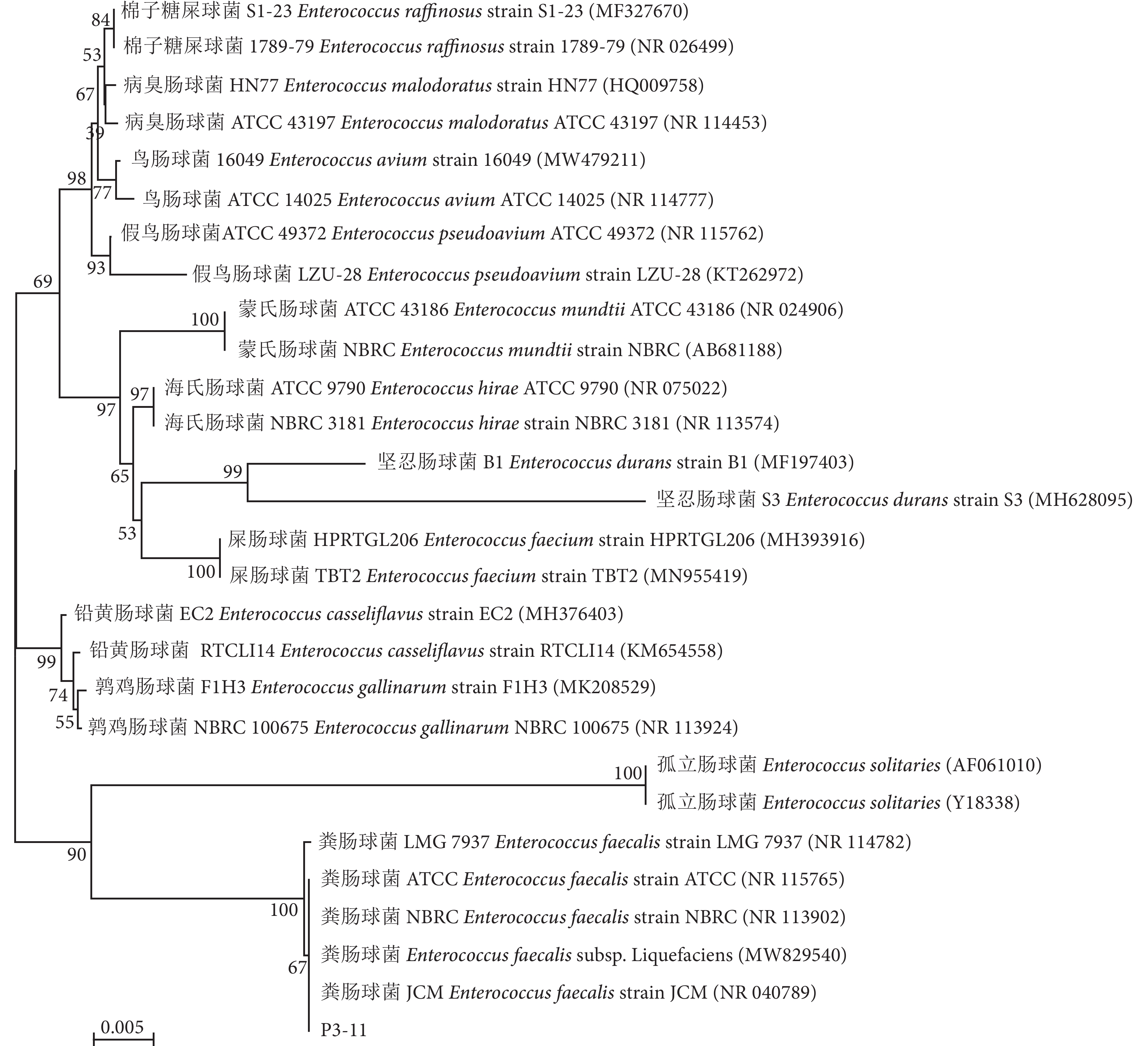
图 3 基于菌株P1-17、P2-33及相关菌株16S rRNA基因序列的系统发育树
注:括号中的序号代表菌株的GenBank登录号;分支点上的数字代表bootstrap值;标尺刻度代表碱基替代率。图4同此。
Figure 3. Phylogenetic tree based on 16S rRNA sequences of Strains P1-17, P2-33 and other related strains
Note: The sequence number in the brackets is the GenBank accession number of the strain; the numbers at the node are the bootstrap values; the scale bar indicates nucleotide substitution ratio. The same case in Fig. 4.
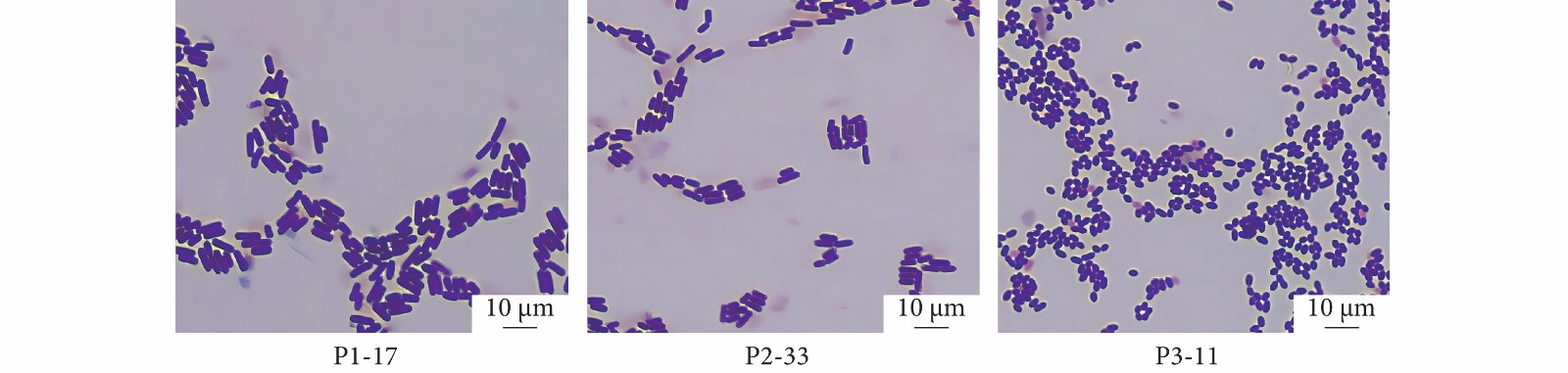
表 1 菌株P1-17、P2-33、P3-11的生理生化特征
Table 1 Physiological and biochemical characteristics of Strains P1-17, P2-33 and P3-11
鉴定项目
Tested item菌株 Strain P1-17 P2-33 P3-11 蛋白胨水 peptone water + + + 蔗糖 Sucrose − − + 甘露糖 Mannose − − − 阿拉伯糖 Pectinose + + + 乳糖 Lactose − − − 葡萄糖氧化发酵试验 OF − − − 邻硝基苯-半乳糖苷酶
o-nitrophenyl-galactosidase− − − 柠檬酸盐 Citrate − − − 硫化氢 H2S − − − 尿素酶 Urease − − − VP试验 Voges-Proskauer test + + + 明胶液化 Gelatin Liquefaction + + + 吲哚试验 Indole test − − + 葡萄糖 Glucose − − + 甘露醇 Mannitol − − + 肌醇 Inositol − − + 山梨醇 Sorbitol − − + 鼠李糖 Rhamnose − − − 密二糖 Melibiose − − − 苦杏仁苷 Amygdalin − − + 氧化酶试验 OX − − − 注:+. 阳性;−. 阴性。 Note: +. Positive; –. Negative. 表 2 菌株P1-17、P2-33 和 P3-11的药敏特性
Table 2 Antibiotic sensitivity of Strains P1-17, P2-33 and P3-11
抗菌药物种类
Type of Antibacterial药物名称
Drug纸片含药量
Drug content抑菌圈直径/敏感性
Diameter of inhibition zone (mm)/SusceptibilityP1-17 P2-33 P3-11 青霉素类
Penicillins氨苄西林 Ampicillin 10 μg·片−1 27.41/S 22.34/S 14.56/R 青霉素 Penicillin 10 μg·片−1 32.65/S 27.54/S 21.37/S 阿莫西林 Amoxicillin 20 μg·片−1 28.57/S 18.26/S 16.22/I 新生霉素 Neomycin 30 μg·片−1 28.57/S 28.57/S 17.30/S 头孢菌素类
Cephalosporins头孢噻肟 Cefotaxime 30 μg·片−1 37.58/S 31.48/S 0/R 头孢曲松 Ceftriaxone 30 μg·片−1 37.45/S 33.56/S 0/R 喹诺酮类
Quinolones诺氟沙星 Norfloxacin 10 μg·片−1 32.36/S 31.43/S 19/S 依诺沙星 Enoxacin 10 μg·片−1 32.54/S 31.64/S 22/S 环丙沙星 Ciprofloxacin 5 μg·片−1 41.05/S 36.55/S 24/S 氧氟沙星 Ofloxacin 5 μg·片−1 41.24/S 39.15/S 25/S 左氧氟沙 Levofloxacin 5 μg·片−1 39.04/S 38.86/S 19/S 恩诺沙星 Enrofloxacine 10 μg·片−1 40.32/S 36.58/S 24/I 洛美沙星 Lomefloxacin 10 μg·片−1 40.27/S 38.36/S 21/S 磺胺类
Sulfonamides磺胺异恶唑 Sulfisoxazole 300 μg·片−1 20.34/S 18.87/S 0/R 复方新诺明 Cotrimoxazole 25 μg·片−1 34.66/S 32.78/S 0/R 四环素类
Tetracyclines四环素 Tetracycline 30 μg·片−1 25.16/S 23.36/S 15/I 多西环素 Doxycycline 30 μg·片−1 33.27/S 33.27/S 26/S 大环内酯类
Macrolides阿奇霉素 Azithromycin 15 μg·片−1 30.26/S 27.67/S 13/R 罗红霉素 Roxithromycin 15 μg·片−1 37.57/S 33.35/S 14/R 氨基苷类
Aminoglycosides链霉素 Streptomycin 10 μg·片−1 26.26/S 25.86/S 0/R 卡那霉素 Kanamycin 30 μg·片−1 35.06/S 34.89/S 0/R 丁胺卡那 Amikacin 30 μg·片−1 37.34/S 31.57/S 0/R 庆大霉素 Gentamycin 10 μg·片−1 34.36/S 31.68/S 0/R 妥布霉素 Tobramycin 10 μg·片−1 36.02/S 34.08/S 0/R 氯霉素类
Chloramphenicol氯霉素 Chloramphenicol 30 μg·片−1 35.24/S 35.24/S 18/I 氟苯尼考 Florfenicol 30 μg·片−1 38.31/S 33.65/S 22/S 肽类抗生素
Peptides万古霉素 Vancomycin 30 μg·片−1 30.10/S 28.35/S 20/S 多黏菌素 B Polymyxin B 300 IU 17.28/S 14.07/S 0/R 多烯类抗真菌抗生素
Polyeneantifungal制霉菌素 Nystatin 100 μg·片−1 10.57/R 11.21/R 0/R 注:S. 高度敏感;I. 中度敏感;R. 耐药。 Note: S. Sensitive; I. Intermediate sensitive; R. Resistance. 表 3 拮抗菌株产酶能力
Table 3 Enzyme production ability of antagonistic strains
菌株名称
Strain水解圈直径/菌落直径
Dh/Dc蛋白酶
Protease脂肪酶
Lipase淀粉酶
AmylaseP1-17 4.23 0 4.20 P2-33 3.82 0 4.46 P3-11 0 0 0 表 4 分离菌株对杀香鱼假单胞菌的拮抗试验
Table 4 Antagonism test of isolated strains against P. plecoglossicida
菌株
Strain抑菌圈直径 Diameter of inhibition zone/mm P1-17 P2-33 P3-11 杀香鱼假单胞菌 1303001 P. plecoglossicida 1303001 10.24±0.41 9.86±0.35 12.57±0.64 杀香鱼假单胞菌 1303002 P. plecoglossicida 1303002 10.53±0.72 10.18±1.14 12.6±0.83 杀香鱼假单胞菌 1306002 P. plecoglossicida 1306002 9.76±0.47 9.58±0.34 11.47±0.54 杀香鱼假单胞菌 202010001 P. plecoglossicida 202010001 10.43±1.40 9.76±0.65 11.85±1.21 杀香鱼假单胞菌 202010002 P. plecoglossicida 202010002 9.45±1.52 9.25±0.74 10.68±0.36 杀香鱼假单胞菌 202105001 P. plecoglossicida 202105001 9.18±0.64 8.74±1.32 10.46±0.54 杀香鱼假单胞菌 202105002 P. plecoglossicida 202105002 10.65±0.22 9.65±0.83 12.34±1.12 杀香鱼假单胞菌 202105003 P. plecoglossicida 202105003 9.34±0.72 9.05±0.63 11.38±1.43 表 5 拮抗菌对其他致病菌的拮抗试验
Table 5 Antagonism test of antagonistic bacteria against other pathogenic bacteria
菌株
Strain抑菌圈直径 Diameter of inhibition zone/mm P1-17 P2-33 P3-11 溶藻弧菌 Vibrio alginolyticus 10.39±0.67 7.78±0.56 0 哈维氏弧菌 V. harveyi 10.65±1.54 10.34±1.56 0 副溶血弧菌 V. parahaemolyticus 11.57±0.34 11.25±0.42 0 美人鱼发光杆菌 Photobacterium damselae 15.13±0.24 14.96±0.53 0 嗜水气单胞菌 Aeromonas hydrophila 19.64±1.56 16.85±0.37 0 迟缓爱德华氏菌 Edwardsiella tarda 21.85±0.24 20.83±0.46 0 注:“0”表示无拮抗作用。 Note: "0" indicates no antagonism. -
[1] 刘家富. 大黄鱼养殖与生物学[M]. 厦门: 厦门大学出版社, 2013: 1-6. [2] 农业农村部渔业渔政管理局, 全国水产技术推广总站, 中国水产学会. 2021中国渔业统计年鉴[M]. 北京: 中国农业出版社, 2021: 44, 48. [3] ZHOU Q J, WANG J, MAO Y, et al. Molecular structure, expression and antibacterial characterization of a novel antimicrobial peptide NK-lysin from the large yellow croaker (Larimichthys crocea)[J]. Aquaculture, 2019, 500: 315-321. doi: 10.1016/j.aquaculture.2018.10.012
[4] 许斌福, 程海华, 池洪树, 等. 大黄鱼内脏白点病的病原分析与鉴定[J]. 福建农业学报, 2015, 30(7): 631-635. doi: 10.3969/j.issn.1008-0384.2015.07.002 [5] ZHANG J T, ZHOU S M, AN S W, et al. Visceral granulomas in farmed large yellow croaker, Larimichthys crocea (Richardson), caused by a bacterial pathogen, Pseudomonas plecoglossicida[J]. J Fish Dis, 2014, 37(2): 113-121. doi: 10.1111/jfd.12075
[6] LI C W, WANG S L, REN Q L, et al. An outbreak of visceral white nodules disease caused by Pseudomonas plecoglossicida at a water temperature of 12° C in cultured large yellow croaker (Larimichthys crocea) in China[J]. J Fish Dis, 2020, 43(11): 1353-1361. doi: 10.1111/jfd.13206
[7] 刘亚楠, 习丙文, 梁利国, 等. 水产动物病原菌拮抗菌的研究进展[J]. 江苏农业科学, 2013, 41(5): 208-212. doi: 10.3969/j.issn.1002-1302.2013.05.079 [8] 任竹玲. 罗非鱼舒伯特气单胞菌疾病及其益生菌防治研究[D]. 海口: 海南大学, 2020: 16-17. [9] KUEBUTORNYE F K A, ABARIKE E D, LU Y, et al. Mechanisms and the role of probiotic Bacillus in mitigating fish pathogens in aquaculture[J]. Fish Physiol Biochem, 2020, 46(3): 819-841. doi: 10.1007/s10695-019-00754-y
[10] 赵龙妹, 聂利芳. 鸡源产蛋白酶乳酸菌的筛选与鉴定[J]. 黑龙江畜牧兽医, 2020(10): 89-93, 98, 151-152. [11] 柯轲, 方端, 高福, 等. 凝结芽孢杆菌在动物饲料中的应用[J]. 中国微生态学杂志, 2022, 34(8): 988-993. doi: 10.13381/j.cnki.cjm.202208023 [12] OLSSON J C, WESTERDAHL A, CONWAY P L, et al. Intestinal colonization potential ofturbot (Scophthalmus maximus) and dab (Limanda limanda) associated bacteria with inhibitory effects against Vibrio anguillarum[J]. Appl Environ Microbiol, 1992, 58(2): 551-556. doi: 10.1128/aem.58.2.551-556.1992
[13] WANG W W, WU S G, ZOU H, et al. Characterization of cellulose-decomposing bacteria in the intestine of grass carp, Ctenopharyngodon idella (Val.)[J]. Acta Hydrobiologica Sinica, 2014, 38(2): 291-297.
[14] 王凌利, 杨亲, 祝瑶, 等. 猪链球菌细菌素的筛选及理化特性分析[J]. 中国预防兽医学报, 2022, 44(4): 363-368. [15] 布坎南R E, 吉本斯N E. 伯杰细菌鉴定手册[M]. 北京: 科学出版社, 1984: 729-731. [16] 东秀珠, 蔡妙英. 常见细菌系统鉴定手册 [M]. 北京: 科学出版社, 2001: 25-26. [17] MUKHERJEE A, CHANDRA G, GHOSH K. Single or conjoint application of autochthonous Bacillus strains as potential probiotics: effects on growth, feed utilization, immunity and disease resistance in Rohu, Labeo rohita (Hamilton)[J]. Aquaculture, 2019, 512: 734302. doi: 10.1016/j.aquaculture.2019.734302
[18] PENNSYLVANIA U. Clinical and Laboratory Standards Institute[J]. Zambia, 2021, 35(3): 44-106.
[19] 孟小亮. 黄颡鱼肠道益生菌的筛选及其应用研究[D]. 武汉: 华中农业大学, 2010: 12-17. [20] 夏京津, 陈建武, 宋怿, 等. 解淀粉芽孢杆菌HE活性成分鉴定及抗菌特性分析[J]. 南方水产科学, 2019, 15(3): 41-49. doi: 10.12131/20190054 [21] XU B H, YE Z W, ZHENG Q W, et al. Isolation and characterization of cyclic lipopeptides with broad-spectrum antimicrobial activity from Bacillus siamensis JFL15[J]. 3 Biotech, 2018, 8(10): 1-10.
[22] LI X X, GAO X J, ZHANG S M, et al. Characterization of a Bacillus velezensis with antibacterial activity and inhibitory effect on common aquatic pathogens[J]. Aquaculture, 2020, 523: 735165. doi: 10.1016/j.aquaculture.2020.735165
[23] 刘婷, 尹启蒙, 周滟晴, 等. 一株副溶血性弧菌拮抗菌的筛选、鉴定及其抑菌物质特性研究[J]. 食品与发酵工业, 2022, 48(1): 76-83. [24] 徐春霞. 网箱养殖大黄鱼内脏白点病病原菌分离鉴定及致病性研究[J]. 水产科学, 2021, 40(5): 670-678. [25] 王娟, 封永辉, 蔡立胜, 等. 来自大黄鱼 (Pseudosciaena crocea) 肠道的弧菌拮抗菌的筛选与鉴定[J]. 海洋与湖沼, 2010, 41(5): 707-713. doi: 10.11693/hyhz201005007007 [26] 王梦霞, 朱晓玲, 王君如, 等. 大黄鱼病原哈维氏弧菌 (Vibrio harveyi) 拮抗菌的筛选及益生菌特征的分析[J]. 饲料工业, 2015, 36(2): 52-58. [27] 傅超英, 王建平, 孙琛, 等. 大黄鱼主要致病菌拮抗菌株的分离鉴定、抑菌谱及安全性分析[J]. 生物技术通报, 2019, 35(1): 67-75. doi: 10.13560/j.cnki.biotech.bull.1985.2018-0565 [28] HUYBEN D, NYMAN A, VIDAKOVIĆ A, et al. Effects of dietary inclusion of the yeasts Saccharomyces cerevisiae and Wickerhamomyces anomalus on gut microbiota of rainbow trout[J]. Aquaculture, 2017, 473: 528-537. doi: 10.1016/j.aquaculture.2017.03.024
[29] LIU Q, WEN L, PAN X H, et al. Dietary supplementation of Bacillus subtilis and Enterococcus faecalis can effectively improve the growth performance, immunity, and resistance of tilapia against Streptococcus agalactiae[J]. Aquac Nutr, 2021, 27: 1160-1172. doi: 10.1111/anu.13256
[30] 王金燕, 李彬, 王印庚, 等. 刺参养殖池塘一株贝莱斯芽孢杆菌的分离及其生理特性[J]. 中国水产科学, 2018, 25(3): 567-575. [31] 任津莹, 陈鹏. 一株贝莱斯芽孢杆菌的分离鉴定及其生物学特性研究[J]. 饲料研究, 2022, 45(2): 79-82. doi: 10.13557/j.cnki.issn1002-2813.2022.02.017 [32] KANG M R, SU X, YUN L L, et al. Evaluation of probiotic characteristics and whole genome analysis of Bacillus velezensis R-71003 isolated from the intestine of common carp (Cyprinus carpio L.) for its use as a probiotic in aquaculture[J]. Aquac Rep, 2022, 25: 101254. doi: 10.1016/j.aqrep.2022.101254
[33] ZHANG D F, XIONG X L, WANG Y J, et al. Bacillus velezensis WLYS23 strain possesses antagonistic activity against hybrid snakehead bacterial pathogens[J]. J Appl Microbiol, 2021, 131(6): 3056-3068. doi: 10.1111/jam.15162
-
期刊类型引用(11)
1. 李兵部,傅建军,陶易凡,强俊,徐跑. 基于D-loop序列和微卫星标记的4个黄颡鱼群体的遗传变异分析. 黑龙江畜牧兽医. 2024(04): 115-127 .  百度学术
百度学术
2. 胡玉婷,凌俊,江河,汪焕,潘庭双,段国庆,周华兴,杨敏,李彤. 苏皖地区中华绒螯蟹养殖群体微卫星遗传多样性的评估. 渔业科学进展. 2024(06): 178-187 .  百度学术
百度学术
3. 李大命,杨子萍,刘燕山,谷先坤,殷稼雯,蔡永久,唐晟凯,张彤晴. 基于线粒体COI序列的江淮下游湖泊鲢群体遗传多样性和遗传结构分析. 淡水渔业. 2023(04): 3-11 .  百度学术
百度学术
4. 宋立民,王娜,郑英珍,丁子元,刘肖莲,姜巨峰,张韦,耿绪云. 基于微卫星标记技术的5个黄颡鱼群体遗传多样性分析. 经济动物学报. 2023(02): 101-108 .  百度学术
百度学术
5. 葛锐,强壮,聂竹兰,李丽,魏杰. 基于高通量转录组测序的斑重唇鱼SSR分布及序列特征分析. 南方农业学报. 2023(03): 806-814 .  百度学术
百度学术
6. 邹利,王金龙,李传武,王冬武,曾春芳,刘明求,刘丽,谢敏,曾鸣. 稻田适养品种呆鲤的遗传多样性分析. 水产科学. 2023(05): 795-804 .  百度学术
百度学术
7. 黄皓,范嗣刚,王鹏飞,陈佳,赵超,闫路路,邱丽华,潘滢. 基于微卫星标记对6个花鲈群体的遗传多样性分析. 南方水产科学. 2022(01): 99-106 .  本站查看
本站查看
8. 胡玉婷,凌俊,江河,汪焕,潘庭双,周华兴. 中华绒螯蟹4个养殖群体遗传多样性与遗传结构分析. 江苏农业科学. 2022(16): 54-59 .  百度学术
百度学术
9. 胡玉婷,侯冠军. 安徽省翘嘴鲌野生群体的遗传多样性分析. 生物学杂志. 2022(04): 79-83 .  百度学术
百度学术
10. 罗宇婷,方弟安,周彦锋,徐东坡,彭云鑫,彭飞,张桂宁,刘凯,尤洋. 基于微卫星标记对长江下游鲢遗传多样性现状的分析. 南方水产科学. 2021(06): 48-57 .  本站查看
本站查看
11. 张显波,傅建军,胡锦丽,朱文彬,闵倩雯,赵飞,吴俣学,董在杰. 基于D-loop序列和SSR的从江田鱼与6个鲤群体的遗传分析. 贵州农业科学. 2021(12): 76-85 .  百度学术
百度学术
其他类型引用(5)



 下载:
下载:







 粤公网安备 44010502001741号
粤公网安备 44010502001741号
