Molecular cloning and expression analysis of BMP10 gene from pearl oyster (Pinctada fucata)
-
摘要:
骨形态发生蛋白(bone morphogenetic proteins,BMPs)具有成骨活性,在胚胎发育、组织发生、生殖调控等方面也有重要作用,但在贝类中研究还很少。研究克隆了合浦珠母贝(Pinctada fucata)BMP10基因,命名为PfBMP10。该基因cDNA长1 752 bp,包含开放阅读框(ORF)1 344 bp,编码447个氨基酸。编码蛋白包含一个N端信号肽、一个前结构域和成熟蛋白区。成熟区包含转化生长因子β(transforming growth factor beta,TGF-β)结构域,其中含7个半胱氨酸残基。不同发育时期表达分析显示PfBMP10高表达于担轮期和变态期,提示其可能参与幼体担轮期、变态期的发育调控。组织表达分析显示其在鳃和外套膜高表达,贝壳缺刻实验显示缺刻处理24 h后其在鳃和外套膜中的表达量均显著上升,表明PfBMP10可能与贝壳形成相关。研究结果可为BMP家族基因功能及贝壳形成机理研究提供参考。
Abstract:Bone morphogenetic proteins (BMPs) have functions of inducing bone formation as well as regulating embryo development, histogeny and reproduction. However, there are only a few researches on BMPs in molluscs. The BMP10 gene was cloned from pearl oyster (Pinctada fucata) and thus named as PfBMP10. The full length cDNA of PfBMP10 is 1 752 bp, including an open reading frame (ORF) of 1 344 bp which encodes 447 amino acids. The protein sequence includes an N-terminal signal peptide, a pro-domain and a mature protein region. The mature protein contains transforming growth factor-β (TGF-β) domain which has seven cysteine residues. PfBMP10 expressed higher at the trochophore stage and metamorphosis during the larval development as indicated by qPCR, suggesting that it may involve in larval developmental regulation of trochophore stage and metamorphosis. Compared with other tissues, PfBMP10 showed high relative expression level in the gill and mantle, and rose in 24 h after shell notching, indicating that it probably involves in shell formation. The study provides references for functional study of BMP family genes and mechanism study of shell formation.
-
Keywords:
- Pinctada fucata /
- BMP10 /
- shell formation /
- shell notching experiment
-
URIST等[1]在1965年将脱钙骨基质注入肌肉,一定时间后发现植入部位有大量软骨及骨组织形成。进一步提取出一种物质,发现其能够使间充质干细胞定向分化为骨细胞,从而形成骨组织,这种物质被命名为骨形态发生蛋白(bone morphogenetic proteins,BMP)。BMP广泛分布于低等的蠕虫到高等的哺乳动物等各物种中,构成BMP蛋白家族,包括BMP1~BMP18以及BMP2B、BMP8B等,除BMP1外均属于转化生长因子β(transforming growth factor beta,TGF-β)超家族[2-4]。
BMP家族蛋白结构高度保守,前体蛋白包含信号肽、前结构域和成熟区。活性蛋白是由2个单体依靠一个链间二硫键形成的二聚体,可以同种聚合,也可异种聚合[5-6]。研究显示,BMP大多具有成骨活性[7-9],在胚胎发育[10]、组织发生[11]、生殖调控[12]等方面也有重要作用。其作用方式符合TGF-β超家族蛋白的典型作用途径,在胞外作为配体和跨膜受体结合,进而调节Smads蛋白(Brosophila mothers against decapentaplegic proteins)羧基末端,使其磷酸化而活化,活化的Smads形成复合体进入细胞核,结合到DNA的特定部位,调节下游基因的转录[13-15]。
合浦珠母贝(Pinctada fucata)是生产海水珍珠的主要贝类,目前面临种质退化、育珠技术水平低等问题,严重制约了产业发展。开展珍珠贝基础生理特别是生长、育珠过程相关研究将为良种选育和育珠技术改进提供理论支撑。贝壳和珍珠分别是外套膜和珍珠囊分泌物质进行生物矿化的产物,形成过程类似,另外贝壳拥有优美的外观、良好的生物相容性和强度特性,具有医学和材料学方面的价值,因此贝壳理化特性和形成机理一直是研究热点[16-17]。借助于早期的贝壳成分研究、形貌观察及近年几十种参与贝壳形成基质蛋白的分子克隆,有关贝壳的结构框架和物质组成逐步得到阐明。但由于参与贝壳形成的物质种类繁多,调控过程高度复杂,目前贝壳形成的具体过程依然存在诸多未知点[18-22]。特别是针对起调控作用的物质研究较少,目前研究比较深入的只有Pif基因[23],另外在小鼠(Mus musculus)体内的实验发现合浦珠母贝Smad3基因过表达会增强基质蛋白KRMP编码基因启动子的活性,提示TGF-β通路可能参与贝壳形成的调控[24]。
BMP在软体动物中的潜在作用已得到关注。合浦珠母贝BMP2[25]和BMP7[26]已被鉴定,组织表达均显示其在外套膜和鳃中高表达,推测其和贝壳形成相关。进一步对参与贝壳形成的相关基因进行发掘是阐明贝壳形成过程的基础,另外鉴于BMP家族基因的多功能性,其在软体动物生长发育等方面的潜在作用也具有研究价值。该实验克隆得到合浦珠母贝BMP10基因,通过实时荧光定量PCR分析其在不同发育时期和成体各组织的表达情况,并进行贝壳缺刻实验以探究其在贝壳缺损修复中的作用,以为BMP在软体动物发育中的作用和贝壳形成机理研究提供参考。
1. 材料与方法
1.1 实验材料
实验用合浦珠母贝取自中国水产科学研究院南海水产研究所海南热带水产研究中心陵水基地。取健康成贝,放水泥池暂养1周。对贝进行解剖,分别取外套膜、闭壳肌、鳃、性腺、珍珠囊、足、血细胞、肝胰腺。取性成熟贝进行解剖取精卵、体外受精,借助显微镜观察辨别幼体发育时期,在贝苗发育的担轮期、D-形期、壳顶期、眼点期、变态期分别取样。以上样品均加RNA保护液(TaKaRa,大连)于-80 ℃保存。
1.2 实验方法
1.2.1 RNA的提取与cDNA的合成
取外套膜组织样品,按Tirzol说明书(Invitrogen,USA)提取总RNA,用1%琼脂糖凝胶电泳检测完整性,紫外分光光度计检测浓度和纯度。以总RNA为模板,利用M-MLV反转录试剂(TaKaRa,大连)合成cDNA,利用5′-Full RACE Kit(TaKaRa,大连)合成5′RACE-cDNA,-20 ℃保存。
1.2.2 目的基因cDNA的获得
查询笔者课题组的合浦珠母贝转录组序列,获得2条注释为BMP10基因的片段。经过和其他物种BMP10基因的比对初步确定2条序列的先后顺序。以此为基础设计3对引物S1/A1、S2/A2、S3/A3(表 1),其中S1/A1和S3/A3分别用于验证2条已知序列,S2/A2用于扩增两序列间的未知序列。以1.2.1中得到的cDNA为模板进行PCR扩增,对已知序列进行验证,对未知序列进行补充。扩增得到的DNA片段进行回收,连入T载体(TaKaRa,大连),再转入大肠杆菌,筛选阳性克隆进行测序,测序所得序列进行拼接,获得一条连续序列。再依据所得序列设计5′特异性引物(表 1),以5′RACE-cDNA为模板,利用5′-Full RACE Kit(TaKaRa,大连)进行RACE扩增,所得序列进行克隆测序和拼接,方法同上。
表 1 实验所用引物序列Table 1 Sequences of primers used in the study引物名称
primer name引物序列(5′→3′)
primer sequence用途
usageS1 TAAGGAGGAGTTGAGGAA 序列验证 A1 AGGCTGGACTGTAGGATC 序列验证 S2 GTTAGGTCTTTATCGCTTAG 中间片段扩增 A2 ACAACCATTCCCTCATA 中间片段扩增 S3 GATTTTGGAGATTCGTAT 序列验证 A3 CTGTTGTCCTTGCCTTTT 序列验证 5′GSP-out ATAGTCAGGTGCCTCTTT 5′RACE 5′GSP-in TTACTAAGCGATAAAGACC 5′RACE qRT-S ATGACCACAAGACCCACTA 荧光定量 qRT-A CTTCTCCTCTATGATTCCA 荧光定量 18S TGTCTGCCCTATCAACTTTC 内参引物 18A TGTGGTAGCCGTTTCTCA 内参引物 1.2.3 目的基因序列分析
用ORF Finder (http://www.ncbi.nlm.nih.gov/gorf/gorf.html) 和Translate (http://web.expasy.org/translate) 查找开放阅读框和预测编码蛋白序列;用PortParam (http://web.expasy.org/cgi-bin/protparam/protparam) 预测编码蛋白的理化性质;用TMHMM (http://www.cbs.dtu.dk/services/TMHMM-2.0/) 预测蛋白的跨膜结构域;用Signal P (http://www.cbs.dtu.dk/services/SignalP/) 预测蛋白信号肽;用SMART (http://smart.embl-heidelberg.de/smart/set_mode) 分析蛋白的功能结构域;利用DNAMAN软件进行同源序列比对。利用MEGA 6.0采用Neighbor-joining (NJ) 法建立进化树。
1.2.4 目的基因在不同发育时期和不同组织的表达分析
依据1.2.2所得序列设计并合成实时荧光定量PCR反转录引物qRT-S/A(表 1)。取合浦珠母贝5个发育时期和8个组织的样品提取总RNA,用反转录试剂盒(TaKaRa,大连)反转录得到cDNA。以18S rRNA为内参,用SYBR real-time PCR Master Mix(TaKaRa,大连)进行实时荧光定量PCR反应。先进行模板浓度梯度实验,确定最佳浓度。每个组或时期样品取3个重复,每个样品进行3次重复,并进行熔解曲线分析。用相对定量2-ΔΔCt法计算基因相对表达水平。用Origin 9.1进行单因素方差分析,P<0.05为差异显著。
1.2.5 贝壳缺刻实验
选取壳长约6 cm的健康成贝48只,随机分为2组,每组24只。各装入一个贝笼,再将所有贝放于一个水泥池中暂养,定期饲喂藻类并换水,保持水质稳定。暂养3 d后随机挑选24只贝进行缺刻处理,方法参照MOUNT等[27]的处理,在足的对面将贝壳剪出V形缺口,确保损伤珍珠层而不破坏外套膜。处理后将贝放回原贝笼,继续饲养。另24只贝不做处理,作为对照组。分别在处理后第0、第6、第12、第24、第36、第48小时各取4只处理贝和4只未处理贝。观察处理贝切口变化,解剖取鳃和外套膜组织,提取总RNA,反转录并进行实时荧光定量PCR反应,方法同1.2.4,对照组和处理组在每个时间点做3个重复。
2. 结果与分析
2.1 目的基因cDNA序列分析
实验获得的目的基因cDNA长1 752 bp,包含开放阅读框(ORF)1 344 bp,5′非编码区(UTR)293 bp,3′非编码区115 bp,命名为PfBMP10(GeneBank登录号KU529148)。该基因编码447个氨基酸,预测分子量为51.92 kD,理论等电点(pI)6.14。跨膜结构预测表明该蛋白位于胞外且无跨膜结构。信号肽预测其有一个N端信号肽(1~23 aa)。功能结构域预测显示该蛋白含有一个前结构域(24~337 aa)和成熟蛋白区(338~447 aa),成熟区包含一个TGF-β结构域(344~447 aa),TGF-β结构域包含7个半胱氨酸残基(345 aa,374 aa,378 aa,410 aa,411 aa,444 aa,446 aa)(图 1)。
![]() 图 1 PfBMP10基因的cDNA序列和编码的氨基酸序列左侧正常和加粗数字分别标明PfBMP10基因cDNA序列和氨基酸残基位置; cDNA序列中加粗部分分别为起始和终止密码子,氨基酸序列中带细下划线的是信号肽序列,带粗下划线的是前结构域;灰色部分为成熟区,方框标记7个半胱氨酸残基。Fig. 1 cDNA and amino acid sequences of PfBMP10 geneThe bold and normal numbers on the left indicate the positions of PfBMP10 gene cDNA sequence and amino acid residues, respectively. The bold letters of cDNA sequence are initiation codon and termination codon. The putative signal peptide and pro-domain are indicated with fine and heavy underline, respectively. The mutation protein which includes seven boxed cysteine residues are shaded in gray.
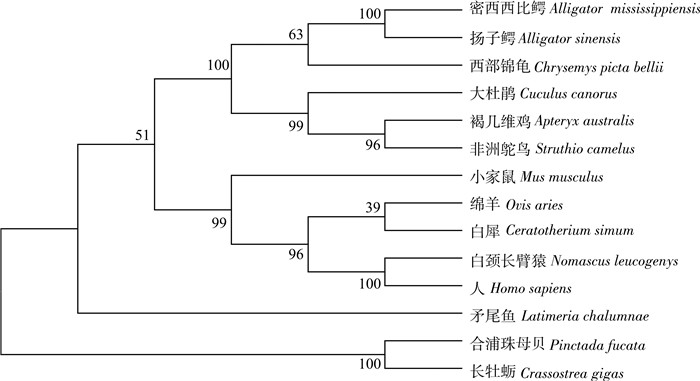
图 1 PfBMP10基因的cDNA序列和编码的氨基酸序列左侧正常和加粗数字分别标明PfBMP10基因cDNA序列和氨基酸残基位置; cDNA序列中加粗部分分别为起始和终止密码子,氨基酸序列中带细下划线的是信号肽序列,带粗下划线的是前结构域;灰色部分为成熟区,方框标记7个半胱氨酸残基。Fig. 1 cDNA and amino acid sequences of PfBMP10 geneThe bold and normal numbers on the left indicate the positions of PfBMP10 gene cDNA sequence and amino acid residues, respectively. The bold letters of cDNA sequence are initiation codon and termination codon. The putative signal peptide and pro-domain are indicated with fine and heavy underline, respectively. The mutation protein which includes seven boxed cysteine residues are shaded in gray.通过DNAMAN软件将PfBMP10蛋白成熟区与人(Homo sapiens)、原鸡(Gallus gallus)等物种的BMP10蛋白对应序列进行比对,结果显示较高的一致性,成熟区7个半胱氨酸残基高度保守(图 2)。选取人、长牡蛎(Crassostrea gigas)等典型物种的BMP10蛋白序列构建系统进化树,结果显示聚类特征符合传统的物种进化分类,PfBMP10蛋白和爬行类、鸟类、两栖类、哺乳类、鱼类BMP10蛋白处于不同分支,和长牡蛎BMP10蛋白聚为一支(图 3)。
2.2 PfBMP10基因在合浦珠母贝不同发育时期和成体不同组织的表达结果
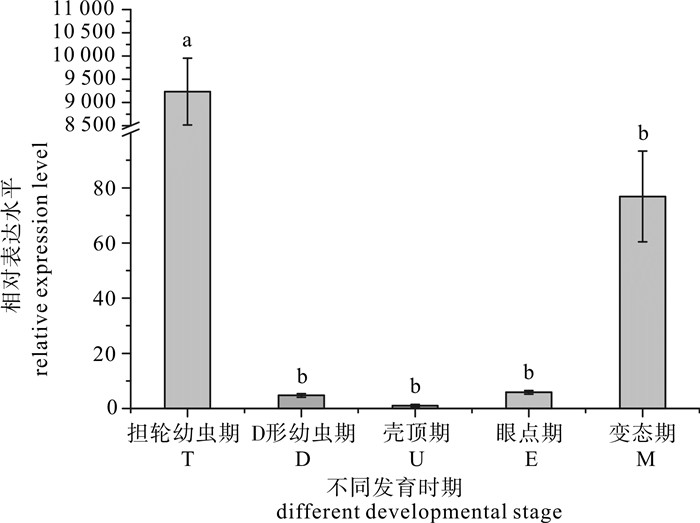
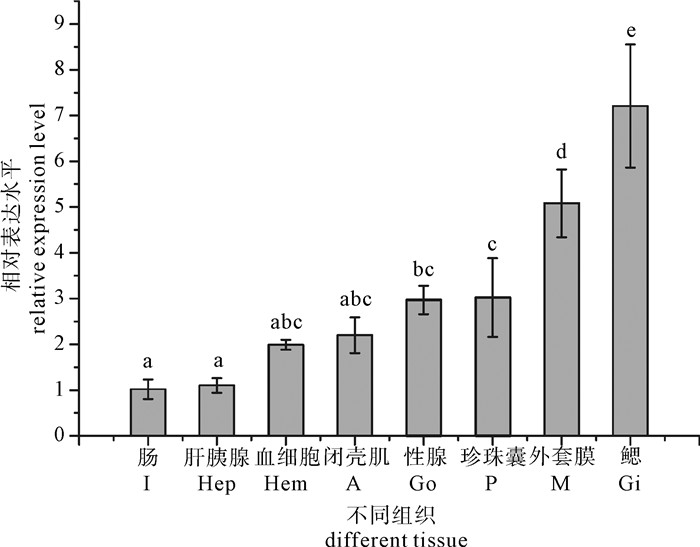
不同发育时期的实时荧光定量PCR分析显示,PfBMP10在5个时期都有表达,表达量从高到低依次为担轮幼虫期>变态期>眼点期>D-形期>壳顶期(图 4)。其中在担轮幼虫期表达量和其他时期差异显著(P<0.05),且达到变态期的120倍,变态期表达量也达到眼点期的13倍。不同组织的实时荧光定量PCR分析显示,PfBMP10在8个组织都有表达,不同组织的表达水平呈现明显差异,在鳃中表达量最高,与其他7种组织差异显著,其次在外套膜也呈现高表达,显著高于其他6种组织(P<0.05)(图 5)。
![]() 图 4 PfBMP10在合浦珠母贝不同发育时期的相对表达量不同字母表示差异显著(P<0.05),后图同此。Fig. 4 Relative expression level of PfBMP10 in different tissues of P.fucataT. trochophore; D. D-veliger larvae; U. umbo veliger larvae; E. eyebot larvae; M. metamorphosis. The significant difference is indicated by different letters (n=3, P < 0.05). The same case in the following figures.
图 4 PfBMP10在合浦珠母贝不同发育时期的相对表达量不同字母表示差异显著(P<0.05),后图同此。Fig. 4 Relative expression level of PfBMP10 in different tissues of P.fucataT. trochophore; D. D-veliger larvae; U. umbo veliger larvae; E. eyebot larvae; M. metamorphosis. The significant difference is indicated by different letters (n=3, P < 0.05). The same case in the following figures.2.3 贝壳在缺刻处理后的形态变化和PfBMP10的表达分析
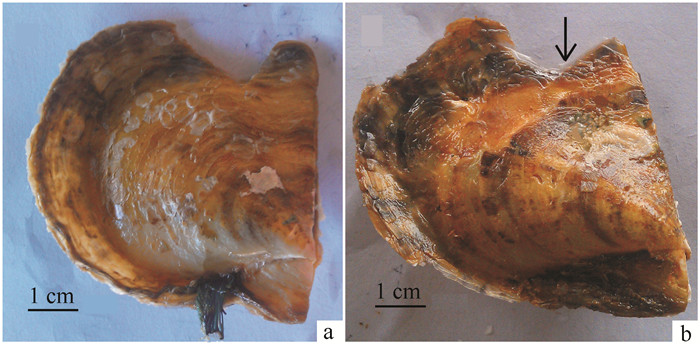
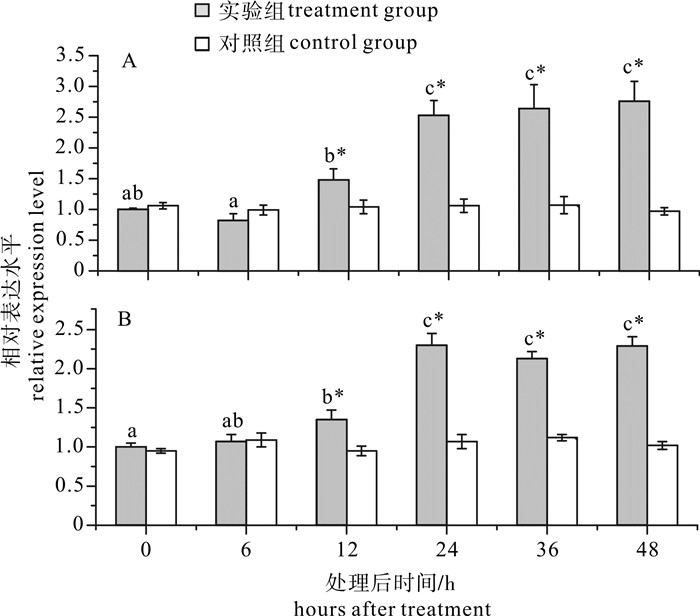
缺刻处理后观察缺口变化,处理48 h后缺口部位可观察到半透明的新生薄层(图 6)。实时荧光定量PCR反应显示,缺刻6 h、12 h后鳃的PfBMP10表达量相比0 h未出现显著变化,缺刻24 h后PfBMP10表达量显著升高,和前几个时间点差异显著(P<0.05),外套膜的PfBMP10表达变化趋势和鳃类似(图 7)。
![]() 图 7 贝壳缺刻处理后不同时间鳃和外套膜中PfBMP10的相对表达量A和B分别为鳃和外套膜实验结果,不同小写字母代表不同处理时间下基因表达量差异显著(n=3,P<0.05),*表示与对照组相比差异显著(n=3,P<0.05)。Fig. 7 Relative expression level of PfBMP10 at different time after shell notchingA and B are experiment results of gill and mantle. Different lower-case letters indicate significant difference of gene expression level among different treatment groups (n=3, P < 0.05). * indicate significant difference compared with control (n=3, P < 0.05).
图 7 贝壳缺刻处理后不同时间鳃和外套膜中PfBMP10的相对表达量A和B分别为鳃和外套膜实验结果,不同小写字母代表不同处理时间下基因表达量差异显著(n=3,P<0.05),*表示与对照组相比差异显著(n=3,P<0.05)。Fig. 7 Relative expression level of PfBMP10 at different time after shell notchingA and B are experiment results of gill and mantle. Different lower-case letters indicate significant difference of gene expression level among different treatment groups (n=3, P < 0.05). * indicate significant difference compared with control (n=3, P < 0.05).3. 讨论
该研究克隆得到合浦珠母贝BMP10基因,编码蛋白包含信号肽、前结构域和成熟区,成熟区包含TGF-β结构域,TGF-β结构域包含7个半胱氨酸残基。这些结构均符合BMP家族成员的典型特征。成熟区序列比对结果显示PfBMP10蛋白和鸟类、哺乳动物等物种的BMP10蛋白具有较高相似性,7个半胱氨酸位置高度保守,说明了远缘物种BMP10蛋白的高度保守性,也预示了其功能的相似性。
关于BMP家族基因参与贝类幼体发育的研究已有报道。KIN等[28]研究刺牡蛎(Saccostrea kegaki)dpp(Decapentapletic)基因在胚胎发育时期的表达,发现其表达于贝壳绞合线部位,认为其可能参与贝壳绞合线结构的建立。NEDERBRAGT等[29]发现欧洲帽贝(Patella vulgata)的dpp-BMP2/4基因在胚胎外胚层周围表达,认为其可能参与胚胎贝壳边界的确定及贝壳形成。封利颖等[30]研究栉孔扇贝(Chlamys farreri)BMP2基因不同发育时期的表达情况,发现其在囊胚期表达量最高,从受精卵到壳顶期的其他时期表达量均较低。该实验中PfBMP10在担轮幼虫期和变态期高表达,和封利颖等[30]的结果略有差异,但相似之处是目的基因均在幼体发育早期大量表达。KAKOI等[31]、SILBERFELD和GROS [32]分别研究了刺牡蛎和马珂三角文蛤(Tivela mactroides)的胚胎发育过程,认为在担轮幼虫期幼体形成壳腺并开始分泌基质,壳形成区外翻逐渐包围整个胚胎。PfBMP10在担轮期极端高表达,暗示了其在担轮期的重要作用。另外PfBMP10在变态期表达量也较高,贝类在变态期会发生一系列剧烈的形态和生理变化,例如面盘消失,转为利用鳃呼吸,这些变化涉及器官形成、细胞分化和凋亡。由此推测PfBMP10可能参与幼体担轮期和变态期的发育调控,可能和壳、鳃等器官的发生有关。该实验只研究了从担轮幼虫期开始的5个发育时期,要得到更全面的结果,可以针对受精卵至完成变态的全过程,缩短取样间隔进行充分研究。另外有待通过原位杂交确定基因表达的具体区域,以期得到PfBMP10在早期发育阶段起作用的更详细证据。
有研究者对BMP家族基因在成体贝类不同组织的表达情况进行过研究。封利颖等[30]研究栉孔扇贝BMP2基因在不同组织的表达情况,显示其在鳃中表达量最高,而外套膜次之。MIYASHITA等[25]克隆了合浦珠母贝的BMP2基因,表达分析显示其在外套膜、鳃和肝中表达量较高,外套膜原位杂交分析显示BMP2基因在外套膜外侧上皮和外褶的内侧上皮高表达,这些部位被认为是合成珍珠层的主要部位,所以推测合浦珠母贝BMP2基因参与贝壳珍珠层的形成。该研究显示PfBMP10在所有组织中均有表达,在鳃中表达量最高,其次为外套膜。和上述研究结果相似,由此推测PfBMP10与贝壳的形成有关。另外该实验和前述研究也都发现目的基因在鳃中有高表达,这一现象值得探讨。鳃是滤食性贝类的呼吸取食器官,罗文等[33]研究了三角帆蚌(Hyriopsis cumingii)珍珠形成初期钙的代谢特征,发现植片后鳃、外套膜、围心腔的钙含量都有上升,鳃中的含量最高,说明三角帆蚌主要是通过鳃吸收和转运钙离子。推测这种机制在贝类的生物矿化中普遍存在。由此推测高表达于鳃和外套膜的PfBMP10可能通过参与钙离子转运而影响贝壳和珍珠形成,相关的证据有待进一步探寻。
ZHOU等[34]进行了合浦珠母贝的缺刻实验,半定量PCR分析显示在缺刻6 h后外套膜中PfBMP2表达量显著上升,证明PfBMP2参与贝壳的损伤修复。该研究中贝壳缺刻处理后缺口位置产生明显变化,至第48小时可见明显的新生半透明薄层,应是新生贝壳,证明贝壳缺损后即开始再生。实验显示在处理24 h后鳃和外套膜中PfBMP10表达量都显著上升,推测PfBMP10参与贝壳损伤的修复。
PfBMP10基因是继BMP2、BMP7之后鉴定的第3个合浦珠母贝BMP家族基因。有研究者对重组人BMP2蛋白和自然提取BMP蛋白的成骨活性进行比较,发现在同等剂量下,重组BMP2的成骨活性不及后者1/10。对此的一种解释是自然提取BMP蛋白是混合蛋白,包含多种BMP,不同BMP蛋白之间有协作效应,导致其成骨活性强于单成分的重组BMP2[3, 35]。因此有待进一步开发合浦珠母贝的其他BMP家族基因,为研究其协同作用进而利用混合蛋白加快育珠提供条件。该研究结果可为BMP家族基因的功能研究及贝壳形成机理研究提供参考。
-
图 1 PfBMP10基因的cDNA序列和编码的氨基酸序列
左侧正常和加粗数字分别标明PfBMP10基因cDNA序列和氨基酸残基位置; cDNA序列中加粗部分分别为起始和终止密码子,氨基酸序列中带细下划线的是信号肽序列,带粗下划线的是前结构域;灰色部分为成熟区,方框标记7个半胱氨酸残基。
Figure 1. cDNA and amino acid sequences of PfBMP10 gene
The bold and normal numbers on the left indicate the positions of PfBMP10 gene cDNA sequence and amino acid residues, respectively. The bold letters of cDNA sequence are initiation codon and termination codon. The putative signal peptide and pro-domain are indicated with fine and heavy underline, respectively. The mutation protein which includes seven boxed cysteine residues are shaded in gray.
图 4 PfBMP10在合浦珠母贝不同发育时期的相对表达量
不同字母表示差异显著(P<0.05),后图同此。
Figure 4. Relative expression level of PfBMP10 in different tissues of P.fucata
T. trochophore; D. D-veliger larvae; U. umbo veliger larvae; E. eyebot larvae; M. metamorphosis. The significant difference is indicated by different letters (n=3, P < 0.05). The same case in the following figures.
图 7 贝壳缺刻处理后不同时间鳃和外套膜中PfBMP10的相对表达量
A和B分别为鳃和外套膜实验结果,不同小写字母代表不同处理时间下基因表达量差异显著(n=3,P<0.05),*表示与对照组相比差异显著(n=3,P<0.05)。
Figure 7. Relative expression level of PfBMP10 at different time after shell notching
A and B are experiment results of gill and mantle. Different lower-case letters indicate significant difference of gene expression level among different treatment groups (n=3, P < 0.05). * indicate significant difference compared with control (n=3, P < 0.05).
表 1 实验所用引物序列
Table 1 Sequences of primers used in the study
引物名称
primer name引物序列(5′→3′)
primer sequence用途
usageS1 TAAGGAGGAGTTGAGGAA 序列验证 A1 AGGCTGGACTGTAGGATC 序列验证 S2 GTTAGGTCTTTATCGCTTAG 中间片段扩增 A2 ACAACCATTCCCTCATA 中间片段扩增 S3 GATTTTGGAGATTCGTAT 序列验证 A3 CTGTTGTCCTTGCCTTTT 序列验证 5′GSP-out ATAGTCAGGTGCCTCTTT 5′RACE 5′GSP-in TTACTAAGCGATAAAGACC 5′RACE qRT-S ATGACCACAAGACCCACTA 荧光定量 qRT-A CTTCTCCTCTATGATTCCA 荧光定量 18S TGTCTGCCCTATCAACTTTC 内参引物 18A TGTGGTAGCCGTTTCTCA 内参引物 -
[1] URIST M R. Bone: formation by autoinduction[J]. Science, 1965, 150 (3698): 893-899. doi: 10.1126/science.150.3698.893
[2] CHIN D, BOYLE G M, PARSONS P G, et al. What is transforming growth factor-beta (TGF-β)?[J]. Brit J Plast Surg, 2004, 57 (3): 215-221. doi: 10.1016/j.bjps.2003.12.012
[3] CARREIRA A C, ALVES G G, ZAMBUZZI W F, et al. Bone morphogenetic proteins: structure, biological function and therapeutic applications[J]. Arch Biochem Biophys, 2014, 561 (6): 64-73. https://www.sciencedirect.com/science/article/abs/pii/S0003986114002549
[4] BRAGDON B, MOSEYCHUK O, SALDANHA S, et al. Bone morphogenetic proteins: a critical review[J]. Cell Signal, 2010, 23 (23): 609-720. https://pubmed.ncbi.nlm.nih.gov/20959140/
[5] GRIFFITH D L, KECK P C, SAMPATH T K, et al. Three-dimensional structure of recombinant human osteogenic protein 1: structural paradigm for the transforming growth factor beta superfamily[J]. P Natl Acad Sci USA, 1996, 93 (2): 878-883. doi: 10.1073/pnas.93.2.878
[6] BUTLER S J, DODD J. A role for BMP heterodimers in roof plate-mediated repulsion of commissural axons[J]. Neuron, 2003, 38 (3): 389-401. doi: 10.1016/S0896-6273(03)00254-X
[7] CHENG H, JIANG W, PHILLIPS F M, et al. Osteogenic activity of the fourteen types of human bone morphogenetic proteins (BMPs)[J]. J Bone Joint Surg Am, 2003, 85 (8): 79-80. doi: 10.2106/00004623-200308000-00017
[8] RAHMAN M S, AKHTAR N, JAMAIL H M, et al. TGF-β/BMP signaling and other molecular events: regulation of and bone formation[J]. Bone Res, 2015, 3 (1): 11-30. https://pubmed.ncbi.nlm.nih.gov/26273537/
[9] BIBBO C, NELSON J, EHRLICH D, et al. Bone morphogenetic proteins: indications and uses[J]. Clin Podiatr Med Sur, 2015, 32 (1): 35-43. doi: 10.1016/j.cpm.2014.09.005
[10] JIA S J, DAI F Y, WU D, et al. Protein phosphatase 4 cooperates with smads to promote BMP signaling in dorsoventral patterning of zebrafish embryos[J]. Dev Cell, 2012, 22 (5): 1065-1078. doi: 10.1016/j.devcel.2012.03.001
[11] LIU A M, NISWANDER L. Bone morphogenetic protein signalling and vertebrate nervous system development[J]. Nat Rev Neurosci, 2005, 6 (12): 945-954. doi: 10.1038/nrn1805
[12] 王伟, 王少兵, 徐银学. BMP/Smad信号通路与哺乳动物卵泡发生[J]. 遗传, 2009, 31 (3): 245-254. doi: 10.3760/cma.j.issn.1003-9406.2009.03.002 [13] CHEN D, ZHAO M, MUNDY G R. Bone morphogenetic proteins[J]. Growth Factors, 2004, 22 (4): 233-241. doi: 10.1080/08977190412331279890
[14] FENG X H, DERYNCK R. Specificity and versatility in TGF-β signaling through Smads[J]. Annu Rev Cell Dev Bi, 2005, 21 (1): 659-693. doi: 10.1146/annurev.cellbio.21.022404.142018
[15] SHI Y G, MASSAGUE J. Mechanisms of TGF-beta signaling from cell membrane to the nucleus[J]. Cell, 2003, 113 (6): 685-700. doi: 10.1016/S0092-8674(03)00432-X
[16] 邹柯姝, 张殿昌, 郭华阳, 等. 4种壳色合浦珠母贝贝壳棱柱层和珍珠质层7种金属元素质量分数的比较分析[J]. 南方水产科学, 2015, 11 (3): 74-79. doi: 10.3969/j.issn.2095-0780.2015.03.012 [17] WEINER S, ADDADI L. Design strategies in mineralized biological materials[J]. J Mater Chem A, 1997, 7 (5): 689-702. doi: 10.1039/a604512j
[18] ZHANG C, ZHANG R Q. Matrix proteins in the outer shells of molluscs[J]. Mar Biotechnol, 2006, 8 (6): 572-586. doi: 10.1007/s10126-005-6029-6
[19] MARIE B, JOUBERT C, TAYALE A, et al. Different secretory repertoires control the biomineralization processes of prism and nacre deposition of the pearl oyster shell[J]. P Natl Acad Sci USA, 2012, 109 (51): 20986-20991. doi: 10.1073/pnas.1210552109
[20] OLIVEIRA D V, SILVA T S, CORDEIRO O D, et al. Identification of proteins with potential osteogenic activity present in the water-soluble matrix proteins from Crassostrea gigas nacre using a proteomic approach[J]. Sci World J, 2012, 1252 (2): 361-367.
[21] WANG X T, LI L, ZHU Y B, et al. Oyster shell proteins originate from multiple organs and their probable transport pathway to the shell formation front[J]. PLoS ONE, 2013, 8 (6): e66522. doi: 10.1371/journal.pone.0066522
[22] FREER A, BRIDGETT S, JIANG J H, et al. Biomineral proteins from Mytilus edulis mantle tissue transcriptome[J]. Mar Biotechnol, 2014, 16 (1): 34-45. doi: 10.1007/s10126-013-9516-1
[23] MICHIO S, KAZUKO S, TOSHIHIRO K, et al. An acidic matrix protein, Pif, is a key macromolecule for nacre formation[J]. Science, 2009, 325 (5946): 1388-1390. doi: 10.1126/science.1173793
[24] 周玉娟. 合浦珠母贝TGFβ信号通路相关基因的克隆及功能研究[D]. 北京: 清华大学, 2010: 95-96.https://cdmd.cnki.com.cn/Article/CDMD-10003-1011280531.htm [25] MIYASHITA T, HANASHITA T, TORIYAMA M, et al. Gene cloning and biochemical characterization of the BMP-2 of Pinctada fucata[J]. Biosci Biotech Bioch, 2008, 72 (1): 37-47. doi: 10.1271/bbb.70302
[26] YAN F, LUO S, JIAO Y, et al. Molecular characterization of the BMP7 gene and its potential role in shell formation in Pinctada martensii[J]. Int J Mol Sci, 2014, 15 (11): 21215-21228. doi: 10.3390/ijms151121215
[27] MOUNT A S, WHEELER A P, PARADKAR R P, et al. Hemocyte-mediated shell mineralization in the eastern oyster[J]. Science, 2004, 304 (5668): 297-300. doi: 10.1126/science.1090506
[28] KIN K, KAKOI S, WADA H. A novel role for dpp in the shaping of bivalve shells revealed in a conserved molluscan developmental program[J]. Dev Biol, 2009, 329 (1): 152-166. doi: 10.1016/j.ydbio.2009.01.021
[29] NEDERBRAGT A J, van LOON A E, DICTUS W J A G. Expression of Patella vulgata orthologs of engrailed and dpp-BMP2/4 in adjacent domains during molluscan shell development suggests a conserved compartment boundary mechanism[J]. Dev Biol, 2002, 246 (2): 341-55. doi: 10.1006/dbio.2002.0653
[30] 封利颖, 郭慧慧, 李雪, 等. 栉孔扇贝骨形态发生蛋白2基因的克隆及表达分析[J]. 中国海洋大学学报(自然科学版), 2013, 43 (12): 37-43. doi: 10.16441/j.cnki.hdxb.2013.12.008 [31] KAKOI S, KIN K, MIYAZAKI K, et al. Early development of the japanese spiny oyster (Saccostrea kegaki): characterization of some genetic markers[J]. Zool Sci, 2008, 25 (5): 455-464. doi: 10.2108/zsj.25.455
[32] SILBERFELD T, GROS O. Embryonic development of the tropical bivalve Tivela mactroides (Born, 1778) (Veneridae: subfamily Meretricinae) : a SEM study[J]. Cah Biol Mar, 2006, 47 (3): 243-251.
[33] 罗文, 杨受保, 丁盈, 等. 三角帆蚌(Hyriopsis cumingii)珍珠形成初期钙代谢的特征分析[J]. 海洋与湖沼, 2010, 41 (6): 895-900. https://www.doc88.com/p-4903125196446.html [34] ZHOU Y J, HE Z X, LI C Z, et al. Correlations among mRNA expression levels of Engrailed, BMP2 and smad3 in mantle cells of pearl oyster Pinctada fucata[J]. Prog Biochem Biophys, 2010, 37 (7): 737-746. doi: 10.3724/SP.J.1206.2010.00040
[35] BESSHO K, KUSUMOTO K, FUJIMURA K, et al. Comparison of recombinant and purified human bone morphogenetic protein[J]. Brit J Oral Max Surg, 1999, 37 (1): 2-5. doi: 10.1054/bjom.1998.0379
-
期刊类型引用(6)
1. 陈一格,李文娟,申晓雅,朱琪,王贺,李家乐,白志毅. 三角帆蚌IGF2BP通过AKT通路促进外套膜矿化相关研究. 水生生物学报. 2024(11): 1943-1951 .  百度学术
百度学术
2. 农珍珍,蒙扬辉,陈肇聪,蒋美君,孙若鹏,朱鹏,覃波忠,钟方杰,张斌,严雪瑜. 基于不同生长时期香港牡蛎转录组数据分析BMP蛋白家族的表达变化. 农业与技术. 2023(12): 109-112 .  百度学术
百度学术
3. 李莹莹,崔东遥,程操,孙景贤,李丹彤,李家祥,常亚青,湛垚垚. 水产动物TGF-β超家族基因系统进化及生物学功能研究进展. 大连海洋大学学报. 2019(06): 853-862 .  百度学术
百度学术
4. 范嗣刚,周代志,刘宝锁,邓正华,郭奕惠,喻达辉. 合浦珠母贝BMP7b基因的克隆与表达分析. 南方水产科学. 2018(01): 121-126 .  本站查看
本站查看
5. 何文耀,范嗣刚,刘宝锁,张博,周童,苏家齐,邓正华,喻达辉. 合浦珠母贝胸腺素β4(thymosin beta4)插核损伤和发育时期的表达研究. 南方水产科学. 2018(02): 66-74 .  本站查看
本站查看
6. 周童,刘宝锁,张博,孟子豪,何文耀,喻达辉. 类胡萝卜素对合浦珠母贝热休克蛋白基因HSP22表达的影响. 南方水产科学. 2018(05): 60-69 .  本站查看
本站查看
其他类型引用(3)



 下载:
下载:


 粤公网安备 44010502001741号
粤公网安备 44010502001741号
