Effect of pearl culture on N19 and Prismalin-14 genes expression in the pearl oyster Pinctada fucata
-
摘要:
为探讨育珠对壳基质蛋白基因表达的影响,开展了合浦珠母贝(Pinctada fucata)N19和Prismalin-14基因在育珠贝和非育珠贝中的荧光定量表达研究。结果显示,N19和Prismalin-14在育珠贝与非育珠贝的闭壳肌、肝胰脏、性腺、肠、鳃等组织中均无表达,在外套膜均有表达;N19在珍珠囊表达,而Prismalin-14则不表达。N19与Prismalin-14在育珠贝外套膜的表达量均大于非育珠贝,表明育珠可能对某些壳基质蛋白的表达具有一定的促进作用。其中Prismalin-14在育珠贝外套膜的表达量最高(P<0.01),是非育珠贝外套膜Prismalin-14的1.23倍,是育珠贝外套膜N19的25.04倍,是非育珠贝外套膜N19的41.04倍,推测在珍珠贝生长过程中,对Prismalin-14的需求量可能比N19大。N19在育珠贝珍珠囊中表达量最高(P<0.01),是育珠贝N19外套膜的11.58倍,是非育珠贝外套膜的18.97倍,推测在珍珠形成的过程中,对N19的需求量可能比贝壳自身生长对N19的需求量大。
Abstract:To investigate the effect of pearl culture on the expressions of genes encoding shell matrix proteins, the relative expression levels of N19 and Prismalin-14 genes in the pearl-culturing oysters (PC) and non-pearl-culturing oysters (NPC) Pinctada fucata were investigated by using RT-FQ-PCR. The results show that the expressions of both N19 and Prismalin-14 are extremely weak or negative in muscle, liver, gonad, bowel or gill of PC and NPC, but positive in the mantle. The expression of N19 is obviously in pearl sac, while of Prismalin-14 is negative. The expressions of both N19 and Prismalin-14 in PC mantle are higher than that in NPC, which indicate pearl culture may promote expressions of some shell matrix protein genes. The relative expression level of Prismalin-14 is the highest (P < 0.01) in PC mantle, which is 1.23-fold greater than that of NPC mantle, 25.04-fold greater than N19 in PC mantle, 41.04-fold greater than N19 in NPC mantle, suggesting that pearl oysters may demand much more Prismalin-14 than N19 does. Meanwhile, the relative expression level of N19 is the highest in the pearl sac (P < 0.01), being 11.58-fold greater than that of the PC mantle, 18.97-fold greater than that of the NPC mantle, suggesting that pearl growth may demand much more N19 than shell growth does.
-
Keywords:
- shell matrix protein /
- RT-FQ-PCR /
- gene expression /
- pearl culture
-
生物胺作为神经递质在动物的生理过程起到重要的作用,大多数海洋无脊椎动物的附着变态都与神经系统密切相关,在浮游幼虫充分发育至附着变态时,神经递质甚至被认为开启了附着变态的发生。COON等[1-2]发现肾上腺素、左旋多巴、多巴胺和章鱼胺能够诱导牡蛎的附着变态。张涛等[3]也在海湾扇贝(Argopecten irradias)中发现了类似现象,且发现其幼虫体内多巴胺含量在附着变态期间显著上升。柯才焕等[4-5]也发现肾上腺素对翡翠贻贝(Perna viridis)的附着变态有显著诱导作用。相对于肾上腺素、左旋多巴和多巴胺,目前对软体动物章鱼胺及其受体的研究还比较少见。
章鱼胺(Octopamine,OA)广泛存在于无脊椎动物中,是神经系统中存在的多种微量生物胺之一,是脊椎动物的去甲肾上腺素同类物,在无脊椎动物中可作为神经递质或神经调节物,通过与章鱼胺受体(Octopamine receptor, OAR)相结合传递神经信号[6-7]。至今,已有4名诺贝尔奖获得者从事关于章鱼胺的研究,分别涉及章鱼胺受体识别、线虫体内章鱼胺功能以及脊椎动物体内痕量生物胺探寻等领域。章鱼胺和去甲肾上腺素均属于酪氨酸衍生物,它们在贝类生物中作为神经信号分子被利用,脊椎动物则充分利用了去甲肾上腺素及其衍生物肾上腺素,却很少利用章鱼胺[8]。章鱼胺通过受体实施各种各样的功能,很多种受体的分子功能与特征不仅在果蝇(Drosophila)中已经弄清,在昆虫中也进行了研究[9]。在果蝇中,章鱼胺涉及到攻击性行为[10-11]、睡眠[12]和生殖过程[13-16];在昆虫中,章鱼胺及其受体则具有控制运动与栖息的功能[17]。目前,章鱼胺受体已经从软体动物腹足类的椎实螺属(Lymnaea)[18-19]和海兔螺属(Aplysia)[20]中被鉴定出来,且发现其在生命活动中能够行使控制行为发生、参与新陈代谢以及信号转导等多种生理功能[21-23]。BLAIS等[24]报道了大西洋浪蛤(Spisula solidissima)的一种章鱼胺受体,认为该受体是组成型表达,但并未对其功能作进一步研究。王敏等[25]在海洋贝类中检测到丰富的章鱼胺,其含量是海洋鱼类等其他生物的几十倍之多。PRYCE等[26]通过免疫印迹分析技术、HPLC分析技术,在美洲牡蛎(Crassostrea virginica)的鳃、外套膜、心脏、血淋巴以及神经系统中检测到了OA和OAR的存在,并证实了其在美洲牡蛎的心脏活动中行使重要功能。
合浦珠母贝(Pinctada fucata)又称马氏珠母贝,隶属于软体动物门中的双壳纲,是生产海水珍珠的主要贝类。合浦珠母贝在它们复杂的生命活动中,需要经历一个自由游动的早期变态发育过程[27],按照时间顺序分为受精卵-担轮期-D型期-壳顶期[28]。在变态发育的过程中,其幼虫在形态、生理、结构和功能方面都会发生显著变化[29],推测该过程可能受到OAR基因的调控作用。贝壳是贝类重要的组成部分,贝的生长发育和珍珠的形成都与贝壳密不可分,而贝壳的形成又受到基因的调控,关于贝壳的理化特性和形成机理一直是研究的热点[30-31]。鉴于OAR基因的多功能性,该实验进行了合浦珠母贝OAR基因的克隆,通过实时荧光定量PCR技术分析其在不同发育时期和不同组织中的表达情况,并通过贝壳缺刻实验探究其在贝壳形成及修复中的调控作用。对合浦珠母贝OAR基因的研究将有助于了解贝类生长发育和贝壳形成的分子机制,为贝类育种研究提供理论基础。
1. 材料与方法
1.1 实验材料
实验所用合浦珠母贝取自海南陵水养殖基地,壳长为60~80 mm,解剖后分别取合浦珠母贝外套膜、闭壳肌、肝胰腺、鳃、珍珠囊和肠6个组织。另取性成熟贝进行解剖取精卵、体外受精,随后取5个发育时期样品,包括担轮期(受精后约8 h)、D型期(受精后24 h)、壳顶期(受精后4~7 d)、眼点期(受精后9~15 d)和变态期幼虫(受精后11~20 d)。样品放入RNA保护液(TaKaRa,大连)中,-80 ℃保存备用。
1.2 实验方法
1.2.1 总RNA提取与cDNA合成
用上述所取得外套膜组织样品,按照Trizol(Invitrogen,USA)传统方法提取外套膜组织的总RNA,在DEPC水中溶解,用1%的琼脂糖凝胶电泳检测其完整性,核酸微量定量仪ND-2000检测A260/A280比值和浓度。按照cDNA合成试剂盒进行反转录,产物在-20 ℃冰箱保存备用。
1.2.2 转录组序列的分析及基因克隆
根据合浦珠母贝转录组序列中的OAR基因片段,设计3′引物(OAR-3′-S1:ACAAGGCGACGCAGGTTAGT和OAR-3′-S2:ACTATCTATCAGTGACGGGTAC)。根据SMART-RACE试剂盒(Clontech,广州),通过巢式PCR扩增OAR基因cDNA全长。然后通过NCBI中ORF finder预测开放阅读框(ORF)序列,并设计引物(ORF-S:ATGTCAGTGATTAATTTATC和ORF-A:TGCAGGACGTTTTTGTTTGG)扩增验证。用1.5%的琼脂糖凝胶进行电泳,对目的片段胶回收。将目的片段与pMD18-T载体经连接、转化等步骤(参照傅明骏等[32]的方法),挑选阳性克隆,送英潍捷基(上海)有限公司测序。
1.2.3 生物信息学分析
利用NCBI数据库中的Blast (http://blast.ncbi.nlm.nih.gov/Blast.cgi)、OFR Finder(http://www.ncbi.nlm.nih.gov/projects/gorf/orfig.cgi)、VecScreen (http://www.ncbi.nlm.nih.gov/VecScreen/VecScreen.html)等软件对测序结果进行分析,验证该目的基因并将其拼接得到该基因的全长cDNA序列;使用ProtParam (http://web.expasy.org/protparam/)预测编码蛋白的理化性质;使用NetPhos 2.0 Server (http://www.cbs.dtu.dk/services/NetPhos/)预测磷酸化位点;使用SMART(http://smart.embl-heidelberg.de/)预测其蛋白结构域;用SignalP 4.1(http://www.cbs.dtu.dk/services/SignalP/)预测信号肽序列;使用PSORT Ⅱ Prediction (http://psort.hgc.jp/form2.html)进行细胞定位的预测;利用MatGAT 2.02(http://ww3.bergen.edu/faculty/jsmalley/matgat.html)进行序列相似性和一致性分析;利用DNAMAN软件进行同源序列比对。用NetNGlyc 1.0(http://www.cbs.dtu.dk/services/NetNGlyc/)预测OAR的糖基化位点。利用Mega 6.0软件,用临位相接法(neighbor-joining)构建系统进化树。
1.2.4 目的基因在不同组织和不同发育时期的表达
提取合浦珠母贝不同组织(鳃、肝胰腺、外套膜、闭壳肌、珍珠囊、肠道)和不同发育时期(担轮期、D型期、壳顶期、眼点期、变态期)总RNA,按照荧光定量反转录试剂盒(TaKaRa,大连)的说明合成cDNA。根据OAR的cDNA序列设计特异性引物为qOAR-S和qOAR-A(qOAR-S:GGAACCATCAACCGAATC和qOAR-A:CCGTCAA-TACTACGCAAG),内参基因18S的引物为q18S-S和q18S-A(q18S-S:TGTCTGCCCTATCAACTTT和q18S-A:TGTGGTAGCCGTTTCTCA)。按照TaKaRa SYBR PremixExtaqTMKit(Perfect Real Time)试剂盒进行PCR反应。每个组织或时期样品取3个重复,每个样品进行3次重复,用蒸馏水作为阴性对照。实验结果采用相对荧光定量法(△CT法即2-ΔΔCt)[33]分析OAR基因在合浦珠母贝各个组织及不同发育时期的相对表达量。
1.2.5 缺刻实验
选取28只壳长为(6±0.5)cm的健康成贝装入一个贝笼,再将所有贝暂养于水泥池中,定期饲喂并每天换水,保持水质稳定。3 d后随机挑选24只进行缺刻处理,方法参照MOUNT等[34],在足的对面用手术剪将贝壳剪出深度约1 cm的V形缺口,确保损伤珍珠层而不破坏外套膜。处理后将贝放回原贝笼,继续饲养。另4只贝不做处理,作为对照组。分别在处理0 h、6 h、12 h、24 h、36 h、48 h后各取4只处理贝。观察处理贝切口变化,解剖后取外套膜组织,提取总RNA,反转录并进行实时荧光定量PCR反应,方法同1.2.4。
2. 结果
2.1 OAR的cDNA序列特征
使用基因特异性引物进行RACE扩增,经过拼接之后获得目的基因cDNA序列全长(图 1),命名为pfOAR。pfOAR cDNA全长为1 890 bp,开放阅读框(ORF)序列长度为1 659 bp,5′UTR为113 bp,3′UTR为118 bp。预测OAR基因编码552个氨基酸,理论分子量为63.167 kD,理论等电点为9.19。SMART结构域预测显示,PfOAR具有7个跨膜结构域(74~96 aa、109~131 aa、146~168 aa、189~211 aa、243~265 aa、476~498 aa和508~530 aa)。SignalP 4.1软件分析表明,PfOAR不存在信号肽序列。PSORT Ⅱ Prediction细胞定位显示,PfOAR位于细胞膜上,属于跨膜蛋白。NetPhos 2.0 Server分析显示PfOAR具有42个磷酸化位点,其中丝氨酸位点28个,苏氨酸位点8个,酪氨酸位点6个。NetNGlyc 1.0显示PfOAR编码蛋白有6个糖基化位点。
![]() 图 1 OAR基因cDNA全长序列及氨基酸序列左侧正常和加粗数字分别标明OAR基因cDNA序列和氨基酸残基位置;cDNA序列中方框内分别为起始和终止密码子;阴影部分为跨膜结构域(TM)。Figure 1. Nucleotide and deduced amino acid sequences of OARThe bold and normal numbers on the left indicate the positions of the OAR gene cDNA sequence and the amino acid residues, respectively; the boxed letters of cDNA sequence are initiation codon and termination codon; the transmembrane domains (TM) are highlighted.
图 1 OAR基因cDNA全长序列及氨基酸序列左侧正常和加粗数字分别标明OAR基因cDNA序列和氨基酸残基位置;cDNA序列中方框内分别为起始和终止密码子;阴影部分为跨膜结构域(TM)。Figure 1. Nucleotide and deduced amino acid sequences of OARThe bold and normal numbers on the left indicate the positions of the OAR gene cDNA sequence and the amino acid residues, respectively; the boxed letters of cDNA sequence are initiation codon and termination codon; the transmembrane domains (TM) are highlighted.通过DNAMAN软件将合浦珠母贝OAR蛋白序列与太平洋牡蛎(C.gigas)、静水椎实螺(Lymnaea stagnalis)等物种的OAR蛋白序列进行比对,结果显示PfOAR与其他软体动物的OAR蛋白序列具有较高的一致性,7个跨膜结构域高度保守(图 2)。选取哺乳类、爬行类等脊椎动物的肾上腺素受体(AR)蛋白序列与合浦珠母贝等无脊椎动物物的OAR蛋白序列构建系统进化树。结果显示,PfOAR与太平洋牡蛎OAR蛋白聚为一支,然后与海蜗牛(Aplysia californica)、静水椎实螺的OAR蛋白聚为一支(图 3)。
2.2 OAR基因在不同组织中的表达
荧光定量PCR检测结果显示,pfOAR在合浦珠母贝6个被检测的组织中均有表达,表达水平存在显著的组织差异性。pfOAR在外套膜中表达水平最高,显著高于其他组织(P < 0.05),其他5个被检测组织中pfOAR表达水平差异不显著(图 4)。
![]() 图 4 OAR基因在合浦珠母贝不同组织中的表达水平M.外套膜;P.珍珠囊;I.肠;H.肝胰腺;G.鳃;A.闭壳肌; 不同字母表示差异显著(P < 0.05), 后图同此。Figure 4. Relative expression of pfOAR in different tissues of P.fucataM. mantle; P. pearl sac; I. intestine; H. hepatopancreas; G. gill; A. adductor muscle. The significant difference is indicated by different letters (P < 0.05).The same case in the following figures.
图 4 OAR基因在合浦珠母贝不同组织中的表达水平M.外套膜;P.珍珠囊;I.肠;H.肝胰腺;G.鳃;A.闭壳肌; 不同字母表示差异显著(P < 0.05), 后图同此。Figure 4. Relative expression of pfOAR in different tissues of P.fucataM. mantle; P. pearl sac; I. intestine; H. hepatopancreas; G. gill; A. adductor muscle. The significant difference is indicated by different letters (P < 0.05).The same case in the following figures.2.3 OAR基因在不同发育时期中的表达
利用qRT-PCR技术对合浦珠母贝5个不同发育时期(担轮期、D型期、壳顶期、眼点期、变态期)的pfOAR mRNA表达量进行检测,结果显示,pfOAR mRNA在变态期的表达量显著高于其他4个发育时期(P < 0.05)(图 5)。
2.4 OAR基因缺刻实验分析
利用qRT-PCR技术,在进行缺刻实验后对pfOAR mRNA在合浦珠母贝外套膜组织中的表达情况进行检测,结果显示,pfOAR mRNA在贝壳缺刻后0~24 h表达量缓慢上升,但差异不显著,缺刻36 h后表达量显著升高(P < 0.05),随后表达量下降(图 6)。
3. 讨论
章鱼胺受体广泛存在于无脊椎动物中,主要为G蛋白偶联受体家族成员[17],具有调节神经肌源性节奏收缩、调节cAMP水平、参与磷酸化途径等重要作用[7]。该研究利用RACE技术克隆得到合浦珠母贝OAR基因的cDNA全长序列。生物信息学分析结果显示,PfOAR蛋白具有7个跨膜结构域,属于典型的G蛋白偶联受体,这与BLAIS等[24]的研究结果相符。多序列比对结果显示,PfOAR与太平洋牡蛎、静水椎实螺等其他软体动物的OAR蛋白序列具有较高的相似性,特别是在跨膜结构域部分具有高度的保守性,研究表明,跨膜区的保守有利于维持受体结构的稳定,为受体发挥功能提供保障[35]。系统进化树显示,PfOAR与太平洋牡蛎的OAR聚为一支,表明它们之间具有较高的同源性,哺乳类、爬行类、鱼类、节肢类等各自聚为一支,这与它们的生物学分类相符。
qRT-PCR表达分析结果显示,pfOAR mRNA在6个组织(外套膜、珍珠囊、肠、肝胰腺、鳃、闭壳肌)中均有表达,这与大西洋浪蛤[24]、美洲牡蛎[36]和紫贻贝(Mytilus galloprovincialis)[37]中的研究结果相似。但是这些研究并没有指出组织表达的差异性,该研究结果显示,pfOAR在不同组织中的表达量存在显著差异,在外套膜中的表达量显著高于其他组织。外套膜是参与生物矿化形成贝壳的重要组织,推测pfOAR可能在生物矿化过程中起到一定的调控作用。为了证实推测,该研究进一步做了合浦珠母贝贝壳的缺刻实验。qRT-PCR表达分析结果显示,破壳后pfOAR的表达量在0~24 h缓慢上调,在第36小时显著升高达到最大值,随后表达量下降。有研究报道,OAR基因的亚型oct1在受到刺激时,细胞内钙离子的浓度会明显增加,钙离子是参与贝壳形成的重要离子,这在一定程度上证明了该基因可能具有矿化功能[18-19]。该研究结果表明pfOAR可能参与了贝壳修复的调控过程。
章鱼胺及其受体与海洋贝类幼虫的附着变态密切相关。COON等[2]发现章鱼胺能够诱导牡蛎的附着变态,张涛等[3]在海湾扇贝中也发现了类似现象。该实验中pfOAR基因在合浦珠母贝不同发育时期(担轮期、D型期、壳顶期、眼点期、变态期)中的表达结果显示,pfOAR mRNA在变态期(即幼虫附着的时期)的表达量显著高于其他时期,这与上述研究结果相一致,表明pfOAR在合浦珠母贝的变态发育时期可能起重要作用。在变态发育过程中,也涉及到贝壳的发育[28, 38],包括胚壳Ⅰ(D型期早期)和胚壳Ⅱ(D型期后期和壳顶期)的形成,胚壳Ⅱ已经开始出现珍珠层和棱柱层的分层[39],因此变态发育过程也涉及到生物矿化的发生。此外,合浦珠母贝在变态发育过程中除了壳的发生以外,形态也发生了很大的变化,包括面盘完全退化、唇瓣形成、足由原来位于身体中后部转向前部,鳃丝明显向贝壳腹缘延伸等,因此,pfOAR在合浦珠母贝变态期的高表达表明该基因除了可能参与生物矿化、诱导附着外,也可能参与变态期相关器官的发生。
该研究通过pfOAR在合浦珠母贝各组织以及不同发育时期的相对表达情况以及缺刻实验,对其功能做了初步研究,表明pfOAR参与了贝壳生物矿化和附着变态等过程,但其具体调控通路有待进一步研究。
-
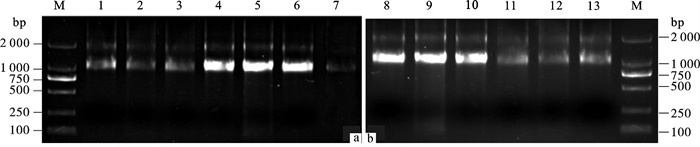
图 1 合浦珠母贝不同组织总RNA
a.育珠贝;b.非育珠贝;M.DNA分子质量标准DL2000;1~7. 鳃、性腺、肠、肝胰脏、外套膜、珍珠囊、闭壳肌;8~13.肝胰脏、外套膜、鳃、闭壳肌、性腺、肠
Figure 1. Total RNA extracted from P.fucata tissues
a. pearl-culturing oyster (PC); b. oyster without pearl-culture (NPC); M. DNA molecular weight marker (DL2000);1~7. gill, gonad, bowel, liver, mantle, pearl sac and muscle; 8~13. liver, bowel, gill, mantle, muscle and gonad
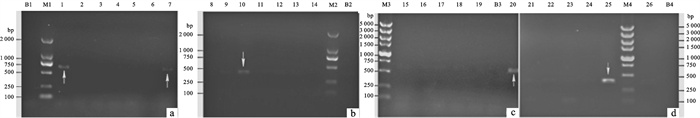
图 2 合浦珠母贝N19、Prismalin-14基因的组织表达谱[育珠贝(a、b)与非育珠贝(c、d)]
M1~M2. DNA分子质量标准DL2000;M3~M4. DNA分子质量标准DL5000;B1~B4.空白对照;1~7(N19).珍珠囊、鳃、性腺、肠、肝胰脏、闭壳肌、外套膜;8~14(Prismalin-14).肝胰脏、肠、外套膜、鳃、珍珠囊、闭壳肌、性腺;15~20(N19).闭壳肌、性腺、鳃、肠、肝胰脏、外套膜;21~26(Prismalin-14).肝胰脏、肠、鳃、闭壳肌、外套膜、性腺
Figure 2. The expression of N19 and Prismalin-14 genes in different tissues of P.fucata [PC(a, b) and NPC(c, d)]
M1~M2. DNA molecular weight marker (DL2000);M3~M4. DNA molecular weight marker (DL5000);B1~B4. blank control group; lane 1~7 (N19): pearl sac, gill, gonad, bowel, liver, muscle and mantle; lane 8~14 (Prismalin-14). liver, bowel, mantle, gill, pearl sac, muscle and gonad; lane 15~20 (N19). muscle, gonad, gill, bowel, liver, and mantle; lane 21~26 (Prismalin-14).liver, bowel, gill, muscle, mantle and gonad
表 1 引物序列
Table 1 Sequences of primers designed in this study
引物primer 引物序列(5′→3′)sequences PCR引物 N19-F CGGAATTCATGGTGTCCCATGCTCACAT primer for PCR N19-R CCCAAGCTTCTACGGCTTACAGCAACGGT Prismalin-14-F CGGAATTCATGCGATCTCTGCTAGTCCT Prismalin-14-R CCCAAGCTTTTAATCATCAAATCCGCCAT Real-Time FQ-PCR引物 N19-F-RT[16-17] TGGCAACAAAGCAGTCATAACCG primer for Real-Time FQ-PCR N19-R-RT[16-17] GGCGTCGTTGTAGCATTGAAGG Prismalin-14-F-RT[17] CAATGCGATCTCTGCTAGTCC Prismalin-14-R-RT[17] ATAGGAGAAACGCGGGAAATA 18S rRNA-F-RT TGTCTGCCCTATCAACTTTC 18S rRNA-R-RT TGTGGTAGCCGTTTCTCA -
[1] 胡曦璇, 石安静. 珍珠囊研究概况[J]. 水生生物学报, 1994, 18(1): 76-81. https://xueshu.baidu.com/usercenter/paper/show?paperid=b269449b51da12d8e9b0c71788c228f3&site=xueshu_se&hitarticle=1 [2] SUZUKI M, SARUWATARI K, KOGURE T, et al. An acidic matrix protein, Pif, is a key macromolecule for nacre formation[J]. Science, 2009, 325(5946): 1388-1390. doi: 10.1126/science.1173793
[3] 尹立鹏, 邓岳文, 杜晓东, 等. 贝龄对马氏珠母贝植核贝生长, 成活率和育珠性状的影响[J]. 中国水产科学, 2012, 19(4): 715-720. doi: 10.3724/SP.J.1118.2012.00715 [4] 金武. 三角帆蚌生长性状与育珠性状的遗传参数估计[D]. 上海: 上海海洋大学, 2012. https://d.wanfangdata.com.cn/thesis/ChhUaGVzaXNOZXdTMjAyNDA5MjAxNTE3MjUSB0QzNjYyNDUaCHR1cnE5c2du [5] 张希, 邢孔武, 喻达辉, 等. 合浦珠母贝不同来源群体小片贝的育珠效果比较[J]. 广东农业科学, 2011, 38(5): 153-154. doi: 10.3969/j.issn.1004-874X.2011.05.053 [6] 邓岳文, 邓陈茂, 杜晓东, 等. 细胞小片处理对马氏珠母贝育珠效果影响[J]. 海洋湖沼通报, 2011 (3): 46-49. doi: 10.3969/j.issn.1003-6482.2011.03.007 [7] YANO M, NAGAI K, MORIMOTO K, et al. A novel nacre protein N19 in the pearl oyster Pinctada fucata[J]. Biochem Biophys Res Commun, 2007, 362(1): 158-163. doi: 10.1016/j.bbrc.2007.07.172
[8] 张立娟, 何毛贤. 生长激素与胰岛素样生长因子-Ⅰ对马氏珠母贝壳生长形成相关基因表达的影响[J]. 热带海洋学报, 2012, 31(2): 96-101. doi: 10.3969/j.issn.1009-5470.2012.02.013 [9] SUZUKI M, MURAYAMA E, INOUE H, et al. Characterization of Prismalin-14, a novel matrix protein from the prismatic layer of the Japanese pearl oyster (Pinctada fucata)[J]. Biochem J, 2004, 382(Pt 1): 205-213.
[10] SUZUKI M, NAGASAWA H. The structure-function relationship analysis of Prismalin-14 from the prismatic layer of the Japanese pearl oyster, Pinctada fucata[J]. FEBS J, 2007, 274(19): 5158-5166. doi: 10.1111/j.1742-4658.2007.06036.x
[11] TAKEUCHI T, ENDO K. Biphasic and dually coordinated expression of the genes encoding major shell matrix proteins in the pearl oyster Pinctada fucata[J]. Mar Biotechnol, 2006, 8(1): 52-61. doi: 10.1007/s10126-005-5037-x
[12] INOUE N, ISHIBASHI R, ISHIKAWA T, et al. Gene expression patterns and pearl formation in the Japanese pearl oyster (Pinctada fucata): a comparison of gene expression patterns between the pearl sac and mantle tissues[J]. Aquaculture, 2010, 308: S68-S74. doi: 10.1016/j.aquaculture.2010.06.036
[13] INOUE N, ISHIBASHI R, ISHIKAWA T, et al. Comparison of expression patterns of shell matrix protein genes in the mantle tissues between high-and low-quality pearl-producing recipients of the pearl oyster, Pinctada fucata[J]. Zool Sci, 2011, 28(1): 32-36. doi: 10.2108/zsj.28.32
[14] YOKO M, TAKUMA N, HIDEO A, et al. Expression of genes responsible for biomineralization of Pinctada fucata during development[J]. Comp Biochem Physiol Part B: Biochem Mol Biol, 2010, 155(3): 241-248. doi: 10.1016/j.cbpb.2009.11.009
[15] 杨桂梅, 鲍宝龙, 任大明. 3-磷酸甘油醛脱氢酶、β-肌动蛋白和18S rRNA作为相对定量的内标在牙鲆发育阶段的稳定性比较[J]. 上海水产大学学报, 2005, 14(1): 84-88. [16] WANG N, KINOSHITA S, RIHO C, et al. Quantitative expression analysis of nacreous shell matrix protein genes in the process of pearl biogenesis[J]. Comp Biochem Physiol Part B: Biochem Mol Biol, 2009, 154(3): 346-350. doi: 10.1016/j.cbpb.2009.07.012
[17] ZHANG L, HE M. Quantitative expression of shell matrix protein genes and their correlations with shell traits in the pearl oyster Pinctada fucata[J]. Aquaculture, 2011, 314(1): 73-79. doi: 10.1016/j.aquaculture.2011.01.039
[18] SATO-OKOSHI W, OKOSHI K. Characteristics of shell microstructure and growth analysis of the Antarctic bivalve Laternula elliptica from Lützow-Holm Bay, Antarctica[J]. Polar Biol, 2008, 31(2): 131-138. doi: 10.1007/s00300-007-0340-9
[19] INOUE N, ISHIBASHI R, ISHIKAWA T, et al. Can the quality of pearls from the Japanese pearl oyster (Pinctada fucata) be explained by the gene expression patterns of the major shell matrix proteins in the pearl sac?[J]. Mar Biotechnol, 2011, 13(1): 48-55. doi: 10.1007/s10126-010-9267-1
[20] INOUE N, ISHIBASHI R, ISHIKAWA T, et al. Gene expression patterns in the outer mantle epithelial cells associated with pearl sac formation[J]. Mar Biotechnol, 2011, 13(3): 474-483. doi: 10.1007/s10126-010-9318-7
[21] 严俊, 邓小琼, 胡丹静, 等. 淡水养殖珍珠棱柱层的微结构形貌特征[J]. 南方水产科学, 2013, 9(1): 48-52. doi: 10.3969/j.issn.2095-0780.2013.01.008 [22] MA H, LI R, YANG L, et al. A modified integrated model of the internal structure of Chinese cultured pearls[J]. J Wuhan Univ Technol: Mater Sci Ed, 2011, 26(3): 510-513. doi: 10.1007/s11595-011-0258-5
[23] MA H, SU A, ZHANG B, et al. Vaterite or aragonite observed in the prismatic layer of freshwater-cultured pearls from South China[J]. Prog Nat Sci, 2009, 19(7): 817-820. doi: 10.1016/j.pnsc.2008.11.005
[24] MA H, ZHANG B, LEE I S, et al. Aragonite observed in the prismatic layer of seawater-cultured pearls[J]. Front Mater Sci China, 2007, 1(3): 326-329. doi: 10.1007/s11706-007-0061-6
-
期刊类型引用(6)
1. 张磊, 王锦旭, 杨贤庆, 魏涯, 杨少玲. 合浦珠母贝糖胺聚糖泡腾片制备工艺优化及其质量分析. 大连海洋大学学报. 2018(05): 644-650 .  百度学术
百度学术
2. 王珍珍, 黄桂菊, 范嗣刚, 刘宝锁, 张博, 苏家齐, 喻达辉. 合浦珠母贝matrilin-1基因的克隆和表达分析. 南方水产科学. 2017(01): 76-84 .  本站查看
本站查看
3. 谭才钢, 刘宝锁, 张东玲, 牛志凯, 张博, 陈明强, 范嗣刚, 姜松, 黄桂菊, 李有宁, 陈素文, 喻达辉. 合浦珠母贝主要形态性状与体质量的灰色关联分析. 南方水产科学. 2015(02): 35-40 .  本站查看
本站查看
4. 邹柯姝, 张殿昌, 郭华阳, 张楠, 江世贵. 4种壳色合浦珠母贝贝壳棱柱层和珍珠质层7种金属元素质量分数的比较分析. 南方水产科学. 2015(03): 74-79 .  本站查看
本站查看
5. 毕晓敏, 黄桂菊, 范嗣刚, 朱文杰, 喻达辉. 合浦珠母贝矿化基因Pearlin重组蛋白的表达条件优化. 南方水产科学. 2015(06): 100-106 .  本站查看
本站查看
6. 肖雅元, 徐姗楠, 刘永, 李纯厚. 0~#柴油水溶液对翡翠贻贝CYP4基因表达的影响研究. 南方水产科学. 2014(02): 61-66 .  本站查看
本站查看
其他类型引用(1)




 下载:
下载:






 粤公网安备 44010502001741号
粤公网安备 44010502001741号
