Structure and evolution of mitochondrial control regions in Evynnis cardinalis
-
摘要:
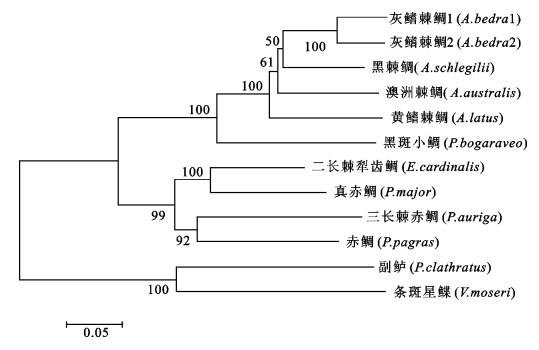
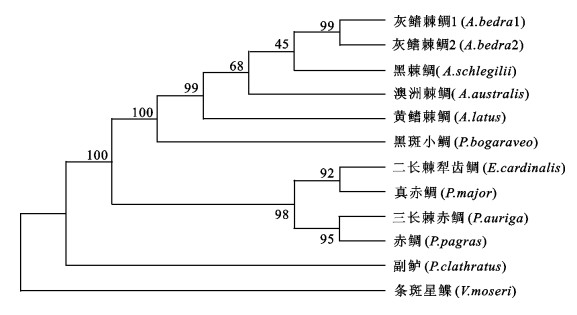
测定了二长棘犁齿鲷(Evynnis cardinalis)线粒体控制区全序列,并结合从GenBank中下载的9种鲷科鱼类的相应序列,采用Clustal X对控制区结构进行了分析,识别了相应的保守序列,包括终止相关序列ETAS、中央保守区的CSB-F、CSB-E、CSB-E、CSB-C、CSB-B和CSB-A序列,以及保守序列区的CSB1、CSB2和CSB3序列。以副鲈(Paralabrax clathratus)和条斑星鲽(Verasper moseri)为外群,用最大简约法(MP)和邻接法(NJ)构建了系统发育树。结果显示鲷科鱼类构成一个单系类群,二长棘犁齿鲷应该归入赤鲷属,建议有必要对犁齿鲷属和赤鲷属的分类地位重新评估。
Abstract:We determined the complete sequences of the mitochondrial DNA control regions in Evynnis cardinalis using long polymerase chain reaction (PCR), and aligned them by Clustal X with 9 related Sparidae sequences in GenBank. Several conserved sequence blocks were identified in the mitochondrial DNA control regions, including extended terminal associated sequences (ETAS), blocks of central domain (CSB-F, CSB-E, CSB-D, CSB-C, CSB-B, CSB-A) and conserved sequence blocks (CSB1, CSB2, CSB3). Taking Paralabrax clathratus and Verasper moseri as outgroups, the molecular phylogenetic trees of Sparidae were constructed based on neighbour-joining and maximum-parsimony methods, which reveal that Sparidae is a monophyletic group, and E.cardinalis should be grouped with Pagrus. Therefore, it is necessary to reevaluate the taxonomic status of Evynnis and Pagrus.
-
Keywords:
- Evynnis cardinalis /
- control region /
- structure /
- phylogeny
-
剩余产量模型因其简单的形式和所需数据较少的特点为渔业资源评估广泛采用[1]。当渔获物的年龄组成无法精确获得时,年龄结构模型(age-structured model)便不能应用,而剩余产量模型只需多年的渔获量和捕捞努力量或单位捕捞努力量渔获量的渔业统计数据。剩余产量模型也可作为年龄结构模型的辅助工具,为渔业动态研究提供不同的思路。这类模型的优点在于其形式简单便于评估和评估结果比较易于理解[2]。通常较难获得现实渔业的相关参数真值,文章主要利用蒙特卡罗(Monte Carlo)模拟方法来检验连续Fox产量模型在几种不同渔业史条件下的模拟渔业中评估结果的有效性,借此来探索能使连续Fox产量模型产生最佳评估效果所需要的渔业条件,为渔业管理部门和渔业种群数量变动的研究人员使用该种模型对真实渔业进行管理和研究时提供参考。
1. 材料与方法
1.1 模型
Fox模型[3]是一个连续的微分方程,假定生长率与生物量的关系为Gompertz分布,方程如下:
$$ d B_t/d t=r B_t \ln \left(K/B_t\right)-C_t $$ (1) 式中Bt表示t时间的生物量,dBt/dt为种群增长率,r为种群内禀生长率,K为种群最大生物量或称环境容纳量,Ct为t时间的捕捞量。
设时间间隔为一年对方程(1)进行积分,得到一个连续的Fox形生物量动态方程:
$$ \begin{aligned} & \ln B_{t+1}=\left(1-\mathrm{e}^{-r}\right) \ln (K)+\mathrm{e}^{-r} \ln \left(B_t\right)-(1- \\ & \left.\mathrm{e}^{-1}\right) r^{-1} F_t \end{aligned} $$ (2) $$ \begin{aligned} &\text { 渔获量: } C_t=\int_t^{t+1} F_t B_t d t=F_t \int_{B_t}^{B_{t+1}} d B_t/[rln (K)\\ &\left.-r \ln \left(B_t\right)-F_t\right] \end{aligned} $$ (3) $$ \begin{aligned} & \quad C_t=F_t B_t\left[\left(1+\mathrm{e}^{-r}/r-r^{-1}\right) \times\left(\ln (K)-\ln \left(B_t\right)\right.\right. \\ & \left.\left.-r^{-1} F_t\right)+1\right] \end{aligned} $$ (4) Ft为t时刻的捕捞死亡系数。Ft=qft,q为可捕系数,ft为t年中的捕捞努力量。
1.2 评估方法
文章所用的评估方法是假设生物量方程(2) 没有误差,误差仅出现在渔获量方程(4)中。因此,此模型估计参数的方法为观测误差估计法,假设渔获量方程存在相乘的误差结构,目标函数表示为:
$$ \min \sum\limits_{t=1}^{\mathrm{T}}\left[\ln \left(C_t\right)-\ln \left(\hat{C}_t\right)\right]^2 $$ 其中t表示年份,Ct表示t年中渔获量的观测值,Ĉt表示t年中渔获量的估计值。应用Excel中的规划求解计算出参数的估计值。直接计算的参数包括B1 (第一年的生物量)、r (种群内禀增长率)、K (环境容纳量)和q (可捕系数)。具体算法如下:
(1) 给出4个参数(B1、r、K、q)的起始估计值。
(2) 利用方程(2)计算出逐年的生物量,再利用方程(4)计算逐年的渔获量估计值。
(3) 最小化目标函数。
(4) 监测目标函数收敛性,如果收敛, 计算结束。否则,调整各个参数起始值后,从步骤2重新做,直至目标函数收敛。
当种群的参数(B1、r、K和q)通过非线性回归方法得出后,相应重要的管理参数可以计算得出fMSY=r/(2q),MSY=rKe-1,其中MSY表示最大可持续产量(maximal stay yield),fMSY表示获得最大可持续产量时的捕捞努力量。
文章通过以下各项指标来比较模型在各个渔业中的评估效果,参数估计值与真值之间的偏差(relative bias of median,RBM%)=|真实值-估计值|×100%/真实值,各个参数的四分位数间距(interquartile range,IQR),相对四分位数间距(relative interquartile range,RIQR%)=四分位数间距×100%/中值。预测误差百分数(percentage prediction error,PPE%)=|CPUE真实值-CPUE估计值|×100%/CPUE真实值[3]。为了计算真实渔业各参数的各项指标,文章采用了非参数化Bootstrap分析法[4],Bootstrap分析过程重复了1 000次,在随机抽取数据过程中,采用的是成对抽取数据。
1.3 模拟渔业
文章采用蒙特卡罗模拟方法产生数据,该方法需要一个操作模型和一个评估模型,采用的操作模型和评估模型都是连续形Fox产量模型。假设不同的渔业史或渔业过程,即给出不同的捕捞努力量变化过程,根据连续形Fox产量模型方程(2)、(4),计算出相应渔业的模拟生物量和渔获量,在此基础上叠加正态分布的随机数[5]作为该渔业的观测值。文章设置了不同的白色噪音水平(变异系数为1%、10%、30%和50%)。种群参数的真值都设为r=0.4,K=1 000,q=0.01,B1的真值随不同的渔业过程而不同。根据相应的计算公式主要的管理参数真值为fMSY=20,MSY=147.15。图 1-a显示的是一种过度捕捞型渔业,以下称为渔业一;该渔业从开始就经历了高的捕捞努力量,并且捕捞努力量逐年增加,生物量逐渐减少。图 1-b显示的是一种恢复型渔业,以下称为渔业二;该渔业由于经历了过度捕捞的缘故,初始阶段生物量较低, 随后采取了保护措施,捕捞努力量一直被限制在较低的水平,生物量随着时间不断增加。图 1-c显示的是一种波动型渔业,也可称为管理良好的渔业,以下称为渔业三;该渔业开始于资源未开发阶段,加入高捕捞努力量生物量下降,然后限制捕捞努力量,生物量逐渐增加,生物量和捕捞努力量随时间上下波动。
1.4 真实渔业
北大西洋剑鱼渔业评估所用数据是ICCAT对该渔业产量和生物量丰度指标——单位捕捞努力渔获量(catch per unit of effort,CPUE)的统计[6],共有1975~1998年24年间的数据,该渔业1988年以前产量逐渐升高,但是CPUE却不断降低,随后产量逐渐降低,CPUE降低趋势趋于平缓(图 2)。
2. 结果
文章对每种渔业在不同白色噪音水平下分别进行了1 000次模拟分析计算,去除部分无效结果(包括参数值< 0、q>=1、B1>K 3种情况)按白色噪音0.01、0.1、0.3、0.5的顺序,各渔业的有效结果次数,渔业一为524、577、639、172;渔业二为496、355、114、22;渔业三为485、445、425、90。取各估计参数的中值为参数的估计值。RBM%、IQR、RIQR%的最终计算结果列于表 1~表 3。
表 1 参数估计值与真实值之间偏差RBM%Table 1. Bias between the estimated and true parameter values白色噪音 white noise 0.01 0.1 0.3 0.5 B1 渔业一 7.1×10-10 2.2×10-4 10.2 21.4 渔业二 1.1×10-6 2.61 0.3 0.3 渔业三 1.9×10-6 0.64 0.9 2.9 r 渔业一 0.001 0.73 2.7 1.8 渔业二 0.03 0.02 1.4 7.8 渔业三 0.01 0.57 0.9 11.5 K 渔业一 1.1×10-8 0.3 2.6×10-4 4.3 渔业二 6.11×0-7 9.3×10-4 0.6 6.0 渔业三 6.3×10-10 0.9 7.5 17.2 q 渔业一 0.2 1.31 0 7.5 渔业二 0.2 0.26 3.4 32.8 渔业三 0.3 2.8 16.7 2.4 最大可持续产量
MSY渔业一 0.01 0.5 3.8 2.0 渔业二 0.02 1.07 0.1 6.9 渔业三 6.5×10-4 0.08 5.8 1.9 最大可持续产量时的捕捞努力量
fMSY渔业一 0.2 1.4 0.4 3.8 渔业二 0.2 0.04 8.6 36.7 渔业三 0.3 3.3 12.0 15.2 表 2 参数估计值的四分位数间距IQRTable 2. Interquartile range of the estimated parameters白色噪音 white noise 0.01 0.1 0.3 0.5 B1 渔业一 2 222 976 1 306 渔业二 3 139 268 325 渔业三 13 136 411 799 r 渔业一 0.001 0.04 0.20 0.26 渔业二 0.002 0.10 0.20 0.20 渔业三 0.002 0.04 0.14 0.20 K 渔业一 0.2 192 878 1 172 渔业二 0.7 565 1 036 828 渔业三 9.0 209 621 1 287 q 渔业一 6.6×10-5 0.002 0.01 0.013 渔业二 9.9×10-5 0.006 0.01 0.012 渔业三 6.4×10-5 0.002 0.006 0.007 最大可持续产量
MSY渔业一 0.5 14 56 83 渔业二 0.6 40 81 66 渔业三 0.6 14 44 81 最大可持续产量时的捕捞努力量
fMSY渔业一 0.1 2.6 11.7 14.5 渔业二 0.3 6.5 12.7 8.6 渔业三 0.09 1.3 3.3 4.8 表 3 参数估计值的相对四分位数间距RIQR%Table 3. Relative IQR(RIQR%)of the estimated parameters白色噪音 white noise 0.01 0.1 0.3 0.5 B1 渔业一 0.3 4.9 110.8 134.5 渔业二 1.4 67.7 133.6 162.2 渔业三 1.6 17.1 51.9 97.1 r 渔业一 0.3 9.8 52.3 65.3 渔业二 0.4 24.2 48.9 44.8 渔业三 0.5 11.2 34.5 51.9 K 渔业一 0.2 19.2 87.8 112.4 渔业二 0.07 56.5 104.2 88.1 渔业三 0.9 20.7 67.1 109.9 q 渔业一 0.7 18.3 101.7 125.3 渔业二 0.9 57.0 137.9 92.6 渔业三 0.6 16.7 53.6 72.1 最大可持续产量
MSY渔业一 0.4 9.6 39.5 55.4 渔业二 0.3 26.8 55.1 48.5 渔业三 0.4 9.6 31.6 54.4 最大可持续产量时的捕捞努力量
fMSY渔业一 0.6 13.2 58.1 75.1 渔业二 1.3 32.5 69.3 68.3 渔业三 0.5 6.9 18.5 28.2 图 3、图 4体现了在白色噪音为10%的情况下3种模拟渔业的观测产量和估计产量的对比关系。
文章还计算了各渔业的预测误差百分数PPE%(图 5)。从总体上看,当白色噪音小于10%的情况下,所有模拟渔业的各个参数估计值接近于真实值。当白色噪音达到30%时,评估结果偏差较大,模型表现不好。从各参数的评估结果来看,渔业起始生物量B1和最大环境容纳量K的评估结果偏差较小,但是RIQR%较大,参数r和q的评估结果比较接近真值。MSY和fMSY的评估结果最为准确,总体RIQR%小于75.1%。从不同渔业来看,用RBM%来衡量,模型在渔业一中的表现为最好,其次为渔业三,各个渔业24个被估参数的偏差平均值,渔业一为2.6%、渔业为二4.6%、渔业三为4.3%;用RIQR%来衡量,模型在渔业三中表现最好,各渔业RIQR%的平均值,渔业一为46.6%、渔业二为55.1%、渔业三为31.6%。
对北大西洋剑鱼渔业的1 000次Bootstrap分析结果列于表 4中。为证明可靠渔业信息对评估结果准确性的影响,文章在白色噪音为0.1的情况下对参数加以约束,假设B1=0.2K,将评估参数减少为3个,对渔业2进行了分析计算,并将计算结果与参数未加约束时作了比较,比较结果见表 5。
表 4 北大西洋剑鱼渔业参数估计值和参数四份位数间距及相对四份位数间距Table 4. Median and IQR, IQR% for the North Atlantic swordfish fishery参数名称
parameter参数中值
median四分位数间距
IQR相对四分位数间距
RIQR%B1 30 773 6 021 19.6 r 0.94 0.05 5.6 K 44 733 2 697 6.0 q 0.013 0.001 9.2 最大可持续产量 MSY 15 678 621 3.9 最大可持续产量时的捕捞努力量 fMSY 37 4.5 12.3 表 5 约束参数与不约束参数评估结果比较Table 5. Comparison of the results with constrained and unconstrained parametersr K q 最大可持续产量
MSY最大可持续产量时的捕捞努力量
fMSY四分位数间距
IQR无约束 0.10 565 0.006 40.0 6.5 约束 0.03 116 0.001 8.9 2.1 相对四分位数间距
RIQR%无约束 24.2 56.5 57.0 26.8 32.5 约束 8.2 11.9 11.2 6.1 10.7 3. 讨论
采用连续Fox模型对渔业初始生物量B1和环境容纳量K的评估结果表明,虽然估计值与真实值之间的偏差较小,但是RIQR%较大,这是因为将中值作为最终参数估计值时个别较大值和较小值对中值影响很小,因此,建议将中值作为最终估计值来平衡个别较大值和较小值对结果的影响。B1和K的RIQR%较大,证明模型对这2个参数的评估结果不够理想,个别渔业甚至出现B1估计值高于环境容纳量K真实值的情况,在最终计算参数时将这样的结果归为无效结果删除,以防止影响结果的准确性,产生这种情况主要是由于白色噪音的影响及该模型对数据较为敏感。但是当B1估计值较大时,K估计值也偏高,B1估计值准确性较差可能主要是由于模型为非线性的,且同时要估计4个参数所导致的,在模拟分析计算时假设B1值与K值呈线性关系;如渔业二中假设B1=0.2K,这样评估参数减少为3个,如表 5所示,添加约束时各参数的IQR和RIQR%仅为未添加约束时的1/3左右,所得结果准确性明显好于同时评估4个参数时的结果。
具体的渔业种群评估中,如果能合理利用渔业信息以确定某个参数,则可以简化计算,并且能提高评估结果的准确性。
模型估计管理参数MSY和fMSY的准确性高于其它参数,这可能由于被估各参数间存在一定的相关性[7],MSY和fMSY的估计值是参数r、K、q以相乘或相除形式结合在一起,这样降低了单个参数估计偏差的影响程度。因此,管理参数MSY和fMSY的估计值更为准确[8]。
当白色噪音大于30%时,参数评估偏差较大,模型表现不好,这可能主要因为较高的白色噪音干扰了种群动态变动的内在规律。例如,噪音较大时曾出现观测产量为负值的不合理情况,使得模型不能捕捉渔业在没有噪音情况下的真实关系。所以,高质量的渔业统计数据对资源评估结果十分重要。
渔业三评估结果好于其它渔业可能因为该渔业经历了过度捕捞和恢复2个渔业阶段,渔业过程较为完整,所含数据信息丰富。而其它2种渔业的信息相对缺乏,导致模型对这2种渔业评估表现较渔业三差。
文章评估北大西洋剑鱼(Xiphias gladius)渔业的MSY为15 678 t,fMSY为37×106钩次·a-1;PRAGER[6]评估该渔业的MSY为14 070和14 510 t;孙利东[8]评估该渔业的MSY为12 493 t,fMSY为53×106钩次·a-1。文章评估的MSY与其它不同模型评估的MSY结果相差不大,证明连续性Fox模型对北大西洋剑鱼渔业的评估结果具有参考价值。该渔业的捕捞努力量从20世纪80年代开始就超过了文章评估的fMSY,这种情况造成了北大西洋剑鱼渔业的衰退,评估结果与图 2反映的情况吻合。从20世纪80年代开始该渔业的CPUE不断降低,为了保护和合理利用该渔业资源,应该将捕捞努力量严格控制到合理水平。
-
图 1 二长棘犁齿鲷线粒体DNA控制区序列结构
阴影表示保守序列及polyT序列,方框表示核心序列和反向互补序列,后同
Figure 1. Structure of mitochondrial control regions in E.cardinalis
The shadowed sequences are conserved region and polyT region, and the boxed ones are core sequence of ETAS and reverse complementary sequences.The same case in the following table.
表 1 数据来源
Table 1 Source of DNA data
科
family属
genera种类
speciesNCBI登录号
NCBI accession No.鲷科Sparidae 棘鲷属(Acanthopagrus) 灰鳍棘鲷1(A.berda 1) AM992212 灰鳍棘鲷2(A.berda 2) AM992246 黄鳍棘鲷(A.latus) EF506764 黑棘鲷(A.schlegelii) AF381106 澳洲棘鲷(A.australis) AF381061 赤鲷属(Pagrus) 赤鲷(P.pagrus) AY439117 真赤鲷(P.major) AP002949 三长棘赤鲷(P.auriga) AB124801 小鲷属(Pagellus) 黑斑小鲷(P.bogaraveo) AB305023 科Serranidae 副鲈属(Paralabrax) 副鲈(P.clathratus) DQ192395 鲽科Pleuronectidae 星鲽属(Verasper) 条斑星鲽(V.moseri) NC-008461 表 2 11种鱼类的ETAS序列
Table 2 ETAS sequence of 11 fishes
种类
speciesETAS序列
ETAS sequence来源
source二长棘犁齿鲷(E.cardinalis) ACAT TA GTA TATCATCATTAAAATATATACACCAA 此文 暗纹东方鲀(T.fasciatus) TACAT T TGTA TATCCCCATTCATATATATTAAACA 文献[8] 康氏似鲹(S.commersonianus) TACAT T TGTA TATCACCATTACCTTATATTAACCA 文献[11] 马舌鲽(R.hippoglossoides) TACAT T TGTA TAACACCATATATTTATAGAACC 文献[12] 鳜(S.chuatsi) TGCAT T TGTA TTACACCATAAAACTATATTAACCA 文献[21] 中华乌塘鳢(B.sinensis) TACAT T TGTA TTACACCATATATTTATGTAAAC 文献[22] 带纹条鳎(P.olivaceus) TACAT T TGTA TTACACCATATATTTTATAGTAAACA 文献[23] 松江鲈鱼(T.fasciatus) TACA GT TGTA TATCACCATAATTTATATTAACCA 文献[24] 鲟(Acipenser sturio) ACAT TAAGACATGC ATG TAATCCCCATTAATTTCTAGCCACCAT 文献[25] 鱼类通式common sequences in fish TACAT T------ATGTATTATCACCAT--ATATTATATTAACCA 文献[26] 鲤科鱼类Cyprinids TACAT T--------ATGTATTATCACCAT-----TATATTAACCA 文献[27] 紫红笛鲷(L.argentimaculatus) ACAT ATG ATTTACACCATACATTTATATCAAACA 文献[28] -
[1] 谢佳燕. 齐口裂腹鱼线粒体DNA控制区结构分析[J]. 动物学杂志, 2011, 46(2): 97-101. http://dwxzz.ioz.ac.cn/dwxb/article/abstract/20110214 XIE Jiayan. Structure analysis of mtDNA control region in Schizothorax prenanti[J]. J Zool, 2011, 46(2): 97-101. (in Chinese) http://dwxzz.ioz.ac.cn/dwxb/article/abstract/20110214
[2] SBIS E, TANZARIELLO F, REYES F, et al. Mammalian mitochondrial D-loop region structural analysis: identification of new conserved sequences and the functional and evolutionary implications[J]. Gene, 1997, 205(1/2): 125-140. doi: 10.1016/S0378-1119(97)00404-6
[3] WALBERG M W, CLAYTON D A. Sequence and properties of the human KB cell and mouse L cell D-loop regions of mitochondrial DNA[J]. Nucleic Acids Res, 1981, 9(20): 5411-5421. doi: 10.1093/nar/9.20.5411
[4] 伍汉霖, 邵广昭, 赖春福. 拉汉世界鱼类名典[M]. 基隆: 台湾水产出版社, 1999: 7-977. WU Hanlin, SHAO Guangzhao, LAI Chunfu. Latin-Chinese dictionary of fishes names[M]. Keelung: The Sueichan Press, 1999: 7-977. (in Chinese)
[5] 成泰庆, 郑葆珊. 中国鱼类系统检索[M]. 北京: 科学出版社, 1987: 334-336. CHENG Taiqing, ZHENG Baoshan. Systematic synopsis of Chinese fishes[M]. Beijing: Science Press, 1987: 334-336. (in Chinese)
[6] 杨慧荣, 江世贵. 用RAPD技术探讨5种鲷科鱼类的亲缘关系[J]. 水产学报, 2006, 30(4): 469-474. doi: 10.3724/SP.J.1231 YANG Huirong, JIANG Shigui. Study on genetic relationships of Sparidae by RAPD[J]. J Fish China, 2006, 30(4): 469-474. (in Chinese) doi: 10.3724/SP.J.1231
[7] 张殿昌, 邵燕卿, 苏天凤, 等. 二长棘犁齿鲷线粒体细胞色素b基因序列和分子系统发育分析[J]. 南方水产, 2007, 3(2): 1-7. https://www.schinafish.cn/article/id/8ae35a81-fc80-4d86-ae94-af2e722dc0ee ZHANG Dianchang, SHAO Yanqin, SU Tianfeng, et al. The sequence analysis of mitochondrial cytochrome b gene and molecular phylogeny of Parargyrops edita[J]. South China Fish Sci, 2007, 3(2): 1-7. (in Chinese) https://www.schinafish.cn/article/id/8ae35a81-fc80-4d86-ae94-af2e722dc0ee
[8] 邵爱华, 朱江, 史全良, 等. 暗纹东方鲀线粒体DNA控制区结构和系统发育分析[J]. 中国水产科学, 2007, 14(3): 352-360. doi: 10.3321/j.issn:1005-8737.2007.03.002 SHAO Aihua, ZHU Jiang, SHI Quanliang, et al. Characterization and phylogenetic analysis of control region of mitochondrial genome from Takifugu fasciatus[J]. J Fish Sci China, 2007, 14(3): 352-360. (in Chinese) doi: 10.3321/j.issn:1005-8737.2007.03.002
[9] 张艳春, 孔晓瑜, 王忠明. 大口鳒线粒体DNA控制区结构和鲽形目鱼类的系统进化初步研究[J]. 热带海洋学报, 2010, 29(6): 71-78. https://lib.cqvip.com/Qikan/Article/Detail?id=37150763&from=Qikan_Article_Detail ZHANG Yanchun, KONG Xiaoyu, WANG Zhongming. Characterization of mitochondrial control region sequence of Psettodes erumei and phylogenetic analysis of Pleuronectiformes[J]. J Trop Oceanogr, 2010, 29 (6): 71-78. (in Chinese) https://lib.cqvip.com/Qikan/Article/Detail?id=37150763&from=Qikan_Article_Detail
[10] 张燕, 张鹗, 何舜平. 中国鲿科鱼类线粒体DNA控制区结构及其系统发育分析[J]. 水生生物学报, 2003, 27(5): 463-467. doi: 10.3321/j.issn:1000-3207.2003.05.004 ZHANG Yan, ZHANG E, HE Shunping. Studies on the structure of the control region of the Bagridae in China and its phylogenetic significance[J]. Acta Hydrobiological Sinica, 2003, 27(5): 463-467. (in Chinese) doi: 10.3321/j.issn:1000-3207.2003.05.004
[11] 朱世华, 郑文娟, 邹记兴, 等. 鲹科鱼类线粒体DNA控制区结构及系统发育关系[J]. 动物学研究, 2007, 28(6): 606-614. doi: 10.3321/j.issn:0254-5853.2007.06.005 ZHU Shihua, ZHENG Wenjuan, ZHOU Jixing, et al. Mitochondrial DNA control region structure and molecular phylogenetic relationship of Carangidae[J]. Zool Res, 2007, 28(6): 606-614. (in Chinese) doi: 10.3321/j.issn:0254-5853.2007.06.005
[12] 赫崇波, 曹杰, 刘卫东, 等. 圆斑星鲽线粒体基因组全序列结构及其进化[J]. 遗传, 2007, 29(7): 829-836. http://www.fishscichina.com/zgsckx/article/abstract/3863?st=article_issue HE Chongbo, CAO Jie, LIU Weidong, et al. Structure analysis of mtDNA control region of spotted halibut (Verasper variegatus) and its related species[J]. Hereditas, 2007, 29(7): 829-836. (in Chinese) http://www.fishscichina.com/zgsckx/article/abstract/3863?st=article_issue
[13] 柳淑芳, 吴仁协, 朱玲, 等. 小黄鱼(Larimichthys polyactis)线粒体基因组结构与特征[J]. 海洋与湖沼, 2010, 41(6): 885-894. https://lib.cqvip.com/Qikan/Article/Detail?id=37132376&from=Qikan_Search_Index LIU Shufang, WU Renxie, ZHU Ling, et al. Complete sequence and gene organization of mitochondrial DNA of the small yellow croaker Larimichthys polyactis[J]. Oceanologica et Limnologia Sinica, 2010, 41(6): 885-894. (in Chinese) https://lib.cqvip.com/Qikan/Article/Detail?id=37132376&from=Qikan_Search_Index
[14] 秦钦, 许志强, 边文冀, 等. 斑点叉尾线粒体DNA控制区结构和群体遗传多样性分析[J]. 淡水渔业, 2011, 41(4): 50-54. QIN Qin, XU Zhiqiang, BIAN Wenji, et al. Structure of mitochondrial DNA control region and population genetic diversity analysis of channel catfish (Ictalurus punctatus)[J]. Freshw Fish, 2011, 41(4): 50-54. (in Chinese)
[15] 荣朝振, 祖国掌, 胡建华, 等. 泥鳅线粒体DNA控制区结构分析及遗传多样性研究[J]. 南方水产科学, 2011, 7(5): 55-62. doi: 10. 3969/j. issn.2095 - 0780. 201 1. 05. 009 RONG Chaozhen, ZU Guozhang, HU Jianhua, et al. Structure of mitochondrial DNA control region and genetic diversity of Misgurnus anguillicaudatus[J]. South China Fish Sci, 2011, 7(5): 55-62. (in Chinese) doi: 10. 3969/j. issn.2095 - 0780. 201 1. 05. 009
[16] 陆星, 梁日深, 陈竹, 等. 斜带髭鲷线粒体DNA控制区序列的结构分析[J]. 水生态学杂志, 2011, 32(6): 78-83. https://sstxzz.ihe.ac.cn/ch/reader/create_pdf.aspx?file_no=201107130151 LU Xing, LIANG Rishen, CHEN Zhu, et al. Structure of mitochondrial control region sequence of Hapalogenys nitens[J]. J Hydroecol, 2011, 32(6): 78-83. (in Chinese) https://sstxzz.ihe.ac.cn/ch/reader/create_pdf.aspx?file_no=201107130151
[17] 苏天凤, 江世贵. 竹筴鱼属鱼类线粒体DNA控制区结构及其系统发育分析[J]. 南方水产科学, 2011, 7(1): 18-25. doi: 10.3969/j.issn.2095-0780.2011.01.003 SU Tianfeng, JIANG Shigui. Structure and phylogenetic analysis of mitochondrial DNA control region of genus Trachurus[J]. South China Fish Sci, 2011, 7(1): 18-25. (in Chinese) doi: 10.3969/j.issn.2095-0780.2011.01.003
[18] THOMPSON J D, GIBSON T J, PLEWNIAK F, et al. The CLUSTAL_X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Res, 1997, 25(24): 4876-4882. doi: 10.1093/nar/25.24.4876
[19] XIA Junhong, XIA Kuaifei, GONG Jinbo, et al. Complete mitochondrial DNA sequence, gene organization and genetic variation of control regions in Parargyrops edita[J]. Fish Sci, 2007, 73(5): 1042-1049. doi: 10.1111/j.1444-2906.2007.01434.x
[20] SOUTHERN S O, SOUTHERN P J, DIZON A E. Molecular characterization of a cloned dolphin mitochondrial genome[J]. J Mol Evol, 1988, 28(1/2): 32-40. doi: 10.1007/BF02143495
[21] ZHAO Jinliang, WANG Weiwei, LI Sifa, et al. Structure of the mitochondrial DNA control region of the Sinipercine fishes and their phylogenetic relationship[J]. Acta Genetica Sinica, 2006, 33(9): 793-799. doi: 10.1016/S0379-4172(06)60112-1
[22] 王登玉. 中华乌塘鳢线粒体DNA控制区结构分析与群体遗传学研究[D]. 厦门: 厦门大学, 2008: 27-43. 10.7666/d.y1444429 WANG Dengyu. Length and sequence variation in mitochondrial DNA eontrol region and genetic structure of Bostrichthy sinesis[D]. Xiamen: Xiamen University, 2008: 27-43. (in Chinese) 10.7666/d.y1444429
[23] 周立石. 鳎亚目鱼类系统学关系及带纹条鳎线粒体DNA控制区结构的初步研究[D]. 青岛: 中国海洋大学, 2005: 50-59. 10.7666/d.y828458 ZHOU Lishi. Pilot study for phylogenetic analysis of Soleoidei fishes and structure of Zebrias zebra mtDNA control region[D]. Qingdao: Ocean University of China, 2005: 50-59. (in Chinese) 10.7666/d.y828458
[24] 刘海林, 章群, 唐优良, 等. 黄渤海松江鲈鱼线粒体控制区结构与序列多态性分析[J]. 海洋通报, 2010, 29(3): 283-289. doi: 10.3969/j.issn.1001-6392.2010.03.008 LIU Hailin, ZHANG Qun, TANG Youliang, et al. Structure and genetic diversity of mtDNA D-Loop sequences among Trachidermus fasciatus stocks in Yellow Sea and Bohai Sea of China[J]. Mar Sci Bull, 2010, 29(3): 283-289. (in Chinese) doi: 10.3969/j.issn.1001-6392.2010.03.008
[25] 张四明, 吴清江, 张亚平. 中华鲟(Acipenser sinensis)及相关种类的mtDNA控制区串联重复序列及其进化意义[J]. 中国生物化学与分子生物学, 2000, 16(4): 458-461. doi: 10.3969/j.issn.1007-7626.2000.04.008 ZHANG Siming, WU Qingjiang, ZHANG Yaping. Tandem repeats of Chinese sturgeon (Acipenser sinensis) and related species and its significance in evolution[J]. Chin J Biochem Mol Biol, 2000, 16(4): 458-461. (in Chinese) doi: 10.3969/j.issn.1007-7626.2000.04.008
[26] 刘焕章. 鱼类线粒体DNA控制区的结构与进化: 以鳑鲏鱼类为例[J]. 自然科学进展, 2002, 12(3): 266-270. doi: 10.3321/j.issn:1002-008X.2002.03.008 LIU Huanzhang. The structureand evolution of mitochondrial DNA control region of fish: a case study to bitterlings[J]. Prog Nat Sci, 2002, 12(3): 266-270. (in Chinese) doi: 10.3321/j.issn:1002-008X.2002.03.008
[27] GUO Xinhong, LIU Shaojun, LIU Yun. Comparative analysis of the mitochondrial DNA control region in cyprinids with different ploidy level[J]. Aquaculture, 2003, 224(1/2/3/4): 25-38. doi: 10.1016/S0044-8486(03)00168-6
[28] 谭围, 郭昱嵩, 王中铎. 笛鲷鱼类的线粒体DNA控制区结构及其系统发育分析[J]. 海洋学报, 2010, 32(1): 139-145. https://xueshu.baidu.com/usercenter/paper/show?paperid=5813ee59fa665947e5122c401ecd6e58&site=xueshu_se&hitarticle=1 TAN Wei, GUO Yusong, WANG Zhongduo. Structure of the mitochondrial DNA control region of snapper species and their phylogenetic relationship[J]. Acta Oceanologica Sinica, 2010, 32(1): 139-145. (in Chinese) https://xueshu.baidu.com/usercenter/paper/show?paperid=5813ee59fa665947e5122c401ecd6e58&site=xueshu_se&hitarticle=1
[29] HE Chongbo, HAN Jiabo, GE Longli, et al. Sequence and organization of the complete mitochondrial genomes of spotted halibut (Verasper variegatus) and barfin flounder (Verasper moseri)[J]. DNA Seq, 2008, 19(3): 246-55. doi: 10.1080/10425170701563303
[30] 江世贵, 刘红艳, 苏天凤, 等. 4种鲷科鱼类的线粒体细胞色素b基因序列及分子系统学分析[J]. 中国水产科学, 2003, 10(3): 184-188. https://www.doc88.com/p-8176141285829.html JIANG Shigui, LIU Hongyan, SU Tianfeng, et al. Molecular phylogeny of mitochondrial cytochrome b gene sequences from four Sparidae fishes[J]. J Fish Sci China, 2003, 10(3): 184-188. (in Chinese) https://www.doc88.com/p-8176141285829.html
[31] XIA Junhong, XIA Kuaifei, JIANG Shigui. Complete mitochondrial DNA sequence of the yellowfin seabream Acanthopagrus latus and a genomic comparison among closely related sparid species[J]. Mitochondrial DNA, 2008, 19(4): 385-393. doi: 10.1080/19401730802350998
-
期刊类型引用(6)
1. 张磊, 王锦旭, 杨贤庆, 魏涯, 杨少玲. 合浦珠母贝糖胺聚糖泡腾片制备工艺优化及其质量分析. 大连海洋大学学报. 2018(05): 644-650 .  百度学术
百度学术
2. 王珍珍, 黄桂菊, 范嗣刚, 刘宝锁, 张博, 苏家齐, 喻达辉. 合浦珠母贝matrilin-1基因的克隆和表达分析. 南方水产科学. 2017(01): 76-84 .  本站查看
本站查看
3. 谭才钢, 刘宝锁, 张东玲, 牛志凯, 张博, 陈明强, 范嗣刚, 姜松, 黄桂菊, 李有宁, 陈素文, 喻达辉. 合浦珠母贝主要形态性状与体质量的灰色关联分析. 南方水产科学. 2015(02): 35-40 .  本站查看
本站查看
4. 邹柯姝, 张殿昌, 郭华阳, 张楠, 江世贵. 4种壳色合浦珠母贝贝壳棱柱层和珍珠质层7种金属元素质量分数的比较分析. 南方水产科学. 2015(03): 74-79 .  本站查看
本站查看
5. 毕晓敏, 黄桂菊, 范嗣刚, 朱文杰, 喻达辉. 合浦珠母贝矿化基因Pearlin重组蛋白的表达条件优化. 南方水产科学. 2015(06): 100-106 .  本站查看
本站查看
6. 肖雅元, 徐姗楠, 刘永, 李纯厚. 0~#柴油水溶液对翡翠贻贝CYP4基因表达的影响研究. 南方水产科学. 2014(02): 61-66 .  本站查看
本站查看
其他类型引用(1)




 下载:
下载:





 粤公网安备 44010502001741号
粤公网安备 44010502001741号
