DGGE fingerprinting of bacterial community in aquaculture water of Crassostrea ariakensis from Guandu district, Zhanjiang city
-
摘要:
2011年3月~12月利用变性梯度凝胶电泳(denatured gradient gel electrophoresis, DGGE)技术对湛江官渡地区近江牡蛎(Crassostrea ariakensis)养殖水细菌群落组成进行了监测研究。UPGAMA聚类分析显示3月~12月的样品细菌主要分属2大类群;多样性指数结果显示3月~7月细菌多样性高于8月~12月;系统发育分析显示牡蛎养殖区水体优势菌群主要由以下种群组成:未可培养的变形菌门(Proteobacteria)(α、γ、δ、ε)、未可培养的拟杆菌门(Bacterioidetes)、未可培养的疣微菌门(Verrucomicrobiae bacterium)、未可培养的放线菌门(Actinobacteria)和未可培养的蓝细菌门(Cyanobacter bacterium)。最优势菌群为α-变形菌纲和拟杆菌门的黄杆菌属(Flevobacterium)。
Abstract:We had monitored and investigated the bacterial community composition in aquaculture water for Crassostrea ariakensis in Guandu of Zhanjiang by denaturing gradient gel electrophoresis (DGGE) from March to December, 2011. UPGMA clustering analysis shows that there are 2 major populations of bacterial species. Diversity index reveals that the diversities in March~July were higher than those in August~December. Phylogenetic analysis indicates that the dominant microbial populations in the water are uncultured Proteobacteria (α, γ, δ and ε), uncultured Bacterioidetes, uncultured Verrucomicrobiae bacterium, uncultured Actinobacteria and uncultured Cyanobacter bacterium. Uncultured α-proteobacterium and Flevobacterium of uncultured Bacteroidetes are the most dominant bacteria.
-
Keywords:
- Crassostrea ariakensis /
- aquaculture water /
- bacterial community /
- DGGE
-
中国近江牡蛎(Crassostrea ariakensis)养殖有300多年的悠久历史,具有广泛的产业基础,养殖规模大、产量高。近江牡蛎在广东省汕头牛田洋、珠海的横琴和南水高栏、阳江程村、湛江官渡等地区已成为其海水贝类养殖的支柱和品牌。随着近江牡蛎养殖历史的延长和集约化生产的发展,逐渐出现养殖规模与环境容量不相适应、养殖密度过大、养殖技术缺乏规范等问题。近几年各地陆续出现近江牡蛎大规模死亡事件,导致严重的经济损失,如广东、广西等地近年牡蛎采苗及养成过程病害发生,尤其由于环境变化、盐度升高及在春夏或秋冬的季节更替时节,在采苗场保苗死亡异常严重,达80%以上,导致这几年本地苗源供应紧张,各核心养殖区大量引进外地苗种,养成过程大规模死亡现象时有发生。因此,研究养殖环境与病害发生的关系,建立牡蛎病害预警预报系统和病害防控技术体系,是满足牡蛎健康养殖及产业可持续发展的迫切需求。目前,PCR-DGGE技术主要应用于土壤微生物多样性[1-2]、肠道菌群[3-4]、海洋环境微生物多样性[5]、养殖水环境中微生物多样性[6-8]等领域,而针对水产养殖动物水环境中微生物的研究大多是进行异养细菌总数的宏观监测,或者通过传统分离培养的方法获得其中的优势细菌。该过程步骤繁琐、工作量大,且生态环境中未可培养的微生物易被遗漏[8]。而变性梯度凝胶电泳(denatured gradient gel electrophoresis, DGGE)技术对微生物多样性的分析不依赖于微生物的培养过程,而是直接提取环境样品中总的DNA,由此包括了样品中可培养微生物和未可培养微生物的总遗传信息,从而真实地反映微生物群落的原始组成[9-10]。此研究采用PCR-DGGE技术分析牡蛎养殖区微生物的数量和种群变化规律,为进一步探讨牡蛎养殖系统内微生物的生态变化与疾病发生的关系提供理论依据。
1. 材料与方法
1.1 水样采集
样品为湛江官渡牡蛎养殖区水体,于2011年3月~12月进行采样,采集固定站位表层水体1 L水样过滤(所用滤膜为Millpore 0.2 μm,GTTP04700),收集菌体后迅速放冰盒内保存,运回实验室于-80 ℃冻存备用。每约30 d采样1次,共采样10次。
1.2 水样细菌总DNA的提取
采用Omega E.Z.N.A.® BWater DNA Kit试剂盒提取,其操作方法参照说明书执行。
1.3 16S rDNA-V3区域的PCR扩增
采用16S rDNA基因V3可变区具有特异性的8引物对341f+GC(5′- CGCCCGCCGCGCGCGGCGGG-CGGGGCGGGGGCACGGGGG G CC TA GGGGAGGCAGCAG-3′)(下划线处为GC夹序列)534r(5′-ATTACCGCGGCTGCTGG-3′)[11],扩增产物长约234 bp(含GC),用于DGGE分析。以不添加模板为阴性对照组,模板体积由ddH2O补充。
16S V3区PCR反应条件为10 μL 5×PCR Buffer,0.25μL DNA STAR Polymerase酶,4 μL dNTP mix,引物各1 μL,模板2 μL,无菌去离子水补足体积至50 μL。引物由上海生工生物工程有限公司合成。
PCR反应程序为模板预变性条件94 ℃ 4 min,反应条件为94 ℃ 45 s,60 ℃45 s,72 ℃ 1 min,26次循环,72 ℃ 10 min,4 ℃保存模板备用。用1.5%的琼脂糖凝胶电泳检测,DNA green染色观察。
1.4 DGGE
采用Bio-Rad公司DGGE分析系统进行试验。V3区PCR产物经浓缩后上样于8%的聚丙烯酰胺凝胶(PAGE)(变性梯度为50%~65%)进行电泳,恒压100 V,10.3 h。溴化乙锭染液中染色60 min后拍照分析。
1.4.1 DGGE条带的切割和DNA回收
将DGGE图谱中清晰的优势条带标记后割胶回收、捣碎后放入EP管中加入30 μL灭菌去离子水,4 ℃过夜保存,取1 μL上清液作为PCR的模板进行16S rDNA V3区扩增,条件同1.3。
1.4.2 连接、转化以及序列测定
经纯化后的PCR产物连接到pMD18-T Simple Vector载体(TaKaRa,大连产),转化Top10细胞,将阳性克隆株交上海英捷公司测序。
1.5 序列分析
将测序所得的20条优势细菌的16S rDNA序列通过NCBI在线投递工具BankIt(http://www.ncbi.nlm.nih.gov/BankIt/)提交至GenBank。利用BLAST功能(http://www.ncbi.nlm.nih.gov/BLAST/)进行DNA序列匹配,建立系统进化树。
1.5.1 聚类分析及多样性指数计算
采用凝胶定量软件Quantity One对DGGE图谱进行UPGAMA聚类分析,同时利用输出的数据计算3月~12月样品的香农威尔多样性指数(Shanno-Wiener index)。
$$ H=-\sum\limits_{i=1}^s P_i \ln P_i $$ 其中s为每个样品的条带数目,Pi=Ni/N,Ni为第i条带的峰面积,N为所有峰的总面积[12]。
1.5.2 细菌系统进化分析
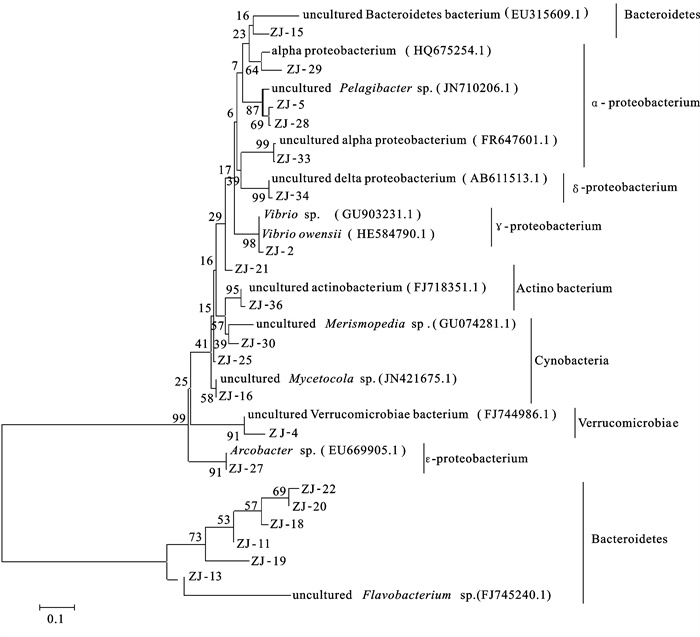
将测序得到的序列通过NCBI数据库的BLASTn(http://blast.Ncbi.nlm.nih.gov/blast.cgi)进行同源性比对搜索,选择最高同源性的序列作为参比序列,并同时记其序列录登陆号。利用MEGA 4.0软件的邻接法(neighbor-joining)构建系统发育树,其中自举值Bootstrap重复1 000次。
2. 结果与分析
2.1 16S rDNA-V3区PCR扩增结果
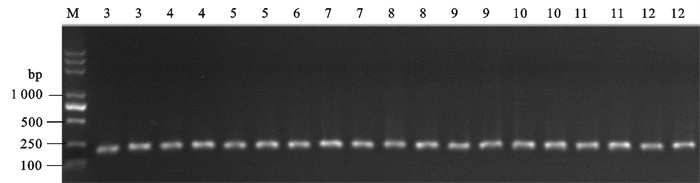
通过采用紫外分光光度计检验水体细菌总DNA提取纯度,光密度OD260/OD280为1.75~1.91,符合下步反应要求。以提取的总DNA为模板进行16S rDNA PCR扩增,扩增产物经凝胶试剂盒切胶回收后以此回收产物为模板进行16S rDNA -V3区PCR片段扩增,PCR产物经2%的琼脂糖凝胶110 V电泳20 min后拍照(图 1),可见PCR扩增后样品的16S rDNA-V3区片段约为234 bp,符合16S rDNA-V3区高变区片段大小,可以进行下步试验。
2.2 DGGE指纹图谱分析结果
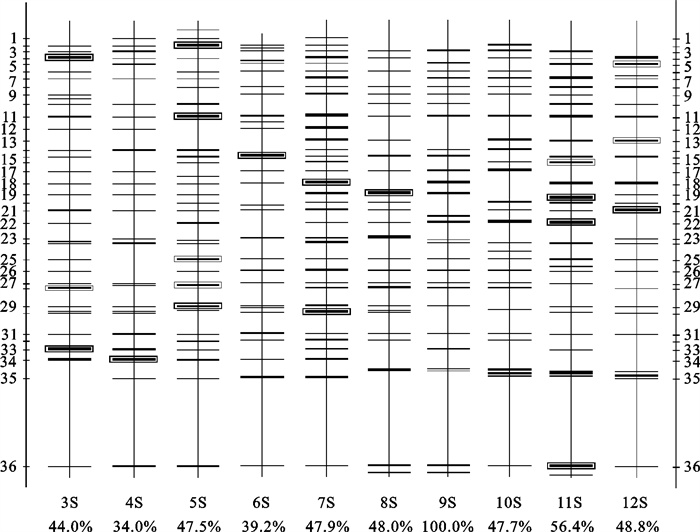
湛江牡蛎养殖区不同月份水体样品的PCR产物经DGGE后,从图 2可以直观地了解各个样品时空分布规律。依据DGGE技术能分离长度相同而碱基组成不同的DNA序列的原理,认为DGGE指纹图谱中位于同一位置的条带看可以作为同一种细菌种群[13],因此,此试验选取20条不同位置上的DGGE特征条带用于后续克隆测序(图 2)。
![]() 图 2 不同月份细菌DGGE分析示意图带边框20处为被切割的条带用于后续测序;切割条带编号依次为ZJ-2、4、5、11、13、15、16、18、19、20、21、22、25、27、28、29、30、33、34、36Fig. 2 Sketch map of DGGE analysis of bacteria in different monthsThe 20 framed cutting bands are for later sequencing; cutting band No. from top to bottom is ZJ-2, 4, 5, 11, 13, 15, 16, 18, 19, 20, 21, 22, 25, 27, 28, 30, 33, 34 and 36.
图 2 不同月份细菌DGGE分析示意图带边框20处为被切割的条带用于后续测序;切割条带编号依次为ZJ-2、4、5、11、13、15、16、18、19、20、21、22、25、27、28、29、30、33、34、36Fig. 2 Sketch map of DGGE analysis of bacteria in different monthsThe 20 framed cutting bands are for later sequencing; cutting band No. from top to bottom is ZJ-2, 4, 5, 11, 13, 15, 16, 18, 19, 20, 21, 22, 25, 27, 28, 30, 33, 34 and 36.3月~12月的海水样品DGGE电泳结果见图 2,可见2011年7月站位采集的样品电泳谱带最为丰富,因此,此研究以2011年7月站位1采集的样品即泳道7为标准,用Quantity One软件对10个月的海水样品的DGGE图谱进行分析(图 3)。处于不同位置的条带数为36条,优势条带ZJ-11和ZJ-36在整个采样时间内均存在。
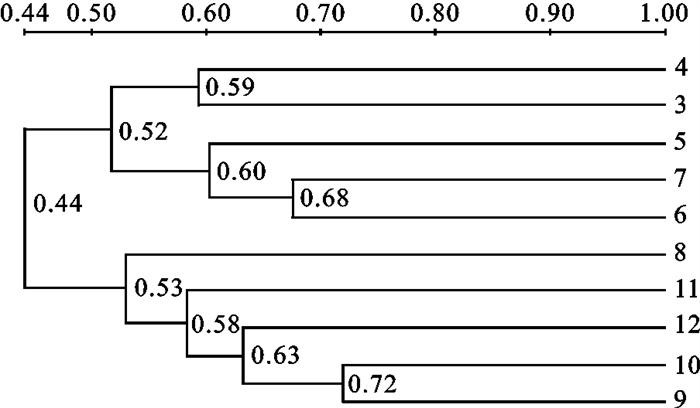
基于各月水体细菌DGGE条带的位置、数量和亮度,采用Quantity One软件对DGGE图谱进行UPGMA聚类分析。各个样品细菌群落按相似程度聚为2个大簇,即2011年3月~7月样品聚为另一大簇,2011年8月~12月样品聚为另一大簇(图 3)。
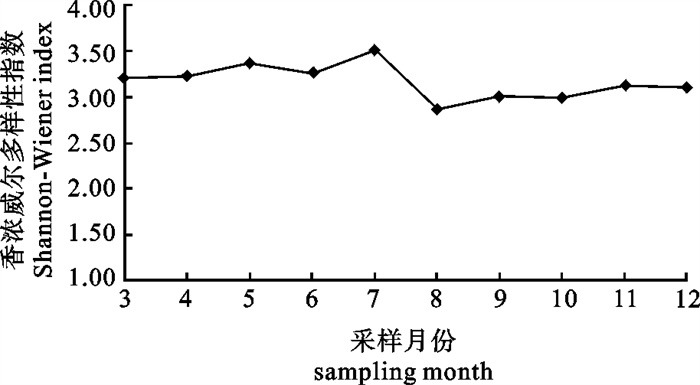
不同的采样月份多样性指数成W状波动变化,3月~7月样品多样性指数为3.21~3.51;8月~12月变化不大,为2.87~3.13,低于3月~7月样品的多样性指数;在监测周期内7月的多样性指数最高(图 4)。
2.3 系统发育分析
选取DGGE示意图中不同位置处22条特征条带切胶回收、测序。将所测条带的序列在GenBank数据库中用Blast进行检索和同源性比对,并下载相近序列。运用ClustalX程序进行排序,采用MEGA 4.0软件进行分析,用邻接法构建系统进化树(图 5)。
湛江牡蛎养殖区共成功克隆20个条带,在已测定出的20种主要类型中未可培养的变形菌门(Proteobacteria)占40%,其中α-变形菌纲占20%,γ-变形菌纲占10%,δ-变形菌纲占5%,ε-变形菌纲占5%;未可培养的拟杆菌门(Bacterioidetes)占35%;未可培养的蓝细菌门(Cyanobacteria)占15%,包括未可培养的片球藻属(Merismopedia sp.)和未可培养的Mycetocda sp.;未可培养的放线菌门(Actinobacteria)占5%;未可培养的疣微菌门(Verrucomicrobiae bacterium)占5%。最优势群落为变形菌门和拟杆菌门,其中未可培养的放线菌门和未可培养的拟杆菌门的黄杆菌属(Flevobacterium)在全年采样样品中均存在。
3. 讨论
3.1 多样性指数变化与环境的关系
多样性指数又称差异指数(discrepancy index),是指应用数理统计方法求得表示生物群落和个体数量的数值,用以评价环境质量。分析结果显示,多样性指数在整个监测周期内呈周期性波动变化,说明了牡蛎养殖水系统内微生物群落结构在时间上的演替变化。此研究发现在疾病的高发季节3月~7月样品的多样性指数均高于8月~12月,这可能与湛江牡蛎养殖区开放式的养殖系统内特殊地理位置、环境等影响相关。杨彩霞等[14]对沙子口细菌群落多样性分析发现,夏季温度较高时由于某些高温性浮游生物迅速增长抑制了其他微生物的生长,夏季细菌群落多样性最低;而冬季温度相对较低,各种浮游生物的增长速率较缓慢,夏季被抑制的细菌种类得到增殖,冬季细菌群落多样性最为丰富。刘勇勤等[15]研究发现冬季沉积雪中微生物数量最少,但多样性最高,而夏季恰恰相反。对于微生物多样性的影响并不是单一因子而是多因子的综合作用,浮游生物的变化对微生物群落结构也存在很大的影响,此结果的出现是生物变化与环境条件的复杂反应[16]。
3.2 细菌群落结构组成及优势菌群变化规律
KELLY和CHISTOSERDOV[17]通过克隆文库的方法对美国大南湾夏季细菌群落的结构变化进行研究,发现大南湾海区细菌群落主要由变形菌门(α、β、γ)、屈挠杆菌(Flexibacter)和蓝细菌组成;SCHAUER等[18]研究表明布拉内斯湾海域细菌群落组成主要为α-变形菌亚门、拟杆菌群(Bacteroides)和蓝细菌;姚延丹等[19]对复合池塘循环水养殖系统微生物群落结构分析发现,池塘优势菌群分属放线菌门、变形菌门、蓝细菌门和拟杆菌门4个门。邓德波等[20]利用16S rRNA克隆文库的方法对养殖水系统研究,指出牡蛎壳上的附着细菌主要分属于变形菌门(包括α-、β-和γ-变形菌纲)、拟杆菌门等。章洁香[20]研究湛江湾微生物群落结构在营养盐中的变化发现,湛江湾水体第一优势菌群为变形菌门,其次为拟杆菌门,最后是放线菌门。杨彩霞等[21]对清河湾扇贝养殖海区细菌群落结构的季节变化研究发现,20个序列代表的细菌分属于α-变形菌纲、β-变形菌纲、γ-变形菌纲、拟杆菌门、放线菌门和疣微菌门。此研究获得的细菌类群为变形菌门(α、γ、δ和ε)、拟杆菌门、疣微菌门、放线菌门和蓝细菌门,与以上国内外对不同海域或养殖系统的研究结果大体一致,但不同生境中细菌群落优势类群也存在差异[22-24]。
变形细菌(又称紫色细菌)是所有细菌中最大和最具多样性的一个族群,由5个亚门(α、β、γ、δ和ε)组成,其中α-和γ-变形菌纲在海水浮游细菌中尤为重要。一般而言,海洋浮游真细菌中能利用光能生长的细菌以α-变形菌纲为主[25-27],而与人类生活密切相关的假单胞菌(Pseudomonas)、弧菌(Vibrio)及广泛分布于海洋厌氧环境中的硫酸盐还原细菌、海洋动物体内的氧化硫内共生菌等,绝大部分属于γ-变形菌纲和δ-变形菌纲[28],并且多数易于培养的细菌都在γ-变形菌纲类群中,被认为是最大优势的海洋细菌。ε-变形菌纲的细菌常存在于海洋的厌氧和热液环境中,部分与该环境中的动物形成共生关系,对碳(C)、硫(S)循环起重要作用[29]。此研究发现变形菌纲中的α-变形菌纲在克隆测序序列中占20%,为变形菌门中的优势类亚群,而γ-变形菌纲占10%,为次优势亚群。据报道,α-变形菌纲的代表种类是海水中最大优势的细菌,而γ-变形菌纲是培养方法中最常见的种类,它在分子生物学方法构建的数据库中却最不常见[30],这与此研究结果相一致。此研究中序列ZJ-5和ZJ-28与α-变形菌纲的SAR11簇的一个成员Pelagibacter sp.亲缘关系极近。据报道16S rDNA序列的SAR11几乎分布于所有的浮游环境中,可能是海洋中最丰富的微生物,从近岸浅海到深海中均有发现,几乎占表层水微生物数量的1/3和中层区域微生物数量的1/5[31]。此研究中克隆条带ZJ-2与弧菌属的V.owensii AP55处于同一进化支上,该弧菌分离自澳大利亚发病极严重的人工养殖甲壳动物体内[32]。已有研究表明,珊瑚、鱼、虾、软体动物、海绵和浮游动物等体表和体内常生存着高密度的弧菌,弧菌具有丰富多样的生理生化功能,对生物和生态系统可能会产生重要的影响,如有些弧菌具有致病性、发光现象、固氮作用及降解几丁质和琼脂等复杂多糖类的能力[33]。
黄杆菌属(Flavobacterium)是好氧或兼性厌氧的化能异养菌,归属于细菌域中的一个主要分支拟杆菌门,其最显著的特征是能够滑动并产生可降解多聚物如琼脂、纤维素和几丁质等各种胞外酶,这些酶的产生对降解复杂的有机物质(如甲壳动物的外壳)具有非常重要的生态学意义,有些种类的胞外酶对鱼类和无脊椎动物有致病性[31]。黄杆菌属是某些海洋生物环境中的优势类群,占细菌总量的15%~25%[34]。此研究中黄杆菌属所占比例为30%,比前人的研究结果略高,这可能是因为不同生境中细菌群落类群比例存在一定的差距,另一个原因可能是16S rDNA的分类方法尚具有一定的缺陷所致,具体原因还待进一步研究。
近江牡蛎的养殖环境是开放式生态养殖系统,养殖微生态系统的可持续稳定平衡与其生长及病害发生息息相关,且养殖系统内细菌群落组成中肠道菌群绝大多数可以从水环境中找到来源[35]。因此,整个环境微生态系统的平衡直接影响牡蛎体内细菌生态系统,研究养殖海区微生态系统的平衡是探讨牡蛎养殖系统内微生物的生态变化与预防疾病发生的基础。
-
图 2 不同月份细菌DGGE分析示意图
带边框20处为被切割的条带用于后续测序;切割条带编号依次为ZJ-2、4、5、11、13、15、16、18、19、20、21、22、25、27、28、29、30、33、34、36
Figure 2. Sketch map of DGGE analysis of bacteria in different months
The 20 framed cutting bands are for later sequencing; cutting band No. from top to bottom is ZJ-2, 4, 5, 11, 13, 15, 16, 18, 19, 20, 21, 22, 25, 27, 28, 30, 33, 34 and 36.
-
[1] WATANABE T, ASAKAWA S, NAKAMURA A, et al. DGGE method for analyzing 16S rDNA of methanogenic archaeal community in paddy field soil[J]. FEMS Microbiol Lett, 2004, 232(2): 153-163. doi: 10.1016/S0378-1097(04)00045-X
[2] de JOURNETT T D, ARNOLD W A, la PARA T M. The characterization and quantification of methanotrophic bacterial populations in constructed wetland sediments using PCR targeting 16S rRNA gene fragments[J]. Appl Soil Ecol, 2007, 35(3): 648-659. doi: 10.1016/j.apsoil.2006.09.006
[3] PLUSKE J R, DURMIC Z, PAYNE H G, et al. Microbial diversity in the large intestine of pigs born and reared in different environments[J]. Livestock Sci, 2007, 108(1/2/3): 113-116. doi: 10.1016/J.LIVSCI.2007.01.010
[4] ZOETENDAL E G, AKKERMANS A D, de VOS W M. Temperature gradient gel electrophoresis analysis of 16S rRNA from human fecal samples reveals stable and host-specific communities of active bacteria[J]. Appl Environ Microbiol, 1998, 64(10): 3854-3859. doi: 10.1128/AEM.64.10.3854-3859.1998
[5] DÍEZ B, PEDRÓS-ALIÓ C, MARSH T L, et al. Application of denaturing gradient gel electrophoresis (DGGE) to study the diversity of marine picoeukaryotic assemblages and comparison of dgge with other molecular techniques[J]. Appl Environ Microbiol, 2001, 67 (7): 2942-2951. doi: 10.1128/AEM.67.7.2942-2951.2001
[6] 罗鹏, 胡超群, 张吕平, 等. 凡纳滨对虾海水养殖系统内细菌群落的PCR-DGGE分析[J]. 中国水产科学, 2009, 16(1): 31-38. doi: 10.3321/j.issn:1005-8737.2009.01.005 LUO Peng, HU Chaoqun, ZHANG Lüping, et al. PCR-DGGE analysis of bacterial communities in marine Litopenaeus vannamei culture system[J]. Fish Sci China, 2009, 16(1): 31-38. (in Chinese) doi: 10.3321/j.issn:1005-8737.2009.01.005
[7] 关晓燕, 周遵春, 姜冰, 等. DGGE分析不同盐度仿刺参养殖环境中菌群多样性[J]. 水产科学, 2011, 30(5): 276-280. doi: 10.3969/j.issn.1003-1111.2011.05.006 GUAN Xiaoyan, ZHOU Zunchun, JIANG Bing, et al. Environmental bacterial diversity in sea cucumber (Apostichopus japonicus) culture under different salinities based on DGGE fingerprint[J]. Fish Sci, 2011, 30(5): 276-280. (in Chinese) doi: 10.3969/j.issn.1003-1111.2011.05.006
[8] 关晓燕, 周遵春, 陈仲, 等. 应用PCR-DGGE指纹技术分析高温季节仿刺参养殖水环境中菌群多样性[J]. 海洋湖昭通报, 2010(1): 82-88. https://qikan.cqvip.com/Qikan/Article/Detail?id=33484928 GUAN Xiaoyan, ZHOU Zunchun, CHEN Zhong, et al. Bacteria diversity analysis on the water of sea cucumber (Apostichopus japonicus) culture pond under high-temperature season based on PCR-DGGE fingerprint technology[J]. Trans Oceanol Limnol, 2010(1): 82-88. (in Chinese) https://qikan.cqvip.com/Qikan/Article/Detail?id=33484928
[9] MUYZER G, SMALLA K. Application of denaturing gradient gele lectrophoresis (DGGE) and temperature gradient gel electrophoresis (TGGE) in microbial ecology[J]. Antome Van Leeuwenhoek, 1998, 73(1): 127-141. doi: 10.1023/A:1000669317571
[10] MUYZER G. DGGE/TGGE a method for identifying genes from natural ecosystems[J]. Curr Opin Microbiol, 1999, 2(3): 317-322. doi: 10.1016/S1369-5274(99)80055-1
[11] 刘淮德, 王雷, 王宝杰, 等. 应用PCR-DGGE分析南美白对虾肠道微生物多样性[J]. 饲料工业, 2003(20): 55-58. doi: 10.3969/j.issn.1001-991X.2008.20.018 LIU Huaide, WANG lei, WANG Baojie, et al. Microbial diversity analysis on Panaeus vannamei intestinal under high-temperature season based on PCR-DGGE fingerprint technology[J]. Feed Ind, 2003(20): 55-58. (in Chinese) doi: 10.3969/j.issn.1001-991X.2008.20.018
[12] EIEHNER C A, ERB R W, TIMMIS K N, et al. Thermal gradient gel electrophoresis analysis of bioprotection from pollutant shocks in the activated sludge microbial community[J]. Appl Environ Microbiol, 1999, 65(1): 102-109. doi: 10.1128/AEM.65.1.102-109.1999
[13] SIGLER W V, MINIACI I C, ZEYER J. Electrophoresis time impacts the denaturing gradient gel electrophoresis-based assessment of bacterial community structure[J]. J Microbiol Meth, 2004, 57(1): 17-22. doi: 10.1016/j.mimet.2003.11.011
[14] 杨彩霞. 两个养殖海区及栉孔扇贝消化盲囊细菌群落多样性分析[D]. 青岛: 中国海洋大学, 2011. 10.7666/d.y1927694 YANG Caixia. Seasonal changes of bacterial community composition in two culture area and digestive diverticula of scallops, Chlamys farreri[D]. Qingdao: Ocean University of China, 2011. (in Chinese) 10.7666/d.y1927694
[15] 刘勇勤, 姚檀栋, 康世昌, 等. 珠穆朗玛峰地区东绒布冰川冰雪微生物群落及其季节变化[J]. 科学通报, 2006, 51(11): 1287-1296. doi: 10.3321/j.issn:0023-074X.2006.11.009 LIU Yongqin, YAO Tandong, KANG Shichang, et al. Microbial community and seasonal variation of east Flannelette glacier ice and snow in Mount Everest region[J]. Sci Bull, 2006, 51(11) : 1287-1296. (in Chinese) doi: 10.3321/j.issn:0023-074X.2006.11.009
[16] 张俊逸. 珠江广州城市河段水质和微生物多样性特征[D]. 广州: 暨南大学, 2011. https://xueshu.baidu.com/usercenter/paper/show?paperid=a182b7478cfe8f1c2b02e11e1f6be713&site=xueshu_se&hitarticle=1 ZHANG Junyi. Characteristics of water quality and microbial diversity in Guangzhou city segment of Pearl River[D]. Guangzhou: Jinan University, 2011. (in Chinese) https://xueshu.baidu.com/usercenter/paper/show?paperid=a182b7478cfe8f1c2b02e11e1f6be713&site=xueshu_se&hitarticle=1
[17] KELLY K M, CHISTOSERDOV A Y. Phylogenetic analysis of the succession of bacterial communities in the Great South Bay (Long Island)[J]. FEMS Microbiol Ecol, 2001, 35(1): 85-95. doi: 10.1111/j.1574-6941.2001.tb00791.x
[18] SCHAUER M, BALAGUÉ V, PEDRÓS-ALIÓ C, et al. Seasonal changes in the taxonomic composition of bacterioplankton in a coastal oligotrophic system[J]. Aquat Microb Ecol, 2003, 31(2): 163-174. https://emm.icm.csic.es/publication/schauer-seasonal-2003/
[19] 姚延丹, 李谷, 陶玲, 等. 复合池塘循环水养殖系统微生物群落结构分析[J]. 中国水产科学, 2011, 18(2): 407-415. doi: 10.3724/SP.J.1118.2011.00407 YAO Yandan, LI Gu, TAO Ling, et al. Analysis of microbial community structure in an integrated pond recirculating aquaculture system[J]. Fish Sci China, 2011, 18(2): 407-415. (in Chinese) doi: 10.3724/SP.J.1118.2011.00407
[20] 章洁香. 湛江湾微生物群落在不同营养梯度水体中的变化[D]. 湛江: 广东海洋大学, 2011. https://xueshu.baidu.com/usercenter/paper/show?paperid=836e47b37ceedd088bb1557bb215008f&site=xueshu_se&hitarticle=1 ZHANG Jiexiang. The changes of microbial community along the different trophic gradient of seawater in Zhanjiang Bay[D]. Zhanjiang: Guangdong Ocean University, 2011. (in Chinese) https://xueshu.baidu.com/usercenter/paper/show?paperid=836e47b37ceedd088bb1557bb215008f&site=xueshu_se&hitarticle=1
[21] 杨彩霞, 王崇明, 李赟, 等. 应用DGGE技术分析流清河湾扇贝养殖海区细菌群落结构的季节变[J]. 水产学报, 2012, 36(3): 407-414. doi: 10.3724/SP.J.1231.2012.27767 YANG Caixia, WANG Chongming, LI Yun, et al. Seasonal changes of bacterial community composition in the scallop culture area of Liuqinghe Bay studied by PCR-DGGE[J]. J Fish China, 2012, 36(3): 407-414. (in Chinese) doi: 10.3724/SP.J.1231.2012.27767
[22] GRAY J P, HERWIG R P. Phylogenetic analysis of the bacteria communities in marine sediments[J]. Appl Environ Microbiol, 1996, 62(11): 4049-4059. doi: 10.3724/SP.J.1231.2012.27767
[23] LI L, KATO C, HORIKOSHI K. Microbial diversity in sediments collected from the deepest cold-seep area, the Japan Trench[J]. Mar Biotechnol, 1999, 1(4) : 391-400. doi: 10.1007/PL00011793
[24] RAVENSCHLAG K, SAHM K, PERNTHALER J, et al. High bacterial diversity in permanently cold marine sediments[J]. Appl Environ Microbiol, 1999, 65(9): 3982-3989. doi: 10.1128/AEM.65.9.3982-3989.1999
[25] COTTRELL M T, KIRCHMAN D L. Community composition of marine bacterioplankton determined by 16S rRNA gene clone libraries and fluorescence in situ gybridization[J]. Appl Environ Microbiol, 2000, 66(12): 5116-5122. doi: 10.1128/AEM.66.12.5116-5122.2000
[26] SUZUKI T, MUROGA Y, TAKAHAMA M, et al. Roseigium denhamense gen. nov, sp. nov. and Roseibium hemelinense sp. nov., aerobic bacteriochlorophyll-containing bacteria isolated from the east and west coasts of Australia[J]. Int J Syst Evol Microbiol, 2000, 50(6): 2151-2156. doi: 10.1099/00207713-50-6-2151
[27] GONZÁLEZ J M, KIENE R P, MORAN M A. Transformation of sulfur compounds by an abundant lineage of marine bacteria in the α-subclass of the class Proteobacteria[J]. Appl Environ Microbiol, 1999, 65(9): 3810-3819. doi: 10.1128/AEM.65.9.3810-3819.1999
[28] DWI S, HIRATA K, ASADA Y, et al. Utilization of cyanobacterial biomass from water bloom for bioproduction of lactic acid[J]. World J Microbiol Biotechnol, 2001, 17(3), 259-264. doi: 10.1023/A:1016638619700
[29] CAMPBELL B J, CARY S C. Characterization of a novel spirochete associated with the hydrothermal vent polychaete annelid, Alvinella pompejana[J]. Appl Environ Microbiol, 2001, 67(1): 110-117. doi: 10.1128/AEM.67.1.110-117.2001
[30] AUSTIN B. Marine microbiology[M]. Australia: Cambridge University Press, 1988: 29-32.
[31] 张晓华, 李筠, 纪伟尚, 等. 海洋微生物学[M]. 青岛: 中国海洋大学出版社, 2007: 68-73. https://xueshu.baidu.com/usercenter/paper/show?paperid=d089bfab4c9b9b2c1f26831cb45e9587&site=xueshu_se&hitarticle=1 ZHANG Xiaohua, LI Jun, JI Weishang, et al. Marine microbiology[M]. Qingdao: China Ocean University Press, 2007: 68-73. (in Chinese) https://xueshu.baidu.com/usercenter/paper/show?paperid=d089bfab4c9b9b2c1f26831cb45e9587&site=xueshu_se&hitarticle=1
[32] CANO-GÓMEZ A, GOULDEN E F, OWENS L, et al. Vibrio owensii sp. nov., isolated from cultured crustaceans in Australia[J]. FEMS Microbiol Lett, 2010, 302(2): 175-181. doi: 10.1111/j.1574-6968.2009.01850.x
[33] 王淑贤. 海洋弧菌中五类溶血素基因分布及其生物学活性研究[D]. 青岛: 中国海洋大学, 2007. 10.7666/d.y1112038 WANG Shuxian. Distribution of five kinds of haemolysin genes in Vibrios and its correlation with haemolytic and phospholipase activities[D]. Qingdao: Ocean University of China, 2007. (in Chinese) 10.7666/d.y1112038
[34] EILERS H, PERNTHALER J, GLÖCKNER F O, et al. Culturability and in situ abundance of pelagic bacteria from the North Sea[J]. Appl Environ Microbiol, 2000, 66(7): 3044-3051. doi: 10.1128/AEM.66.7.3044-3051.2000
[35] 王轶南, 朱世伟, 常亚青. 刺参肠道及养殖池塘茵群组成的PCR-DGGE指纹图谱分析[J]. 渔业科学进展, 2010, 31(3): 119-122. doi: 10.3969/j.issn.1000-7075.2010.03.017 WANG Yinan, ZHU Shiwei, CHANG Yaqing, et al. PCR-DGGE analysis of bacterial community composition in the intestine and aquaculture pond of Aostichopus japonicus[J]. Prog Fish Sci, 2010, 31(3): 119-122. (in Chinese) doi: 10.3969/j.issn.1000-7075.2010.03.017
-
期刊类型引用(5)
1. 赵旺,黄星美,邓正华,温为庚,陈明强,王雨,沈铭辉,夏光远,张钰伟,于刚. 夏季美济礁潟湖养殖区海水细菌多样性及其与环境因子的相关性. 中国水产科学. 2022(05): 720-731 .  百度学术
百度学术
2. 吴燕燕,钱茜茜,李来好,杨贤庆,马海霞. 鱼类腌制品加工过程微生物群落多样性研究进展. 生物技术通报. 2015(07): 40-44 .  百度学术
百度学术
3. 王瑞旋,林韵锶,郭志勋,吴开畅,王江勇. 海南陵水企鹅珍珠贝肠道及其养殖水体中异养细菌耐药性研究. 热带海洋学报. 2013(06): 96-100 .  百度学术
百度学术
4. 程翔,杨景云,马淑霞,王春敏. 变性梯度凝胶电泳技术在微生态学研究中应用的进展. 中国微生态学杂志. 2013(04): 475-477+481 .  百度学术
百度学术
5. 赵旺,姜敬哲,王江勇,陈韬,刘广锋,王瑞旋,杨蕊. 杂色鲍幼体附着基藻际细菌群落的PCR-DGGE分析. 中国水产科学. 2013(06): 1225-1233 .  百度学术
百度学术
其他类型引用(5)



 下载:
下载:
 粤公网安备 44010502001741号
粤公网安备 44010502001741号
