Sequence analysis of mitochondrial 16S rRNA of three dolphin species, Tursiops truncatus, Sousa chinensis and Steno bredanensis
-
摘要:
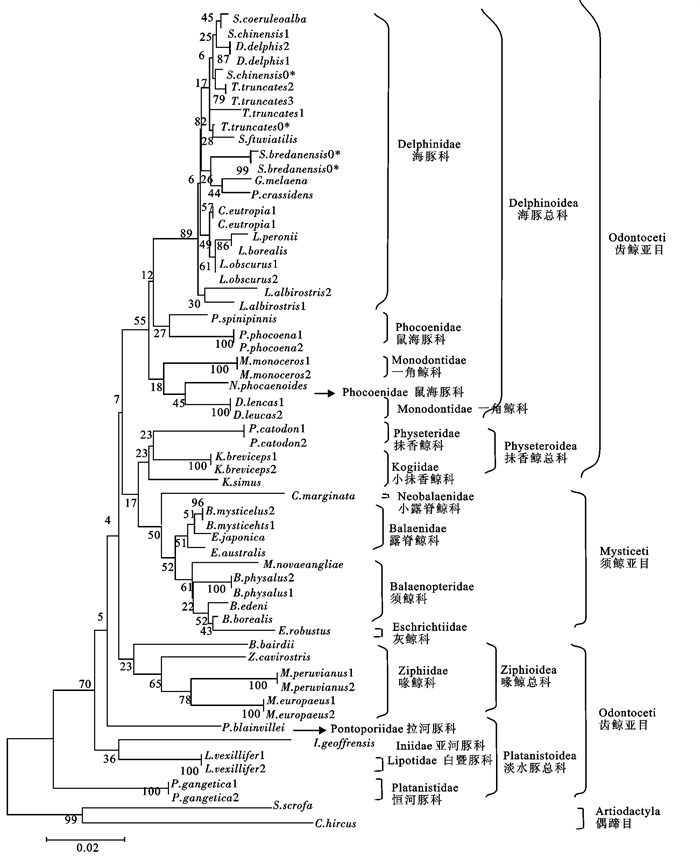
采用聚合酶链式反应(PCR)技术扩增瓶鼻海豚、中华白海豚和糙齿海豚的线粒体DNA 16S rRNA基因,获得了大约600 bp的片段。扩增产物直接测序,去除引物序列后分别获得515、519和529 bp的核苷酸片段。碱基组成平均为T 25.8%,C 20.7%,A 32.1%,G 21.4%,GC含量为42.1%。与35种鲸豚类的55条同源序列比对,去除部分端部序列后得到497个比对位点,包括14个插入/缺失位点和127个变异位点(104个简约信息位点,23个单突变子)。序列比较表明,我国水域的瓶鼻海豚和中华白海豚与国外种存在一定的差异。NJ聚类分析结果与目前的主流观点基本相同,即须鲸亚目形成单系群而齿鲸亚目则为多系群,后者包括海豚总科、抹香鲸总科、喙鲸总科和淡水豚总科。其中抹香鲸总科与须鲸亚目聚合在一起,然后与海豚总科聚合再与喙鲸总科相聚。淡水豚总科为一并系群并处于整个鲸豚类的基部,其中恒河豚科是最早分化的一支。但在海豚总科中,鼠海豚科的江豚则与一角鲸科的白鲸聚合一起,与形态分类不同。
Abstract:Mitochondrial 16S rRNA gene fragments were amplified from dolphins Tursiops truncatus, Sousa chinensis and Steno bredanensis using PCR technique. The PCR products were about 600 bp and were directly sequenced subsequently. Finally, 515, 519 and 529 bp unambiguous nucleotide sequences were obtained respectively after excluding the primer sequences. The average base compositions were T 25.8%, C 20.7%, A 32.1% and G 21.4% with GC content being 42.1%. Alignment with 55 homologous sequences from 35 cetacean species in GenBank revealed 14 insertion/deletions and 127 variable sites (104 parsimony-informative sites and 23 singletons) among 497 truncated alignment sites. Sequence comparison suggested that Chinese T.truncatus and S.chinensis are different from the corresponding species abroad. NJ cluster analysis indicated that subclass Mysticeti is a monophyly whereas subclass Odontoceti is a polyphyly, including Delphinoidea, Physeteroidea (sperm whales), Ziphioidea and Platanistoidea. The sperm whales in Odontoceti was closely related with baleen whales in Mysticeti, and then clustered with Delphinoidea and then with Ziphioidea. The Platanistoidea was paraphyletic and located at the base of the tree, among which Platanistidae constitutes the base clade. Neophocaena phocaenoides(Phocoenidae) clustered with Delphinapterus leucas(Monodontidae) in Delphinoidea, inconsistent with morphological classification.
-
Keywords:
- dolphins /
- 16S rRNA gene /
- sequence analysis /
- molecular phylogenetics
-
随着大黄鱼(Pseudosciaena crocea Richardson)网箱养殖的蓬勃发展,各种病虫害的发生也越来越频繁。近年来,浙江省沿海各大黄鱼养殖场相继发生了一种溃疡病,该病发病率高、流行范围广,危害非常严重。为了搞清浙江沿海养殖大黄鱼溃疡病的病原,找到有效的防治方法,本文以典型症状的病鱼作为材料,通过菌株的分离鉴定、人工感染试验、电镜观察等手段,对大黄鱼溃疡病的致病菌进行了研究,并分析了致病菌的胞外产物,进行了药物抑菌试验,以期为大黄鱼病害的防治提供科学依据。
1. 材料与方法
1.1 材料
病鱼取自浙江宁波象山和温州洞头养殖海区,健康鱼取自浙江宁波奉化养殖海区。
1.2 病原菌分离鉴定
取症状典型的濒死病鱼,无菌条件下从病鱼肝、肾、脾等组织取样,划线接种于普通营养琼脂培养基、IP培养基[1]和TCBS培养基上,27℃恒温培养24 h后,挑取形态特征一致的优势菌落进行克隆培养,转接斜面保存备用。
按照常见细菌系统鉴定手册[2]、伯杰细菌鉴定手册(第9版)[3]的方法进行细菌学鉴定。
1.3 人工感染试验
用60 cm×50 cm×40 cm水体的玻璃缸,每缸放养体长15~20 cm的健康大黄鱼10尾,连续充气,暂养1 d后进行感染试验。试验菌株培养18~24 h,菌悬液浓度按McF浊度管结合活菌计数方法确定。
1.3.1 注射感染试验
将培养好的菌株以无菌生理盐水制成浓度为7.51×107 CFU · mL-1的菌悬液,以肌肉注射(0.3 mL · 尾-1)的方法进行人工感染试验。对照组注射等量无菌生理盐水。
1.3.2 创伤感染试验
以无菌针头对试验鱼体进行穿刺、刮鳞创伤,然后置于试验菌浓度为7.51×104 CFU · mL-1的海水中,对照组不加菌液。
1.3.3 浸泡感染试验
以体表无损伤的健康大黄鱼进行,置于试验菌浓度为7.51×104 CFU ·mL-1的海水中,对照组不加菌液。
1.4 病原菌胞外产物分析
挑取单菌落接种于IP斜面活化24 h,用磷酸盐缓冲液(PBS)pH 7.0洗下,取菌悬液涂于同样的平板培养基上,25℃培养24 h,每皿中加入PBS 5 mL将菌洗入无菌三角瓶中,磁力搅拌器搅拌30 min,4℃,12 000 rpm离心20 min除去菌体,上清液经孔径0.22 μm的纤维素膜过滤,滤液置4℃冰箱保存备用。胞外产物活性分析采用杯碟法进行[4]。
1.5 药敏试验
在IP固体培养基上以纸片法测定病原菌对不同药物的敏感性,经28℃恒温培养24 h,观察抑菌圈的有无及大小。药敏纸片(直径6 mm)购自上海疾病控制中心。
1.6 中草药抑菌作用
15种中草药均为市售,按照文献[5]制成中药原汁(W/V)。将培养24 h的病原菌以无菌生理盐水制成菌悬液(约107 CFU · mL-1),取0.1 mL菌液于无菌平皿中,将IP培养基灭菌冷却后加入不同体积药液倒入含菌平皿制成平板,25℃恒温培养24 h,观察计数细菌生长情况,能够完全抑制细菌生长的最大稀释度即为该药的最小抑菌浓度(MIC)。继续培养48 h,以无菌生长的最低浓度为最小杀菌浓度(MBC)。
2. 结果与分析
2.1 病鱼临诊症状
患病大黄鱼前期症状不明显。中后期主要症状为:鱼体表脱鳞、溃疡,吻部充血,有的鱼吻部断裂,烂鳍、烂尾;解剖后可见肝肿大,有块状斑,略带土黄色;肾肿;肠空,无食物,有少量液体,肠壁无充血。
在典型的发病网箱内,从出现少量病鱼到大部分发病死亡历时约1周,发病死亡率一般为20%~60%。流行时间以夏季高温期为主,7~8月份为高峰期。发病范围大,感染率高,鱼种和成鱼均能感染发病。在同一养殖海区当年鱼种比2龄以上成鱼更易发病,死亡率明显偏高。
2.2 病原菌的致病性
从患病大黄鱼体内分离到1株优势菌824-1进行人工感染试验,结果见表 1。菌株824-1对健康鱼的肌肉注射感染和创伤浸泡感染的死亡率均为100%,但单纯浸泡感染不能使健康鱼发病。健康鱼经人工感染后出现的症状与自然发病鱼症状基本一致。从发病鱼的血液、肾等处可分离到与试验菌株形态特征、理化性状完全一致的菌株,表明分离菌株是大黄鱼的致病菌。
表 1 人工感染试验结果Table 1. Results of artificial infection test感染方式
method of infection菌液浓度/CFU·mL-1
bacterial concentration注射量/mL
injection volume试验数/尾
total number of
fish challenged累计死亡数/尾
number of mortality死亡率/%
mortality rate肌肉注射
intramuscular injection7.51×107 0.3 10 10 100 对照(生理盐水) 0.3 10 0 0 创伤浸泡
injured immersion7.51×104 - 10 10 100 对照(海水) - 10 0 0 单纯浸泡
immersion7.51×104 - 10 0 0 对照(海水) - 10 0 0 2.3 病原菌的鉴定
菌株824-1的基本特征为革兰氏阴性短杆菌,菌体直或稍弯曲,两端钝圆,单个。极生单鞭毛,具运动性,无荚膜,无芽孢。在IP培养基上27℃培养24 h菌体大小为(0.6~0.9)μm×(1.2~1.5)μm,菌落直径为2 mm左右。圆形白色透明隆起,周缘光滑,无色素。TCBS培养基上呈黄色圆形菌落,表面光滑。根据菌株824-1的生理生化反应(表 2),该菌株应归属于弧菌属中的溶藻弧菌(Vibrio alginolyticus)[2-3]。
表 2 分离菌株的生理生化特性Table 2. Physiological and biochemical characteristics of the isolated bacterium鉴定项目item 824-1 鉴定项目item 824-1 鉴定项目item 824-1 鉴定项目item 824-1 4℃生长
4℃ growth- 甲基红
MR- 柠檬酸盐利用
utilization of itrate- 甘露糖
mannose+ 43℃生长
43℃ growth+ V.P. + 液化明胶
liquefying glutin+ 纤维二糖
cellobiose+ 0%NaCl生长
0%NaCl growth- 产硫化氢
produce H2S- 水解淀粉
hydrolyzing amylum+ 阿拉伯糖
Arabinose- 6%NaCl生长
6%NaCl growth+ 产氨
produce NH3- 葡萄糖产酸
produce acid of glucose+ 麦芽糖
maltose+ 10%NaCl生长
10%NaCl growth+ 赖氨酸脱羧酶
lysine decarbxylase+ 葡萄糖产气
produce gas of glucose- 半乳糖
galactose+ 0/129(10μg) + 精氨酸双水解酶
arginine dihydrolase- 肌醇
inositol- 鼠李糖
rhamnose- 0/129(150μg) + 鸟氨酸脱羧酶
ornithine decarbxylase+ 山梨醇
sorbitol- 甘露醇
mannitol+ 氧化酶
oxidase+ 脲酶
urease- 蔗糖
sucrose+ 密二糖
melibiose- 接触酶
catalase+ β-半乳糖甙酶
ONPG- 乳糖
lactose- 苦杏仁甙
amygdalin+ 吲哚
indole+ 硝酸盐还原
nitrate deoxidizing+ 果糖
fructose+ 2.4 胞外产物
以杯碟法测定溶藻弧菌胞外产物的酶活性表明(表 3),该细菌的胞外产物具有明胶酶、淀粉酶、酪蛋白酶、脂肪酶、卵磷脂酶、几丁质酶等多种酶活性,但没有脲酶活性。相同体积的胞外产物提取液中,淀粉酶、明胶酶的活性最高;其次是酪蛋白酶;脂肪酶、卵磷脂酶和几丁质酶的活性较低。另外,该菌株对兔红细胞具有一定的溶解能力以及溶血活性。
表 3 溶藻弧菌胞外产物的酶活性Table 3. The enzymatic activities of ECP of V.alginolyticus胞外产物
ECP明胶酶
gelatinase淀粉酶
amylase酪蛋白酶
casease脂肪酶
lipase卵磷脂酶
lecithinase几丁质酶
chitinase脲酶
urease溶血活性
haemolysin活性
ECP activity+++ +++ ++ + + + - + 2.5 药敏试验
药敏实验结果见表 4。20种化学药物中,有13种对病原菌具有明显抑制作用,其中以复方新诺明、磺胺+TMP、庆大霉素等3种药物的抑菌作用较强,妥布霉素、多粘菌素等次之,而以前水产养殖中常用的四环素等药物对此病原菌无抑菌作用或不明显,提示病原菌对其可能已具有抗性。
表 4 不同药物对菌株824-1的抑菌作用Table 4. The activities of the 20 antimicrobial agents on strain 824-1药品
antimicrobial agent含药量/μg或
IU·片-1
content抑菌圈直径/mm
inhibitory ring
diameter药品
antimicrobial agent含药量/μg或
IU·片-1
content抑菌圈直径/mm
inhibitory ring
diameter阿莫西林
moxycillin10 0 磺胺+TMP
sulfarmehoxazole/TMP25 25 新生霉素
neomycin30 10 卡那霉素
kanamycin30 16 氟哌酸
norfloxacin10 10 麦迪霉素
midecamycin30 0 庆大霉素
gentamycin10 20 丁胺卡那霉素
amikacin30 15 吡哌酸
pipemidic acid30 12 头孢孟多
cefamandole30 16 青霉素G
penicillin G10 0 妥布霉素
tobradistin10 17 强力霉素
doxycycline30 12 链霉素streptomycin 10 14 克林霉素
clindamycin2 0 四环素tetracycline 30 0 多粘菌素
polymycin300 15 氨苄青霉素
ampicillin10 0 复方新诺明
Co. SMZ25 26 磺胺甲基异噁唑
sulfamethoxazole10 0 2.6 中草药的抑菌作用
15种中草药对病原菌824-1菌株的抑菌效果见表 5。不同药物对病原菌的抑制作用相差较大,其中以石榴皮、地榆、五味子、大黄等4种药物的抑菌能力最强,其最小抑菌浓度(MIC)和最小杀菌浓度(MBC)分别为1∶ 640、1∶ 640、1∶ 320、1∶320和1∶ 320、1∶ 160、1∶ 160、1∶ 160。乌梅、公丁香、连翘、板蓝根等也有较强的抑菌作用;而大青叶、木瓜、威灵仙、地丁等只有在较高浓度时才有抑菌作用。
表 5 中草药对824-1菌株的最小抑菌浓度(MIC)和最小杀菌浓度(MBC)Table 5. MICs and MBCs of the eater extracts of Chinese herbal medicines evaluated药名CHM MIC MBC 药名CHM MIC MBC 药名CHM MIC MBC 乌梅
Fructus prunusis1∶160 1∶80 大黄
Rheum officinale1∶320 1∶160 木瓜
Fructus chaenomelesis1∶20 1∶20 黄芩
Radix scutellariae1∶80 1∶40 白芍
Rhizoma paeoniae1∶20 1∶20 威灵仙
Radix clematidis1∶10 1∶10 地榆
Rhizoma sanguisorbae1∶640 1∶160 大青叶
Folium polygoni1∶20 1∶10 地丁
Corydalis bungeana1∶10 1∶5 石榴皮
Pericarpium granati1∶640 1∶320 五味子
Fructus schizandrae1∶320 1∶160 黄芪
Radix astragalus1∶20 1∶20 公丁香
Flos caryophyllum1∶160 1∶80 连翘
Fructus forsythiae1∶160 1∶160 板蓝根
Rhizoma isatidis1∶160 1∶80 3. 讨论
菌株824-1的生理生化特性虽然与文献上对溶藻弧菌的描述略有差异,文献上溶藻弧菌能利用柠檬酸盐[2-3],而分离菌株却对柠檬酸盐不利用。推测其原因与同一种菌内不同个体之间的差异有关。按照细菌学分类原理,这种微小差异并不影响该种的分类地位的确立。另外,根据全自动细菌鉴定仪VITEK32的鉴定结果,分离菌株归属于溶藻弧菌的可信度在92%以上,因此可将分离菌株鉴定为溶藻弧菌(Vibrio aiginolyticus)。
溶藻弧菌广泛存在于自然海区的海水中,是一种条件致病菌,能引起海水鱼类、贝类、对虾[6-8]等发生疾病,但其致病性取决于宿主的健康状况、环境条件、海水中细菌数量、海水的理化物质等因素。
人工感染试验结果表明,溶藻弧菌对大黄鱼的致病途径不是经口,可能是鱼体受伤后,菌体通过创伤面进入体内,产生毒素,使病灶出血溃烂,造成死亡。对病原菌胞外产物的研究表明,其胞外产物含脂肪酶、蛋白酶、淀粉酶、明胶酶及溶血因子等成分,这与以往的报道基本一致[9]。由于淀粉酶有分解糖原的作用;明胶酶、酪蛋白酶等能分解胶原等蛋白成分;脂肪酶可分解脂类;而溶血因子又能使大黄鱼的血细胞溶解,所以推测胞外产物在溶藻弧菌侵染大黄鱼的过程中起到了一定的作用。因此,在夏季高水温期时,养殖海域中弧菌大量增殖,而由于网箱养殖密度过高,鱼碰撞受伤的机率大大增加,同时还由于寄生虫造成鱼体表损伤,继发细菌感染,进一步导致鱼体受多种因素的侵害而死亡。
药敏试验结果表明,病原菌对复方新诺明、磺胺+TMP、庆大霉素等3种药物较为敏感,可从中进一步筛选预防和治疗药物。
中草药抑菌试验表明,所使用的15种中草药对溶藻弧菌均有不同程度的抑制作用,其中石榴皮、地榆、五味子、大黄等4种药物的抑菌作用尤其显著。
由于抗菌药的大量使用,不但会提高病原菌的抗药性,还可能因为药物残留导致水产品质量安全下降,而中草药具有低毒、低残留、副作用小、不易产生抗药性等特点,且药中某些成分既有抗菌作用,又有免疫作用,能改善机体的免疫状态,提高自身抗菌能力[10],因此,可将上述中草药配制成抗病药物饵料来代替抗生素应用于海水网箱大黄鱼养殖生产。
-
表 1 样品种类、GenBank序列号和碱基组成
Table 1 Species, GenBank accession numbers and base composition of samples
科名
family属名
genus种名/数量
species/nos.序列长度/bp
lengthGenBank序列号
Accession number碱基组成 base composition GC含量/%
GCA T C G 海豚科
Delphinidae宽吻海豚属
Tursiops瓶鼻海豚
T.truncates(4)481 DQ839318* AY770538
U13123 Z1866332.1 26.2 20.2 21.5 41.7 白海豚属
Sousa中华白海豚
S.chinensis(2)481 AY770539 32.2 26.2 20.2 21.4 41.6 糙齿海豚属
Steno糙齿海豚(2)
S.bredanensis481 DQ839319* AF334508 31.8 25.5 21.1 21.6 42.7 真海豚属
Delphinus真海豚
D.delphis(2)481 U13106 Z18637 32.4 26.2 20.2 21.2 41.4 斑纹海豚属
Lagenorhynchus暗色斑纹海豚
L.obscurus(2)481 U13114 Z18649 32.2 26.4 20.2 21.2 41.4 白喙斑纹海豚
L.albirostris(2)481 U13113 AJ554061 32.5 25.8 20.9 20.8 41.7 露脊海豚属
Lissodelphis南露脊海豚
L.peronii(1)481 U13115 32.0 26.4 20.2 21.4 41.6 北露脊海豚
L.borealis(1)481 AF334504 32.4 26.4 20.2 21.0 21.2 喙头海豚属
Cephalorhynchu黑喙头海豚
C.eutropia(2)481 U13105 Z18635 32.2 26.2 20.4 21.2 41.6 原海豚属
Stenella蓝白原海豚(1)
S.coeruleoalba481 AJ010816 32.2 26.6 19.8 21.4 41.2 土库海豚属
Sotalia土库海豚
S.fluviatilis(1)481 AF304061 32.2 26.2 20.6 21.0 41.6 领航鲸属
Globicephala长鳍领航鲸
G.melaena(1)481 U13109 32.4 25.4 21.0 21.2 42.2 伪虎鲸属
Pseudorca伪虎鲸
P.crassidens(1)481 AF334506 32.2 25.6 20.8 21.4 42.2 抹香鲸科
Physeteridae抹香鲸属
Phuseter抹香鲸
P.catodon(2)482 U13119 Z18665 32.0 24.1 23.2 20.7 43.9 鼠海豚科
Phocoenidae鼠海豚属
Phocoena鼠海豚
P.phocoena(2)482, 483 U13121 AJ554063 31.4 26.9 19.7 21.9 41.6 棘鳍鼠海豚(1)
P.spinipinnis480 U13122 32.3 26.7 19.8 21.3 41.1 江豚属
Neophocaena江豚(1)
N.phocaenoides482 AF334503 31.3 26.8 20.1 21.8 41.9 一角鲸科
Monodontidae一角鲸属
Monodon一角鲸(2)
M.monoceros481 AF334502 AJ554062 31.8 26.2 20.6 21.4 42.0 白鲸属
Delphinapterus白鲸
D.leucas(2)481, 480 AF334501 U13107 31.6 25.8 21.0 21.5 42.5 小抹香鲸科
Kogiidae小抹香鲸属
Kogia小抹香鲸
K.breviceps(2)480,481 U13111 Z18643 31.6 24.5 22.5 21.3 43.8 拟小抹香鲸
K.simus(1)480 AF334490 31.5 24.6 22.3 21.7 44.0 喙鲸科
Ziphiidae柯氏喙鲸属
Ziphius剑吻鲸
Z.cavirostris(1)482 U13124 31.3 25.9 21.0 21.8 42.8 喙鲸属
Berardius贝氏喙鲸
B.bairdii(1)482 AF334493 32.2 25.7 21.6 20.5 42.1 中喙鲸属
Mesoplodon小喙鲸(2)
M.peruvianus483 U13118 Z18654
U13116 Z1865132.5 28.6 19.0 19.9 38.9 热氏中喙鲸(2)
M.europaeus482 32.2 27.6 19.5 20.7 40.2 露脊鲸科
Balaenidae弓头鲸属
Balaena北极鲸
B.mysticelus(2)481 U13102 AP006472 31.2 25.8 22.0 21.0 43.0 露脊鲸属
Eubalaena南露脊鲸
E.australis(1)481 AP006473 30.8 25.2 22.7 21.4 44.1 北太平洋露脊鲸
E.japonica(1)481 AP006474 31.4 25.4 22.2 21.0 43.2 小露脊鲸科
Neobalaenidae小露脊鲸属
Caperea小露脊鲸
C.marginata(1)481 AP006475 31.8 26.2 21.6 20.4 42.0 须鲸科
Balaenopteridae须鲸属
Balaenoptera长须鲸
B.physalus(2)481 U13103 Z18633 31.4 25.8 22.0 20.8 42.8 拟大须鲸
B.edeni(1)481 AB201258 31.2 27.0 20.8 21.0 41.8 大须鲸
B.borealis(1)482 AP006470 31.3 26.8 21.0 21.0 42.0 座头鲸属
Megaptera座头鲸(1)
M.novaeangliae482 U13117 31.1 26.6 21.4 21.0 42.4 灰鲸科
Eschrichtiidae灰鲸属
Eschrichtius灰鲸
E.robustus(1)483 U13108 31.5 25.5 21.9 21.1 43.0 恒河豚科
Platanistidae恒河豚属
Platanista恒河豚
P.gangetica(2)483 AF334491 AF248732 32.1 25.9 21.5 20.5 42.0 拉河豚科
Pontoporiidae拉河豚属
Pontopori拉河豚
P.blainvillei(1)483 AF248733 30.8 24.8 22.4 21.9 44.3 白暨豚科
Lipotidae白暨豚属
Lipotes白暨豚
L.vexillifer(2)482 AF304065 AF248731 31.7 26.8 21.0 20.5 41.5 亚河豚科
Iniidae亚河豚属
Inia亚河豚
I.geoffrensis(1)481 AF334497 30.8 25.6 22.5 21.2 43.7 *本研究
*this study -
[1] 王丕烈. 中国鲸豚[M]. 香港: 海洋企业有限公司, 1999: 9-13. [2] IRWIN D M, KOCHER T D, WILSON A C. Evolution of the cytochrome b gene of mammals[J]. J Mol Evol, 1991, 32(2): 128-144. doi: 10.1007/BF02515385
[3] GRAUR D, HIGGINS D G. Molecular evidence for the inclusion of cetaceans within the order Artiodactyla[J]. Mol Biol Evol, 1994, 11(3): 357-364. https://pubmed.ncbi.nlm.nih.gov/8015431/
[4] NIKAIDO M, ROONEY A P, OKADA N. Evolution and phylogenetic relationships among cetartiodactyls based on insertions of short and long interpersed elements: Hippopotamuses are the closest extant relatives of whales[J]. Proc Natl Acad Sci USA, 1999, 96(18): 10 261-10 266. doi: 10.1073/pnas.96.18.10261
[5] ARNASON U. Southern blot hybridization in cetaceans using killer whale restriction fragment as a probe[J]. Hereditas, 1982, 97(1): 47-49. https://pubmed.ncbi.nlm.nih.gov/6290428/
[6] HOELAEL A R, HANCOCK J M, DOVER G A. Evolution of the cetacean mitochondrial D-loop region[J]. Mol Biol Evol, 1991, 8(4): 475-493. https://academic.oup.com/mbe/article/8/4/475/990252
[7] NIKAIDO M, MATSUNO F, HAMILTONH, et al. Retroposon analysis of major cetacean lineages: the monophyly of the toothed whales and the paraphyly of river dolphins[J]. PNAS, 2001, 98(13): 7 384-7 389. doi: 10.1073/pnas.121139198
[8] MAY-COLLADO L, AGNARSSON I. Cytochrome b and Bayesian inference of whale phylogeny[J]. Mol Phylogenet Evol, 2006, 38(2): 344-354. doi: 10.1016/j.ympev.2005.09.019
[9] MILINKOVITCH M C, MEYER A, POWELL J R. Phylogeny of all major groups of Cetaceans based on DNA sequences from three mitochondrial genes[J]. Mol Biol Evol, 1994, 11(6): 939-948. https://pubmed.ncbi.nlm.nih.gov/7755710/
[10] HASEGAWA M, ADACHI J, Milinkovitch M C. Novel phylogeny of whales supported by total molecular evidence[J]. J Mol Evol, 1997, 44(Suppl. 1): 117-120. doi: 10.1007/PL00000062
[11] MESSENGER S, MCGUIRE J. Morphology, molecules, and the phylogenetics of cetaceans[J]. Syst Biol, 1998, 479(1): 90-124. https://pubmed.ncbi.nlm.nih.gov/12064244/
[12] ARNASON U, GULLBERG A. Cytochrome b nucleotide sequences and the identification of five primary lineages of extant cetaceans[J]. Mol Biol Evol, 1996, 13(2): 407-417. doi: 10.1093/oxfordjournals.molbev.a025599
[13] 杨光, 周开亚. 淡水豚类分子系统发生的研究[J]. 兽类学报, 1999, 19(1): 1-9. doi: 10.3969/j.issn.1000-1050.1999.01.001 [14] 杨光, 刘珊, 季国庆, 等. 淡水豚类线粒体DNA 12S rRNA基因的序列变异及其分子系统学研究[J]. 动物学研究, 2000, 21(6): 425-431. doi: 10.3321/j.issn:0254-5853.2000.06.001 [15] YANG G, ZHOU K Y, REN W H, et al. Molecular systematics of river dolphins inferred from complete mitochondrial cytochrome-b gene sequences[J]. Mar Mamm Sci, 2002, l8(1): 20-29. doi: 10.1111/j.1748-7692.2002.tb01015.x
[16] 刘珊, 杨光, 周开亚. 淡水豚类mtDNA 16S rRNA基因的系统发生[J]. 南京师范大学学报: 自然科学版, 2000, 23(4): 74-78. doi: 10.3969/j.issn.1001-4616.2000.03.019 [17] YAN J, ZHOU K Y, YANG G. Molecular phylogenetics of 'river dolphins′and the Baiji mitochondrial genome[J]. Mol Phylogenet Evol, 2005, 37(3): 743-750. doi: 10.1016/j.ympev.2005.06.009
[18] 杨光, 周开亚. 中国水域江豚种群遗传变异的研究[J]. 动物学报, 1997, 43(4): 411-419. doi: 10.3321/j.issn:0001-7302.1997.04.011 [19] YANG G, REN W H, ZHOU K Y, et al. Population genetic structure of finless porpoises, Neophocaena phocaenoides, in Chinese waters inferred from mitochondrial control region sequences[J]. Mar Mamm Sci, 2002, 18(2): 336-347. doi: 10.1111/j.1748-7692.2002.tb01041.x
[20] 夏军红, 郑颈松, 魏卓, 等. 天鹅洲保护区长江江豚AFLP遗传多样性分析[J]. 高技术通讯, 2004, 14(7): 25-28. doi: 10.3321/j.issn:1002-0470.2004.07.006 [21] WANG J Y, CHOU L S, WHITE B N. Mitochondrial DNA analysis of sympatric morphotypes of bottlenose dolphins (genus: Tursiops) in Chinese waters[J]. Mol Ecol, 1999, 8(10): 1 603-1 612. doi: 10.1046/j.1365-294x.1999.00741.x
[22] 季国庆, 杨光, 刘珊, 等. 中国水域瓶鼻海豚的mtDNA控制区序列变异性分析[J]. 动物学报, 2002, 48(4): 487-493. doi: 10.3969/j.issn.1674-5507.2002.04.008 [23] 王加连, 杨光, 刘海, 等. 线粒体DNA序列分析在中国水域真海豚物种鉴定中的初步应用[J]. 兽类学报, 2003, 23(2): 120-126. doi: 10.3969/j.issn.1000-1050.2003.02.006 [24] 杨光, 季国庆, 周开亚, 等. 鲸类c-mos基因的序列变异性及在系统发生分析中应用的初步研究[J]. 兽类学报, 2003, 23(4): 277-282. doi: 10.3969/j.issn.1000-1050.2003.04.001 [25] BERG W J, FERRIS S D. Restriction endonuclease analysis of salmonid mitochondrial DNA[J]. Can J Fish Aquat Sci, 1984, 41(77): 1 041-1 047. doi: 10.1139/f84-121
[26] FRANCE S C, KOCHER T D. Geographic and bathymetric patterns of mitochondrial 16S rRNA sequence divergence among deep-sea amphipods, Eurythenes gryllus[J]. Mar Biol, 1996, 126(4): 633-643. doi: 10.1007/BF00351330
[27] 刘亚军, 喻子牛, 姜艳艳, 等. 栉孔扇贝16S rRNA基因片段序列的多态性研究[J]. 海洋与湖沼, 2002, 33(5): 477-483. doi: 10.3321/j.issn:0029-814X.2002.05.004 [28] 江世贵, 张殿昌, 吕俊霖, 等. 3种野鲮亚科鱼类16S rRNA基因序列分析[J]. 南方水产, 2005, 1(1): 1-5. doi: 10.3969/j.issn.2095-0780.2005.01.001 [29] 郭奕惠, 黄桂菊, 喻达辉. 合浦珠母贝DNA的抽提和RAPD反应体系的优化[J]. 南方水产, 2006, 2(4): 59-64. doi: 10.3969/j.issn.2095-0780.2006.04.010 [30] 王小玉, 喻达辉, 郭奕惠, 等. 七种珍珠贝RAPD鉴别标记的初步研究[J]. 南方水产, 2006, 2(1): 18-22. doi: 10.3969/j.issn.2095-0780.2006.01.004 [31] BAKER C S, CIPRIANO F, PALUMBI S R. Molecular genetic identification of whale and dolphin products from commercial markets in Korea and Japan[J]. Mol Ecol, 1996, 5(5): 671-685. doi: 10.1111/j.1365-294X.1996.tb00362.x
[32] THOMPSON J D, GIBSON T J, PLEWNIAK F, et al. The clustal X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Res, 1997, 25(24): 4 876-4 882. doi: 10.1093/nar/25.24.4876
[33] KUMAR S, TAMURA K, NEI M. MEGA3: an integrated software for molecular evolutionary genetics analysis and sequence alignment[J]. Brief Bioinformatics, 2004, 5(2): 150-163. doi: 10.1093/bib/5.2.150
[34] 周开亚. 中国近海的两种宽吻海豚[J]. 兽类学报, 1987, 7(4): 246-254. https://www.cnki.com.cn/Article/CJFDTotal-SLXX198704001.htm [35] MILINKOVITCH M C, LEDUC R G, ADACHI J. Effects of character weighting and species sampling on phylogeny reconstruction: a case study based on DNA sequence data in cetaceans[J]. Genet, 1996, 144(4): 1 817-l 833. doi: 10.1093/genetics/144.4.1817
[36] HAMILTON H, CABALLERO S, COLLINS A G, et al. Evolution of river dolphins[J]. Royal Soc Proc: Biol Sci, 2001, 268(1 466): 549-558. doi: 10.1098/rspb.2000.1385
[37] CASSENS I, VICARIO S, WADDE V G, et al. Independent adaptation to riverine habitats allowed survival of ancient cetacean lineages[J]. PANS, 2000, 97(21): 11 343-11 347. doi: 10.1073/pnas.97.21.11343
[38] SLIJPERE J. Whales [M]. Ithaca, New York: Cornell University Press, 1979.



 下载:
下载:
 粤公网安备 44010502001741号
粤公网安备 44010502001741号
