Diagnostic RAPD markers of seven Chinese pearl oyster species
-
摘要:
采用RAPD分子标记技术对珠母贝属的大珠母贝、珠母贝、黑珠母贝、白珠母贝、合浦珠母贝、长耳珠母贝和珍珠贝属的企鹅珍珠贝的基因组DNA的特异性遗传标记进行分析。从21个OPM和S系列中筛选出4个引物, 共扩增出57个位点, 每条引物平均产生14·3个位点。扩增片段大小在250~2000bp间, 平均每种贝每条引物产生4·9条带。其中引物S10对7种珍珠贝的RAPD产物呈现出物种的特异性, 可同时将7种珍珠贝分开, 其余引物可以将2种或2种以上的珍珠贝区别开来。引物S10可以作为种间鉴定的标记。
Abstract:Random amplified polymorphic DNA (RAPD) technique was employed in analyzing the specific genetic markers of seven pearl oysters including Pinctada maxima, P.margaritifera, P.nigra, P.albina, P.fucata, P.chemnitzi in the genus Pinctada and Pteria penguin in the genus Pteria. Twenty one primers of S series and OPM series were screened. Four primers produce 57 loci. The average number of loci amplified by each primer is 14.3. The mean number of fingerprints per species amplified by each primer is 4.9 with length varying from 250 to 2 000 bp. Primer S10 generate diagnostic markers for each species and can distinguish the seven species studied simultaneously. The other primers screened in this study can discriminate two or more species from the others.
-
Keywords:
- species identification /
- Pinctada /
- Pteria /
- molecular markers /
- RAPD
-
我国的珍珠贝主要包括珠母贝属(Pinctada) 的大珠母贝(P.maxima)、珠母贝(P.margaritifera)、黑珠母贝(P.nigra)、白珠母贝(P.albina)、合浦珠母贝(P.fucata)、长耳珠母贝(P.chemnitzi)和珍珠贝属的企鹅珍珠贝(Pteria penguin)等[1-2]。这些珍珠贝是生产海水珍珠的重要母贝或具有潜在的重要经济价值,其养殖业是广东、广西和海南最具特色的海洋产业,闻名遐尔的“南珠”即为其中的合浦珠母贝所产。但部分珍珠贝种类之间形态差异小,如射肋珠母贝和合浦珠母贝仅凭形态描述很难鉴定[2]。有的种形态变异大,不同栖息环境、不同发育阶段其形态特征均有所不同,分类鉴定容易出错,因此出现了很多同物异名[3]。幼体发育阶段的材料由于个体小,凭形态特征进行种类分类也容易混淆。此外,合浦珠母贝、长耳珠母贝和大珠母贝种间人工杂交中是真正的杂交,或是雌核发育的结果?目前只有同工酶的实验证据[4],尚无DNA方面的直接证据,由于同工酶是基因表达的产物,还不能完全说明杂交后代的身份性质。对于没有外部形态特征的样品,如肌肉或内脏团组织样品,如何鉴定?因此开发种类特异(species specific)的DNA分子标记对种类鉴定、分子标记辅助育种等方面具有重要的应用价值。
随机扩增多态性DNA(random amplified polymorphic DNA,RAPD)是1990年创建的一种DNA多态检测技术[5-6]。该技术具有简单快速和多态性较高等优点,已广泛运用于分类鉴定[7-11]与亲缘关系分析[12-14]、遗传多样性[15-16]和连锁图谱构建[17]等。阎冰等[18]对马氏珠母贝和长耳珠母贝进行了RAPD分析,发现两者扩增带型差异较大。喻达辉和朱嘉濠[19]对珠母贝属6个种的ITS2序列进行了分析,发现不同种间序列差异较大,但种内差异非常小,并且发现白珠母贝和黑珠母贝的序列差异也较小,认为它们可能是亚种,因此认为ITS2序列可以作为种间鉴别标记。由于DNA序列标记在应用上不是太方便,本文利用RAPD技术对7种珍珠贝进行遗传标记分析,筛选种类特异的、基于PCR的简单快速的分子标记,为种类鉴定、遗传育种等提供简单实用的遗传标记。
1. 材料和方法
1.1 实验材料
分析的种类包括珠母贝属的大珠母贝、珠母贝、黑珠母贝、白珠母贝、合浦珠母贝、长耳珠母贝和珍珠贝属的企鹅珍珠贝。其中白珠母贝采自澳大利亚,长耳珠母贝采自大亚湾,黑珠母贝和部分大珠母贝个体由中国科学院南海海洋研究所何毛贤博士惠赠,其它种采自海南三亚,采于2001年。各个种的个体数见表 1。每个个体取闭壳肌样品保存于95%的酒精中备用。
表 1 4条引物对7种珍珠贝RAPD扩增的条带数及各个种的条带数Table 1. The RAPD fragments amplified from the 7 pearl oyster species using 4 random primers种类(个体数)
species(no. of individuals)各引物的平均带数
average fragments per primer总平均带数
total mean fragmentsS10 S17 S358 OPM17 大珠母贝Pinctada maxima (5) 1.3(1~2) 5.0(4~6) 3.8(2~5) 1.3(1~2) 11.3(10~13) 珠母贝P.maritifera (3) 2.7(2~3) 5.0(4~6) 4.7(3~6) 3.0(2~4) 15.3(12~17) 黑珠母贝P.nigra (3) 2.0(1~2) 4.7(2~7) 1.3(0~3) 0.3(0~1) 8.3(4~13) 白珠母贝P.albina (3) 2.7(2~4) 3.7(3~4) 3.3(2~5) 2.7(2~4) 12.3(10~16) 合浦珠母贝P.fucata (11) 4.0(3~5) 3.8(3~5) 2.2(0~4) 1.8(1~4) 11.8(10~13) 长耳珠母贝P.chemnitzi (4) 3.0(2~4) 6.8(6~8) 1.5(1~2) 2.8(2~3) 14.0(11~17) 企鹅珍珠贝Pteria penguin (3) 2.0(1~3) 6.3(4~8) 1.7(1~2) 3.3(2~5) 13.3(9~17) 引物的平均扩增带数
total mean fragments/primer2.7(1~5) 5.0(2~8) 2.6(0~6) 2.1(1~5) 4.9(0~8) 1.2 模板DNA制备
每个个体DNA提取采用本实验室改进的方法,分别取20 mg左右的闭壳肌,放入1.5 mL离心管内,先加入100 μL TEN9细胞裂解缓冲液(Tris-Cl 50 mmol · L-1,pH 9.0;EDTA 100 mmol · L-1;NaCl 200 mmol · L-1),剪碎,再加入TEN9至600 μL,混匀后于56℃温浴5 min,加入SDS至终浓度为2%,混匀,56℃温浴15 min,加入蛋白酶K(20 mg · mL-1)10 μL,于56℃消化至溶液澄清。加入15 μL RNase A,37℃反应约15 min,冷却后用常规的酚、氯仿方法纯化,乙醇沉淀DNA,干燥后加入200 μL去离子超纯水溶解,4℃存放。提取的基因组DNA样品用1%琼脂糖凝胶电泳、EB染色检测,再测定OD260和OD280,检测DNA的质量和计算浓度,配成20 ng · μL-1的DNA备用。
1.3 RAPD扩增
RAPD反应总体积为25 μL, 包括:1× PCR Buffer, 0.2 mmol · L-1 dNTPs, 2.0 mmol · L-1 MgCl2, 0.25 μmol · L-1引物, 1U Taq DNA合成酶,20 ng DNA模板。PCR循环程序为:94℃变性5 min,然后45个循环,每个循环包括:94℃ 1 min,40℃ 1 min,72℃ 2 min。最后72℃延伸10 min。21条引物筛选出4条用于扩增分析。引物由Operon公司合成。4条引物序列为:S10:CTGCTGGGAC,S17:AGGGAACGAG,S358:TGGTCGCAGA,OPM17:TCGGTCCGGG。
1.4 扩增产物电泳检测
取10 μL扩增产物在1%琼脂糖凝胶上进行电泳分离(0.5×TBE,3 V · cm-1恒压),EB染色,用凝胶成像仪(SynGene)进行分析。
2. 结果
2.1 RAPD扩增结果
4条引物共扩增出57个位点,平均每条引物产生14.3个位点,其中S17和S358产生的位点数较多,分别为19和18个位点,S10和OPM17产生的位点较少,分别为9和11。种内每条引物产生的平均带数在0.3~6.8条之间(表 1),4条引物扩增的总带数平均为8.3~15.3条,片段大小在250~2 000 bp之间(图 1)。其中黑珠母贝4条引物扩增的总带数最少,平均为每个体8.3条(表 1)。种间每条引物平均扩增2.1~5.0条(表 1),其中,S17扩增的带最多,平均为每种5.0条,其余3条引物扩增的平均带数较接近,在2.1~2.7条之间。平均每种每条引物扩增的带数为4.9条。
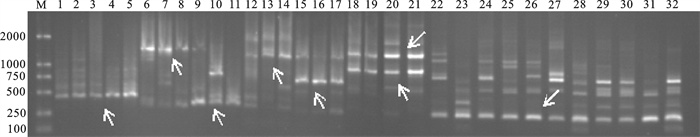
![]() 图 1 引物S10在7种珍珠贝中的RAPD扩增结果M.分子量标记; 1~5.大珠母贝; 6~8.黑珠母贝; 9~11.珠母贝; 12~14.白珠母贝; 15~17.企鹅珍珠贝; 18~21.长耳珠母贝; 22~32.合浦珠母贝Figure 1. RAPD amplification result of the seven pearl oyster species using random primer S10M. DNA ladder; 1~5. Pinctada maxima; 6~8. P.nigra; 9~11. P.margaritifera; 12~14. P.albina; 15~17. Pteria penguin; 18~21. Pincata chemnitzi; 22~32. P.fucata
图 1 引物S10在7种珍珠贝中的RAPD扩增结果M.分子量标记; 1~5.大珠母贝; 6~8.黑珠母贝; 9~11.珠母贝; 12~14.白珠母贝; 15~17.企鹅珍珠贝; 18~21.长耳珠母贝; 22~32.合浦珠母贝Figure 1. RAPD amplification result of the seven pearl oyster species using random primer S10M. DNA ladder; 1~5. Pinctada maxima; 6~8. P.nigra; 9~11. P.margaritifera; 12~14. P.albina; 15~17. Pteria penguin; 18~21. Pincata chemnitzi; 22~32. P.fucata2.2 RAPD分子标记
S17、S358和OPM17引物可在部分种扩增出种类特异的带,可以将2种或2种以上的珍珠贝区别开来,而S10在7种珍珠贝中均可扩增出种类特异的一致性带1~2条(图 1,箭头所示),可作为所研究种类鉴别的分子标记。
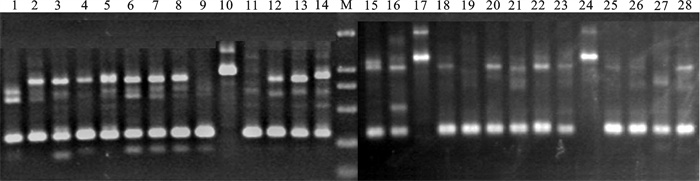
研究用S10引物对大亚湾合浦珠母贝种群28个个体进行扩增发现,有3个个体的扩增谱带明显不同于合浦珠母贝(图 2),与图 1比较,与长耳珠母贝相同。其它个体在250 bp左右均有1条合浦珠母贝特有的一致的扩增带。
3. 讨论
物种的鉴定,以往多根据经验的积累从其外形特征进行研究,进而发展到用生化指标(同工酶等)对物种进行鉴定。然而,这些方法所得到的标记往往是基因表达后的产物,受个体发育情况和外部环境条件的影响变化较大,容易产生误差。而DNA是与身俱来的遗传物质,具有很高的稳定性。因此DNA标记是揭示物种亲缘关系最有效的分子标记。随机扩增多态性DNA(RAPD)技术作为第二代分子标记具有简单、方便、快速、多态性较高等优点,是物种鉴定的首选标记[20]。CROSSLAND等[10]利用RAPD标记将Littorina的形态相似的2个姊妹种L.saxatilis和L.arcana很清楚地分开。KLINBUNGA等[11, 16]对泰国的5种牡蛎:Crassostrea belcheri, C.iredalei, Saccostrea cucullata, S.forskali和Striostrea mytiloides用RAPD技术进行了种类鉴定分析,结果发现C.belcheri, C.iredalei和S.cucullata能扩增出种类特异的带,而另外2种没有发现鉴别条带。可见同一条引物在多个种同时扩增出带并不十分容易。本研究通过采用RAPD技术筛选7种珍珠贝间的种类鉴定标记,4条引物中的1条引物S10可将所研究的7个种都分开,可作为种类鉴别标记。由于这7个种绝大部分都是我国的常见种,因此所获得的种间鉴别标记具有非常重要的实用价值。其它3条引物也可将部分种分开,其中S17可以将长耳珠母贝、合浦珠母贝、企鹅贝与其它贝区别开来,另外2条引物可以鉴别出2种或2种以上的珍珠贝。从扩增情况来看,每条引物扩增的带数并不多,但位点很多,说明不同种间扩增位点差异较大,表明RAPD技术在珍珠贝种类鉴定方面具有重要的开发价值。
-
图 1 引物S10在7种珍珠贝中的RAPD扩增结果
M.分子量标记; 1~5.大珠母贝; 6~8.黑珠母贝; 9~11.珠母贝; 12~14.白珠母贝; 15~17.企鹅珍珠贝; 18~21.长耳珠母贝; 22~32.合浦珠母贝
Figure 1. RAPD amplification result of the seven pearl oyster species using random primer S10
M. DNA ladder; 1~5. Pinctada maxima; 6~8. P.nigra; 9~11. P.margaritifera; 12~14. P.albina; 15~17. Pteria penguin; 18~21. Pincata chemnitzi; 22~32. P.fucata
表 1 4条引物对7种珍珠贝RAPD扩增的条带数及各个种的条带数
Table 1 The RAPD fragments amplified from the 7 pearl oyster species using 4 random primers
种类(个体数)
species(no. of individuals)各引物的平均带数
average fragments per primer总平均带数
total mean fragmentsS10 S17 S358 OPM17 大珠母贝Pinctada maxima (5) 1.3(1~2) 5.0(4~6) 3.8(2~5) 1.3(1~2) 11.3(10~13) 珠母贝P.maritifera (3) 2.7(2~3) 5.0(4~6) 4.7(3~6) 3.0(2~4) 15.3(12~17) 黑珠母贝P.nigra (3) 2.0(1~2) 4.7(2~7) 1.3(0~3) 0.3(0~1) 8.3(4~13) 白珠母贝P.albina (3) 2.7(2~4) 3.7(3~4) 3.3(2~5) 2.7(2~4) 12.3(10~16) 合浦珠母贝P.fucata (11) 4.0(3~5) 3.8(3~5) 2.2(0~4) 1.8(1~4) 11.8(10~13) 长耳珠母贝P.chemnitzi (4) 3.0(2~4) 6.8(6~8) 1.5(1~2) 2.8(2~3) 14.0(11~17) 企鹅珍珠贝Pteria penguin (3) 2.0(1~3) 6.3(4~8) 1.7(1~2) 3.3(2~5) 13.3(9~17) 引物的平均扩增带数
total mean fragments/primer2.7(1~5) 5.0(2~8) 2.6(0~6) 2.1(1~5) 4.9(0~8) -
[1] 王祯瑞. 中国近海珍珠贝科的研究[M]//中国科学院海洋研究所. 海洋科学集刊. 北京: 科学出版社, 1978, 14: 101-117. [2] 王祯瑞. 软体动物门双壳纲珍珠贝亚目. 中国动物志, 无脊椎动物, 第31卷[M]. 北京: 科学出版社, 2002: 68-98. [3] HYND J S. A revision of the Australian pearl-shells, genus Pinctada(Lamellibranchia)[J ]. Aust J Mar Fresh Res, 1955, 6(1): 98-137. doi: 10.1071/MF9550098
[4] 李刚, 姜卫国, 魏贻尧. 合浦珠母贝、长耳珠母贝和大珠母贝种间人工杂交的研究: III同工酶谱的比较研究[J]. 热带海洋, 1983, 2(4): 321-328. https://xueshu.baidu.com/usercenter/paper/show?paperid=d25002588c7ad1d2cb78cdfe7ecc996a&site=xueshu_se&hitarticle=1 [5] WILLIANS J G, KUBELIK A R, LIVAK K J, et al. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers[J]. Nucleic Acids Res, 1990, 18(22): 6531-6535. doi: 10.1093/nar/18.22.6531
[6] WELSH J, MICHAEL M. Fingerprinting genomes using PCR with arbitrary primers[J]. Nucleic Acids Res, 1990, 18(24): 7213-7218. doi: 10.1093/nar/18.24.7213
[7] 邓怀, 张四明, 汪登强, 等. 十种常见淡水鱼类的RAPD鉴定[J]. 淡水渔业, 1998, 28(1): 8-10. doi: 10.3969/j.issn.1000-6907.1998.01.002 [8] 刘萍, 孟宪红, 庄志猛, 等. 3种对虾种间RAPD遗传标记[J]. 海洋水产研究, 2002, 23(2): 1-6. doi: 10.3969/j.issn.1000-7075.2002.02.001 [9] LIU Z, LI P, ARGUE B J, et al. Inheritance of RAPD markers in channel catfish (Ictalurus punctatus), blue catfish (I. furcatus), and their F1, F2 and backcross hybrids[J]. Animal Genetics, 1998, 29(1): 58-62. doi: 10.1046/j.1365-2052.1998.00284.x
[10] CROSSLAND S, COATES D, GRAHAME J, et al. Use of random amplified polymorphic DNAs (RAPDs) in separating two sibling species of Littorina[J]. Mar Ecol Prog Ser, 1993, 96(3): 301-305. doi: 10.3354/meps096301
[11] KLINBUNGA S, AMPAYUP P, TASSANAKAJON A, et al. Development of species specific markers of the tropical oyster (Crassostrea belcheri) in Thailand[J]. Mar Biotechnol, 2000, 2(5): 476-484. doi: 10.1007/s101260000028
[12] 张四明, 邓怀, 汪登强, 等. 7种鲟形目鱼类亲缘关系的随机扩增多态性DNA研究[J]. 自然科学进展, 1999, 9(9): 818-823. doi: 10.3321/j.issn:1002-008X.1999.09.009 [13] 邹曙明, 楼允东, 孙效文, 等. 用RAPD方法研究草鱼、柏氏鲤和3个地理种群鲤的亲缘关系[J]. 中国水产科学, 2000, 7(3): 6-11. doi: 10.3321/j.issn:1005-8737.2000.01.002 [14] 夏德全, 曹萤, 吴婷婷, 等. 用RAPD方法分析太湖大银鱼、太湖新银鱼和寡齿新银鱼的亲缘关系[J]. 中国水产科学, 2000, 7(1): 12-15. doi: 10.3321/j.issn:1005-8737.2000.01.003 [15] LIU Z J, LI P, ARGUE B J, et al. Random amplified polymorphic DNA markers: usefulness for gene mapping and analysis of genetic variation of catfish[J]. Aquac, 1999, 174(1): 59-68. doi: 10.1016/S0044-8486(99)00007-1
[16] KLINBUNGA S, AMPAYUP P, TASSANAKAJON A, et al. Genetic diversity and molecular markers of cupped oysters (genera Crassostrea, Caccostrea, and Striostrea) in Thailand revealed by randomly amplified polymorphic DNA analysis[J]. Mar Biotechnol, 2001, 3(2): 133-144. doi: 10.1007/s101260000057
[17] 孙效文, 梁利群. 鲤鱼的遗传连锁图谱(初报)[J]. 中国水产科学, 2000, 7(3): 2-5. [18] 阎冰, 叶力, 邓凤娇, 等. 马氏珠母贝与解氏珠母贝的随机扩增多态DNA分析[J]. 广西科学, 2001, 8(4): 287-290. doi: 10.3969/j.issn.1005-9164.2001.04.013 [19] 喻达辉, 朱嘉濠. 珠母贝属6个种的ITS2分子标记研究[J]. 南方水产, 2005, 1(4): 6-12. doi: 10.3969/j.issn.2095-0780.2005.04.002 [20] LIU Z J, CORDES J F. DNA marker technologies and their applications in aquaculture genetics[J]. Aquac, 2004, 238(1): 1-37. doi: 10.1016/j.aquaculture.2004.05.027



 下载:
下载:
 粤公网安备 44010502001741号
粤公网安备 44010502001741号
