Preliminary exploration of Balaenoptera edeni edeni population distribution in surrounding waters of Weizhou Island based on environmental DNA
-
摘要:
鲸类作为海洋生态系统中的顶级捕食者,是维持海洋生态平衡和生态稳定的关键物种。广西涠洲岛布氏鲸 (Balaenoptera edeni edeni) 是中国近海唯一稳定出现的须鲸种群,但其栖息地分布现状尚不明确。由于布氏鲸活动能力强、分布范围广,目视监测难以进行稳定跟踪调查。基于此,结合环境DNA (Environmental DNA, eDNA) 技术,监测了不同时期 (2022年4月和2023年1月) 涠洲岛布氏鲸栖息地的分布现状。研究发现,4月在布氏鲸的热点分布海域 (涠洲岛—斜阳岛之间) 目视和eDNA均发现布氏鲸存在 (n=3),同时在涠洲岛西南海域也发现布氏鲸存在 (n=2),其中有1个站位仅eDNA检测到;1月在布氏鲸的热点分布海域目视和eDNA均发现布氏鲸存在 (n=1),涠洲岛东部海域仅eDNA检测到布氏鲸存在 (n=1)。结果表明,eDNA技术相比目视具有更高的灵敏度,可用于验证布氏鲸的分布,同时发现涠洲岛东部和西南部海域是布氏鲸的潜在热点分布海域。该研究验证了eDNA技术在涠洲岛布氏鲸分布监测上的可行性,进一步明确了涠洲岛布氏鲸栖息地的分布现状,为其种群的高效监测和科学保护提供了基线信息。
Abstract:Cetaceans, as apex predators in marine ecosystems, play a key role in maintaining ecological balance and stability. The Eden's whale (Balaenoptera edeni edeni) in Weizhou Island is the only baleen whales population that consistently appears in the waters near China. However, their specific distribution around Weizhou Island remains unclear. Due to their extensive range and active behavior, conducting stable tracking surveys via visual means is challenging. Thus, through environmental DNA (eDNA) technology, we assessed the distribution status of Eden's whales habitats around Weizhou Island during different periods (April 2022 and January 2023). The study reveals that in April, Eden's whales were visually observed and detected via eDNA in the hotspot distribution area (Between Weizhou Island and Xieyang Island) (n=3). They were also found in the southwest waters of Weizhou Island (n=2), with one site solely identified via eDNA. In January, Eden's whales were visually observed through eDNA in the hotspot distribution area (n=1) and detected through eDNA in the eastern waters off Weizhou Island (n=1). The findings indicate that compared with visual observation, eDNA technology exhibited higher sensitivity and could be utilized to verify the distribution of Eden's whales. Furthermore, potential hotspot distribution areas for Eden's whales were identified in the eastern and southwestern waters off Weizhou Island. In conclusion, this research validates the feasibility of utilizing eDNA technology for monitoring the distribution of Eden's whales around Weizhou Island. Besides, it provides further clarification regarding the distribution status of Eden's whales habitats around Weizhou Island, which supplies essential baseline information for the effective monitoring and scientific conservation of this population.
-
鲸类动物作为海洋生态系统中的重要成员,扮演着不可替代的角色;作为顶级捕食者,在调控海洋食物链和生态平衡中具有关键作用。鲸类通过控制中下层生物资源量,对海洋生态系统的稳定性与多样性产生直接影响。鲸类的洄游和迁徙行为使之成为不同海洋生态系统间的桥梁,促使物质循环、能量传递等关键生态过程协调进行。此外,鲸类还在文化、经济领域发挥着巨大作用,譬如鲸豚在绘画、雕塑中的文化象征,赏鲸活动为地区带来经济收益[1]等。然而,截至2021年,世界上已有37%的鲸类被国际自然及自然资源保护联盟(IUCN)列为受威胁物种。鲸类的栖息依赖于猎物的可获得性、安全的环境和特定水温;人类海洋活动和气候变化带来的环境压力,将会造成鲸类分布和行为的改变[2-4]。因此,亟需快速、有效的鲸类生物监测方法,为其管理和保护措施的制定提供参考。
鲸类分布的研究多依赖于目视观测和被动声学调查[5-6],调查方法的局限性导致鲸类分布的基础信息仍显不足,限制了有效保护措施的制定。环境DNA (Environmental DNA, eDNA) 技术是一种非侵入性收集水生动物遗传信息的方法,具有灵敏、准确、可靠的优势。该技术通过采集水生动物释放到水体中的遗传物质,利用PCR技术可对濒危、稀有物种进行遗传监测,对目标物种和环境的负面影响极小[7-8],已被广泛应用于多种水生动物的监测[9-13]。2016年,中国广西涠洲岛周围海域布氏鲸 (Balaenoptera edeni edeni) 群体被首次发现[14-15]。基于公民科学问卷调查与实地调查相结合的方法对涠洲岛周围海域布氏鲸觅食区的调查表明,该海域布氏鲸的觅食热点区域位于涠洲岛与斜阳岛之间,觅食的高频时间为每年3—4月[14-15]。线上渔民社区和应用程序2种方式收集的布氏鲸群体目击数据显示从11月至次年2月均有其目击记录,分布区域在涠洲岛以南及斜阳岛周围海域[16]。此外,有研究发现2019—2021年涠洲岛—斜阳岛周边海域布氏鲸种群数量较为稳定,无明显差异,并新发现2种捕食行为[17]。
以往对布氏鲸分布的研究集中于涠洲岛—斜阳岛邻近海域,但其在涠洲岛—斜阳岛周围海域分布的现状仍不清晰,且以往研究多使用目视观测数据。因此,本研究拟采用目视与eDNA相结合方法,系统探究布氏鲸在涠洲岛—斜阳岛周边海域的分布现状,尤其是热点栖息地的分布,以期为北部湾布氏鲸的长期监测与保护提供基线信息。
1. 材料与方法
1.1 研究区域
涠洲岛位于北部湾海域中部,是广西最大的两个古火山残堆体岛屿之一[18],面积约26 km2,年均气温23.0 ℃,平均降水量1 297 mm,年均海面温度24.6 ℃,年均海水盐度31.9‰[19],蕴含丰富的海洋生物资源。
1.2 样品采集
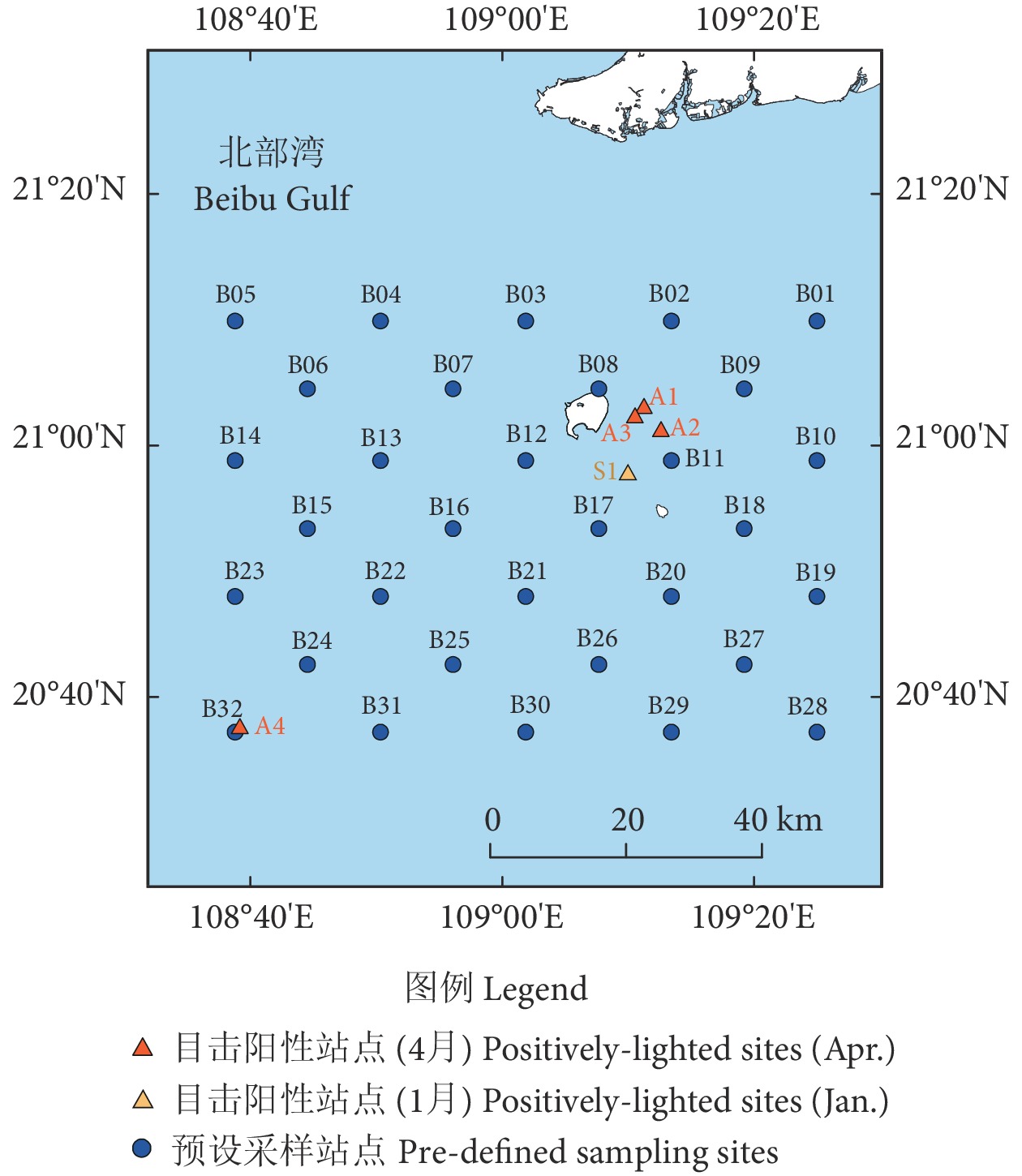
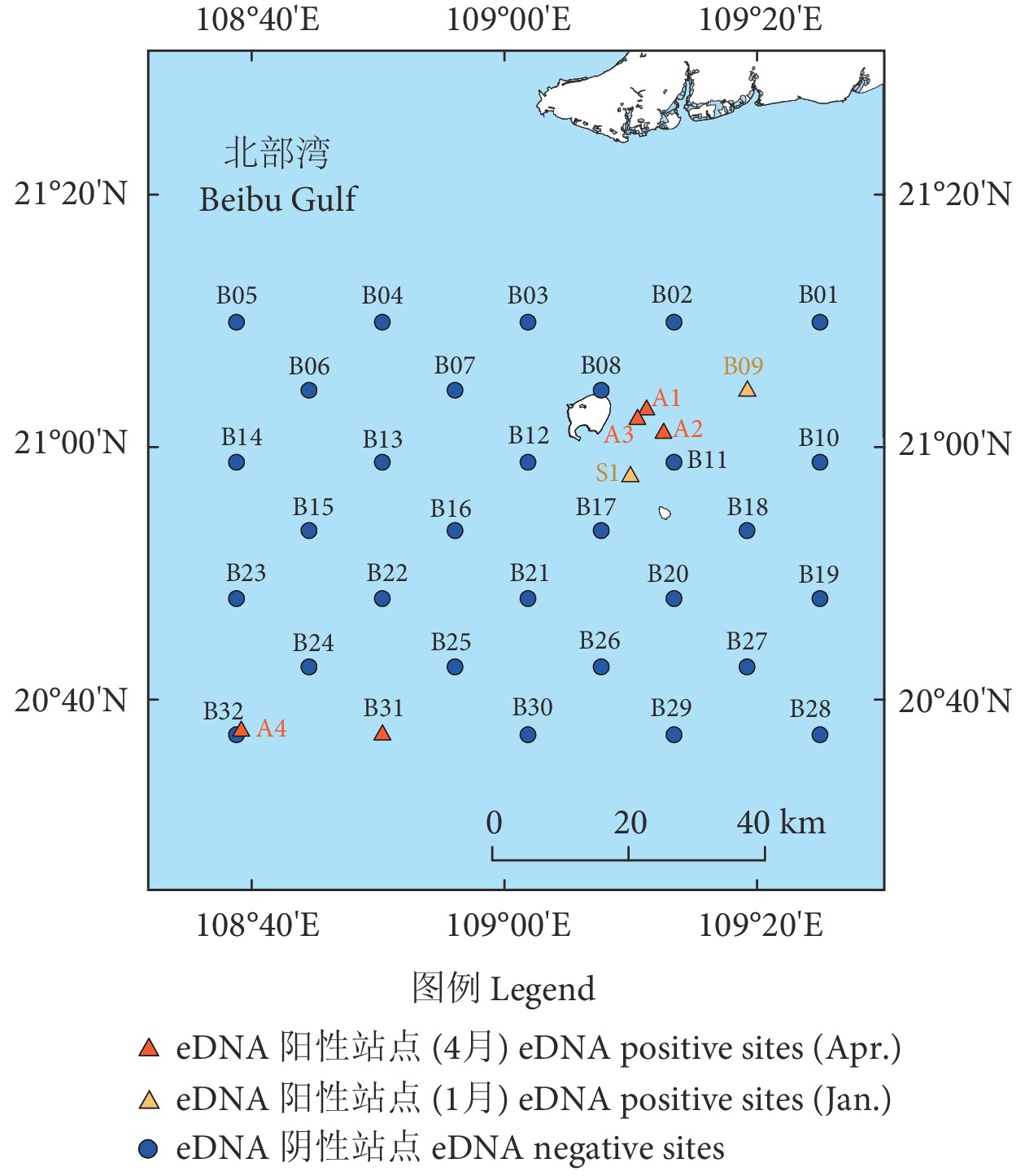
调查站位从涠洲岛—斜阳岛布氏鲸热点分布海域向四周扩张设计,共32个采样站点(图1)。采样时间为2022年4月和2023年1月,其中1月是布氏鲸陆续出现在涠洲岛邻近海域并且数量初步稳定的时期,4月是布氏鲸少部分个体开始离开的时期。按截线走航方式调查,使用快艇“涠洲22018”(长8.27 m,发动机132 kW) 采集eDNA水样,并同步采用目视观测。抵达目标站点后使用采水器采集表层 (水深约1 m) 水4.5 L,同时在观察到布氏鲸的站点采集eDNA水样(图1)。使用直径47 mm、孔径0.45 μm的混合纤维素滤膜 (Pall Corporation,美国) 进行真空过滤。每张滤膜过滤水样1 L,每个站点过滤4张滤膜,装入2 mL无菌冻存管中,置于干冰中低温保存。其中布氏鲸目击阳性位点过滤2张滤膜。为防止交叉污染,过滤装置依次经过5% (φ) 次氯酸钠浸洗、蒸馏水冲洗和所在站点海水润洗。每个采样点均以相同的方式过滤1 L蒸馏水作为阴性对照。地图数据来源于全国地理信息资源目录服务系统 (https://www.webmap.cn/,1∶25万全国基础地理数据库,2019公众版) 。
1.3 引物设计
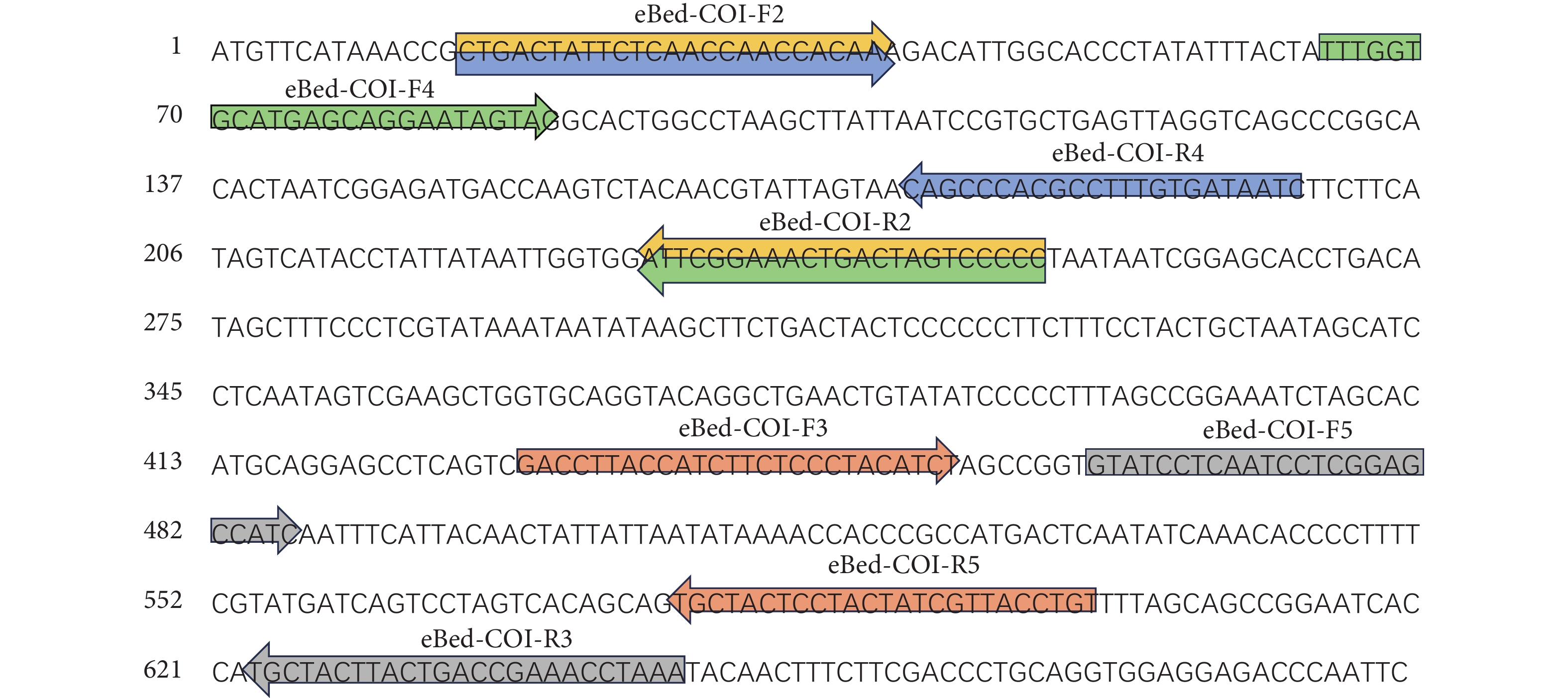
从美国国家生物技术信息中心 (NCBI) 下载布氏鲸线粒体全序列 (序列号:NC_007938),选择物种鉴定常用的COI基因序列作为目的片段,利用Primer Premier软件[20]设计5对布氏鲸扩增引物,扩增长度均在200 bp左右 (表1,图2)。
表 1 布氏鲸线粒体COI目的片段扩增引物信息Table 1 Primers information for target fragment of Eden's whale mitochondrial COI引物
Primer引物序列 (5'—3')
Primer sequence (5'−3')扩增长度
Amplification length/bp延伸时间
Extension time/s退火温度
Annealing temperature/℃P1 F: CTGACTATTCTCAACCAACCACAAA
R: GGGGGACTAGTCAGTTTCCGAAT222 16 58 P2 F: CTGACTATTCTCAACCAACCACAAA
R: GATTATCACAAAGGCGTGGGCTG184 14 58 P3 F: GACCTTACCATCTTCTCCCTACATCT
R: ACAGGTAACGATAGTAGGAGTAGCA173 13 58 P4 F: TTTGGTGCATGAGCAGGAATAGTAG
R: GGGGGACTAGTCAGTTTCCGAAT190 14 58 P5 F: GTATCCTCAATCCTCGGAGCCATC
R: TTTAGGTTTCGGTCAGTAAGTAGCA185 14 58 1.4 引物特异性检验
将5对布氏鲸扩增引物在NCBI的Primer-BLAST平台上分别与广西海域历史搁浅记录的13种鲸类[21]进行引物特异性检验。其中,引物特异性检验的严格性参数设置如下:允许总错配数为2,3' 端允许错配数为1,其他参数使用默认设置。
1.5 eDNA的提取
使用海洋动物组织基因组DNA提取试剂盒DP324 (天根,中国) 提取总eDNA,提取操作步骤参照李红婷等[22]改进的方案。将滤膜剪碎后,浸泡在800 µL的GA缓冲液和80 µL的蛋白酶K中,56 ℃下孵化至膜完全裂解 (约2.5 h),将提取的eDNA洗脱到100 μL的TE缓冲液中,–40 ℃冻存备用。
1.6 引物验证
选取2个布氏鲸目击站点 (A1、A2) 的2张滤膜作为eDNA阳性模板 (n=4) 进行PCR扩增效果检验。另选取7个非目击站点 (B12、B16、B17、B19、B20、B21、B26) 的3张滤膜 (n=21) 进行补充验证。PCR扩增体系为:15 μL 2×Pro Taq Master Mix (AG,中国),3 μL DNA模版,0.5 μL 正反向引物,11 μL 无菌去离子水。PCR扩增程序为:98 ℃预变性3 min;98 ℃变性30 s,58 ℃退火30 s,72 ℃延伸,延伸时间见表1,30个循环;72 ℃延伸5 min。扩增产物经2% (w)琼脂糖凝胶电泳检验。选取目的条带送往北京睿博兴科生物技术有限公司进行双向测序,测序引物与扩增引物相同。所得序列用BLASTn比对,确定引物扩增的特异性。
1.7 布氏鲸的种群分布探究
根据引物验证结果,选取扩增条带单一明亮且阳性扩增比例最高的引物 (P1),进行涠洲岛周围海域所有站点的eDNA检测。后续实验的扩增体系、扩增程序、物种确认与引物验证步骤相同。
eDNA的提取和扩增实验均在中国水产科学研究院南海水产研究所的eDNA专用分子实验室进行,实验室定期采用紫外线和次氯酸钠溶液消毒。
2. 结果
2.1 不同引物特异性检验情况
以北部湾海域历史搁浅的13种鲸类作为引物特异性检验的基础 (表2)。结果显示,5对针对COI基因的引物均能完全匹配布氏鲸的目标序列。除了小鳁鲸 (B. acutorostrata) 外,5对引物未能在其他11种鲸类中匹配到相应的目标序列。对于小鳁鲸,引物P1、P2、P4、P5能在其数据库中检测到目标序列,但引物P2在其数据库中检测到的序列均存在2个或更多的碱基差异,而引物P4、P5检测到的目标序列至少存在1个碱基差异。
表 2 不同引物在北部湾鲸类中的特异性检验Table 2 Specificity testing of different primers for cetacean species in Beibu Gulf搁浅鲸类
Stranded cetacean引物P1
Primer P1引物P2
Primer P2引物P3
Primer P3引物P4
Primer P4引物P5
Primer P5布氏鲸 Balaenoptera edeni edeni √ √ √ √ √ 小鳁鲸 B. acutorostrata √ — × — — 长吻真海豚 Delphinus capensis × × × × × 短吻真海豚 D. delphis × × × × × 里氏海豚 Grampus griseus × × × × × 伪虎鲸 Pseudorca crassidens × × × × × 中华白海豚 Sousa chinensis × × × × × 热带斑海豚 Stenella attenuata × × × × × 长吻飞旋海豚 S. longirostris × × × × × 糙齿海豚 Steno bredanensis × × × × × 印太宽吻海豚 Tursiops aduncus × × × × × 宽吻海豚 T. truncatus × × × × × 印太江豚 Neophocaena phocaenoides × × × × × 注:“×”表示在选定数据库中未匹配到目标序列;“√”表示在选定数据库中匹配到目标序列;“—”表示检测出的所有目标序列均有碱基差异。 Note: "×" represents the absence of the target sequence in the selected database; "√" represents the presence of the target sequence in the selected database; "—" represents that all detected target sequence exhibit variations in nucleotide bases. 2.2 不同引物PCR扩增情况
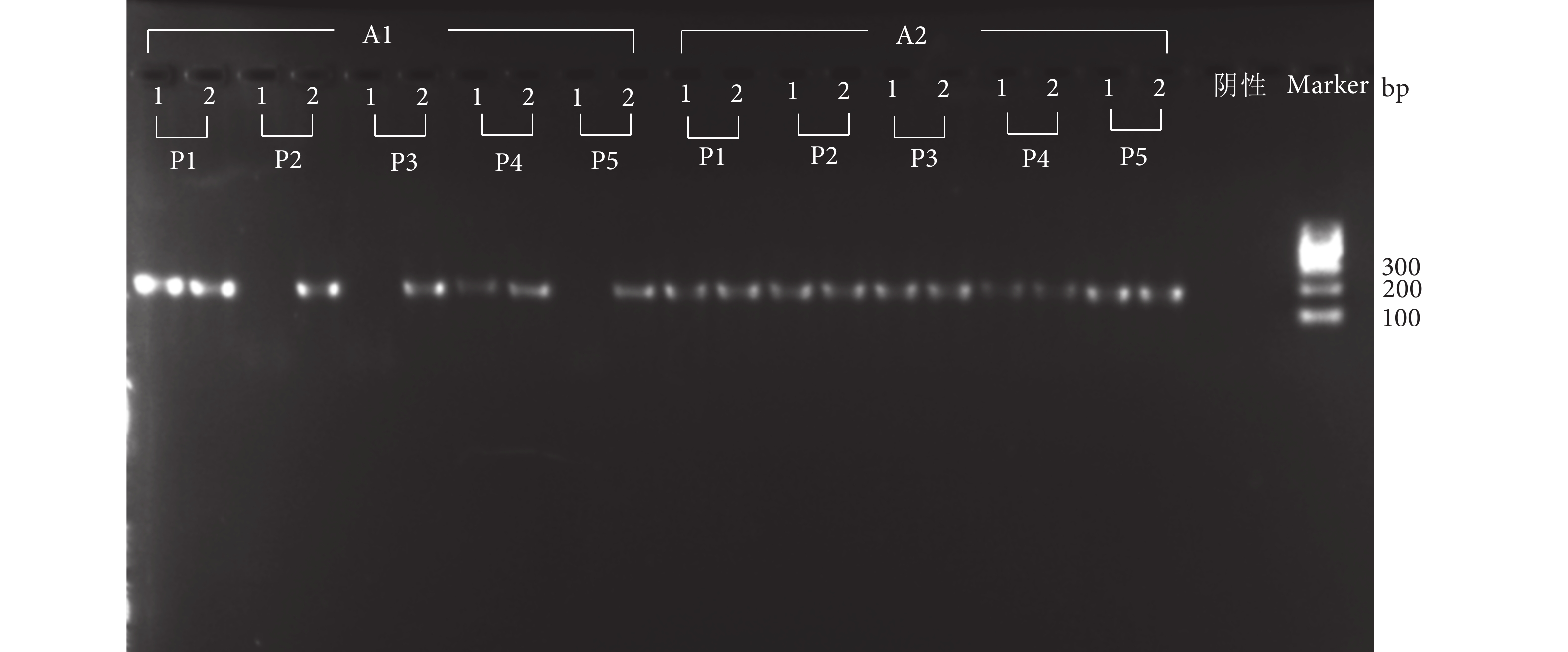
布氏鲸COI基因的引物扩增检测结果 (图3) 显示:在2个目击站点 (A1、A2) 的所有重复样本中,引物P1、P4成功扩增出目的条带。而引物P2、P3和P5在A1站点的第1个重复样本中未能扩增出目的条带。
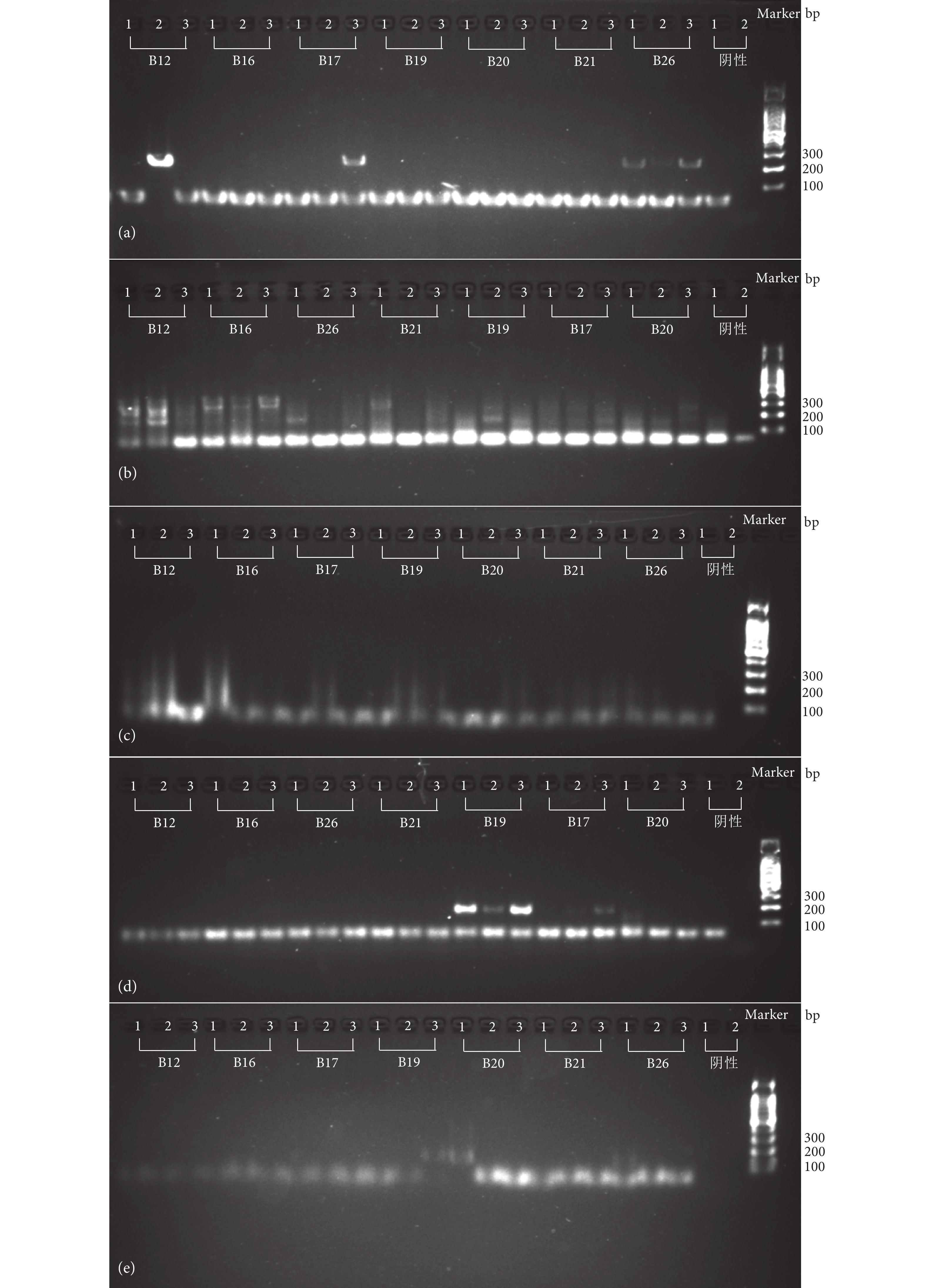
通过对7个预设采样站点的布氏鲸eDNA验证实验 (图4) 发现,引物P1在5个样本中成功扩增出目标条带,阳性扩增率为23.80%。除了B26站点的1个重复扩增条带较弱外,其他4个样本的扩增条带清晰明亮,未出现拖带现象。引物P2的扩增结果普遍出现拖带,目标片段的条带不明显。引物P3未能成功扩增出目标条带。引物P4在4个样本中扩增出目标条带,阳性扩增率为19.04%,其中只有在B19站点的2个重复样本中目标片段的扩增条带较为明显。引物P5仅在2个样本中扩增出目标条带,阳性扩增率为9.52%,但这2个条带的清晰度均不高。鉴于引物P1具有较高的阳性扩增比例且条带清晰,因此决定采用引物P1作为涠洲岛布氏鲸分布eDNA检测的分子标记。
![]() 图 4 预设采样站点布氏鲸eDNA扩增结果注:a、b、c、d、e分别为引物P1、P2、P3、P4、P5扩增胶图;1、2、3表示重复样品;B12—B26表示预设采样站点。Fig. 4 Eden's whale eDNA amplification of preset sampling sitesNote: a, b, c, d, e represent primers of P1, P2, P3, P4 and P5 amplification gels; 1, 2, 3 represent replicate samples; B12−B26 represent pre-defined sampling sites.
图 4 预设采样站点布氏鲸eDNA扩增结果注:a、b、c、d、e分别为引物P1、P2、P3、P4、P5扩增胶图;1、2、3表示重复样品;B12—B26表示预设采样站点。Fig. 4 Eden's whale eDNA amplification of preset sampling sitesNote: a, b, c, d, e represent primers of P1, P2, P3, P4 and P5 amplification gels; 1, 2, 3 represent replicate samples; B12−B26 represent pre-defined sampling sites.2.3 目的片段测序筛选情况
布氏鲸COI基因的测序结果 (表3) 表明,引物P1在布氏鲸目击站点的3个样本 (A1-2、A2-1和A2-2) 中均成功获得布氏鲸COI序列。 A1站点的第1个重复样本 (A1-1) 测序结果出现双峰,且序列无匹配结果。在补充验证的样本中,引物P1有3个样本测序结果为池鹭 (Ardeola bacchus),另有1个样本为家鸡 (Gallus gallus)。引物P2和P3在目击站点的3个样本中成功获得布氏鲸COI序列。引物P4也在目击站点的3个样本中成功获得布氏鲸COI序列,但A1-1的测序结果未匹配到任何物种;而在补充验证的样本中,引物P4的2个样本测序结果为沙丁鱼属 (Sardinella sp.)。引物P5只在目击站点的3个样本中成功获得布氏鲸COI序列,补充验证的样本扩增条带模糊,未能进行测序。
表 3 布氏鲸COI基因片段测序比对结果Table 3 Sequencing and alignment results of Eden's whale COI gene fragment站点
Sampling site重复
Repetition引物 Primer P1 P2 P3 P4 P5 A1 1 * — — * — A1 2 布氏鲸 布氏鲸 布氏鲸 布氏鲸 布氏鲸 A2 1 布氏鲸 布氏鲸 布氏鲸 布氏鲸 布氏鲸 A2 2 布氏鲸 布氏鲸 布氏鲸 布氏鲸 布氏鲸 B12 1, 2, 3 池鹭 (2) — — — — B16 1, 2, 3 — — — — — B17 1, 2, 3 家鸡 (3) — — — — B19 1, 2, 3 — — — 沙丁鱼属 (1, 3) — B20 1, 2, 3 — — — — — B21 1,2,3 — — — — — B26 1, 2, 3 池鹭 (1, 3) — — — — 注:“—”表示未扩增出布氏鲸;“*”表示扩增出目标条带但测序失败。 Note: "—" represents that B. edeni edeni have not been amplified; "*" represents successful amplification of the target band, but sequencing was unsuccessful. 2.4 涠洲岛周围海域布氏鲸eDNA的分布监测
采用引物P1检测2022年4月和2023年1月涠洲岛周围海域布氏鲸的eDNA存在情况。研究显示,在4月的调查航次中,采水站点数共36个,目击阳性率为11.11% (n=4),eDNA阳性率为13.89% (n=5),其中B31站点未目击到布氏鲸,但成功检出了布氏鲸的eDNA (图5)。在1月调查航次共33个采样站点中,除了S1目击站点外, B09预设采样站点也检测到了布氏鲸COI分子标记,目击率为3.03%,eDNA检出率为6.06%。上述结果表明,布氏鲸的分布并不局限于已知的涠洲岛和斜阳岛之间海域,在涠洲岛东部和西南海域也能通过eDNA证实其存在痕迹,说明其可能是布氏鲸潜在分布海域。
3. 讨论
3.1 eDNA技术可用于检测珍稀物种的存在
eDNA技术已被广泛运用于水生生物的监测中[23-27]。eDNA技术的高灵敏度促使其能够检测到目标生物的微量DNA,从而使其成为监测稀有或低密度种群的有效手段[28]。例如,有研究利用eDNA技术对弗吉尼亚州北部溪流中的木龟 (Glyptemys insculpta) 分布进行监测,即使在传统方法未能发现其踪迹的情况下,也能在预期位点检测到木龟的eDNA[29]。此外,作为一种非侵入性监测方法,eDNA技术还被用于包括长江江豚 (Neophocaena asiaeorientalis asiaeorientalis)[30]、港湾鼠海豚 (Phocoena Phocoena)[31]以及鳐 (Zearaja maugeana)[32]等珍稀濒危物种的保护研究上,显著减少了对生态环境和目标物种的干扰[33-34]。eDNA技术的另一大优势在于其监测门槛较低,不仅成本相对较低,对人员技术的要求也不高,因此更适用于大规模的监测项目[35]。
本研究基于eDNA技术的检测结果不仅覆盖了2个调查航次的所有目击站点,而且在4月航次的B31站点和1月航次的B09站点,即使无目击记录,也成功检测到了布氏鲸的存在。这一发现突显了eDNA技术在物种分布研究中的预测能力,为其研究提供了更大的时间窗口。已有关于虎鲸 (Orcinus orca) eDNA的研究表明[27],即使是在虎鲸经过后的2 h内,仍可检测到其eDNA,展现了eDNA技术的潜力。另一项新近研究[36]通过eDNA技术推测了中国东南沿海布氏鲸可能的迁徙路线,进一步证实了eDNA技术在监测布氏鲸分布方面的预测价值。本研究中,4月航次目击站点A4与预设采样站点B32地理位置接近,A4站点成功检测到了布氏鲸的COI分子标记,而B32站点未检测到,这一结果表明eDNA技术能够在小尺度空间内进行物种分布研究,体现了其在空间分辨率上的可靠性。
3.2 eDNA特异性引物的选择
在eDNA技术中,引物的选择对成功检出目标物种至关重要。引物的特异性是准确识别目标物种的关键,考虑到水环境中含有来自各类生物的eDNA,设计引物时要保证特异性,以确保扩增得到的DNA片段来源于目标物种而非其他物种。此外,引物的灵敏度和检测限同样重要,优质的引物能够提升eDNA技术的灵敏度,使其在低浓度条件下也能检出目标物种的DNA,这对追踪稀有物种尤为关键[37]。同时,引物反应的稳定性也是一个重要因素,引物选择应保证在不同环境条件下可实现稳定可靠的反应[38]。
本研究选取线粒体COI基因片段作为布氏鲸检测的分子标记。线粒体DNA (mtDNA) 在细胞中存在多个拷贝,并被线粒体膜所包裹,研究显示mtDNA的降解速度相对较慢[39],因此eDNA中的mtDNA片段被捕获和检测到的概率更高。此外,线粒体COI基因在进化过程中高度保守,但在足够的变异位点上仍能反映物种间的差异,这确保了基于eDNA检测布氏鲸的特异性和稳定性。虽然其保守性导致近缘物种间变异较小,限制了对同一区域内近缘物种的特异性检测,但本研究通过对研究区域历史搁浅鲸类数据库的引物特异性反比分析显示,本研究设计的引物在除小鳁鲸外的11种鲸类中均具有良好的特异性。尽管在区分同属布氏鲸属的小鳁鲸时特异性较弱,但在整个研究期间未目击到小鳁鲸,且实验所得序列均经过物种鉴定确认,未发现小鳁鲸在研究区域存在。因此,可认为小鳁鲸对本次研究结果的影响可以忽略不计。另外,为避免假阴性导致的实验误差,本研究选择了扩增长度约200 bp、阳性扩增率高的引物进行布氏鲸分布检测。短扩增片段能提高扩增灵敏度,但引物特异性也会受到影响。因此,本研究以测序结果作为最终的检测标准,而非仅依赖于扩增结果。未来可寻找特异性与灵敏性的最佳平衡,或考虑开发新的分子标记,如线粒体D-loop基因、核DNA分子标记等,结合多种分子标记的共同检测可能成为解决这一挑战的有效途径。
3.3 环境因素对eDNA监测的影响
水样中eDNA的完整性是影响扩增结果的主要因素。eDNA在水环境中以溶解、颗粒吸附、细胞内和细胞器内4种形式存在,受水体环境参数影响,每种状态各具特定的衰减率[40]。此外,水样中的天然PCR抑制剂,比如海水中的高盐度[31]和淡水中的腐殖质[41],均可能对eDNA检测结果产生影响,甚至导致假阴性结果。
在4月的调查航次中,A1目击站点就出现了假阴性情况,而重复样本成功检出了布氏鲸COI序列,表明增加样本量可提高eDNA检出率,降低假阴性风险[42]。同时,假阴性样本使用引物P1和P4扩增出条带的结果,排除了水样中天然PCR抑制剂导致假阴性的可能性,推测假阴性可能由eDNA不完整造成,这可能源于样本提取过程中的捕获失败或保存不当。为避免假阴性出现,可通过增加水样体积和过滤水量[43],或优化DNA提取流程[22],来提高eDNA的捕获率。
此外,4月航次中B12、B17、B19和B26站点出现了非特异扩增。鉴于水环境中eDNA的衰减特性,选择较短的DNA片段进行检测虽可以提高目标物种的检出率,但也可能导致物种识别错误和偏差[8]。在PCR扩增过程中,非目标物种的eDNA与目标物种的eDNA会产生竞争,导致目标物种eDNA的扩增机会减少,从而产生非特异性扩增。为减少非特异性扩增,本研究对PCR产物进行测序验证,确保扩增的DNA片段源自目标物种。另有研究利用在PCR反应中适当提高退火温度[44]或采用嵌套式PCR方法[45]以增强扩增反应的特异性。
3.4 涠洲岛布氏鲸的分布现状和保护建议
此前的研究指出,2017—2018年,涠洲岛布氏鲸种群主要分布在涠洲岛西南海域及斜阳岛周围,且全年均有出现,3—4月的出现频率最高[14];2019—2020年的调查显示,从9月到次年4月均有布氏鲸的目击记录,其中1月的目击频率最高,布氏鲸的分布范围主要在涠洲岛至斜阳岛之间海域[16]。然而,船基调查的准确性在很大程度上依赖于调查努力量和调查人员的经验,这导致不同调查者得到的结果可能存在偏差。eDNA技术作为一种低门槛、程序化的监测方法,能达到更客观和统一的调查标准。本研究中,4月和1月航次的eDNA检测结果涵盖了所有已知的布氏鲸目击点,在未目击布氏鲸的B31站点(4月航次)和B09站点(1月航次)也检测到了布氏鲸的存在,这些eDNA检测结果覆盖了先前研究指出的布氏鲸种群热点分布区域,进一步验证了eDNA技术在确认物种分布方面的有效性。
1月的调查结果表明布氏鲸的活动范围主要集中在已知的热点分布区及其周边海域,而4月的调查结果不仅在热点分布区内3次目击到布氏鲸,在涠洲岛西南方向约49 km的海域(A4站点)也目击到了布氏鲸,且A4和B31站点的eDNA检测结果为阳性,验证了该区域存在布氏鲸活动。据目视观测结果可知,布氏鲸经常傍晚才出现在涠洲岛及斜阳岛之间的热点分布区捕食,推测布氏鲸在其他时段可能迁移到了更深的西南部海域,该区域可能是布氏鲸的潜在热点分布区域。eDNA技术的应用不仅拓展了对布氏鲸分布范围的认识,还提高了分布评估的准确性。将eDNA技术与定量PCR技术如荧光定量PCR (qPCR)[46] 和数字液滴PCR (ddPCR)[47]相结合,可进行更为精确的布氏鲸eDNA浓度测定,进而对核心栖息地进行客观评估。此外,有基于卫星数据分析的研究建议在涠洲岛周围20 km的海域内建立布氏鲸保护区[48] ,但这忽略了涠洲岛西南海域也可能存在布氏鲸的核心栖息地。利用卫星数据预测的分布模型虽为保护区划定提供了依据,但实际观测表明,布氏鲸极少出现在涠洲岛邻近的西北海域,且在涠洲岛南部海域20 km以外存在潜在的核心栖息地。因此,建议通过eDNA技术进一步扩大调查范围,以确定布氏鲸保护区的准确边界,为建立合理的保护区提供科学依据。
4. 结论与展望
大型鲸类的分布研究一直是个难点问题。eDNA技术的出现为该问题的解决提供了新的方案。本研究针对北部湾布氏鲸的研究初步验证了该技术的有效性并筛选出特异性的检测引物,还发现了涠洲岛布氏鲸种群可能存在多个热点分布栖息地,但仍有待更大时空尺度的研究验证。同时,本研究所使用的传统PCR技术在微量eDNA扩增检测方面仍不够灵敏,而qPCR技术能实时监测DNA的合成过程,并通过与标准曲线对比,实现对初始模板DNA浓度的相对定量检测[49]。此外, ddPCR技术作为第三代PCR技术可以准确计算初始模板DNA的数量,规避PCR抑制等问题[50]。结合eDNA技术与新兴PCR定量技术,将为提升eDNA技术的灵敏度和应用能力提供有力支撑。
-
图 4 预设采样站点布氏鲸eDNA扩增结果
注:a、b、c、d、e分别为引物P1、P2、P3、P4、P5扩增胶图;1、2、3表示重复样品;B12—B26表示预设采样站点。
Figure 4. Eden's whale eDNA amplification of preset sampling sites
Note: a, b, c, d, e represent primers of P1, P2, P3, P4 and P5 amplification gels; 1, 2, 3 represent replicate samples; B12−B26 represent pre-defined sampling sites.
表 1 布氏鲸线粒体COI目的片段扩增引物信息
Table 1 Primers information for target fragment of Eden's whale mitochondrial COI
引物
Primer引物序列 (5'—3')
Primer sequence (5'−3')扩增长度
Amplification length/bp延伸时间
Extension time/s退火温度
Annealing temperature/℃P1 F: CTGACTATTCTCAACCAACCACAAA
R: GGGGGACTAGTCAGTTTCCGAAT222 16 58 P2 F: CTGACTATTCTCAACCAACCACAAA
R: GATTATCACAAAGGCGTGGGCTG184 14 58 P3 F: GACCTTACCATCTTCTCCCTACATCT
R: ACAGGTAACGATAGTAGGAGTAGCA173 13 58 P4 F: TTTGGTGCATGAGCAGGAATAGTAG
R: GGGGGACTAGTCAGTTTCCGAAT190 14 58 P5 F: GTATCCTCAATCCTCGGAGCCATC
R: TTTAGGTTTCGGTCAGTAAGTAGCA185 14 58 表 2 不同引物在北部湾鲸类中的特异性检验
Table 2 Specificity testing of different primers for cetacean species in Beibu Gulf
搁浅鲸类
Stranded cetacean引物P1
Primer P1引物P2
Primer P2引物P3
Primer P3引物P4
Primer P4引物P5
Primer P5布氏鲸 Balaenoptera edeni edeni √ √ √ √ √ 小鳁鲸 B. acutorostrata √ — × — — 长吻真海豚 Delphinus capensis × × × × × 短吻真海豚 D. delphis × × × × × 里氏海豚 Grampus griseus × × × × × 伪虎鲸 Pseudorca crassidens × × × × × 中华白海豚 Sousa chinensis × × × × × 热带斑海豚 Stenella attenuata × × × × × 长吻飞旋海豚 S. longirostris × × × × × 糙齿海豚 Steno bredanensis × × × × × 印太宽吻海豚 Tursiops aduncus × × × × × 宽吻海豚 T. truncatus × × × × × 印太江豚 Neophocaena phocaenoides × × × × × 注:“×”表示在选定数据库中未匹配到目标序列;“√”表示在选定数据库中匹配到目标序列;“—”表示检测出的所有目标序列均有碱基差异。 Note: "×" represents the absence of the target sequence in the selected database; "√" represents the presence of the target sequence in the selected database; "—" represents that all detected target sequence exhibit variations in nucleotide bases. 表 3 布氏鲸COI基因片段测序比对结果
Table 3 Sequencing and alignment results of Eden's whale COI gene fragment
站点
Sampling site重复
Repetition引物 Primer P1 P2 P3 P4 P5 A1 1 * — — * — A1 2 布氏鲸 布氏鲸 布氏鲸 布氏鲸 布氏鲸 A2 1 布氏鲸 布氏鲸 布氏鲸 布氏鲸 布氏鲸 A2 2 布氏鲸 布氏鲸 布氏鲸 布氏鲸 布氏鲸 B12 1, 2, 3 池鹭 (2) — — — — B16 1, 2, 3 — — — — — B17 1, 2, 3 家鸡 (3) — — — — B19 1, 2, 3 — — — 沙丁鱼属 (1, 3) — B20 1, 2, 3 — — — — — B21 1,2,3 — — — — — B26 1, 2, 3 池鹭 (1, 3) — — — — 注:“—”表示未扩增出布氏鲸;“*”表示扩增出目标条带但测序失败。 Note: "—" represents that B. edeni edeni have not been amplified; "*" represents successful amplification of the target band, but sequencing was unsuccessful. -
[1] HOYT E. Whale watching 2001: wordwide tourism number, expenditures, and expanding socioeconomic benefits[M]. Yarmouth Port, MA, USA: International Fund for Animal Welfare (IFAW), 2001: 13-14.
[2] POLOCZANSKA E S, BROWN C J, SYDEMAN W J, et al. Global imprint of climate change on marine life[J]. Nat Clim Chang, 2013, 3(10): 919-925. doi: 10.1038/nclimate1958
[3] PECL G T, ARAUJO M B, BELL J D, et al. Biodiversity redistribution under climate change: Impacts on ecosystems and human well-being[J]. Science, 2017, 355: 6332.
[4] WEELDEN C V, TOWERS J R, BOSKER T. Impacts of climate change on cetacean distribution, habitat and migration[J]. Clim Chang Ecol, 2021, 1: 100009.
[5] LI M, WANG X X, HUNG S K, et al. Indo-Pacific humpback dolphins (Sousa chinensis) in the Moyang River Estuary: the western part of the world's largest population of humpback dolphins[J]. Aquat Conserv: Mar Freshw Ecosyst, 2019, 29(5): 798-808. doi: 10.1002/aqc.3055
[6] ZURIEL Y E, AVSHALOM N L, RIJN I V, et al. Multi-year passive acoustic monitoring of coastal dolphins along the Israeli Mediterranean shallow shelf reveals the impact of marine fish farms and trawling patterns on their habitat utilization[J]. Mar Environ Res, 2023, 188: 106014. doi: 10.1016/j.marenvres.2023.106014
[7] BOHMANN K, EVANS A, GILBERT M T, et al. Environmental DNA for wildlife biology and biodiversity monitoring[J]. Trends Ecol Evol, 2014, 29(6): 358-367. doi: 10.1016/j.tree.2014.04.003
[8] BENG K C, CORLETT R T. Applications of environmental DNA (eDNA) in ecology and conservation: opportunities, challenges and prospects[J]. Biodivers Conserv, 2020, 29(7): 2089-2121. doi: 10.1007/s10531-020-01980-0
[9] KUMAR G, EBLE J E, GAITHER M R. A practical guide to sample preservation and pre-PCR processing of aquatic environmental DNA[J]. Mol Ecol Resour, 2020, 20: 29-39. doi: 10.1111/1755-0998.13107
[10] BESSEY C, JARMAN S N, SIMPSON T, et al. Passive eDNA collection enhances aquatic biodiversity analysis[J]. Commun Biol, 2021, 4: 236-248. doi: 10.1038/s42003-021-01760-8
[11] TANG Y K, WU Y S, LIU K, et al. Investigating the distribution of the Yangtze finless porpoise in the Yangtze River using environmental DNA[J]. PLoS One, 2019, 14(8): e0221120. doi: 10.1371/journal.pone.0221120
[12] VALSECCHI E, BYLEMANS J, GOODMAN S J, et al. Novel universal primers for metabarcoding environmental DNA surveys of marine mammals and other marine vertebrates[J]. Environ DNA, 2020, 2(4): 460-476. doi: 10.1002/edn3.72
[13] ZOU K S, CHEN J W, RUAN H T, et al. eDNA metabarcoding as a promising conservation tool for monitoring fish diversity in a coastal wetland of the Pearl River Estuary compared to bottom trawling[J]. Sci Total Environ, 2020, 702: 134704. doi: 10.1016/j.scitotenv.2019.134704
[14] CHEN M, HUANG S L, WU H P, et al. Occurrence of Bryde's whales, Balaenoptera edeni, in the northern Beibu Gulf, China[J]. Mar Mammal Sci, 2019, 35(4): 1643-1652. doi: 10.1111/mms.12607
[15] CHEN B Y, ZHU L, JEFFERSON T A, et al. Coastal Bryde's whales' (Balaenoptera edeni) foraging area near Weizhou Island in the Beibu Gulf[J]. Aquat Mamm, 2019, 45(3): 274-279. doi: 10.1578/AM.45.3.2019.274
[16] ZHANG Y Y, CHEN M, CHEN M, et al. Community-based population monitoring for large baleen whales: the case study of Bryde's whale in Beibu Gulf of China[J]. Integr Zool, 2021, 16(4): 626-635. doi: 10.1111/1749-4877.12525
[17] 吴采雯. 广西布氏鲸的种群动态及捕食策略研究[D]. 南京: 南京师范大学, 2021: 1-6. [18] 何如, 黄梅丽, 罗红磊, 等. 近五十年来广西海岛的气候变化与气象灾害特征分析[J]. 气象研究与应用, 2015, 36(2): 31-35, 39. doi: 10.3969/j.issn.1673-8411.2015.02.006 [19] 黎广钊, 梁文, 农华琼, 等. 涠洲岛珊瑚礁生态环境条件初步研究[J]. 广西科学, 2004(4): 379-384. [20] YE J, COULOURIS G, ZARETSKAYA I, et al. Primer-BLAST: a tool to design target-specific primers for polymerase chain reaction[J]. BMC Bioinform, 2012, 13: 134. doi: 10.1186/1471-2105-13-134
[21] LIU M M, LIN M L, LI S H. Species diversity and spatiotemporal patterns based on cetacean stranding records in China, 1950–2018[J]. Sci Total Environ, 2022, 822: 153651. doi: 10.1016/j.scitotenv.2022.153651
[22] 李红婷, 张帅, 邹柯姝, 等. 珠江河口水体环境DNA提取方法的建立及优化[J]. 南方水产科学, 2022, 18(3): 30-37. doi: 10.12131/20210304 [23] VISSER F, MERTEN V J, BAYER T, et al. Deep-sea predator niche segregation revealed by combined cetacean biologging and eDNA analysis of cephalopod prey[J]. Sci Adv, 2021, 7: eabf5908. doi: 10.1126/sciadv.abf5908
[24] 王学昉, 孟维钊, 王丛丛, 等. 环境DNA技术在长江口水生生物监测中的应用潜力[J]. 应用海洋学学报, 2021, 40(3): 547-554. [25] PARSONS K M, EVERETT M, DAHLHEIM M, et al. Water, water everywhere: environmental DNA can unlock population structure in elusive marine species[J]. R Soc Open Sci, 2018, 5(8): 180537. doi: 10.1098/rsos.180537
[26] SZÉKELY D, CORFIXEN N L, MØRCH L L, et al. Environmental DNA captures the genetic diversity of bowhead whales (Balaena mysticetus) in West Greenland[J]. Environ DNA, 2021, 3(1): 248-260. doi: 10.1002/edn3.176
[27] BAKER C S, STEEL D, NIEUKIRK S, et al. Environmental DNA (eDNA) from the wake of the whales: droplet digital PCR for detection and species identification[J]. Front Mar Sci, 2018, 5: 133. doi: 10.3389/fmars.2018.00133
[28] JERDE C L, MAHON A R, CHADDERTON W L, et al. "Sight-unseen" detection of rare aquatic species using environmental DNA[J]. Conserv Lett, 2011, 4(2): 150-157. doi: 10.1111/j.1755-263X.2010.00158.x
[29] AKRE T S, PARKER L D, RUTHER E, et al. Concurrent visual encounter sampling validates eDNA selectivity and sensitivity for the endangered wood turtle (Glyptemys insculpta)[J]. PLoS One, 2019, 14(4): e0215586. doi: 10.1371/journal.pone.0215586
[30] 吴昀晟, 唐永凯, 李建林, 等. 环境DNA在长江江豚监测中的应用[J]. 中国水产科学, 2019, 26(1): 124-132. [31] FOOTE A D, THOMSEN P F, SVEEGAARD S, et al. Investigating the potential use of environmental DNA (eDNA) for genetic monitoring of marine mammals[J]. PLoS One, 2012, 7(8): e41781. doi: 10.1371/journal.pone.0041781
[32] WELTZ K, LYLE J M, OVENDEN J, et al. Application of environmental DNA to detect an endangered marine skate species in the wild[J]. PLoS One, 2017, 12(6): e0178124. doi: 10.1371/journal.pone.0178124
[33] GOLDBERG C S, STRICKLER K M, PILLIOD D S. Moving environmental DNA methods from concept to practice for monitoring aquatic macroorganisms[J]. Biol Conserv, 2015, 183: 1-3. doi: 10.1016/j.biocon.2014.11.040
[34] JIANG P W, ZHANG S, XU S N, et al. Comparison of environmental DNA metabarcoding and bottom trawling for detecting seasonal fish communities and habitat preference in a highly disturbed estuary[J]. Ecol Indic, 2023, 146: 109754. doi: 10.1016/j.ecolind.2022.109754
[35] THOMSEN P F, KIELGAST J, IVERSEN L L, et al. Monitoring endangered freshwater biodiversity using environmental DNA[J]. Mol Ecol, 2012, 21: 2565-2573. doi: 10.1111/j.1365-294X.2011.05418.x
[36] ZHANG S, CAO Y T, CHEN B Y, et al. Assessing the potential use of environmental DNA for multifaceted genetic monitoring of cetaceans: example of a wandering whale in a highly disturbed bay area[J]. Ecol Indic, 2023, 148: 110125. doi: 10.1016/j.ecolind.2023.110125
[37] DEINER K, BIK H M, MACHLER E, et al. Environmental DNA metabarcoding: transforming how we survey animal and plant communities[J]. Mol Ecol, 2017, 26(21): 5872-5895. doi: 10.1111/mec.14350
[38] GOLDBERG C S, TURNER C R, DEINER K, et al. Critical considerations for the application of environmental DNA methods to detect aquatic species[J]. Methods Ecol Evol, 2016, 7(11): 1299-1307. doi: 10.1111/2041-210X.12595
[39] ALLENTOFT M E, COLLINS M, HARKER D, et al. The half-life of DNA in bone: measuring decay kinetics in 158 dated fossils[J]. Proc Biol Sci, 2012, 279(1748): 4724-4733.
[40] MAUVISSEAU Q, HARPER L R, SANDER M, et al. The multiple states of environmental DNA and what is known about their persistence in aquatic environments[J]. Environ Sci Technol, 2022, 56(9): 5322-5333. doi: 10.1021/acs.est.1c07638
[41] MATHESON C D, GURNEY C, ESAU N, et al. Assessing PCR inhibition from humic substances[J]. Open Enzym Inhib J, 2010, 3: 38-45.
[42] SHAW J L A, CLARKE L J, WEDDERBURN S D, et al. Comparison of environmental DNA metabarcoding and conventional fish survey methods in a river system[J]. Biol Conserv, 2016, 197: 131-138. doi: 10.1016/j.biocon.2016.03.010
[43] REES H C, MADDISON B C, MIDDLEDITCH D J, et al. Review: the detection of aquatic animal species using environmental DNA−a review of eDNA as a survey tool in ecology[J]. J App Ecol, 2014, 51(5): 1450-1459. doi: 10.1111/1365-2664.12306
[44] 刘玉香, 宋晓琛, 江香梅. 润楠ISSR-PCR优化反应体系建立及引物筛选[J]. 林业科技开发, 2013, 27(5): 24-28. [45] 彭雷, 赵艳, 马银花. 基于巢式PCR的重叠延伸PCR优化[J]. 安徽农业科学, 2016, 44(20): 126-127. [46] SIGSGAARD E E, CARL H, MØLLER P R, et al. Monitoring the near-extinct European weather loach in Denmark based on environmental DNA from water samples[J]. Biol Conserv, 2015, 183: 46-52. doi: 10.1016/j.biocon.2014.11.023
[47] 李莎, 刘雪清, 姜伟, 等. 环境DNA技术在宜昌江段四大家鱼自然繁殖中的应用[J]. 应用生态学报, 2021, 32(6): 2241-2248. [48] ZHANG Y Y, SUN X D, NONG Z W, et al. The first baleen whale marine protected area proposed for Bryde's whales in the Beibu Gulf, China[J]. Mar Mammal Sci, 2023: 1-17.
[49] BUSTIN S A, BENES V, GARSON J A, et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments[J]. Clin Chem, 2009, 55(4): 611-622. doi: 10.1373/clinchem.2008.112797
[50] WHALE A S, HUGGETT J F, COWEN S, et al. Comparison of microfluidic digital PCR and conventional quantitative PCR for measuring copy number variation[J]. Nucleic Acids Res, 2012, 40(11): e82. doi: 10.1093/nar/gks203



 下载:
下载:
 粤公网安备 44010502001741号
粤公网安备 44010502001741号
