Comparison of fish species detection effect of three sets of commonly used eDNA metabarcoding primers on Sanya water samples
-
摘要:
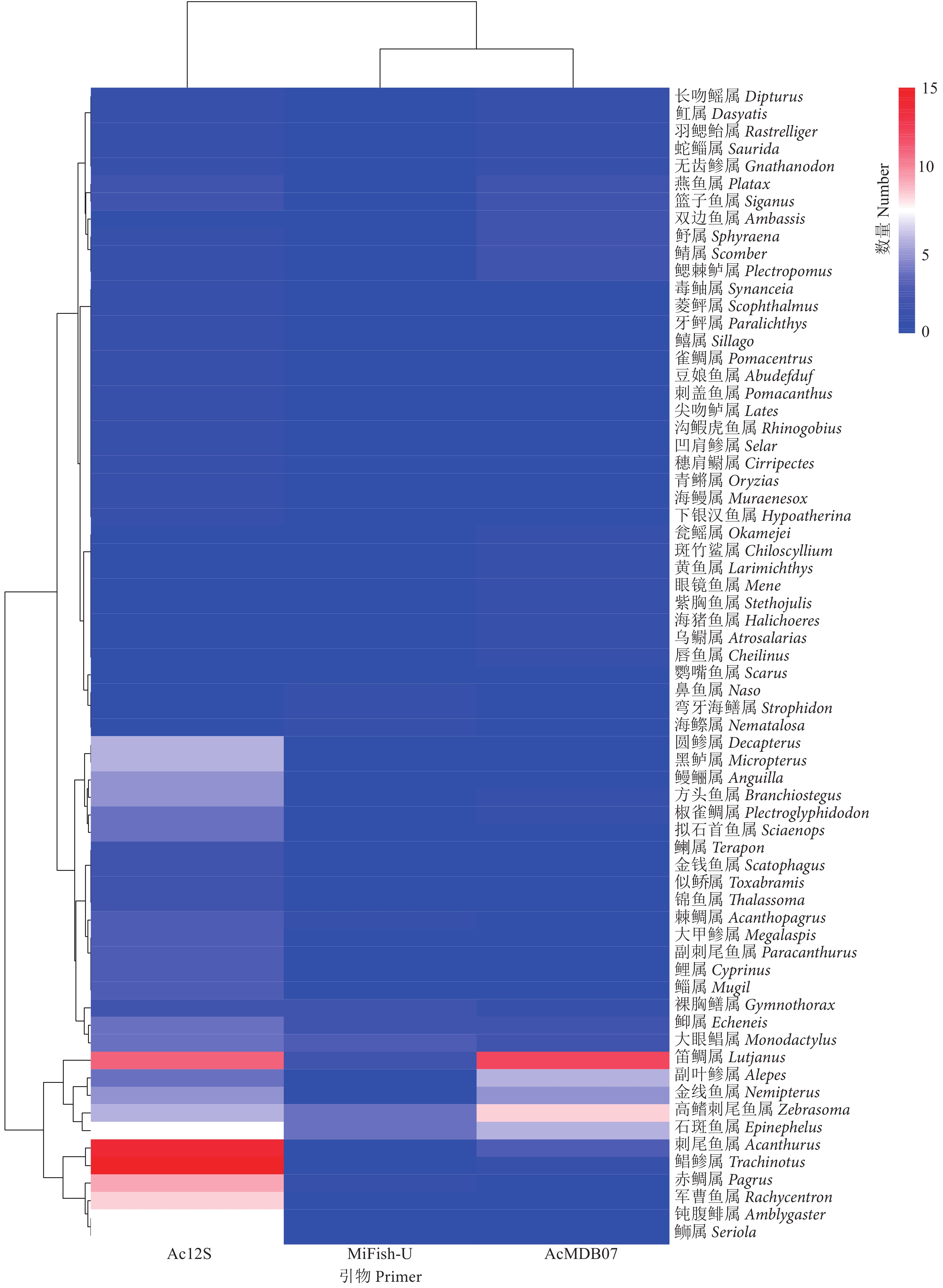
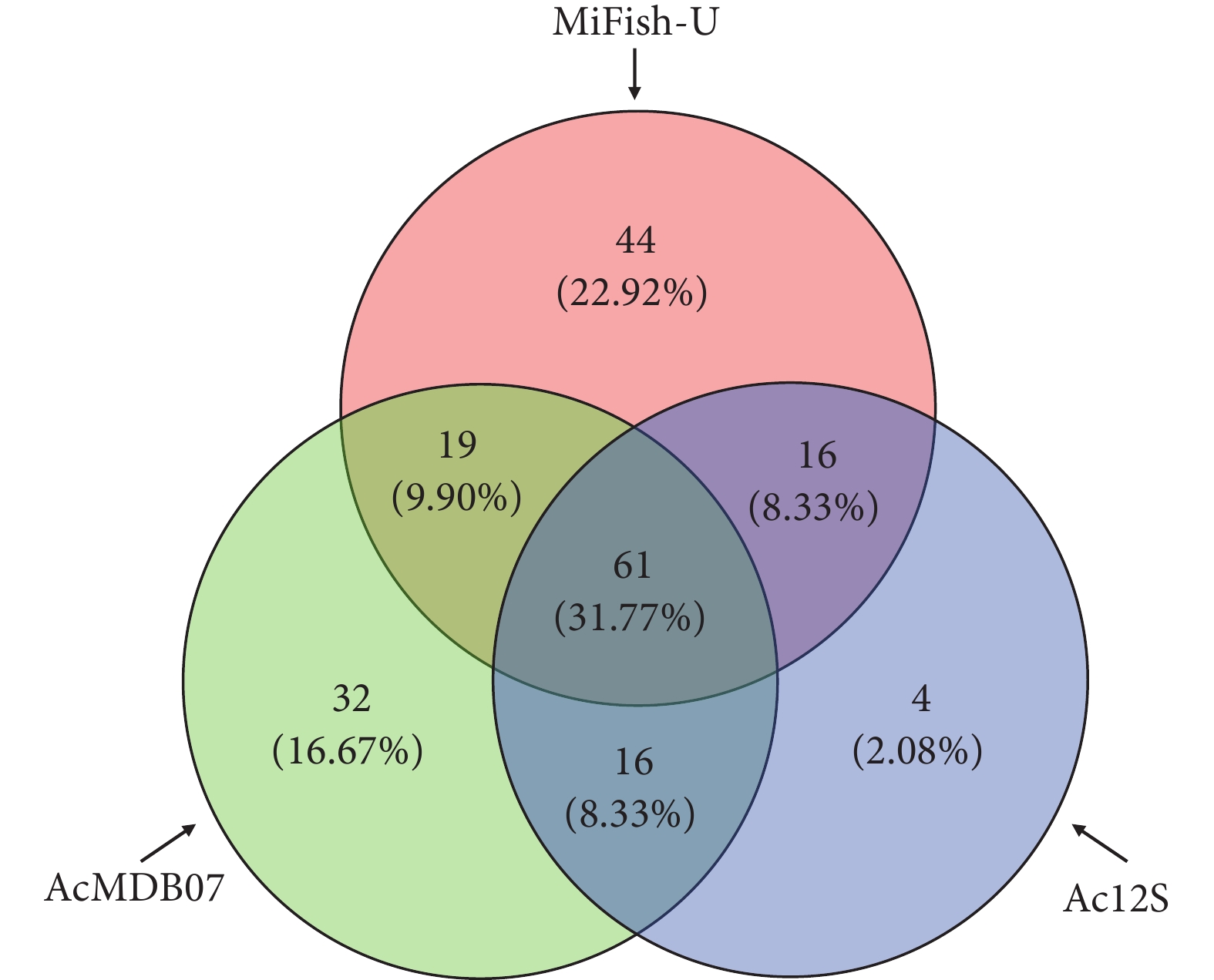
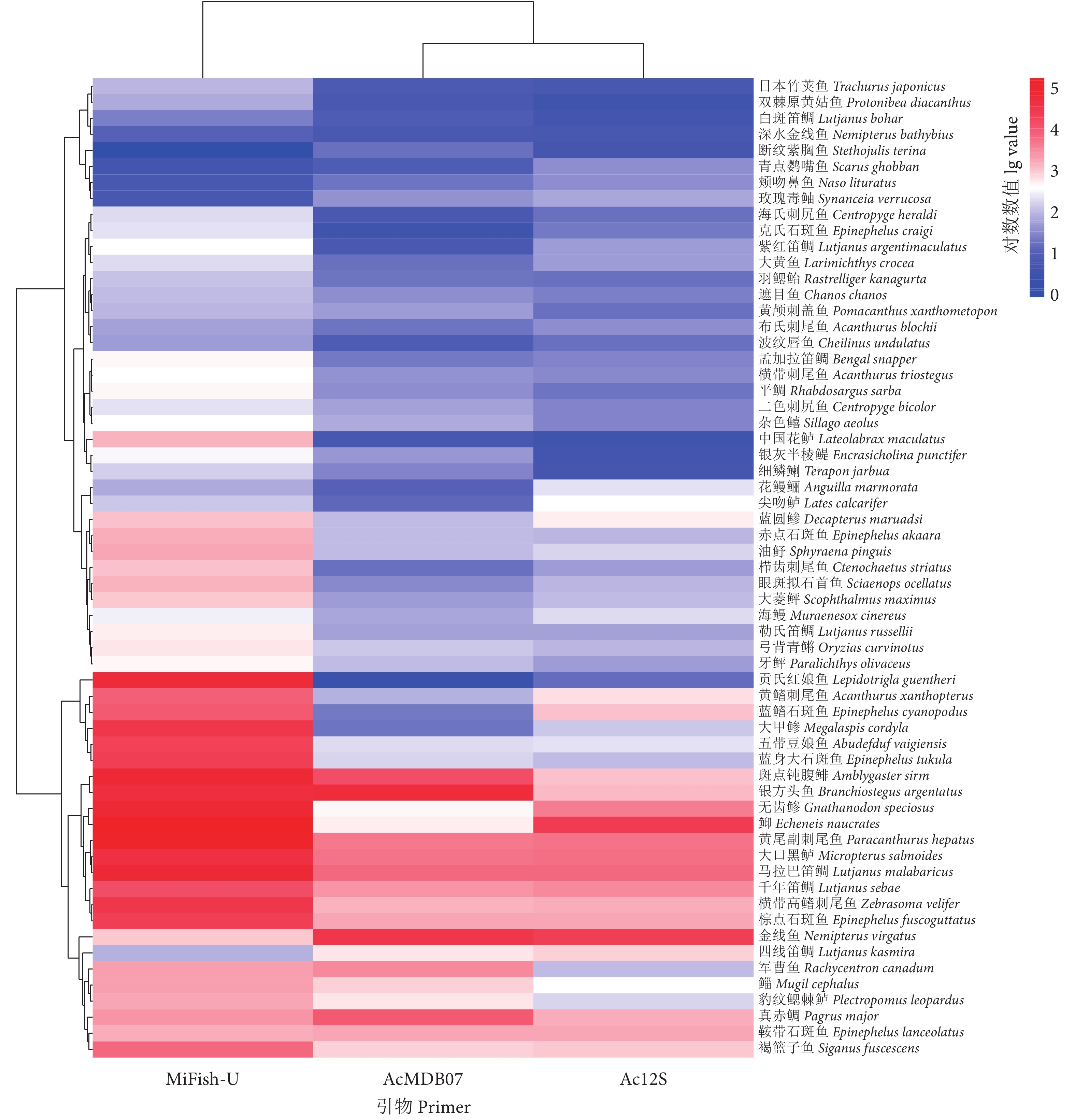
环境DNA (Environmental DNA, eDNA) 宏条形码技术是一种高效率、高灵敏度且无侵入性的物种调查工具。目前基于eDNA宏条形码技术调查鱼类多样性的研究众多,但是该技术发展尚不完善,对不同引物的实际使用效果缺乏共识。为降低测序成本、筛选出实际使用效果最优的通用引物,选取三亚市鱼市场和亚特兰蒂斯水族馆共8个站位的水样,比较了3组常用鱼类eDNA宏条形码通用引物 (MiFish-U、AcMDB07、Ac12S) 在检测鱼类多样性方面的差异。结果表明:1) 3组引物在质控后总序列数、鱼类序列数、总可操作分类单元 (Operational taxonomic units, OTUs)数、鱼类OTUs数和鱼类序列占比方面均存在极显著性差异(p<0.01),MiFish-U的扩增成功率和对鱼类物种的靶向性最高;2) MiFish-U检出的物种数最多 (140种),AcMDB07和Ac12S则分别检出128和97种;3) Ac12S和AcMDB07的参考数据库尚不完善,两者分别有72.76%和42.11%的OTUs无法注释到种水平;4) Ac12S检出的特有鱼类很少 (仅4种),暗示在实际使用过程中更容易被另外2组引物替代,而MiFish-U的可替代性最低;5) 3组引物反映鱼类丰度的总趋势相似,但对具体物种却存在一定差异。结果表明,综合OTUs注释等多种因素,特别是现有参考数据条件下,MiFish-U的物种检出效果优于AcMDB07和Ac12S。
Abstract:Environmental DNA (eDNA) metabarcoding is a high-efficiency, high-sensitivity and non-invasive species investigation tool. At present, there are many studies on the investigation of fish diversity based on eDNA metabarcoding which however is not well-developed, with a lack of consensus on the actual use of different primers. In order to reduce the cost of sequencing and screen out the universal primers with the best practical effect, we selected the water samples from eight sites in Sanya fish markets and Atlantis Aquarium, then compared the differences of three sets of universal primers (MiFish-U, AcMDB07 and Ac12S) for fish eDNA. The results show that: 1) There were significant differences in the reads number after quality control, the fish reads number, the total Operational taxonomic units (OTUs) number, the fish OTUs number and the ratio of fish reads among three sets of primers. MiFish-U had the highest amplification efficiency and targeting to fish species; 2) MiFish-U had the highest number of species (140 species), while AcMDB07 and Ac12S had 128 and 97 species, respectively; 3) The reference databases of Ac12S and AcMDB07 were not perfect, and 72.76% and 42.11% of the OTUs belonging to Ac12S and AcMDB07 could not be annotated to the species level, respectively; 4) There are very few endemic fish detected by Ac12S (Only four species), suggesting that it was easier to be replaced by the other two sets of primers in the actual use process, and MiFish-U had the lowest substitutability; 5) The three sets of primers reflected similar general trends in the fish abundance, but there were some differences in the specific species. The results show that MiFish-U is superior to AcMDB07 and Ac12S in species detection, considering various factors such as OTUs annotation, especially the conditions of existing reference data.
-
Keywords:
- eDNA metabarcoding /
- Fish species diversity /
- Universal primers /
- MiFish-U /
- Hainan
-
河流栖息地是对水生生物有直接或间接影响的多种尺度下物理化学条件的组合[1],随着人类涉水活动的增加,鱼类等水生生物的栖息地正遭受严重破坏。河流生物栖息地评估在河流生态修复中具有重要作用,通过栖息地评估可为河流生态修复提供基础信息和依据[2]。针对不同水动力条件下鱼类生境的变化,国内外学者进行了大量研究[3-5]。小尺度产卵场的地形特征是研究产卵场水动力特征的基础。河流的水动力特征与水下地形、地貌存在密切关系。特殊的河床形态决定了特殊的水力学特征,如坡度与底部流速存在正相关关系[6],这些因素决定了鱼类的产卵条件[7]。河流地形地貌在河流水量和水质不变的基础上,与生物群落的多样性存在正相关性,可影响其功能和结构[8]。河流地形地貌方面的研究集中在河道和河段尺度上,多为定性描述[9]。定量的、微尺度的高程、高程变异系数、地形复杂度、坡度、坡向等地形因子,多应用于海洋河口或中华鲟 (Acipenser sinensis) 等珍稀濒危鱼类的产卵场研究,而在“四大家鱼”产卵场方面的研究运用较少[7,10-12],珠江流域尚未见报道。
西江水系约有70多处经济鱼类产卵场,尤其以郁、黔、浔三江最为集中。东塔产卵场是珠江流域最大的产卵场,也是斑鳠 (Mystus guttatus)、卷口鱼 (Ptychidio jordani) 等珠江众多珍稀特有鱼类的重要栖息地,具有重要的生态功能 [13-14]。近年来受水利水电开发、航道整治、挖沙等涉水活动影响,鱼类栖息地遭到严重破坏,表现为生境丧失、破碎化和同质化,导致鱼类多样性降低、个体小型化,鱼类群落组成发生巨大变化[15-16]。主要渔业对象由80年代的鲥 (Tenualosa reevesii)、花鰶 (Clupanodon thrissa)、青鱼 (Mylopharyngodon piceus)、草鱼 (Ctenopharyngodon idella)、赤眼鳟 (Squaliobarbus curriculus) 等变为目前的广东鲂 (Megalobrama terminalis)、赤眼鳟、鲮 (Cirrhinus molitorella)、䱗 (Hemicculter Leucisculus),“四大家鱼”仔鱼资源在总鱼苗量中所占比例从46.6%下降至4.51%[17-18]。
大藤峡水利枢纽位于东塔产卵场上游约8 km,于2020年建成。该电站的运行,势必会改变下游江段的水下地形结构,导致鱼类栖息地的生境发生变化,从而影响其生存和繁衍[19-20]。要准确认识工程对鱼类栖息地的影响,必须对自然条件下栖息地状况,鱼类与栖息地地形、地貌间的关系有清楚而充分的认识。因此,有必要对该江段水下地形特征进行研究,评估水坝建设及运行对下游鱼类栖息地的影响。
目前,珠江流域关于鱼类微观栖息地地形、地貌的研究尚未见报道,相关研究仅限于定性描述[21],栖息地功能现状缺少科学评估。本研究首次采用声学方法对东塔产卵场栖息地现状进行评估,并构建了栖息地适宜性指数 (Habitat suitability index, HSI) 模型,以期为江河鱼类栖息地功能评价、生态修复提供技术支撑。
1. 材料与方法
1.1 研究区域
东塔产卵场位于浔江上游,自黔、郁两江汇合口起至东塔村止,长约7 km,为珠江最大“四大家鱼”产卵场。该研究区域上起大藤峡水利枢纽下游,下至石咀镇 (图1),具体地理位置为 110.057°E—110.16°E、23.432°N—23.467°N。研究区域长约14 km,水深介于1.5~32.0 m。采用ArcGIS 10.1软件绘制地图,数据来源于全国地理信息资源目录服务系统 (https://www.webmap.cn/,1∶25 万全国基础地理数据库,2019公众版) 。
1.2 数据采集
分别于2016年4月、2017年6月、2018年4月和2019年4月对该江段进行了声学走航探测。使用仪器为分裂波束渔探仪EY60 (Simrad,挪威),换能器中心频率120 kHz,发射功率200 W,−3 dB波束宽7.0°,脉冲宽度256 μs。在每次调查前,参照校准流程,使用碳化钨校准球 (Φ23 mm) 对系统收发增益进行校准[22]。调查船只使用当地渔船,长约8 m,换能器垂直向下固定于船舷,吃水约0.5 m。船速约4.4~5.5 kn,走航方向为顺流而下,设计航线为“之”字形,在实际航程中根据调查江段情况进行实时调整。
2019年4月使用声学多普勒流速剖面仪 (ADCP,WHR600,美国) 对产卵场进行流场、地形同步监测。在测量前,记录待测量区域内上下游断面中心点的坐标,以及弯道处断面中心点坐标,通过配置引导物,画出测量区域的计划线。在实际测量时,根据导航测量软件,记录、校正测船航迹,使测船沿着计划线依次测量。该次测量断面间距为150 m,共94个断面,通过ADCP实测,监测期间浔江流量约5 000 m3·s−1。
1.3 数据分析
1.3.1 地形因子提取
测量的原始数据在ADCP辅助测量软件WinRiver以ASCII码文件导出,读取每个测量断面、流速矢量、水深、经纬度等数据,使用ArcGIS 10.1 软件构建河床不规则三角网和数字高程模型,提取产卵场高程、坡度、坡向、地形起伏度、高程变异系数、地表粗糙度共6个地形因子[11]。
地形因子之间可能存在共线性关系,分析数据时,采用 Pearson相关系数判定两者之间的相关性,当两者存在共线性关系时,仅选取其中一个变量为解释变量。
1.3.2 热点分析
热点分析用于识别具有统计显著性的高值 (热点) 和低值 (冷点) 的空间聚类,是根据在一定分析规模内的所有要素,计算每个要素Getis-Ord Gi*统计值,得到每个要素的z得分和P值。统计学上的显著性正值z得分表示热点,z得分越高,表示热点聚集越紧密;负值表示冷点,z得分越低,冷点聚集就越紧密。通过z得分和P值,可以得到产卵场鱼类资源密度高值或低值在空间上发生聚类的位置[23]。
与传统的鱼类空间分布相关研究不同,本研究侧重从地理空间视角去解释鱼类的空间聚类。首先使用声学数据后处理软件Echoview 5.4 (Myriax Pty Ltd. 澳大利亚) 对原始数据进行处理,获得不同调查时间的鱼类密度信息。其次利用ArcGIS 10.1软件中的不等边三角形TIN方法建立密度数据范围,通过建立TIN能够剔除无数据的水域;然后利用空间统计分析模块中的空间自相关和热点分析对鱼类密度数据进行分析和结果可视化输出。
1.3.3 栖息地模型建立
2016、2018和2019年采样时间均为4月,2017年为6月。为降低在热点分析和模型建立中季节变化造成的影响,本文利用2016、2018和2019年数据构建模型,以2017年数据验证模型。
本文假设2年及以上鱼类热点分布重叠区域 (网格) 为鱼类适宜分布区域,提取热点区域中的地形因子。根据频率分布法,统计各因子分布范围,然后对结果进行归一化处理,估算适宜度指数值 (Suitability index, SI),出现频次最高的值赋予SI=1,SI∈[0, 1]。
由于几何平均值考虑了各因子之间的补偿影响,对生境适宜性的估计更为保守,因此HSI模型采用几何平均法建立,计算公式为[4]:
$$ S_{\mathrm{HSI},i}=\left(\mathrm{SI}_{1 i} \times \mathrm{SI}_{2 i} \times \ldots \times \mathrm{SI}_{n i}\right)^{{}^{1} \diagup {}_{n}\;} $$ (1) 式中:
$S_{\mathrm{HSI},i}$ 为第i个网格的栖息地适宜性指数值;i为研究区域网格序号;${\mathrm{S}\mathrm{I}}_{{n}{i}}$ 为第n个地形因子第i个网格的SI值。1.4 HIS模型验证
利用2017年6月的声学调查数据及水深数据,分析该江段鱼类资源密度分布情况,验证是否HIS值越高的网格鱼类密度越高。
2. 结果
2.1 鱼类密度及空间热冷点分布格局
2016—2019年鱼类平均密度见表1,鱼类密度呈逐年下降趋势,使用非参数检验进行分析,Kurskal-Wallis结果显示,不同调查时间鱼类密度存在显著性差异 (P<0.05)。 从偏度值可以看出数据左偏,表明低密度单元格较多。
表 1 东塔产卵场不同调查时间鱼类密度分布信息Table 1. Fish density distribution in Dongta spawning ground in different periods调查时间
Survey year鱼类密度
Fish density/(尾·m−3)范围
Range偏度
Skewness峰度
Kurtosis变异系数
Coefficient of variance2016年 0.057 4±0.128 7 (0.000 3, 1.018 4) 6.146 41.269 2.24 2017年 0.043 4±0.056 5 (0.007 2, 0.405 3) 3.744 17.185 1.30 2018年 0.031 6±0.037 7 (0.000 4, 0.196 7) 2.004 4.265 1.19 2019年 0.009 4±0.007 1 (0.000 4, 0.035 6) 1.492 2.536 0.75 将鱼类密度导入GIS中进行空间自相关和热点分析,得到鱼类在东塔产卵场的热点分布图 (图2)。空间上,研究区域鱼类资源的空间分布具有一定的聚集特征,即不同年份均出现“热点区域”(红色区域),标志为z>+1.65 (P<0.1),如东塔产卵场部分区域3年结果均为“热点区域”;而大藤峡下游及石咀段出现“冷点区域”(蓝色区域);其他江段绝大部分z值介于−1.65~+1.65,这些区域内渔业资源密度高值和低值之间的空间自相关性弱,为随机性分布。总体来看,东塔产卵场部分区域局部自相关性强,整体自相关性表现并不强烈,鱼类资源密度呈现“热”“冷”不均的局部性分布特征。
2.2 适宜度曲线构建
根据上述方法,从鱼类“热点分布”区域中选取2年及以上重合区域网格,共80个,提取各网格地形因子,利用频度分布,拟合建立地形因子的单因子适宜度曲线。
本文共获取了6个解释变量,包括高程、坡度、坡向、地形起伏度、高程变异系数、地表粗糙度。高程变异系数、地形起伏度和坡度的相关系数大于0.85,因高程变异系数和地形起伏度是基于高程 (水深) 计算得出的,因此只保留坡度进行分析;地表粗糙度数据主要集中在1左右,无法拟合。因此选择水深、坡度、坡向3个地形因子作为预测变量参与分析。由图3可见,热点区域水深介于1.5~25.0 m,鱼类适宜水深为5.0~8.0 m;坡度介于0°~27°,鱼类适宜范围介于1°~1.5°,依据坡度等级,鱼类适宜区域为微斜坡;鱼类适宜的坡向介于90°~180°,即东南方向。
2.3 HSI值计算
根据公式 (1) 计算得出鱼类密度的HSI值。5 000 m3·s−1流量下HSI值分布介于0.02~0.95,平均值为0.31±0.22。其中,HSI值大于0.5的区域占总数的26.67%。由图4可知,研究江段HSI最大值出现在产卵场沙洲两侧,河流两侧也有零星分布;另外,主航道基本上为HSI低值区域。
2.4 栖息地模型验证
使用2017年6月声学数据对HSI模型进行验证。通过Pearson相关性分析可知,鱼类密度与HSI值呈正相关关系 (R=0.794, P=0.011)。拟合结果显示,HSI与鱼类密度的关系为:
$ y=0.02x+ 0.04 $ (R2=0.63, RMSE=0.005),符合本文假设。3. 讨论
3.1 地形因子的选择性特征
东塔产卵场位于郁江、黔江和浔江三江交汇口,鱼类种类丰富,物种多样性高。文献资料显示,该江段鱼类优势种为䱗、鲤 (Cyprinus carpio)、鲮、赤眼鳟、广东鲂、卷口鱼等[15-16]。河流物种多样性和河床特征存在着一定程度的响应关系,同时生物群落的结构和功能受之影响。然而,目前尚不清楚地形因子如何影响鱼类[24]。本研究提取了水深、坡度、坡向、地形粗糙度、高程变异系数、曲率等地形因子进行分析,通过判定各因子之间的相关性及各因子取值的分布情况,筛选出了水深、坡度、坡向作为构建适宜性模型的因子。
水深是鱼类生活空间的反映,鱼的体型大小、生活习性等决定了鱼类对水深的需求[25]。Miranda和Killgore [26]研究表明,鱼类分布与水深呈非随机关系,约75%的物种分布在12 m以浅。Sheldon[27]认为鱼类分布与水深有着紧密联系,随季节和种类的不同而变化。本研究区域最大水深为31.0 m,鱼类适宜水深介于5.0~8.0 m。从图1可以看出,深水区主要为航道,航道在扩能升级过程中改变了河床结构,破坏了鱼类栖息地;另外,鱼类会对航道中的行船噪声产生回避行为[28-29],因此深水区基本为HSI低值区。
水下地形结构影响着鱼类的分布和多样性[24]。相关研究表明,疏浚通过改变水下地形,显著影响鱼类群落及空间分布。鱼类丰度和多样性从与自然河口陡峭的坡度相关,转变为与疏浚河口平缓的坡度相关[12]。西江航道经过二次扩能升级,河流水下地形地貌已发生了变化[30],因此鱼类偏好栖息于坡度较缓的区域[31]。
坡向是坡度所面对的方向,用于识别表面上某一位置处的最陡下坡方向。相关研究表明,中华鲟产卵场平均坡向分别为219.92°和207.63°,长江四大家鱼产卵场平均坡向介于150.16°~200.6°[10-11],本研究中栖息地坡向适宜范围介于90°~180°。上述产卵场坡向分布范围不同,但根据河流地形走势来看,坡向均为水流方向;因此推测鱼类偏好栖息于河流中向着水流方向的微斜坡。
3.2 栖息地适宜度
HSI模型是通过适宜度方程评价各个生态因子对某一特定物种的适宜度值和综合值,以此来评价环境对这个特定物种生存和繁殖的适合程度。以往研究使用的生物因子来源于专家经验或者室内实验[3]。近年来,相关研究越来越重视探讨在自然状态下鱼类分布与生境因子之间的关系,以此来确定鱼类适宜栖息地范围[31]。本研究根据鱼类对生境的利用情况来绘制适宜度曲线,鱼类时空分布及水下地形数据均为实测,地形因子的频率分布是基于3年声学调查结果,利用局部空间自相关方法对鱼类空间分布进行分析,定位到鱼类分布的高密度区域,能较为准确地反映野外实际情况。因此栖息地适宜性指数具有较高的可靠度。另外,模型验证结果显示,HSI值较高的区域,鱼类密度较大,表明该模型适用于东塔江段鱼类栖息地适宜性评价。
在数据分析中,经常会遇到少量的鱼类高密度值,这些值是研究者关注的热点;但从经典统计学角度讲,这些值却是造成评估不确定性的原因[32]。鱼类以个体、种群、群落的形式分布在特定空间上,具有高度的空间自相关性。经典统计学受基本假设的限制,在研究个体、种群和群落空间自相关方面具有较多缺陷[23]。热点分析可划分鱼类资源高低属性值的空间聚集,能够直观清晰地表现渔业资源分布情况,结果具有统计学意义。在渔业生态学中,热点分析已应用于栖息地变动趋势、物种丰富度等方面[33-35]。本研究尝试应用该法来识别鱼类适宜栖息地,取得了较好的效果。由于能够较为直接清晰地表现渔业资源分布情况,热点分析的应用前景将会被不断拓展。
3.3 研究不足及未来展望
本研究以河流地形特征作为变量研究鱼类栖息地适宜情况。坡度、坡向为常量,不会随流量的变化而改变。水深作为表征高程的因子,会随径流量的变化而增减。随着流量增大,水深加深,河流两岸漫滩及中心沙洲会被淹没,适宜鱼类栖息的区域也会发生改变。但是本研究区域边界是基于5 000 m3·s−1流量建立的,河流边界无法随流量的增加而变化。因此本文所建模型无法模拟高流量下的河流栖息地状况。
未来在开展此类工作时,野外采样应选择在水位较高时,尽可能将河流两岸漫滩及中心沙洲淹没。漫滩作为河流生态系统的重要组成部分,为鱼类提供了多样化的栖息生境,具有高生产力和微生境异质性,对维持区域鱼类生物多样性具有重要作用[36-37]。本研究探明了大藤峡水利枢纽蓄水前东塔产卵场水下地形特征及鱼类时空分布,今后仍需持续监测,以评估该电站运行对坝下江段鱼类栖息地的影响。
-
表 1 本研究所用引物基本信息
Table 1 Primer pairs information used in this study
引物名称
Primer name所在基因
Target gene目标类群
Target group碱基组成 (5'—3')
Base composition (5'−3')扩增片段长度
Amplicon length/bp退火温度
Tm/℃参考文献
ReferenceMiFish-U 12S rRNA 硬骨鱼纲 F: GTCGGTAAAACTCGTGCCAGC
R: CATAGTGGGGTATCTAATCCCAGTTTG~170 60.8 [18] AcMDB07 12S rRNA 辐鳍亚纲 F: GCCTATATACCGCCGTCG
R: GTACACTTACCATGTTACGACTT~280 52.4 [19] Ac12S 12S rRNA 辐鳍亚纲 F: ACTGGGATTAGATACCCCACTATG
R: GAGAGTGACGGGCGGTGT~390 57.3 [20] 表 2 高通量测序基本结果
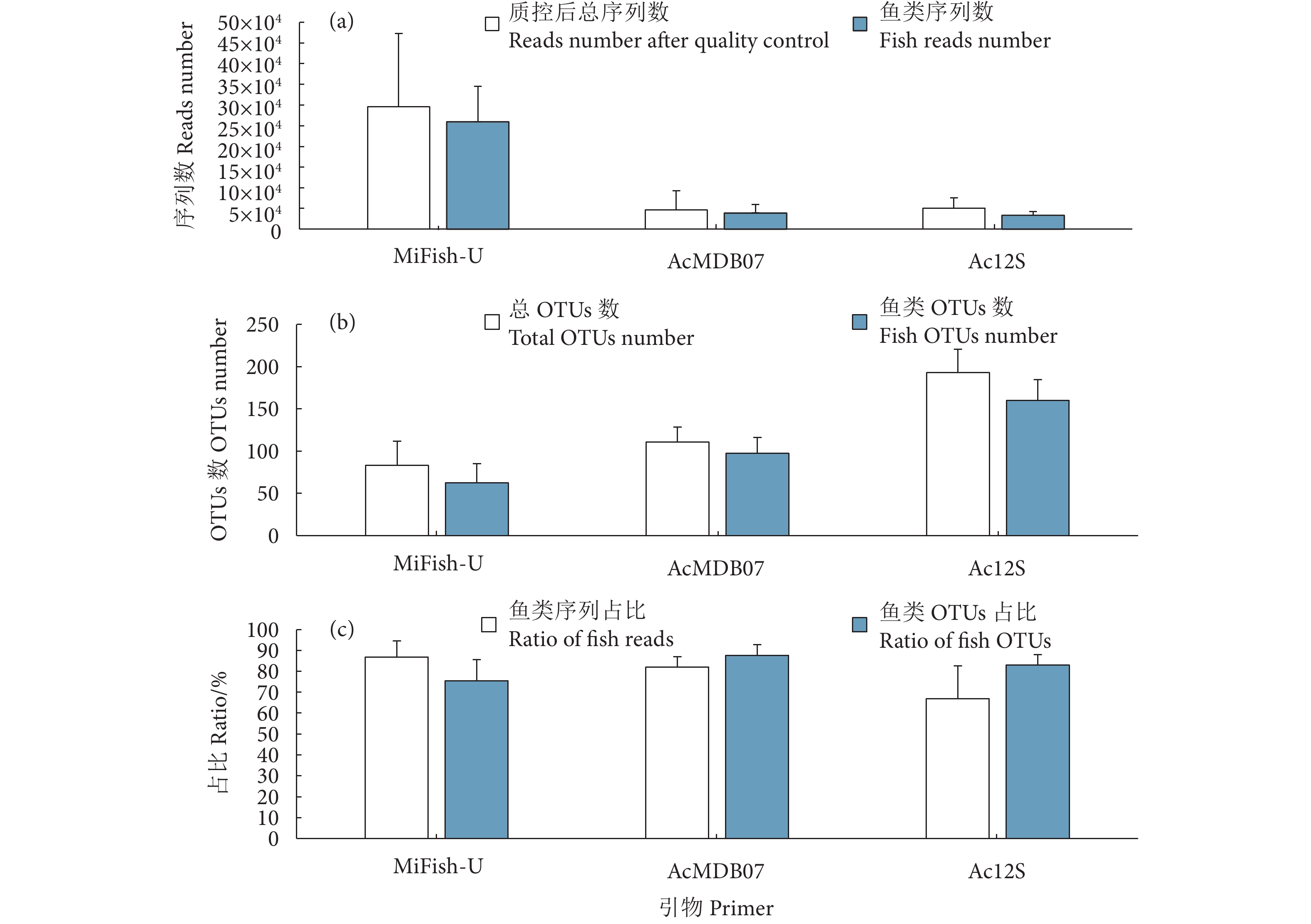
Table 2 Basic results of high-throughput sequencing
样品名称
Sample name原始序列数
Raw reads
number高质量
序列数
Clean reads
number质控后
总序列数
Reads number
after quality
control鱼类
序列数
Fish reads
number总OTUs数
Total OTUs
number鱼类OTUs数
Fish OTUs
number鱼类序列占比
Ratio of fish
reads/%鱼类OTUs占比
Ratio of fish
OTUs/%MiFish-U-AQA 718 262 717 186 345 761 326 679 96 57 94.48 59.38 MiFish-U-AQB 808 828 807 368 385 564 327 617 128 94 84.97 73.44 MiFish-U-AQC 761 826 760 700 366 271 326 214 117 102 89.06 87.18 MiFish-U-AQD 508 982 507 958 246 848 243 533 48 42 98.66 87.50 MiFish-U-FMA 794 224 793 250 382 968 343 957 77 56 89.81 72.73 MiFish-U-FMB 497 380 496 790 238 992 200 604 77 55 83.94 71.43 MiFish-U-FMC 518 074 517 332 245 917 191 987 70 47 78.07 67.14 MiFish-U-FMD 323 782 323 306 152 911 115 375 53 45 75.45 84.91 AcMDB07-AQA 132 732 132 320 51 399 43 648 144 135 84.92 93.57 AcMDB07-AQB 89 570 89 342 33 227 26 141 120 104 78.67 86.67 AcMDB07-AQC 69 200 69 038 26 385 20 958 110 95 79.43 86.36 AcMDB07-AQD 219 354 218 704 91 112 81 552 97 80 89.51 82.47 AcMDB07-FMA 112 654 112 276 42 494 33 249 107 98 78.24 91.59 AcMDB07-FMB 94 404 94 052 34 905 26 195 88 83 75.05 94.32 AcMDB07-FMC 101 346 101 100 40 281 34 569 97 77 85.82 79.38 AcMDB07-FMD 139 406 139 040 54 770 46 438 123 106 84.79 86.18 Ac12S-AQA 235 740 234 438 53 234 43 769 244 205 82.22 84.02 Ac12S-AQB 263 654 262 290 57 228 36 721 203 168 64.17 82.76 Ac12S-AQC 199 250 198 292 59 661 18 762 181 138 31.45 76.24 Ac12S-AQD 240 852 239 524 62 456 42 393 179 152 67.88 84.92 Ac12S-FMA 214 932 213 912 42 855 29 857 219 179 69.67 81.74 Ac12S-FMB 195 986 195 030 38 550 26 058 180 166 67.60 92.22 Ac12S-FMC 192 132 191 200 43 625 29 646 158 134 67.96 84.81 Ac12S-FMD 217 252 216 144 49 461 41 178 178 137 83.25 76.97 表 4 鱼类OTUs参考序列完备度
Table 4 Reference sequence coverage of fish OTUs
序列相似度
Sequence identificationOTUs数 OTUs number MiFish-U AcMDB07 Ac12S 相似度Identification=100% 274 221 186 99%≤相似度Identification<100% 68 98 115 98%≤相似度Identification<99% 12 48 87 97%≤相似度Identification<98% 10 27 103 相似度Identification<97% 14 157 614 -
[1] SALA E, MAYORGA J, BRADLEY D, et al. Protecting the global ocean for biodiversity, food and climate[J]. Nature, 2021, 592(7854): 397-402. doi: 10.1038/s41586-021-03371-z
[2] ALMOND R E A, GROOTEN M, JUFFE B D, et al. Living planet report 2022: building a nature-positive society[M]. Gland: World Wide Fund for Nature, 2022: 32-34.
[3] DÍAZ-FERGUSON E E, MOYER G R. History, applications, methodological issues and perspectives for the use environmental DNA (eDNA) in marine and freshwater environments[J]. Rev Biol Trop, 2014, 62(4): 1273-1284. doi: 10.15517/rbt.v62i4.13231
[4] THOMSEN P F, KIELGAST J, IVERSEN L L, et al. Monitoring endangered freshwater biodiversity using environmental DNA[J]. Mol Ecol, 2012, 21(11): 2565-2573. doi: 10.1111/j.1365-294X.2011.05418.x
[5] ROURKE M L, FOWLER A M, HUGHES J M, et al. Environmental DNA (eDNA) as a tool for assessing fish biomass: a review of approaches and future considerations for resource surveys[J]. Environ DNA, 2022, 4(1): 9-33. doi: 10.1002/edn3.185
[6] 言柯程, 李建超, 田永军, 等. 基于环境DNA metabarcoding和底拖网调查的南黄海西部鱼类多样性比较[J]. 中国海洋大学学报 (自然科学版), 2023, 53(5): 71-81. [7] RUPPERT K M, KLINE R J, PAST M S R. Present, and future perspectives of environmental DNA (eDNA) metabarcoding: a systematic review in methods, monitoring, and applications of global eDNA[J]. Glob Ecol Conserv, 2019, 17: e00547.
[8] 赵娜, 杨刚, 吴祖立, 等. 环境DNA技术发展及其在长江流域水生生态学领域的应用研究进展[J]. 海洋渔业, 2024, 46(1): 119-128. doi: 10.3969/j.issn.1004-2490.2024.01.013 [9] 张方圆, 王汝贤, 杨刚, 等. 环境DNA技术在长江口中华绒螯蟹亲蟹资源监测中的应用[J]. 水生生物学报, 2024, 48(6): 950-957. doi: 10.7541/2024.2023.0337 [10] THOMSEN P F, WILLERSLEV E. Environmental DNA-an emerging tool in conservation for monitoring past and present biodiversity[J]. Biol Conserv, 2015, 183: 4-18. doi: 10.1016/j.biocon.2014.11.019
[11] BENG K C, CORLETT R T. Applications of environmental DNA (eDNA) in ecology and conservation: opportunities, challenges and prospects[J]. Biodivers Conserv, 2020, 29(7): 2089-2121. doi: 10.1007/s10531-020-01980-0
[12] 仝亚东, 匡箴, 刘鹏飞, 等. 基于环境DNA技术的东平湖鱼类多样性研究[J]. 中国水产科学, 2023, 30(12): 1530-1542. doi: 10.12264/JFSC2023-0265 [13] 李晨虹, 凌岚馨, 谭娟, 等. 环境DNA技术在水生生物监测中的挑战、突破和发展前景[J]. 上海海洋大学学报, 2023, 32(3): 564-574. doi: 10.12024/jsou.20221104019 [14] ZHANG S, ZHAO J D, YAO M. A comprehensive and comparative evaluation of primers for metabarcoding eDNA from fish[J]. Methods Ecol Evol, 2020, 11(12): 1609-1625. doi: 10.1111/2041-210X.13485
[15] 舒璐. 基于环境DNA技术的鱼类多样性评估: 胚胎发育动力学、引物评价及洱海鱼类多样性监测[D]. 重庆: 西南大学, 2022: 37-60. [16] 吕宏森, 王安香, 董智玲, 等. 长江上游鱼类环境DNA通用引物的选择与验证[J]. 水产学报, 2024, 48(6): 72-84. [17] 周严, 童璐, 胡文静, 等. 淡水鱼类环境DNA宏条形码引物的筛选及其在千岛湖的应用[J]. 湖泊科学, 2024, 36(1): 187-199. doi: 10.18307/2024.0131 [18] MIYA M, SATO Y, FUKUNAGA T, et al. MiFish, a set of universal PCR primers for metabarcoding environmental DNA from fishes: detection of more than 230 subtropical marine species[J]. R Soc Open Sci, 2015, 2(7): 150088. doi: 10.1098/rsos.150088
[19] BYLEMANS J, GLEESON D M, HARDY C M, et al. Toward an ecoregion scale evaluation of eDNA metabarcoding primers: a case study for the freshwater fish biodiversity of the Murray-Darling Basin (Australia)[J]. Ecol Evol, 2018, 8(17): 8697-8712. doi: 10.1002/ece3.4387
[20] EVANS N T, OLDS B P, RENSHAW M A, et al. Quantification of mesocosm fish and amphibian species diversity via environmental DNA metabarcoding[J]. Mol Ecol Resour, 2016, 16(1): 29-41. doi: 10.1111/1755-0998.12433
[21] VALENTINI A, TABERLET P, MIAUD C, et al. Next-generation monitoring of aquatic biodiversity using environmental DNA metabarcoding[J]. Mol Ecol, 2016, 25(4): 929-942. doi: 10.1111/mec.13428
[22] MILAN D T, MENDES I S, DAMASCENO J S, et al. New 12S metabarcoding primers for enhanced neotropical freshwater fish biodiversity assessment[J]. Sci Rep, 2020, 10(1): 17966. doi: 10.1038/s41598-020-74902-3
[23] TABERLET P, BONIN A, ZINGER L, et al. Environmental DNA-for biodiversity research and monitoring[M]. Oxford: Oxford University Press, 2018: 206.
[24] WILCOX T M, MCKELVEY K S, YOUNG M K, et al. Environmental DNA particle size distribution from brook trout (Salvelinus fontinalis)[J]. Conserv Genet Resour, 2015, 7: 639-641. doi: 10.1007/s12686-015-0465-z
[25] JO T, ARIMOTO M, MURAKAMI H, et al. Particle size distribution of environmental DNA from the nuclei of marine fish[J]. Environ Sci Technol, 2019, 53(16): 9947-9956. doi: 10.1021/acs.est.9b02833
[26] JOSEPH C, FAIQ M E, LI Z, et al. Persistence and degradation dynamics of eDNA affected by environmental factors in aquatic ecosystems[J]. Hydrobiologia, 2022, 849(19): 4119-4133. doi: 10.1007/s10750-022-04959-w
[27] WOOD S A, BIESSY L, LATCHFORD J L, et al. Release and degradation of environmental DNA and RNA in a marine system[J]. Sci Total Environ, 2020, 704: 135314. doi: 10.1016/j.scitotenv.2019.135314
[28] 杨泰昌, 李嘉华, 张颖, 等. 香港瘰螈eDNA引物和TaqMan探针的设计与确认[J]. 动物学杂志, 2020, 55(5): 624-636. [29] 闫卉果, 董智玲, 马婷婷, 等. 基于环境DNA的岩原鲤检测及生物量评估[J]. 水产学报, 2022, 46(6): 1018-1026. [30] GWAK W S, NAKAYAMA K. Development of a MGB probe based qPCR protocol for detecting Pacific cod Gadus macrocephalus in eDNA samples[J]. Conserv Genet Resour, 2023, 15(4): 175-177. doi: 10.1007/s12686-023-01320-8
[31] ZHU T, IWASAKI W. MultiBarcodeTools: easy selection of optimal primers for eDNA multi-metabarcoding[J]. Environ DNA, 2023, 5(6): 1793-1808. doi: 10.1002/edn3.499
[32] 蒋佩文, 李敏, 张帅, 等. 基于线粒体COI和12S rDNA基因构建珠江河口鱼类DNA宏条形码数据库[J]. 南方水产科学, 2022, 18(3): 13-21. [33] BYLEMANS J. Monitoring freshwater fish communities with environmental DNA (eDNA) metabarcoding[D]. Canberra: University of Canberra, 2018: 201-204.
[34] 陈治, 马春来, 叶乐, 等. 鱼类环境DNA metabarcoding片段的近缘物种识别差异[J]. 海洋学报, 2022, 44(8): 51-65. doi: 10.12284/j.issn.0253-4193.2022.8.hyxb202208006 [35] MARQUES V, MILHAU T, ALBOUY C, et al. GAPeDNA: assessing and map global species gaps in genetic databases for eDNA metabarcoding[J]. Divers Distrib, 2021, 27(10): 1880-1892. doi: 10.1111/ddi.13142
[36] NELSON J S, GRANDE T C, WILSON M V H. Fishes of the world[M]. New York: John Wiley & Sons, 2016: 1-5.
[37] 王月. 赤水河鱼类环境DNA宏条形码参考数据库的构建及应用[D]. 大连: 大连海洋大学, 2022: 25-39. [38] 邓升铭. 我国南海鱼类的分子参考数据库的建立及宏条形码引物的开发及利用[D]. 海口: 海南大学, 2023: 15-19. [39] LIM S J, THOMPSON L R. Mitohelper: a mitochondrial reference sequence analysis tool for fish eDNA studies[J]. Environ DNA, 2021, 3(4): 706-715. doi: 10.1002/edn3.187
[40] MIYA M, GOTOH R O, SADO T. MiFish metabarcoding: a high-throughput approach for simultaneous detection of multiple fish species from environmental DNA and other samples[J]. Fish Sci, 2020, 86(6): 939-970. doi: 10.1007/s12562-020-01461-x
[41] COUTON M, LÉVÊQUE L, DAGUIN-THIÉBAUT C, et al. Water eDNA metabarcoding is effective in detecting non-native species in marinas, but detection errors still hinder its use for passive monitoring[J]. Biofouling, 2022, 38(4): 367-383. doi: 10.1080/08927014.2022.2075739
[42] 李诣远, DAVID C M, MICHAEL E P. 基于Nextflow构建的宏条形码自动化分析流程EPPS[J]. 生物多样性, 2019, 27: 567-575. doi: 10.17520/biods.2018211 [43] FURLAN E M, DAVIS J, DUNCAN R P. Identifying error and accurately interpreting environmental DNA metabarcoding results: a case study to detect vertebrates at arid zone waterholes[J]. Mol Ecol Resour, 2020, 20(5): 1259-1276. doi: 10.1111/1755-0998.13170
[44] BURIAN A, MAUVISSEAU Q, BULLING M, et al. Improving the reliability of eDNA data interpretation[J]. Mol Ecol Resour, 2021, 21(5): 1422-1433. doi: 10.1111/1755-0998.13367
[45] 刘山林, 邱娜, 张纾意, 等. 基因组学技术在生物多样性保护研究中的应用[J]. 生物多样性, 2022, 30(10): 334-354. doi: 10.17520/biods.2022441 [46] 杨海乐, 吴金明, 张辉, 等. 大型河流中鱼类组成的eDNA监测效率: 以长江武汉江段为例[J]. 中国水产科学, 2021, 28(6): 796-807. doi: 10.12264/JFSC2021-0556 [47] KUMAR G, REAUME A M, FARRELL E, et al. Comparing eDNA metabarcoding primers for assessing fish communities in a biodiverse estuary[J]. PLoS One, 2022, 17(6): e026672.
-
期刊类型引用(9)
1. 傅建军,安睿,朱文彬,王兰梅,罗明坤,董在杰. 套养池塘主要养殖阶段浮游生物的群落结构特征. 水产学杂志. 2024(01): 104-112 .  百度学术
百度学术
2. 向劲,吴启藩,宋锐,彭治桃,李金龙,高峰,谢敏,程小飞. 循环水养殖池塘中浮游动植物功能群特征及水质评价. 水产学杂志. 2024(01): 81-89 .  百度学术
百度学术
3. 闵文武,王龙燕,陈飞雄,周其椿. 集装箱养殖生态净化池塘中浮游植物群落结构多样性分析. 水产科技情报. 2024(05): 302-310 .  百度学术
百度学术
4. 郑颖强,代梨梨,张辉,彭亮,陶玲,李谷,柴毅,廖咏玲. 池塘循环水养殖系统净化单元对水体浮游植物的影响. 中国水产科学. 2024(12): 1497-1510 .  百度学术
百度学术
5. 王小冬,车轩,刘兴国,顾兆俊,丁艳青,陈晓龙. 曝气扰动促进高营养的饲料腐烂液中绿藻生长. 上海海洋大学学报. 2023(01): 142-149 .  百度学术
百度学术
6. 闵文武,王龙燕,陈飞雄,周其椿,赵瑞平. 生态净化池塘浮游动物群落结构特征及其与环境因子的关系. 贵州农业科学. 2023(02): 66-75 .  百度学术
百度学术
7. 赵秀侠,方婷,陈金良,高娜,陈诚,卢文轩. 陆基推水集装箱循环水养殖系统中浮游植物群落结构特征. 农业环境科学学报. 2023(04): 869-878 .  百度学术
百度学术
8. 王龙燕,闵文武,王金乐,陈飞雄,周其椿,吴俣学. 集装箱循环水养殖系统中浮游植物群落结构特征及影响因素. 渔业现代化. 2022(04): 26-36 .  百度学术
百度学术
9. 赵宇曦,刘兴国,周润锋,肖述文,孙照云. 池塘多营养级养殖水体的初级生产力及影响因子分析. 渔业现代化. 2022(06): 91-99 .  百度学术
百度学术
其他类型引用(4)
-
其他相关附件
-
PDF格式
20240216附录A 点击下载(223KB)
-




 下载:
下载:







 粤公网安备 44010502001741号
粤公网安备 44010502001741号
