Improvement of gel strength of fermented tilapia surimi by Lactiplantibacillus plantarum through inhibition of protein hydrolysis
-
摘要:
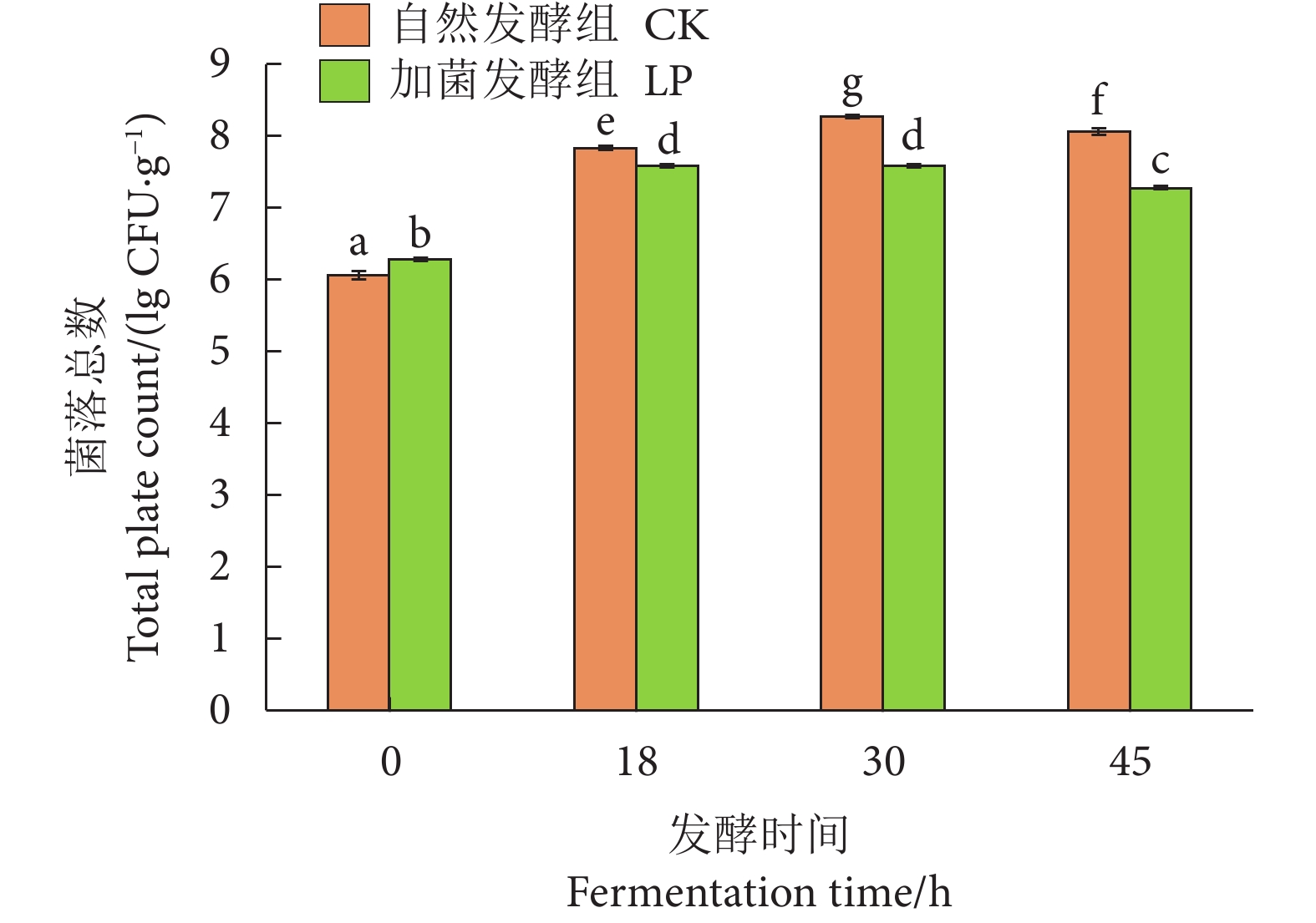
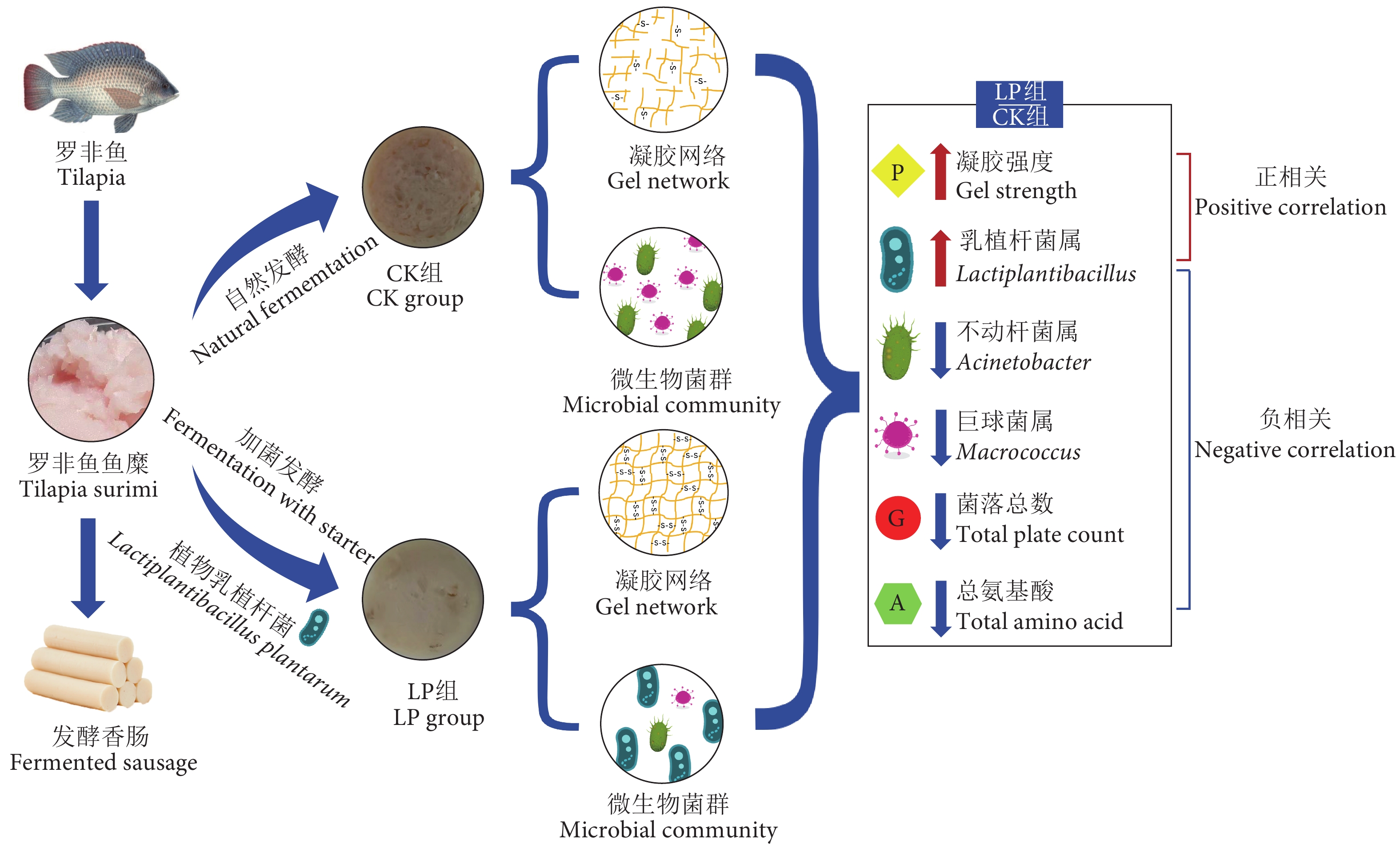
利用微生物发酵能有效改善淡水鱼鱼糜的凝胶强度。罗非鱼鱼糜的凝胶强度低且在加工过程中易出现凝胶劣化。为开发高品质的罗非鱼发酵鱼糜制品,以植物乳植杆菌 (Lactiplantibacillus plantarum) 为发酵菌株,探究了加菌发酵对罗非鱼发酵鱼糜凝胶强度的影响。结果表明,加菌发酵能够显著改善罗非鱼发酵鱼糜的凝胶强度,降低大部分游离氨基酸以及总氨基酸的含量。采用16S rRNA高通量测序技术研究加菌发酵对罗非鱼发酵鱼糜中微生物菌群的影响,结果显示,加菌发酵鱼糜中乳植杆菌属一直为优势菌属,到发酵末期增至63.71%,而其他腐败微生物菌属的丰度显著下降,同时菌落总数明显低于自然发酵鱼糜。构建了不同发酵时期的相关性网络图,结果表明在整个发酵期凝胶强度与菌落总数和总氨基酸含量呈显著负相关,仅乳植杆菌属与菌落总数和总氨基酸含量呈显著负相关,而与凝胶强度呈显著正相关。研究表明,植物乳植杆菌可以通过抑制腐败微生物的生长以及对蛋白质的水解作用来改善罗非鱼发酵鱼糜的凝胶强度。植物乳植杆菌可以作为专用发酵剂开发高品质罗非鱼鱼糜发酵制品。
Abstract:Microbial fermentation can improve the gel strength of freshwater fish surimi effectively. Tilapia surimi has low gel strength and is prone to gel deterioration during processing. In order to develop high-quality fermented surimi products from tilapia, we used Lactiplantibacillus plantarum as a fermentation strain to explore the effect of strain addition on the gel strength of fermented surimi. After adding fermentation strain, the gel strength of fermented surimi was significantly improved, while the contents of most free amino acids and total amino acid significantly decreased. The effect of strain addition on the microbial community in the fermented surimi was studied by 16S rRNA gene high-throughput sequencing. During fermentation with strain, Lactiplantibacillus remained to be the dominant genus, increasing to 63.71% at the end of fermentation, while the abundance of other spoilage microorganisms was significantly inhibited. Besides, the total plate count in the fermented surimi was significantly lower than that in the naturally fermented surimi. The correlation network maps at different fermentation stages show that the gel strength was significantly negatively correlated with the total plate count and total amino acid content during the whole fermentation period. Only Lactiplantibacillus was significantly negatively correlated with the total plate count and total amino acid content but was positively correlated with gel strength. The results indicate that the improved gel strength mainly results from the inhibited growth of spoilage microorganisms, and hydrolysis of surimi proteins by L. plantarum which can be used as a special starter to develop the fermented tilapia surimi products with high quality.
-
Keywords:
- Tilapia /
- Fermented surimi /
- Lactiplantibacillus plantarum /
- Gel strength /
- Correlation network map
-
原产澳大利亚的线纹尖塘鳢(Oxyeleotris lineolatus)与原产东南亚国家的云斑尖塘鳢(O.marmoratus)在我国均俗称笋壳鱼,为虾虎鱼亚目(Gobioidei)中体型最大、极具养殖价值的淡水经济鱼类。云斑尖塘鳢养殖始于20世纪70年代[1-4],线纹尖塘鳢则是近10年才开展[5]。但人工养殖,特别是池塘规模化养殖至今尚未取得突破性进展[6-11],成为制约笋壳鱼养殖产业发展的第二个“瓶颈” (第一个“瓶颈”是苗种培育)。世界各地,特别是华人聚居地所需的笋壳鱼商品,大多来自东南亚国家的野生采捕及囤养式笼箱提供[4, 8, 12];我国的人工养殖产品则是近几年才面市,批次和上市量甚少[4]。这些都表明,笋壳鱼的商品化养殖技术,尤其是池塘养殖技术尚未形成规范,从业者失败多而成功少。
东莞市于1996年引进澳洲北昆士兰线纹尖塘鳢。国内外有关尖塘鳢的生长以及幼、成鱼池塘饲养尚未见报道。本文报道线纹尖塘鳢的池塘养殖生长特性与养成技术,就其存在问题进行讨论、探析。
1. 材料与方法
1.1 养殖条件
1.1.1 养殖池和辅助设施
养殖池塘面积0.1~0.5 hm2,蓄水深1.5~2 m,池埂坚固、不渗漏;壤土底质,淤泥少,在10 cm以下;灌排水方便,水质良好。放养前先排干池水、清淤暴晒,然后注水清塘消毒,进水口用45~60目纱网过滤,防止野杂鱼和敌害生物进入。继续注水使池水深达1.5 m,人工施肥、培水备用。
辅助设施包括网箱(水箱)和网围。网箱以无结节聚乙烯纱网制作,规格3 m×3 m×1.2 m或4 m×4 m×1.5 m,网目20~40,配置有充气泵。水箱用塑料布制作,微透水,规格同网箱,具换水泵和充气泵。网围由20目聚乙烯纱网制作,高1.8 m,长度50~100 m不等,每2~2.5 m处有一固定竹杆,连同网片底杆一起踩在底泥里,形成100~400 m2的围隔水域。
1.1.2 光照和遮光隐蔽物
线纹尖塘鳢喜弱光。网箱和网围内设置有大或凤眼草等浮生植物组成的遮光面,占网箱水面的1/3~1/2、网围的1/5~1/4;池塘离周边1 m外设置多个用竹子做成的浮架,长6~8 m,宽1~1.5 m,种养大供尖塘鳢作隐蔽栖息场所。
1.1.3 越冬池的构建
珠三角地区每年12月至翌年3月,自然水温18℃以下时尖塘鳢停止摄食,12℃以下的低温累积会冻死,需建造简易棚膜越冬池。宜选择长方形、南北走向、面积0.12~0.5 hm2的池塘,先清淤暴晒,搭建“人”字形钢丝绳棚架,后带水清毒塘,水深保持1.5~2.5m。越冬池于11月前进鱼,12月初覆盖农用塑膜。
1.2 苗种来源
线纹尖塘鳢幼鱼由东莞市南方特种水产研究所的生产基地提供,为混合选育多代的亲本经池塘全人工繁育所得,全长2.5~ 3.2 cm,经过驯饵,能摄食鱼糜混合饲料。
1.3 饲养管理
1.3.1 苗种放养与中间培育
线纹尖塘鳢为底栖、具占地和同种相残特性、生长差异大的鱼类。因此,幼鱼必须经中间培育,筛选、分级池养,以使同一饲养池的放养规格基本一致。中间培育在网箱、网围和池塘里进行。网箱也可改为水箱,以较厚的塑料布制作,与网箱一样定置在池塘中,除具充气泵外,还需具换水泵。网箱、水箱放养量为全长2.2~2.7 cm幼鱼150~200 ind·m-2,经10~15 d的适应性暂养,规格达3~3.5 cm时再转入网围。网围放养密度为100~150 ind·m-2,经25~30 d,养成全长4~4.5 cm幼鱼,再行分筛、分级转入较小面积(1 000~2 500 m2)中间培育池。池塘放养密度改为30~40 ind·m-2,饲养至越冬前,时间90~120 d,长成体长12~13.5 cm幼鱼,11月转入越冬池。进池前的幼鱼,个体大小差异甚大,体长变动在5~18 cm间,需进行分筛、分级饲养。越冬池的放养密度为体长12~13.5 cm幼鱼40~55 ind·m-2;个体超大或超小的幼鱼需分开放养,置于越冬池内的网箱或水箱内,容量密度为体长8~11 cm幼鱼75~100 ind·m-2,体长14~17 cm幼鱼30~40 ind·m-2。越冬池内的水温应确保在18℃以上,水温超过30℃时需揭开大棚通风口。水箱需定期换水。深夜或水中溶解氧量低时,网箱、水箱均应启动充气泵。经120~130 d越冬,幼鱼长成体长15~17.5 cm,个体大小差异仍然很大,体长变动范围在12~21.5 cm之间。
1.3.2 人工混合饲料的制作与投喂
线纹尖塘鳢的早期幼鱼已完成驯食人工混合饲料。国内外尖塘鳢饲料的营养配方尚未确立,试验同样沿用人工混合饲料,以低值冰鲜海鱼为主,加工成鱼糜、鱼块,添加5%~10%鳗用配合饲料或面粉作粘合剂制作而成。采用饲料篮定点投喂,每50~60 m2水域设置1个投饲点。饲料篮沿池边均匀放置在水深约60 cm的池底,日投饲量为鱼体总重的5%~7%,晨昏各1次,傍晚占60%。
1.3.3 池塘水质的调控与病害防治
养殖尖塘鳢的池塘水源水质良好。中间培育池经清塘、消毒后即行培水。清毒塘后3~4 d,一次性施放以野生菊科植物为主要成份的绿肥1.5 kg·m-2或以熟化鸡粪为主的有机粪肥0.25 kg·m-2。当浮游动物枝角类等达繁殖高峰期(生物量8~10 ind·mL-1)、水色呈油绿或淡茶褐色、pH值6.5~7.5时,开始投放全长3~4 cm的早期幼鱼。之后,一边投饲,一边保持水色相对稳定,透明度25~30 cm,pH值略偏酸性。池养尖塘鳢常见的病害有寄生虫引致的侵袭性疾病和真菌、致病性细菌等引致的传染性疾病,危及稚幼鱼和幼成鱼。目前,已发现的有车轮虫病、斜管虫病、小瓜子虫病、锚头蚤病、水霉病、气单胞菌点状亚种引致的肌肉溃疡症、嗜水气单胞菌、副溶血弧菌等引致的失血性败血症等。由于尖塘鳢的底栖性、具潜泥习性,患病鱼较难被发现。因而,饲养过程需定期进行食台采样、镜检,必要时需作病原菌的分离、鉴定和药敏试验,对症下药,谨防疾病的发生。
1.4 常规观察、测算与记录
对同一批幼鱼的不同生长龄,包括饲养月龄和年龄,连续定期随机采样,弃大剔小至所需样本数。每次Ⅰ~Ⅱ龄鱼取样25~50尾、Ⅲ~Ⅳ龄鱼取样15~25尾,进行常规生物学活体测量,取其平均值。年龄的划分以每年6月为界,全长2.7~3.2 cm幼鱼为0龄,Ⅰ龄鱼指当年仔鱼饲养1周年个体,Ⅱ龄鱼指饲养2周年个体,依此类推。分级养殖中,期末均进行验收,每次采样120尾以上,弃大剔小,留100尾,再行全长、体长、体重等测算(精确到1 mm、0.1 g);计算养殖成活率及上市率。建立池塘养殖档案,最后作数理统计分析。
2. 结果
2.1 幼成鱼食性与生活习性
自然水域中,线纹尖塘鳢幼成鱼以活动力较弱的小鱼、虾,如沼虾、栉虾虎鱼(Ctonogobias sp)等为食,也摄食底栖水生昆虫幼体和环节动物如摇蚊幼虫和水丝蚓(Limnodrilus sp)等。人工池养条件下,1.5 cm稚鱼已驯食人工混合饲料,2.7 cm以后的早期幼鱼,鱼糜为主的混合饵料摄食率达100%,蛋白质营养需求在38%左右,饵料系数为5.2~6.8。线纹尖塘鳢属占地穴居性鱼类,对食物先窥视后吞食,很少游动掠食,为昏晨摄食型鱼类,晚间觅食活动频繁。
尖塘鳢为热带暖水性鱼类,畏冷,适宜生活水温18~32℃,最适水温24~30℃,18℃以下停止摄食,10℃以下会冻死;喜弱酸性水域,适宜pH值范围5.2~8.2,最适pH值范围6.5~7.5;能正常生活于盐度15以下的咸水和纯淡水。
2.2 幼成鱼生长
2.2.1 周年生长节律
对选择作后备亲鱼的幼鱼群体作连续3年的池养生长测定,结果见表 1。
表 1 池养线纹尖塘鳢的特定生长率和生长指标Table 1 Monthly specific rate and index of growth of pond-reared O.lineolatus年.月
year and month饲养月龄
raising month平均体长/cm
average body length特定生长率/%
special growth rate of length (SGRL)生长指标
growth instruction平均体重/g
average body weight特定生长率/%
special growth rate of weight (SGRW)样品数
number2002.7 0 2.72±0.2903 0.41±0.1178 50 8 1 4.55±0.8285 51.45 1.40 2.38±1.0078 175.87 45 9 2 6.42±1.1509 34.43 1.57 6.05±1.1259 93.30 35 10 3 8.50±0.9800 28.06 1.80 14.20±5.8749 85.32 35 11 4 11.05±0.8574 26.24 2.23 28.36±10.0379 69.17 30 12 5 13.63±0.7509 20.98 2.32 50.85±13.4553 58.39 30 2003.1 6 15.14±0.4751 10.51 1.43 80.12±18.2628 45.46 25 2 7 16.25±0.5726 7.08 1.07 113.87±10.3223 35.15 25 3 8 17.28±0.6284 6.15 1.00 146.85±17.5934 25.44 25 4 9 18.63±0.9546 7.52 1.30 189.27±26.7703 25.38 25 5 10 20.15±0.8594 7.84 1.46 236.97±20.2828 22.48 25 6 11 21.76±0.6023 7.69 1.55 285.61±21.3969 18.67 25 7 12 23.09±0.8786 5.93 1.29 326.88±16.6432 13.50 25 8 13 24.27±2.4167 4.98 1.15 368.51±17.9956 11.99 25 9 14 25.33±0.8673 4.27 1.04 403.40±13.4503 9.05 25 10 15 26.16±0.6120 3.22 0.82 436.69±13.1932 7.93 25 11 16 26.83±1.1694 2.53 0.66 468.74±7.6660 7.08 25 12 17 27.36±0.7000 1.96 0.52 494.83±16.6325 5.42 25 2004.2 19 27.85±0.9819 0.89 0.24 537.25±10.2388 4.11 25 4 21 28.31±0.9477 0.82 0.23 575.72±20.0508 3.45 25 6 23 29.03±0.8817 1.26 0.36 622.38±22.2696 3.90 15 8 25 30.00±1.2486 1.64 0.48 685.88±31.5028 4.86 15 10 27 30.82±1.0363 1.35 0.40 743.02±23.3712 4.00 15 12 29 31.55±1.4601 1.17 0.36 797.68±31.3859 3.55 15 由表 1可见,池养线纹尖塘鳢在1~2周年内的体长生长较快,并呈现阶段性。年间,6~12月生长最快,为大生长期,第1、2年的月均生长指标分别为1.86和1.00;翌年1~5月生长较慢,为小生长期,月均生长指标分别为1.25和0.34。尔后的周年生长趋势与第1、2周年基本相同。体重增长变化与体长增长节律一致。经t检验,tT=0.941 < t0.01=3.355,P>0.01;tW=0.973 < t0.01=3.355,P>0.01。说明线纹尖塘鳢体长、体重在年度生长过程中差异不显著。
2.2.2 体长与体重关系
依表 1对幼成鱼的体长、体重关系作点图分析,体长与体重关系式为W=aLb,经计算求得幂函数回归方程为W=0.02L3.0754,指数b接近3,符合等比生长规律,表明各饲养龄在保持池塘一定的载鱼量条件下,其体长、体重生长均匀(图 1)。经线性回归t检验,Pt=0.00042≪0.05,说明回归方程式是成立的。
2.2.3 肥满度
冬季具保温设施的池养条件下,线纹尖塘鳢每年5~9月份肥满度均较高,Fulton系数(K=100W·L-3)在2.29~2.93之间,此时正是性腺成熟、产卵期。观察表明,肥满度与水温的变化相关,水温上升,Fulton系数增大,详见表 2。
表 2 池养线纹尖塘鳢Fulton系数的周年变化及其与水温关系Table 2 Relationship between Fulton factor of pond-reared O.lineolatus and water temperature during annual cycles月份
month平均水温/℃
average water temperatureFulton系数 Fulton coefficient Ⅰ龄(2002年) Ⅱ龄(2003年) Ⅲ龄(2004年) 1 15.71±1.1894 2.3087±0.3195 2 17.53±1.4060 2.6537±0.0521 2.4871±0.1483 3 21.20±0.8829 2.8461±0.1280 4 24.58±1.6842 2.9271±0.5466 2.5374±0.0988 5 28.25±0.7690 2.8965±0.0533 6 30.11±1.4125 2.7720±0.0288 2.5440±0.2488 7 30.97±1.3126 2.0374±0.3574 2.6553±0.1318 8 29.28±1.1789 2.5266±0.4336 2.5777±0.1872 2.5403±0.1386 9 29.80±1.0960 2.2864±1.0459 2.4822±0.1545 10 26.00±1.6971 2.3122±0.3794 2.4393±0.0672 2.5381±0.1355 11 23.17±1.4637 2.1019±0.3542 2.4270±0.2005 12 18.83±1.2092 2.0052±2.2339 2.4161±0.0807 2.5400±0.1934 t检验 t-test t=0.9998 < t0.01=3.55 t=0.9999 < t0.01=3.55 t=0.9957 < t0.01=3.55 P>0.01 P>0.01 P>0.01 2.2.4 生长参数与生长曲线
线纹尖塘鳢体长与体重关系式W=aLb中的指数接近3(见2.2.2),故可用von Bertalanffy生长方程对其池养生长曲线和生长参数进行拟合,t值使用生长龄(a)为单位,连续测定6周年,其体长与体重关系式为W=0.0186L3.1312,特殊生长方程:
$$ L_t=48.4317\left[1-\mathrm{e}^{-0.3099(t+0.4826)}\right] $$ $$ W_t=3515.5347\left[1-\mathrm{e}^{-0.3099(t+0.4826)}\right]^3 $$ 过渡生长方程:Wt=3515.5347[1-e-0.3099(t+0.4826)]3.1312。
根据上式求出池养线纹尖塘鳢的理论体长和体重,再与实测值比较(表 3),两者较为相近,表明其池养生长型可用von Bertalanffy生长方程表示。由此分别绘制体长和体重生长曲线见图 2。
表 3 池养线纹尖塘鳢体长/体重实测值与理论值比较Table 3 The comparison of actual value with theory value of body length/weight for pond-reared O.lineolatus体长/体重
body length and weight生长龄 growth year Ⅰ Ⅱ Ⅲ Ⅳ Ⅴ Ⅵ 实测体长/cm
actual value of body length17.82±1.7628 25.65±2.3208 32.23±1.7223 36.58±1.4108 39.67±1.2124 41.86±0.8648 理论体长/cm
theory value of body length17.84 25.99 31.97 36.36 39.58 41.94 实测体重/g
actual value of weight134.54±50.4710 592.76±124.7707 1 025.32±158.8059 1 417.83±146.3286 1 625.46±177.7425 2 063.85±119.2088 理论体重/g
theory value of weight154.17 500.59 957.85 1 432.55 1 868.14 2 239.59 图 2可以看出,池养线纹尖塘鳢的体长与年龄呈非线性关系,生长曲线为一不具拐点抛物线,该曲线开始上升较快而后转为缓慢,趋向一个极限;体重生长与年龄也呈非线性关系,生长曲线为一不对称的S形曲线,其生长拐点坐标位置tx=3.2龄,相应的体长L=32.9643 cm,体重W= 1 053.9253 g,属衰老拐点。因而,池养线纹尖塘鳢的起捕规格应在体重1 054 g之前。生长参数与生长曲线的研究将另文发表。
2.3 幼成鱼饲养
2.3.1 载鱼量
对3组12口池塘、面积0.12~0.3 hm2、分别作不同载鱼量的对比试验,经观察和测算,放养越冬线纹尖塘鳢体长12~15 cm、体重60~110 g的幼鱼,密度分别为3、3.8、4.5和5 ind·m-2,同样用鱼糜为主的人工混合饲料投喂7~12个月,捕大留小。结果起捕个体均重分别为618、620、538和502 g;饲料系数(湿重)分别为7.8、7.6、7.6、7.8;上市率分别为92.0%、93.3%、85.4%和82.7%。初步得知线纹尖塘鳢的池塘最佳放养密度为3.5~4 ind·m-2,最佳终载鱼量为1.5~2 kg·m-2。用同样的方法得出,池塘定置网箱放养30~80 g的幼鱼,最佳放养密度为50~60 ind·m-2,最佳终载鱼量20~25 kg·m-2。具微流水、充氧设施的水槽饲养云斑尖塘鳢,放养全长12.5~15.5 cm幼鱼8~15 ind·m-2,终载鱼量17 kg·m-2 [12];最高的澳大利亚泰勒工厂式封闭循环水槽,放养20~50 g的线纹尖塘鳢100~120 ind·m-3,饲养6~8个月,终载鱼量达50 kg·m-3。
2.3.2 幼鱼饲养
2004~2006年间,对地处珠三角地区的东莞、中山、番禺、顺德、斗门、台山和新会等7市区共11个养殖场,统计面积共8.2 hm2的中间培育池,饲养线纹尖塘鳢幼鱼测算结果,6~7月全长2.7~3.2 cm幼鱼,经280~300 d饲养,可长成全长15.0~17.5 cm(变动范围12~21.5 cm),各级成活率分别为定置网箱75%~80%、网围85%~90%、池塘85%~90%、越冬池80%~90%,平均终成活率为43.5%~58.3%。
2.3.3 成鱼饲养
由于线纹尖塘鳢的领地穴居、少游动、懒散、伺攫猎吞鱼、虾食物等特性,人工养殖多采用单养。养殖方式以池塘为主,其次是网箱和水槽。
池塘。每年4月始,放养经中间培育的1龄幼鱼,允养量分别是体长9~12 cm为3.8~4.2 ind·m-2,体长13~15 cm为3.3~3.7 ind·m-2,体长16~18 cm为3~3.2 ind·m-2;搭配尾重350~500 g鳙Hypophthalmichthys molitrix 0.05 ind·m-2,尾重150 g鲢Aristichthys nobilis 0.02 ind·m-2。投喂以鱼糜为主的软团状混合饲料。经6~8个月饲养,个体重可达300~750 g。2002~2005年间,共统计珠三角地区参试池塘面积33.5 hm2,其中南方特种水产研究所生产基地13.5 hm2,单产范围在1~1.8 kg·m-2之间,成活率90% ~98%。
网箱、笼箱。放养经中间培育的1龄幼鱼,全长12.5~15 cm、个体重30~50 g为50~60 ind ·m-2;全长15~17 cm、体重50~80 g为30~50 ind·m-2。在具充气设施的条件下,经150~240 d饲养,个体生长可达200~300 g,单产10~15 kg·m-2。在泰国有一种类似网箱的笼箱[8],安置在河流流速为30 cm·s-1,溶氧量高的水域。放养量尾重100~200 g为70~100 ind·m-2,饲养5~8个月,体重可达450~750 g。网箱、笼箱同样投喂鱼糜为主的软团状混合饲料;均需设置一定水面种养浮生植物,定期清洗、消毒,除去青苔,必要时需作更换。
水槽。尖塘鳢适宜开展室内水槽控、保温养殖。在具换水、充气和排污设施条件下,全长12.5~15.5 cm,体重20~50 g幼鱼,放养量为15~50 ind·m-2,用鱼糜、鱼块饲养180~240 d,个体重250~300 g,单产量最低4~5 kg·m-2,最高12.5~15 kg·m-2。
3. 讨论
(1) 线纹尖塘鳢1~3龄鱼生长比云斑尖塘鳢快[6, 8]。试验观测表明,池养密度、饲养管理基本相同条件下,体长3 cm、体重0.45 g的线纹尖塘鳢、云斑尖塘鳢幼鱼,经29个月龄的饲养,体长月均增长分别为0.99和0.38 cm,体重月均增重27.44和10.70 g[8, 11]。因而,饲养个体重在750 g以内的尖塘鳢商品鱼,应以线纹尖塘鳢为好。相反,饲养750 g以上的商品鱼则应首选云斑尖塘鳢,4龄以后的云斑尖塘鳢个体绝对和相对增重量均比线纹尖塘鳢大。
对池养线纹尖塘鳢29个月龄的体重生长参数作点图分析结果,体重生长曲线也为一不对称的S形曲线,生长拐点位置于1.689龄,相当于514.29 g,属性成熟拐点,此时应是最佳的起捕上市规格。由于线纹尖塘鳢是一年多次产卵型鱼类,每次性腺发育过程成熟系数均偏小,产后仍有一个明显生长期。所以,其拐点出现的年龄并非处于成鱼生长阶段和生长衰老阶段之间,而具衰老拐点生物学意义;其个体年绝对增重量尚很大,延缓其起捕上市时间,饲养更大规格个体仍然可取。这点也为连续养殖6周年,生长拐点在3.2龄,相应的体重为1 054 g所证实。只是饲养3.2龄鱼需要3次越冬,风险大一些。
(2) 尖塘鳢的幼成鱼饲养,特别是池塘饲养一直是决定该养殖业最终能否形成和发展的关键。20年来,珠三角地区开展的池养试验表明,大部份的成活率和上市率在8%以下[11],零收获屡见不鲜。究其原因,①这之前的池塘饲养均是引进苗种,稚幼鱼经原产地采捕、囤养,长途降温空运,成活率比本地自产苗种低;②该鱼生活在底层,传统的拉网起捕率低,尤其是云斑尖塘鳢,饲养过程要观察其摄食、生长,确定各阶段的成活率相当困难;③食性转化过程会有一定损耗,一些云斑尖塘鳢育苗业者主张对体长6~7 cm幼鱼才开始驯饵[6, 11],使原先以枝角类、桡足类、仔虾、野鲮仔稚鱼等为饵改食人工混合饲料,幼成鱼期才驯饵会引致个体生长差异,出现殭鱼(老头鱼),再加上不及时调整规格,就会造成自相残食,降低池养成活率和上市率。因而,除稚鱼提早驯饵外,幼鱼饲养过程的中间培育,定期分筛、分级养殖,是解决池养尖塘鳢幼成鱼能否成功的另一关键。只有这样,才能准确掌握1龄幼鱼各阶段的生长、成活状况。线纹尖塘鳢的池养拉网起捕率高,中间培育的全过程易做到;云斑尖塘鳢在这之前的有效起捕方法是笼装诱捕,因早期幼鱼个体小,实际操作难度大,中间培育、分级饲养就较难以实现[12]。笔者发现晚昏后拉网起捕,云斑尖塘鳢的“三网率”同样很高,将最终解决这一难题。
(3) 文中引入载鱼量也称养殖容量,即单位水体内在保护环境,节约资源和保证应有效益的各个方面都符合可持续发展要求的最大养殖量。参照美国大豆协会斯密托博士观点,把饲养存活率、日间生长率、饲料系数和回报率等作为评价单位水体最佳放养量指标;结合自身生产实践,调研了多个养殖企业的历年放养收获档案,进行数理统计分析,从而推导尖塘鳢幼成鱼池塘单位面积的最佳载鱼量为初放量0.35 kg·m-2,即体长12~15 cm,3.5~4 ind·m-2。终载量1.5~2 kg·m-2,比塘鳢科鱼类中另一池养鱼类——中华乌塘鳢(Bostrichthys sinensis)的池养最佳终载量0.75~1.5 kg·m-2还要高[13]。这也与其耐低氧、活动力不强有关。养殖容量随着养殖方式、养殖技术的改变而有不同。在塘鳢科鱼类中,体长3.1 cm,体重1.4 g的中华乌塘鳢和线纹尖塘鳢,池塘常规饲养20个月龄,平均体长分别达23.6和28.08 cm,平均体重达224.2和556.49 g;月均增长分别为1.03和1.25 cm,月均增重11.14和27.75 g[12],表明尖塘鳢是塘鳢科中个体生长最快的鱼类,其池养业具有广阔的养殖前景。
-
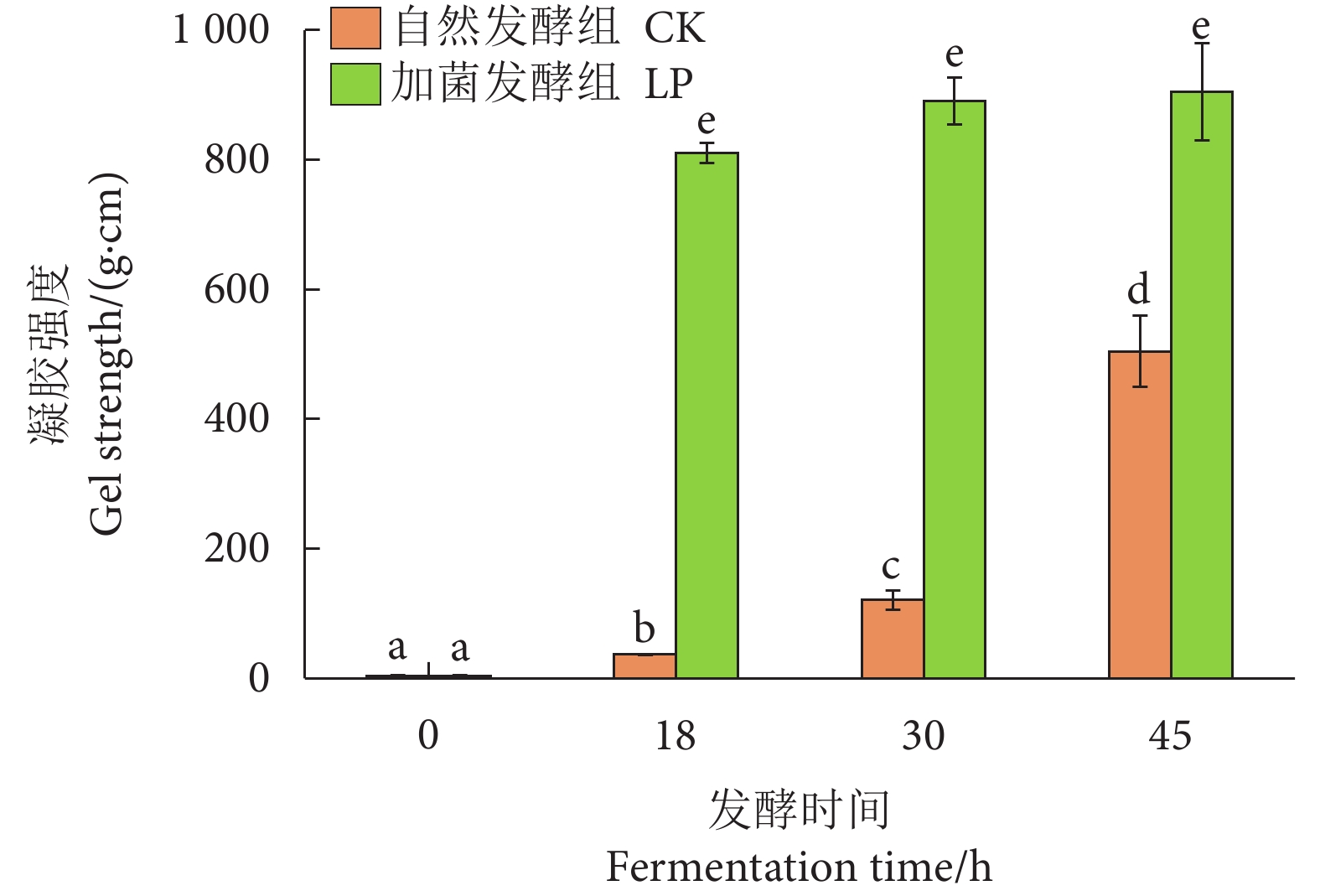
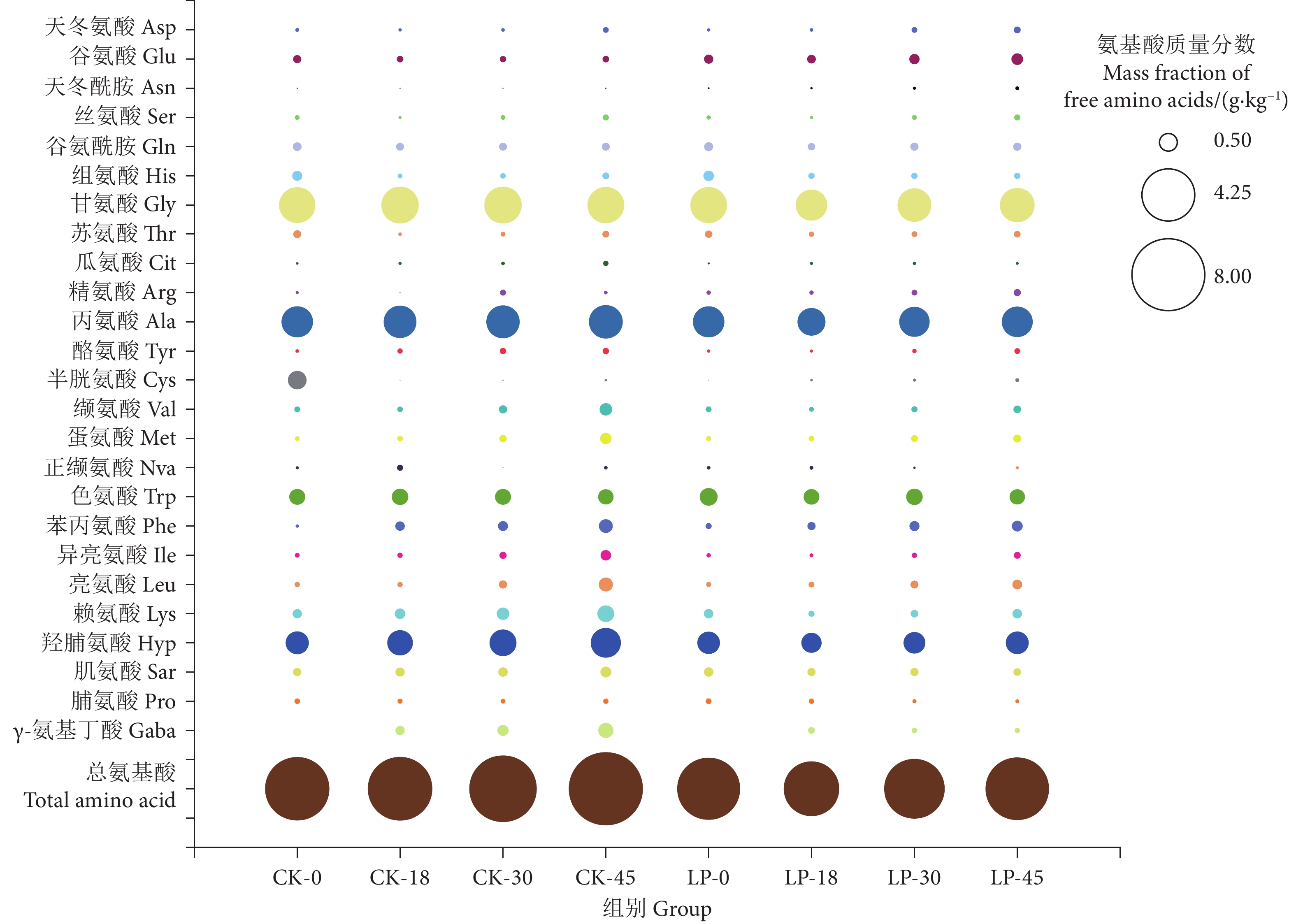
图 2 植物乳植杆菌加菌发酵对罗非鱼鱼糜发酵过程中游离氨基酸含量的影响
注:CK为自然发酵组,LP为加菌发酵组,后面的数字表示发酵时间。
Figure 2. Effect of L. plantarum addition on concentrations of free amino acids of tilapia surimi during fermentation
Note: CK represents the natural fermentation group; LP represents the fermentation group with added strain; the number after it represents the fermentation time.
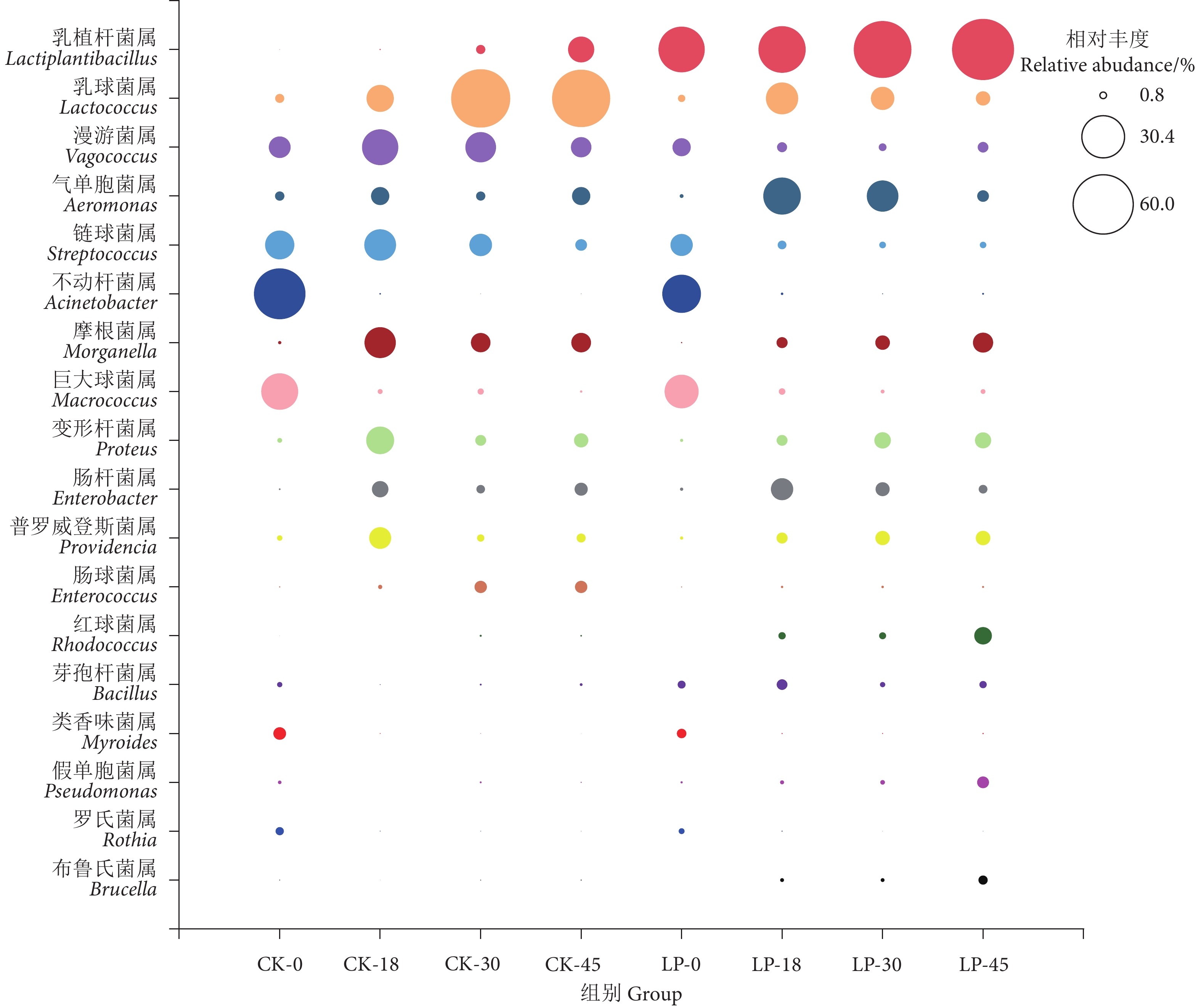
图 4 植物乳植杆菌加菌发酵对罗非鱼鱼糜发酵过程中微生物菌属的影响
注:CK为自然发酵组,LP为加菌发酵组,后面的数字表示发酵时间。
Figure 4. Effect of L. plantarum addition on microbial community at genus level in tilapia surimi during fermentation
Note: CK represents the natural fermentation group; LP represents the fermentation group with added strain; the number after it represents the fermentation time.
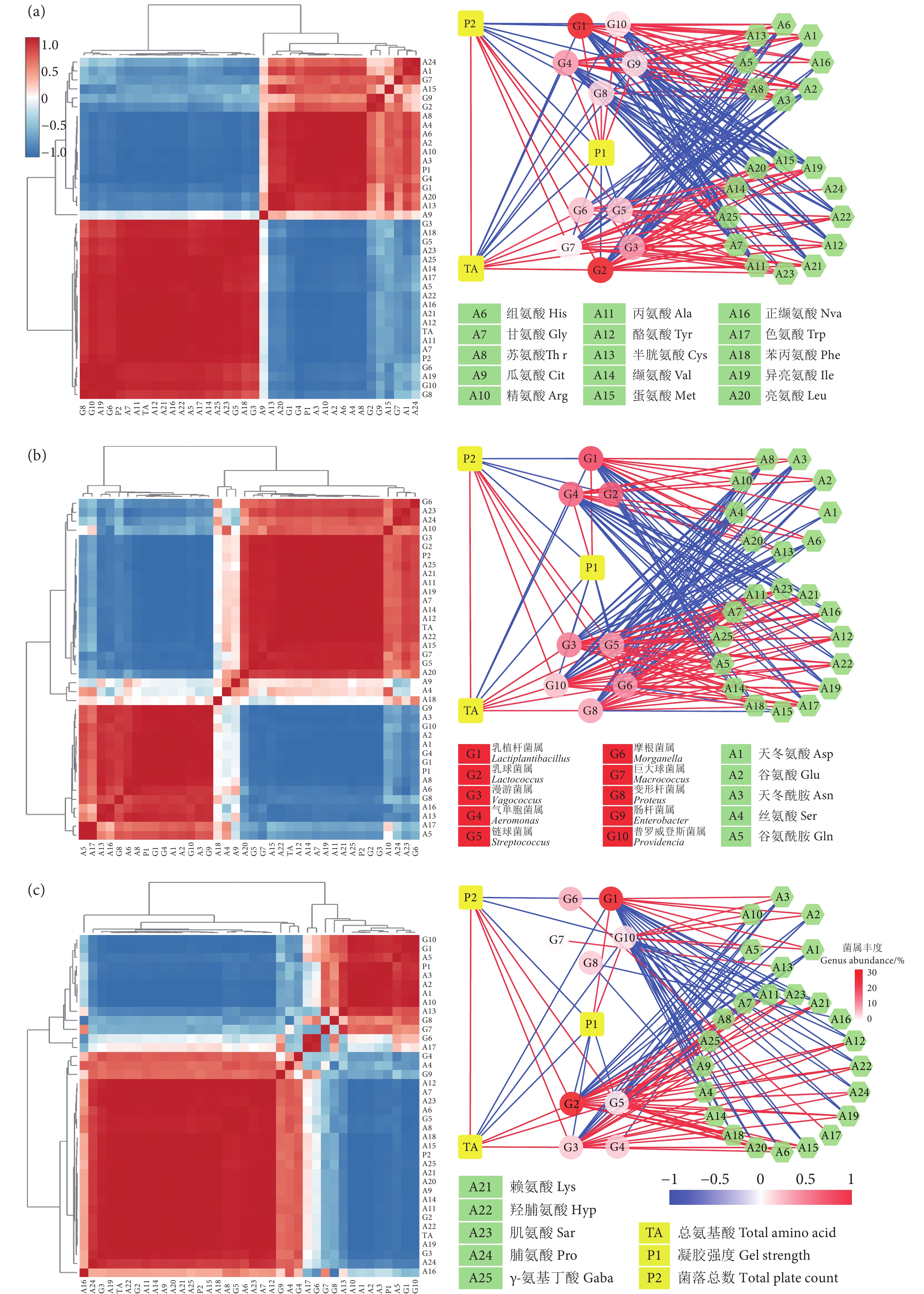
图 5 加菌发酵与自然发酵罗非鱼鱼糜在发酵第18 (a)、第30 (b)、第45 (c) 小时的凝胶强度、菌落总数、核心微生物菌属和游离氨基酸间的Pearson相关性热图和网络图
Figure 5. Pearson correlation heatmaps and correlation network maps among gel strength, total plate count, core microbial genera and free amino acids of tilapia surimi after fermentation for 18 h (a), 30 h (b) and 45 h (c) between LP and CK groups
-
[1] ZHAO Y Q, YANG S L, YANG X Q, et al. Effects of ozonated water treatment on physico-chemical, microbiological and sensory characteristics changes of Nile tilapia (Oreochromis niloticus) fillets during storage in ice[J]. Ozone-Sci Eng, 2020, 42(5): 408-419. doi: 10.1080/01919512.2019.1688133
[2] 农业农村部渔业渔政管理局, 全国水产技术推广总站, 中国水产学会. 2023中国渔业统计年鉴[M]. 北京: 中国农业出版社, 2023: 25. [3] 何晓萌, 黄卉, 李来好, 等. 罗非鱼与海水鱼制备混合鱼糜的凝胶特性研究[J]. 食品工业科技, 2018, 39(2): 5-9. [4] 袁志宁, 霍栓, 张容, 等. 发酵鲢鱼鱼糜香肠加工工艺优化及品质分析[J]. 中国调味品, 2024, 49(2): 157-164 doi: 10.3969/j.issn.1000-9973.2024.02.023 [5] 姜维. 一株耐盐性高效生物胺降解新菌的筛选、分类鉴定及应用研究[D]. 青岛: 中国海洋大学, 2014: 115-138. [6] ZHAO Y, WANG Y Q, LI C S, et al. Novel insight into physicochemical and flavor formation in naturally fermented tilapia sausage based on microbial metabolic network[J]. Food Res Int, 2021, 141: 110122. doi: 10.1016/j.foodres.2021.110122
[7] NIE X H, LIN S L, ZHANG Q L. Proteolytic characterisation in grass carp sausage inoculated with Lactobacillus plantarum and Pediococcus pentosaceus[J]. Food Chem, 2014, 145: 840-844. doi: 10.1016/j.foodchem.2013.08.096
[8] XU Y S, XIA W S, YANG F, et al. Protein molecular interactions involved in the gel network formation of fermented silver carp mince inoculated with Pediococcus pentosaceus[J]. Food Chem, 2010, 120(3): 717-723. doi: 10.1016/j.foodchem.2009.10.068
[9] 赵跃, 李春生, 王悦齐, 等. 罗非鱼鱼糜自然发酵过程中微生物群落结构对其品质形成的影响[J]. 食品科学, 2021, 42(18): 119-126. doi: 10.7506/spkx1002-6630-20200915-194 [10] LI C S, ZHAO Y, WANG Y Q, et al. Microbial community changes induced by Pediococcus pentosaceus improve the physicochemical properties and safety in fermented tilapia sausage[J]. Food Res Int, 2021, 147: 110476. doi: 10.1016/j.foodres.2021.110476
[11] LI C S, CUI Q Y, LI L H, et al. Formation and improvement mechanism of physical property and volatile flavor of fermented tilapia surimi by newly isolated lactic acid bacteria based on two dimensional correlation networks[J]. Food Chem, 2024, 440: 138260. doi: 10.1016/j.foodchem.2023.138260
[12] LI C S, LI W J, LI L H, et al. Microbial community changes induced by a newly isolated salt-tolerant Tetragenococcus muriati cus improve the volatile flavor formation in low-salt fish sauce[J]. Food Res Int, 2022, 156: 111153. doi: 10.1016/j.foodres.2022.111153
[13] 李文静, 李春生, 王悦齐, 等. 发酵盐厌氧菌YL9-2对鱼露发酵过程中品质和风味的改善作用[J]. 南方水产科学, 2022, 18(2): 115-123. doi: 10.12131/20210314 [14] LI Y, LI W J, LI C S, et al. Novel insight into flavor and quality formation in naturally fermented low-salt fish sauce based on microbial metabolism[J]. Food Res Int, 2023, 166: 112586. doi: 10.1016/j.foodres.2023.112586
[15] 赵雯宇, 余达威, 董俊丽, 等. 不同分子量壳聚糖涂膜对冷藏鱼片保鲜效果的影响[J]. 南方水产科学, 2022, 18(2): 150-157. doi: 10.12131/20210333 [16] HU N, LEI M, ZHAO X L, et al. Analysis of microbiota in Hainan Yucha during fermentation by 16S rRNA gene high-throughput sequencing[J]. J Food Process Pres, 2020, 44(7): e14523.
[17] HUANG X N, YU S Z, HAN B Z, et al. Bacterial community succession and metabolite changes during sufu fermentation[J]. LWT, 2018, 97: 537-545. doi: 10.1016/j.lwt.2018.07.041
[18] YIN L J, PAN C L, JIANG S T. Effect of lactic acid bacterial fermentation on the characteristics of minced mackerel[J]. J Food Sci, 2002, 67(2): 786-792. doi: 10.1111/j.1365-2621.2002.tb10677.x
[19] LI C S, ZHAO Y, WANG Y Q, et al. Contribution of microbial community to flavor formation in tilapia sausage during fermentation with Pediococcus pentosaceus[J]. LWT, 2022, 154: 112628. doi: 10.1016/j.lwt.2021.112628
[20] 韦芳, 窦容容, 孙纪录, 等. 鱼糜制品凝胶特性影响因素的研究进展[J]. 食品研究与开发, 2023, 44(19): 208-215. doi: 10.12161/j.issn.1005-6521.2023.19.030 [21] 王嵬, 仪淑敏, 李学鹏, 等. 鱼糜凝胶的形成机制及混合鱼糜研究进[J]. 食品安全质量检测学报, 2016, 7(1): 231-237. [22] PANPIPAT W, CHAIJAN M, BENJAKUL S. Gel properties of croaker-mackerel surimi blend[J]. Food Chem, 2010, 122(4): 1122-1128. doi: 10.1016/j.foodchem.2010.03.096
[23] UEKI N, MATSUOKA Y, WAN J, et al. The effects of endogenous proteases within abdominal muscle parts on the rheological properties of thermally induced gels from white croaker (Pennahia argentata)[J]. Food Chem, 2018, 268: 498-503. doi: 10.1016/j.foodchem.2018.06.080
[24] 王玉林. 淡水鱼鱼糜凝胶特性及凝胶形成过程中肌原纤维蛋白变化的研究[D]. 广州: 广东药科大学, 2020: 3. [25] 李维, 程荻, 杨宏. 二次加热处理对鲢鱼糜制品品质的影响[J]. 华中农业大学学报, 2019, 38(2): 94-101. [26] LI C S, LI L H, CHEN S J, et al. Volatile flavor improvement and spoilage microorganism inhibition in low-salt fish sauce (Yulu) by salt-tolerant Bacillus subtilis[J]. Fermentation-Basel, 2023, 9(6): 515. doi: 10.3390/fermentation9060515
[27] 李琰, 李文静, 李春生, 等. 基于微生物菌群调控的枯草芽孢杆菌改善低盐发酵鱼露品质作用机制[J]. 食品科学, 2023, 44(18): 58-67. doi: 10.7506/spkx1002-6630-20221226-246 [28] ZHANG Q L, LIN S L, NIE X H. Reduction of biogenic amine accumulation in silver carp sausage by an amine-negative Lactobacillus plantarum[J]. Food Control, 2013, 32(2): 496-500. doi: 10.1016/j.foodcont.2013.01.029
[29] BA H V, SEO H W, SEONG P N, et al. Lactobacillus plantarum (KACC 92189) as a potential probiotic starter culture for quality improvement of fermented sausages[J]. Korean J Food Sc An, 2018, 38(1): 189-202.
[30] 杨方. 鱼肉内源酶对发酵鱼糜凝胶和抗氧化特性影响的研究[D]. 无锡: 江南大学, 2016: 34-35. [31] MONTO A R, LI M Z, WANG X, et al. Recent developments in maintaining gel properties of surimi products under reduced salt conditions and use of additives[J]. Crit Rev Food Sci, 2022, 62(30): 8518-8533. doi: 10.1080/10408398.2021.1931024
[32] YANG D Q, LI C S, LI L H, et al. Novel insight into the formation mechanism of umami peptides based on microbial metabolism in Chouguiyu, a traditional Chinese fermented fish[J]. Food Res Int, 2022, 157: 111211. doi: 10.1016/j.foodres.2022.111211
-
期刊类型引用(2)
1. 云万里,郝淑贤,相欢,陈楚欣,黄卉,魏涯,岑剑伟,赵永强,王迪. 面粉类型对油炸裹糊罗非鱼肉品质的影响. 南方水产科学. 2025(02): 174-181 .  本站查看
本站查看
2. 郑妍. 植物蛋白在鱼糜制品中的应用及前景. 粮食与食品工业. 2025(03): 36-40 .  百度学术
百度学术
其他类型引用(0)



 下载:
下载:


 粤公网安备 44010502001741号
粤公网安备 44010502001741号
