Sequencing of whole genome of Bacillus velezensis LG37 and screening of inorganic nitrogen metabolism candidate genes
-
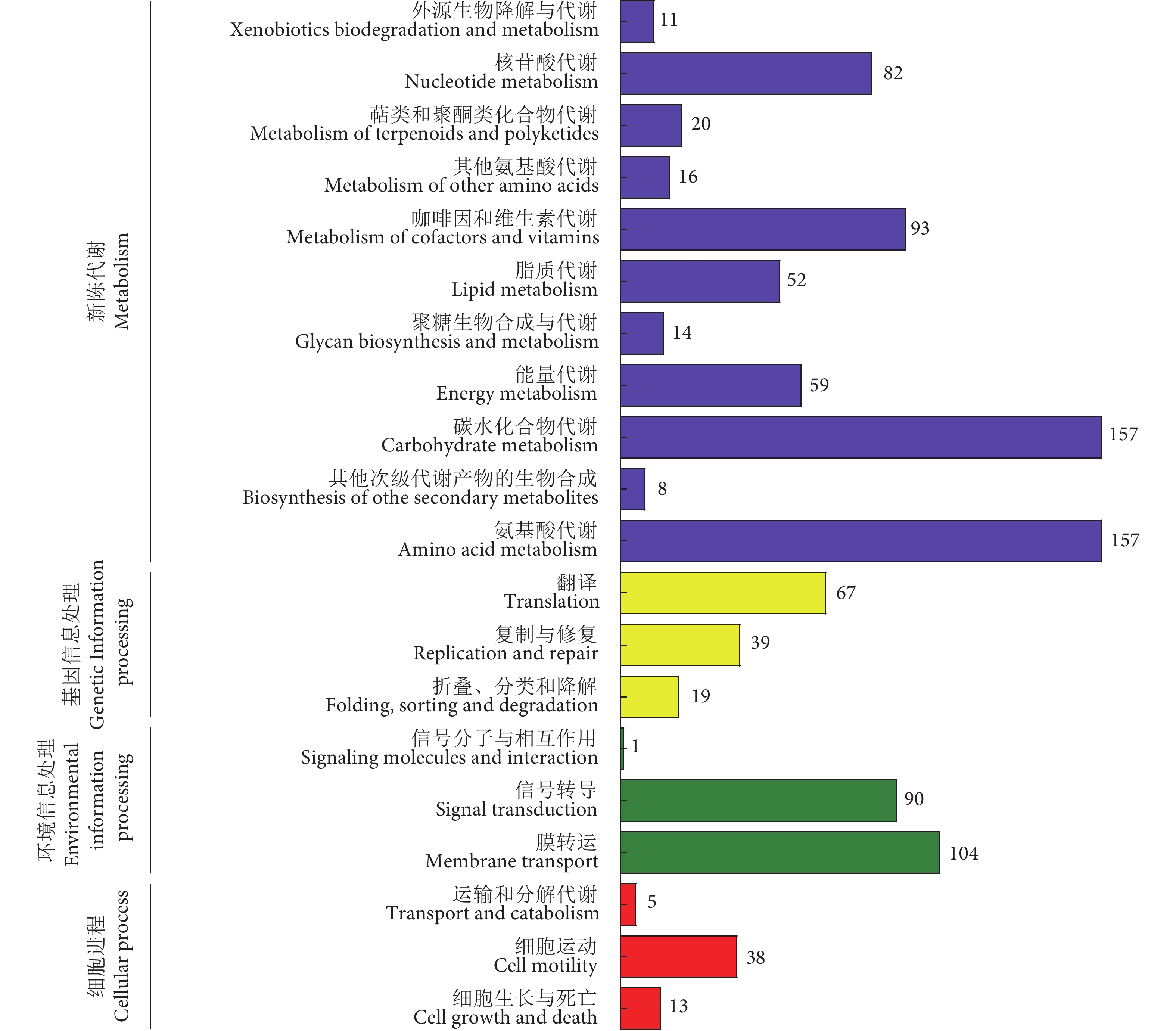
摘要: 前期研究发现贝莱斯芽孢杆菌 (Bacillus velezensis) LG37可高效同化无机氮,但其机理尚不清楚。为解读其高效同化无机氮的机理,结合三代PacBio RS II 和二代Illumina HiSeq 2000 测序技术对贝莱斯芽孢杆菌LG37进行全基因组测序,在此基础上利用NR、KEGG、eggNOG、GO和CARD数据库进行序列注释、分析,并通过本地Blast+对无机氮代谢相关基因进行挖掘。测序结果表明:1) 贝莱斯芽孢杆菌LG37的基因组为3 929 697 bp的环状染色体,GC含量为46.5%,包含3 854个蛋白质编码基因、86个tRNA 基因和27个rRNA基因。2) 共筛选出无机氮代谢相关候选基因94个,主要涉及编码感应蛋白、转录调控因子、转运蛋白、氧化还原酶和同化酶等,并对这些基因的GO功能进行了注释分析。综上,LG37 全基因组测序及无机氮代谢相关基因的分析为芽孢杆菌降低养殖水体中无机氮的研究提供了基因水平数据,为芽孢杆菌微生态制剂降低水体中无机氮的应用研究提供了理论依据。Abstract: It has been found that Bacillus velezensis can assimilate inorganic nitrogen efficiently. However, the underlying mechanism of inorganic nitrogen assimilation remains enigmatic. In order to elucidate the mechanism, we sequenced the complete genome of LG37 by PacBio RS II and Illumina HiSeq 2000, and then annotated and analyzed the sequence by the database of NR, KEGG, eggNOG, GO and CARD. Finally, we screened the genes related to inorganic nitrogen metabolism by local Blast+. The results show that: 1) The genome contained one circular chromosomal with a size of 3 929 697 bp and a GC-content of 46.5%. Gene prediction and annotation was performed to acquire a total of 3 854 protein-coding genes, 86 tRNA genes and 27 rRNA genes. 2) A total of 94 inorganic nitrogen metabolism candidate genes were screened by local Blast+. These genes were involved into coding sensing protein, transcriptional regulator, transporter, oxidoreductase and assimilator, etc.. In conclusion, the whole genome sequencing and data analysis of LG37 provide data at gene level and theoretical basis for functional study and application of Bacillus in reducing inorganic nitrogen in aquaculture water.
-
Keywords:
- Bacillus velezensis /
- Whole genome sequence /
- Inorganic nitrogen /
- Metabolic pathway
-
大口黑鲈 (Micropterus salmoides),俗称加州鲈,隶属鲈形目、鲈亚目、太阳鱼科、黑鲈属。原自然分布于北美洲,于1983年从中国台湾引入广东省[1]。大口黑鲈适温范围较广,生长快且易起捕,同时其肉质鲜美、无肌间刺,因而深受养殖者和消费者欢迎,在我国多个省市与地区得以迅速推广养殖。近几年大口黑鲈人工配合饲料的突破与应用,使大口黑鲈养殖区域与规模迅速扩大。2017年,我国养殖大口黑鲈年产量已达45.7万吨[2]。但近两三年来养殖大口黑鲈抗病性能下降,病害问题频发。大口黑鲈引进中国已三十多年,虽然这期间也从原产地进行过为数不多的小规模引进,但相对于养殖规模而言,亲本的种质更新极为有限。因此抗病性能下降是否与种质质量与遗传多样性下降有关,进一步开展选择育种的种质遗传基础如何,都亟需对中国现有大口黑鲈养殖群体的遗传多样性进行评估。
微卫星 (microsatellite) 标记,又称简单重复序列 (simple sequence repeats, SSR) 或短串联重复序列 (simple tandem repeats, STR),是均匀分布于真核生物基因组中的简单重复序列,由2~6个核苷酸的串联重复片段构成,具有分布广泛、共显性遗传、多态性高等特点,广泛应用于多个生命学科领域[3-4]。20世纪90年代初,微卫星标记开始应用于鱼类遗传及育种研究中[5]。利用微卫星研究大口黑鲈的遗传多样性在近年有所报道。梁素娴等[6]用21对微卫星引物对广东的3个大口黑鲈养殖群体进行遗传多样性分析发现,3个不同群体平均杂合度处于中等偏低水平 (期望杂合度He分别为0.375、0.403和0.368),遗传多样性不高。李镕等[7]利用11对微卫星引物对大口黑鲈第2~第4代选育群体的遗传结构进行分析发现,与F2相比,F4的遗传多样性减少8.76%,表明选育群体的遗传多样性在逐步下降。樊佳佳等[8]用43个微卫星标记对目前国内养殖的大口黑鲈群体、2009年引进的佛罗里达亚种、2010年引进的佛罗里达亚种和北方亚种这4个群体进行遗传多样性检测,发现国内养殖的大口黑鲈群体遗传多样性水平明显低于国外新引进的大口黑鲈群体。以上研究表明,我国大口黑鲈养殖群体出现遗传多样性显著下降、种质退化及种群结构单一等问题。
广东省是中国大口黑鲈成鱼的主养区和主要苗种培育基地。2016年广东省大口黑鲈养殖面积约5 333.4 hm2,产量为23.5万吨,占全国总产量的62.8%。广东省大口黑鲈成鱼养殖主要集中在珠江三角洲的佛山市,其养殖产量近20万吨。佛山市的顺德区和南海区有大口黑鲈鱼苗培育专业村,仅南海区九江镇的大口黑鲈鱼苗生产量就接近40亿尾,约占广东全省生产量的75%,占全国生产量的60%[9]。因此佛山地区的大口黑鲈养殖群体极具代表性,研究其养殖群体的遗传多样性和遗传结构,可以反映出中国大口黑鲈养殖群体的遗传多样性现状。本研究选取广东省佛山市3个不同地方的大口黑鲈养殖群体作为研究对象,利用微卫星标记技术对其进行遗传多样性分析,旨在为当前大口黑鲈养殖群体的种质资源调查和进一步的选育种工作提供支撑。
1. 材料与方法
1.1 实验材料
实验所需大口黑鲈样品分别为佛山市南海区西樵镇的西樵群体 (编号为XJ)、九江镇沙头片区的沙头群体 (编号为ST)、九江镇九江片区烟南村的烟南群体 (编号为YN),这3个养殖群体的苗种来自不同苗种场。从3个养殖群体中各随机选取32尾个体,分别剪取尾鳍,–20 ℃保存于95%的乙醇中,用于DNA的提取。
1.2 基因组DNA的提取与检测
利用天根生化科技 (北京)有限公司的海洋动物组织基因组DNA提取试剂盒,提取大口黑鲈尾鳍的基因组DNA。用1%的琼脂糖凝胶进行电泳,并用酶标仪检测其质量和浓度,−20 ℃冰箱保存。
1.3 微卫星引物的筛选与合成
根据GenBank及文献上登录的大口黑鲈微卫星引物序列[10-14],选择并设计合成51对引物,进行预扩增,并用Qsep100全自动核酸蛋白分析系统 (中国,台湾) 检测引物的特异性和多态性,最终确定其中的12对重复性好、特异性强和多态性高的微卫星引物 (表1) 用于研究。引物委托生工生物工程 (上海) 股份有限公司进行合成。各对引物分别带有TAMRA、HEX和FAM的特异性荧光标记,用于扩增和测算目的条带的大小。
表 1 大口黑鲈微卫星引物序列及退火温度Table 1. Primer sequences and annealing temperatures of microsatellite marker位点
loci引物序列 (5'−3')
primer sequence片段长度/bp
size重复序列
repeat sequence退火温度/℃
annealingtemperature参考文献
Referencemdo3 AGGTGCTTTGCGCTACAAGT
CTGCATGGCTGTTATGTTGG119~143 (CA)20 46.2 [11] mdo4 TCTGAACAACTGCATTTAGACTG
CTAATCCCAGGGCAAGACTG151~155 (CA)11 48.6 [11] mdo7 TCAAACGCACCTTCACTGAC
GTCACTCCCATCATGCTCCT179~205 (CA)12 53 [11] lma120 TGTCCACCCAAACTTAAGCC
TAAGCCCATTCCCAATTCTCC204~224 (GT)28 60 [10, 13] MiSaTPW011 CAACATGGACGCTACTAT
CAACCATCACATGCTTCT170~194 (AGAT)13 60 [14] MiSaTPW038 AGTCAACATTCAAACCACTTTCCCAC
TTGTATGTTAGGATCGGCTGATTGTG227~312 (AGAT)6+5+9+5 60 [14] MiSaTPW025 CCAAGGTCAGGTTTAAC
ACCTTTGTGCTGTTCTGTC277~298 (AGAT)11 55 [14] MiSaTPW060 TATAGTTTGGTCCAGCAGGTGGCGT
TGTGGAATGACATTTAGCCGAGGCC294~561 (AGAT)14+10+3+2+4+9+10 60 [14] MiSaTPW116 CCAAACAGCCTACAGAATGTGCCT
AGTCCCTCGACTGAATGTGTGCAA191~219 (AC)21 55 [14] MiSaTPW001 AGTAAAGGACCACCCTTGTCCA
GCCTGGTCATTAGGTTTCGGAG293~299 (AC)16 60 [14] MiSaTPW058 CATTCCTGAGAGACGTGGTTGCTG
TGTGACTTGCTCTGTGAAAGGTGC143~169 (AAGT)6 60 [14] MiSaTPW154 TGCGGGCTAATAGGCCTGTCTG
GCACCACACAATCACACCTGGTAT182~228 (AC)18 55 [14] 1.4 PCR的反应体系与程序
PCR反应总体积20 μL,含有模板DNA 1 μL、上下游引物 (10 μmol·L–1) 各1 μL、ddH2O 7 μL、Premix rTaq (TaKaRa,中国大连) 10 μL,在96孔板中进行PCR反应。扩增程序为94 ℃预变性3 min;94 ℃变性30 s,退火 (各引物退火温度参照表1中的退火温度) 30 s,72 ℃延伸30 s,35个循环;72 ℃延伸10 min,4 ℃保存。
1.5 扩增产物的检测
引物筛选的预扩增产物通过Qsep100全自动核酸蛋白分析系统进行毛细管电泳检测。将PCR扩增产物室温放置2~3 min,开盖后按要求放在样品托上进行毛细管电泳。电泳参数为6 kV下进样2 s再电泳300 s。电泳完毕后,用Q-ANALYZER软件对结果进行矫正与分析,对扩增产物分子量的差异大小进行个体各微卫星基因座的分型。
荧光引物的扩增产物通过STR基因分型检测,由生工生物工程 (上海) 股份有限公司完成。使用毛细管电泳分离STR片段,激光诱导的荧光检测,由设备检测并数据化存储,自动数据采集并分型。荧光颜色分离,计算片段大小,确定各个等位座的基因分型。整个过程通过ABI 310遗传分析仪 (美国) 完成。
1.6 数据统计与分析
通过STR分型确定标记在个体中的基因型,运用POPGENE 32软件进行统计分析,计算各个群体微卫星位点的等位基因数 (Na)、有效等位基因数 (Ne)、观测杂合度 (Ho)、He、Shannon指数 (I)、遗传相似度 (S) 及群体间遗传距离 (Da)。运用FSTAT软件算出遗传分化指数 (FST)。根据Nei氏遗传距离的大小,运用MEGA 5软件构建系统树[15]。运用CERVUS 3.0软件统计出多态信息含量 (polymorphism information content, PIC)[16]。运用ARLEQUIN 3.1软件进行群体间FST计算、群体分子方差分析 (AMOVA)[17]。利用STRUCTURE 2.3软件分析群体的遗传结构[18]。
2. 结果
2.1 微卫星位点筛选
从不同地区采集到的大口黑鲈养殖群体中,随机选取12个大口黑鲈样本,利用51对微卫星引物进行PCR扩增,通过Qsep100全自动核酸蛋白分析系统进行毛细管电泳检测产物(图1),筛选出12对多态性好、特异性强的微卫星引物用于大群体的扩增分析 (表1)。
![]() 图 1 毛细管电泳检测引物MiSaTPW011部分扩增产物绿色箭头指示20 bp align Marker和1 000 bp align Marker;M. size Marker;1~8. 大口黑鲈8个个体的毛细管电泳结果Figure 1. Detection for partial PCR products of MiSaTPW011 with capillary electrophoresisGreen arrows indicate 20 bp align Marker and 1 000 bp align Marker, respectively; M. size Marker; 1−8. output of eight M. salmoides capillary electrophoresis
图 1 毛细管电泳检测引物MiSaTPW011部分扩增产物绿色箭头指示20 bp align Marker和1 000 bp align Marker;M. size Marker;1~8. 大口黑鲈8个个体的毛细管电泳结果Figure 1. Detection for partial PCR products of MiSaTPW011 with capillary electrophoresisGreen arrows indicate 20 bp align Marker and 1 000 bp align Marker, respectively; M. size Marker; 1−8. output of eight M. salmoides capillary electrophoresis2.2 微卫星位点多态性与群体遗传参数
用筛选出的12对微卫星引物扩增3个大口黑鲈群体共96个个体的DNA,将产物进行STR分型 (图2)。结果表明,各对引物在3个群体中均能扩增到目的条带,并体现了程度不一的多态性(表2)。其中,Na为1~6,Ne为1~3.52,I为0~1.427,Ho为0~0.854,He为0~0.720,PIC为0.02~0.693。
表 2 大口黑鲈12个微卫星位点的等位基因数、杂合度及多态信息含量Table 2. Number of alleles, heterozygosity and polymorphic information content of 12 microsatellite loci of M. salmoides位点
locus等位基因数
Na有效等位基因数
NeShannon指数
I观测杂合度
Ho期望杂合度
He多态信息含量
PICmdo3 1 1.00 0.000 0.000 0.000 0.040 mdo4 4 1.89 0.797 0.086 0.473 0.439 mdo7 2 1.13 0.225 0.118 0.112 0.158 lma120 4 2.89 1.211 0.552 0.658 0.604 MISATPW011 4 2.14 0.838 0.854 0.536 0.425 MISATPW038 4 1.72 0.658 0.523 0.420 0.442 MISATPW025 3 2.49 0.980 0.484 0.601 0.526 MISATPW060 6 3.52 1.427 0.667 0.720 0.693 MISATPW116 1 1.00 0.000 0.000 0.000 0.020 MISATPW001 2 1.12 0.219 0.115 0.109 0.102 MISATPW058 2 1.04 0.101 0.000 0.041 0.040 MISATPW154 4 1.65 0.749 0.208 0.397 0.363 平均 mean 3.08 1.80 0.600 0.301 0.339 0.321 3个大口黑鲈群体的遗传多样性见表3。其中3个群体的Na为2.583 3~2.666 7,Ne为1.659 2~1.911 0,I为0.533 7~0.620 6,Ho为0.294 7~0.304 9,He为0.312 5~0.360 6,PIC为0.294 2~0.335 6。沙头群体的6个遗传多样性参数略高于西樵和烟南群体,西樵和烟南群体的比较接近,且西樵群体的遗传多样性参数最低。
表 3 3个大口黑鲈养殖群体遗传多样性Table 3. Genetic diversity of three populations of M. salmoides群体
population等位基因数
Na有效等位基因数
NeShannon指数
I观测杂合度
Ho期望杂合度
He多态信息含量
PIC西樵 XJ 2.583 3 1.659 2 0.533 7 0.294 7 0.312 5 0.294 2 沙头 ST 2.666 7 1.911 0 0.620 6 0.304 9 0.360 6 0.335 6 烟南 YN 2.583 3 1.736 1 0.554 1 0.301 5 0.328 4 0.296 4 2.3 群体间的遗传分化及遗传距离分析
3个大口黑鲈养殖群体间的遗传分化指数 (FST)为0.012 1~0.028 9 (表4),属低水平遗传分化 (FST<0.05)。其中西樵和烟南群体间的遗传分化水平最低 (FST=0.012 1),沙头群体和烟南群体间的遗传分化水平最高 (FST=0.028 9,表4)。
表 4 3个大口黑鲈养殖群体的遗传分化指数Table 4. Fixation index (FST) of three populations of M. salmoides西樵 XJ 沙头 ST 烟南 YN 西樵 XJ − 沙头 ST 0.017 9 − 烟南 YN 0.012 1 0.028 9 − AMOVA分析显示,14.17%的遗传变异来自群体间,85.83%的遗传变异来自群体内 (P<0.01)。结果表明,遗传变异主要存在个体间,个体间的遗传变异程度远大于群体间 (表5)。
表 5 3个大口黑鲈养殖群体AMOVA分析Table 5. AMOVA analysis among three populations of M. salmoides变异来源
source of variation自由度
df平方和
sum of squares方差组分
variance component% 群体间 among populations 95 192.859 0.296 76 14.17 群体内 within populations 96 146.500 1.526 04 85.83** 总变异 total variation 191 339.359 1.778 07 注:**. 1 023次模拟检验后为极显著 (P<0.01) Note: **. very significant difference after 1 023 permutation tests (P<0.01) 3个群体间Nei's遗传距离 (Da)介于0.013 7~0.024 9,遗传相似度 (S)介于0.975 4~0.986 4 (表6),其中西樵群体和沙头群体的遗传距离最近 (Da=0.013 7)、遗传相似度最高 (S=0.986 4),而烟南群体和沙头群体的遗传距离最远 (Da=0.024 9)、遗传相似度最低 (S=0.975 4)。由遗传距离大小构建了UPGMA聚类树,聚类分析结果显示,3个大口黑鲈群体归为两大类,西樵与烟南群体为一类,沙头群体单独为一类 (图3)。
表 6 3个大口黑鲈养殖群体的遗传相似度S (对角线上) 和遗传距离Da (对角线下)Table 6. Genetic similarity (above diagonal) and genetic distance (below diagonal) of three populations of M. salmoides西樵 XJ 沙头 ST 烟南 YN 西樵 XJ − 0.982 5 0.986 4 沙头 ST 0.017 7 − 0.975 4 烟南 YN 0.013 7 0.024 9 − 2.4 群体遗传结构的分析
运用STRUCTURE软件对3个大口黑鲈群体的遗传结构进行分析,执行K=2~7的假设,设定10次重复,结果显示对数似然函数值lnP(D)随亚群数K的增加而降低,K=1时,lnP(D)最大,从而确定出最适宜的亚群数为1 (图4)。K=2时,3个群体的遗传结构图几乎没有分别 (图5),也说明3个养殖群体都来自一个亚群。
3. 讨论
本研究采用Qsep100全自动核酸蛋白分析系统,通过毛细管电泳筛选微卫星引物,在51对微卫星引物中挑选出12对多态性相对较好的引物进行大群体遗传多样性的检测。在检测阶段则是采用ABI310遗传分析仪通过荧光标记的引物进行PCR扩增,扩增产物经毛细管电泳后进行STR分析。Qsep100全自动核酸蛋白分析系统和ABI310遗传分析仪均采用毛细管电泳系统,2个系统都具有操作简单、速度快、准确性高的优点。不同之处在于,Qsep100系统在电泳介质中加入能结合核酸的荧光物质,利用激光诱导其发光来检测核酸[19],ABI310系统则是先对引物进行荧光标记,使得PCR产物带有荧光,毛细管电泳时直接检测产物荧光来确定产物大小[20]。Qsep100系统成本较高的部分是带有荧光的电泳介质 (即卡夹),ABI310系统成本较高的部分是荧光引物的合成,Qsep100系统的总成本略低。但ABI310系统毛细管的峰图则更清晰,更容易排除杂带的干扰,检测准确性更高。因此综合2套系统的优势,本研究在引物筛选阶段采用Qsep100系统,在检测分型阶段则采用ABI310系统,可以在保证准确性的前提下提高检测效率,节省成本。
对本研究筛选的12对微卫星引物的遗传参数分析发现,12对引物中有5对引物属于低度多态 (PIC<0.25),4对引物属于中度多态 (0.25<PIC<0.5),只有3对引物属于高度多态 (PIC>0.5)。其中有2对引物 (mdo3和MISATPW116)只有1个等位基因 (Na=1),其He均为0,说明这2对引物没有多态性。Malloy等[11]和Lutz-Carrillo等[13-14]通过磁珠富集法筛选到大量的大口黑鲈微卫星引物,本研究选择其中的51对引物用于扩增,并从中筛选出12对多态性高且特异性好的微卫星引物。在Malloy等[11]和Lutz-Carrillo等[13-14]的研究中,这12对引物大都属于高度多态性,其中有10对引物He为0.55~0.94,其遗传多样性参数均显著高于本研究所获得的参数值。另外,在他们的研究中报道有高度多态性的引物,大部分在本研究的3个群体中已不再具有多态性[10-14]。因此,从本研究12对引物在大口黑鲈3个养殖群体扩增所获得的多样性参数来看,本研究中的3个养殖群体的遗传多样性较Malloy等报道过的群体已显著下降。推测当前养殖的大口黑鲈群体大都是由最初引进的大口黑鲈群体繁育的后代,经过多年的近交繁育,遗传多样性已显著下降。
对本研究选取的广东佛山3个大口黑鲈养殖群体的遗传多样性分析发现,3个群体的He为0.312 5~0.360 6。而樊佳佳等[8]用微卫星标记对国内养殖群体和3个引进群体进行遗传多样性检测,其He为0.454 9~0.613 8,高于本研究的分析结果。本研究所选用的MISATPW001和mdo72位点,PIC值已下降为0.102和0.158,而在樊佳佳等[8]的结果中,国内养殖群体中这2个位点的PIC分别为0.141 1和0.239 2,这均说明近几年我国大口黑鲈养殖群体遗传多样性下降明显。另外,3个群体之间,沙头群体 (ST)的遗传多样性最高 (He=0.360 6),各遗传参数均高于另外2个群体,可能与苗种来源有关。
本研究的3个大口黑鲈养殖群体遗传分化指数 (FST)为0.012 1~0.028 9,遗传分化水平较低,说明3个群体遗传差异不明显。3个群体Da介于0.013 7~0.024 9,非常接近,另外,运用STRUCTURE软件进行遗传结构分析也表明3个养殖群体应来自于一个亚群。这佐证了上述观点,即当前大口黑鲈养殖群体极可能均由最初引进的大口黑鲈群体繁育而来。当前大口黑鲈养殖群体结构单一,遗传多样性低,抗病性能下降的问题除了与养殖模式及管理有关外,也极可能与种质退化、遗传多样性下降有关。由于遗传多样性下降,导致其对环境的适应能力变差,进而更容易被病原等侵袭。因此应高度重视提高与维持大口黑鲈种群遗传多样性的问题,并通过分子选育种技术进行优良品种的培育,以促进大口黑鲈养殖产业的健康可持续发展。
-
表 1 贝莱斯芽孢杆菌LG37 基因组特性
Table 1 Genome features of B. velezensis LG37 strain
特性
Feature数值
Value基因组大小 Genome size/bp 3 929 697 GC-含量 GC-content 46.5% 质粒数量 Plasmid number 0 总基因 Total genes 3 967 蛋白编码基因 Protein-coding genes 3 854 转运 RNA tRNA 86 核糖体 RNA rRNA 27 编码区域大小 Coding region size/bp 3 495 864 编码区域GC-含量 GC-content of coding region 47.3% 编码区域/全基因组 Coding region/Genome length 89.0% 间隔区域大小 Intergenic region size/bp 433 833 间隔区域占比 Ratio of intergenic region 11.0% 表 2 LG37 基因组氮代谢通路及其相关基因
Table 2 Related genes of nitrogen metabolism pathways of LG37
通路
Pathway IDKEGG 描述
KEGG description基因
Geneko00910 氮代谢 Nitrogen metabolism orf00490, orf00539, orf00544, orf00545, orf00546, orf00547, orf00817, orf01213, orf01254, orf01968, orf02226, orf02227, orf02368, orf03807, orf03808, orf03809, orf03810, orf03811 M00531 同化硝酸盐还原 Assimilatory nitrate reduction orf00539, orf03807, orf03809 M00530 异化硝酸盐还原 Dissimilatory nitrate reduction orf00544, orf00545, orf00547, orf03808, orf03809, orf03810, orf03811 M00529 反硝化 Denitrification orf00544, orf00545, orf00547, orf03809 M00804 完全硝化 Complete nitrification orf03809 表 3 无机氮代谢候选基因
Table 3 Candidate genes of inorganic nitrogen metabolism
基因
Gene大小
Size/bp蛋白
ProteinGO-分子功能
GO-Molecular functionorf00084 741 Type III pantothenate kinase YacB 泛酸激酶活性 orf00106 246 Putative septation protein SpoVG 分子功能的负调控 orf00148 885 Pyridoxal 5'-phosphate synthase subunit PdxS 谷氨酰胺水解活性 orf00173 2 004 Nitrate reductase YyaE 硝酸还原酶活性 orf00246 1 416 Arginine utilization regulatory protein RocR 转录因子结合 orf00250 1 206 Ornithine aminotransferase RocD 鸟氨酸氧酸转氨酶活性 orf00316 963 Iron(3+)-hydroxamate-binding protein YxeB 无机离子转运与代谢 orf00379 1 017 Respiratory nitrate reductase NarI 硝酸还原酶活性 orf00490 1 287 Glutamate dehydrogenase RocG 谷氨酸脱氢酶 (NAD+) 活性 orf00535 1 377 Cytochrome cd1-nitrite reductase-like YwhL 亚硝酸盐还原酶活性 orf00539 1 131 Nitrate transporter NarT 跨膜转运 orf00544 3 687 Nitrate reductase alpha chain NarG 硝酸还原酶活性 orf00545 1 464 Nitrate reductase beta chain NarH 硝酸还原酶活性 orf00546 558 Nitrate reductase NarJ 未折叠蛋白结合 orf00547 672 Nitrate reductase gamma chain NarI 硝酸还原酶活性 orf00608 318 Urease subunit gamma 氨基酸转运与代谢 orf00609 375 Urease subunit beta 氨基酸转运与代谢 orf00610 1 710 Urease subunit alpha 氨基酸转运与代谢 orf00623 351 Nitrogen regulatory protein P-II GlnB 酶调节活性 orf00624 1 212 Ammonium transporter NrgA 铵跨膜转运蛋白活性 orf00630 282 Stage III sporulation protein D SpoIIID DNA结合转录因子活性 orf00783 726 Glucosamine-6-phosphate deaminase NagB 葡萄糖胺-6-磷酸脱氨酶活性 orf00817 582 YvdA 无机离子转运与代谢 orf00959 2 112 YvgW 阳离子转运ATP酶活性 orf00966 1 716 Sulfite reductase [NADPH] CysI 亚硫酸盐还原酶 (NADPH) 活性 orf00998 1 047 ABC transporter permease protein YvrB 转运体活性 orf01042 357 Uncharacterized protein YusI 氧化还原酶活性 orf01058 1 398 ABC transporter ATP-binding protein ATP结合 orf01105 237 Nitrogen-fixing NifU domain-containing protein 铁硫簇结 orf01175 2 403 Cation:proton antiporter 单价无机阳离子跨膜转运蛋白活性 orf01213 1 032 Nitronate monooxygenase Ncd2 硝酸单加氧酶活性 orf01254 549 YtiB 无机离子转运与代谢 orf01262 813 Nitrate transport system permease protein YtlD 跨膜转运蛋白活性 orf01263 783 Nitrate ABC transporter permease YtlC 跨膜转运 orf01264 1 005 Nitrate ABC transporter periplasmic protein YtlA ATP酶活性 orf01290 753 Quaternary-amine-transporting ATPase 传输ATP酶活性的季铵盐化合物 orf01357 1 191 Nitric oxide dioxygenase 一氧化氮双加氧酶活性 orf01435 1 962 Threonine--tRNA ligase 1 ThrS ATP结合 orf01491 225 Spore germination protein GerE DNA结合 orf01591 657 GlnP 氨基酸转运与代谢 orf01592 651 GlnM 氨基酸转运与代谢 orf01593 828 GlnH 氨基酸转运与代谢 orf01617 801 Formate/nitrite transporter 跨膜转运蛋白活性 orf01659 726 RNA polymerase sigma factor DNA结合转录因子活性 orf01864 855 Nitrogen assimilation regulatory protein nac DNA结合转录因子活性 orf01911 450 Ferric uptake regulation protein DNA结合转录因子活性 orf01968 1 275 Glutamate dehydrogenase RocG 谷氨酸脱氢酶硝酸还原酶活性 orf02215 1 983 Nitrate reductase 硝酸还原酶活性 orf02225 903 HTH-type transcriptional regulator GltC DNA结合转录因子活性 orf02226 903 Glutamate synthase (NADPH/NADH) GltB 谷氨酸合酶 (NADPH) 活性 orf02227 4 560 Glutamate synthase (NADPH/ NADH) GltD 谷氨酸合酶 (NADPH) 活性 orf02368 1 335 Glutamine synthetase GlnA 谷氨酸氨连接酶活性 orf02369 405 HTH-type transcriptional regulator GlnR DNA结合 orf02384 315 Ammonium compound efflux SMR transporter 膜的组成部分 orf02385 354 Ammonium compound efflux SMR transporter 膜的组成部分 orf02417 261 Stage V sporulation protein S 核酸结合 orf02418 795 2',3'-cyclic-nucleotide 2'-phosphodiesterase YmdB 2',3'-环核苷酸2'-磷酸二酯酶活性 orf02465 723 Uridylate kinase PyrH ATP结合 orf02483 363 Chemotaxis protein CheY 磷脂酶信号转导系统 orf02588 783 RNA polymerase sigma factor DNA结合转录因子活性 orf02638 930 Glutaminase 氨基酸转运与代谢 orf02680 666 Potassium uptake protein KtrA 阳离子跨膜转运蛋白活性 orf02747 1 914 YkvW 阳离子转运ATP酶活性 orf02756 342 Putative transcriptional regulator 转录调控,DNA模板 orf02787 1 353 YkrM 阳离子跨膜转运蛋白活性 orf02794 783 Uncharacterized membrane protein YkoY 膜的组成部分 orf02804 742 HTH-type transcriptional regulator TnrA 核心启动子结合 orf02828 315 Ammonium compound efflux SMR transporter 膜的组成部分 orf02830 339 Ammonium compound efflux SMR transporter 膜的组成部分 orf02854 999 Anion permease 无机磷酸盐跨膜转运蛋白活性 orf03008 396 ArsC family transcriptional regulator SpxA 电子转移活性 orf03161 1 215 Cation/H (+) antiporter YhaU 溶质:质子逆向转运活性 orf03186 396 Putative fluoride ion transporter CrcB 无机阴离子跨膜转运蛋白活性 orf03187 360 Putative fluoride ion transporter CrcB 无机阴离子跨膜转运蛋白活性 orf03209 435 HTH-type transcriptional regulator NsrR DNA结合 orf03262 834 ABC-type nitrate transport system 离子跨膜转运 orf03263 990 Putative binding protein SsuA ATP酶活性 orf03264 768 Aliphatic sulfonates import protein SsuB 阴离子跨膜转运蛋白活性 orf03448 720 Probable transcriptional regulatory protein 转录调控,DNA模板 orf03546 330 Ammonium compound efflux SMR transporter 膜的组成部分 orf03547 315 Ammonium compound efflux SMR transporter 膜的组成部分 orf03597 963 Arsenic resistance protein 无机阴离子跨膜转运蛋白活性 orf03677 822 Probable manganese catalase YdbD 无机离子转运与代谢 orf03682 366 Ammonium compound efflux SMR transporter 膜的组成部分 orf03807 1 206 Assimilatory nitrate reductase NasA 硝酸铁氧还蛋白还原酶活性 orf03808 2 328 Nitrite reductase large subunit NasB 亚硝酸盐还原酶 [NAD(P)H] 活性 orf03809 2 133 Assimilatory nitrate reductase NasC 硝酸还原酶活性 orf03810 2 418 Nitrite reductase [NAD(P)H] NasD 亚硝酸盐还原酶 [NAD(P)H] 活性 orf03811 321 Assimilatory nitrite reductase [NAD(P)H] NasE 亚硝酸盐还原酶 [NAD(P)H] 活性 orf03812 1 440 NasF 辅酶代谢 orf03840 1 257 Transport system atp-binding protein opuaa 传输ATP酶活性的季铵盐化合物 orf03889 984 Glutaminase 1 GlsA1 谷氨酰胺酶活性 orf03890 1 437 GlnT 假定的钠/谷氨酰胺转运体 orf03963 1 434 Sodium-independent anion transporter 次级活性硫酸盐跨膜转运蛋白活性 -
[1] 胡晓娟, 文国樑, 田雅洁, 等. 不同培养条件下菌株NB5 对氨氮的去除效果研究[J]. 南方水产科学, 2020, 16(6): 89-96. [2] 方金龙, 王元, 房文红, 等. 氨氮胁迫下白斑综合征病毒对凡纳滨对虾的致病性[J]. 南方水产科学, 2017, 13(4): 52-58. doi: 10.3969/j.issn.2095-0780.2017.04.007 [3] 肖炜, 李大宇, 徐杨, 等. 慢性氨氮胁迫对吉富罗非鱼幼鱼生长、免疫及代谢的影响[J]. 南方水产科学, 2015, 11(4): 81-87. doi: 10.3969/j.issn.2095-0780.2015.04.012 [4] 郭国强, 孙红飞, 张永耀. 分子氨对草鱼鱼种红细胞渗透脆性的影响[J]. 水产科学, 2010, 29(8): 489-491. doi: 10.3969/j.issn.1003-1111.2010.08.011 [5] 孙侦龙, 朱永祥, 刘大勇, 等. 非离子氨氮和亚硝酸盐氮对暗纹东方鲀稚鱼的急性毒性试验[J]. 水产科学, 2015, 34(3): 135-139. [6] KUEBUTORNYE F K A, ABARIKE E D, LU Y. A review on the application of Bacillus as probiotics in aquaculture[J]. Fish Shellfish Immunol, 2019, 87: 820-828. doi: 10.1016/j.fsi.2019.02.010
[7] LIU G X, VIJAYARAMAN S B, DONG Y J, et al. Bacillus velezensis LG37: transcriptome profiling and functional verification of GlnK and MnrA in ammonia assimilation[J]. BMC Genom, 2020, 21(1): 215. doi: 10.1186/s12864-020-6621-1
[8] GOODWIN S, MCPHERSON J D, MCCOMBIE W R. Coming of age: ten years of next-generation sequencing technologies[J]. Nat Rev Genet, 2016, 17(6): 333-51.
[9] GALPERIN M Y, KRISTENSEN D M, MAKAROVA K S, et al. Microbial genome analysis: the COG approach[J]. Brief Bioinform, 2019, 20(4): 1063-1070. doi: 10.1093/bib/bbx117
[10] CASTAÑEDA C D, GAMBLE J N, WAMSLEY K G S, et al. In ovo administration of Bacillus subtilis serotypes effect hatchability, 21-day performance, and intestinal microflora[J]. Poult Sci, 2021, 100(6): 101125. doi: 10.1016/j.psj.2021.101125
[11] TAKAMI H, TOYODA A, UCHIYAMA I, et al. Complete genome sequence and expression profile of the commercial lytic enzyme producer Lysobacter enzymogenes M497-1[J]. DNA Res, 2017, 24(2): 169-177.
[12] MOLTON J S, LEE I R, BERTRAND D, et al. Stool metagenome analysis of patients with Klebsiella pneumoniae liver abscess and their domestic partners[J]. Int J Infect Dis, 2021, 107: 1-4. doi: 10.1016/j.ijid.2021.04.012
[13] 朱玉玲, 彭晶, 唐诗哲, 等. 宏基因组技术在极端环境酯酶挖掘中的应用[J]. 生命科学研究, 2021, 25(2): 169-175. [14] 魏大鹏, 单洪伟, 马甡, 等. 混料设计优化复合菌剂比例的研究[J]. 南方水产科学, 2014, 10(1): 86-91. doi: 10.3969/j.issn.2095-0780.2014.01.013 [15] TAMURA K, STECHER G, PETERSON D, et al. MEGA6: molecular evolutionary genetics analysis version 6.0[J]. Mol Biol Evol, 2013, 30(12): 2725-2729. doi: 10.1093/molbev/mst197
[16] NAKANO K, SHIROMA A, SHIMOJI M, et al. Advantages of genome sequencing by long-read sequencer using SMRT technology in medical area[J]. Hum Cell, 2017, 30(3): 149-161. doi: 10.1007/s13577-017-0168-8
[17] GAN Y Q, ZHANG T, GAN Y Q, et al. Complete genome sequences of two Enterococcus faecium strains and comparative genomic analysis[J]. Exp Ther Med, 2020, 19(3): 2019-2028.
[18] BENNETT J P Jr, KEENEY P M, BROHAWN D G. RNA sequencing reveals small and variable contributions of infectious agents to transcriptomes of postmortem nervous tissues from amyotrophic lateral sclerosis, Alzheimer's disease and Parkinson's disease subjects, and increased expression of genes from disease-activated microglia[J]. Front Neurosci, 2019, 13: 235. doi: 10.3389/fnins.2019.00235
[19] EMSLEY C, KING S, NYULASI I, et al. A GLIMmer of insight into lung transplant nutrition: enhanced detection of malnutrition in lung transplant patients using the GLIM criteria[J]. Clin Nutr, 2021, 40(5): 2521-2526. doi: 10.1016/j.clnu.2021.02.047
[20] CHAN P P, LOWE T M. tRNAscan-SE: searching for tRNA genes in genomic sequences[J]. Methods Mol Biol, 2019, 1962: 1-14.
[21] LAGESEN K, HALLIN P, RØDLAND E A, et al. RNAmmer: consistent and rapid annotation of ribosomal RNA genes[J]. Nucleic Acids Res, 2007, 35(9): 3100-8. doi: 10.1093/nar/gkm160
[22] CAMACHO C, COULOURIS G, AVAGYAN V, et al. BLAST+: architecture and applications[J]. BMC Bioinformatics, 2009, 10: 421. doi: 10.1186/1471-2105-10-421
[23] NAYAK V S. Nonelectrolytic production of caustic soda and hydrochloric acid from sodium chloride[J]. Ind Eng Chem Res, 1996, 35(10): 3808-3811. doi: 10.1021/ie960043h
[24] 张静, 高婷婷, 李勇, 等. 蛋白营养对工业养殖大菱鲆 (Scophthatmus maximus L.)幼鱼生长、氨氮排泄及肌肉氨基酸的效应[J]. 渔业科学进展, 2016, 37(6): 34-41. doi: 10.11758/yykxjz.20151124001 [25] EBELING J M, TIMMONS M B, BISOGNI J J. Engineering analysis of the stoichiometry of photoautotrophic, autotrophic, and heterotrophic removal of ammonia-nitrogen in aquaculture systems[J]. Aquaculture, 2006, 257(1/2/3/4): 346-358.
[26] 雷阳, 张倩, 陈钰, 等. 对虾养殖高效降解氨氮微生态菌的筛选与鉴定[J]. 福建农业科技, 2019(10): 16-20. [27] ZHU X H, ZHANG S M, ZHOU L Y, et al. Probiotic potential of Bacillus velezensis: antimicrobial activity against non-O1 Vibrio cholerae and immune enhancement effects on Macrobrachium nipponense[J]. Aquaculture, 2021, 541: 736817. doi: 10.1016/j.aquaculture.2021.736817
[28] 张德锋, 高艳侠, 可小丽, 等. 贝莱斯芽孢杆菌LF01基因组序列分析及其代谢产物的生防作用[J]. 水产学报, 2022, 46(2): 196-206. [29] FAN B, BLOM J, KLENK H P, et al. Bacillus amyloliquefaciens, Bacillus velezensis, and Bacillus siamensis form an "operational group B. amyloliquefaciens" within the B. subtilis species complex[J]. Front Microbiol, 2017, 8: 22.
[30] MURUKE M S H, CAMP H J O D, SEMESI A K, et al. The level of enzymes involved in the allantoin metabolism of Bacillus fastidiosus grown under different conditions[J]. Curr Microbiol, 1995, 30(1): 45-7. doi: 10.1007/BF00294523
[31] NIU T, LV X, LIU Z, et al. Synergetic engineering of central carbon and nitrogen metabolism for the production of N-acetylglucosamine in Bacillus subtilis[J]. Biotechnol Appl Biochem, 2020, 67(1): 123-132. doi: 10.1002/bab.1845
[32] WANG B, ZHANG D, CHU S, et al. Genomic analysis of Bacillus megaterium NCT-2 reveals its genetic basis for the bioremediation of secondary salinization soil[J]. Int J Genomics, 2020, 2020: 4109186.
[33] MASSAD R S, LOUBET B, TUZET A, et al. Relationship between ammonia stomatal compensation point and nitrogen metabolism in arable crops: current status of knowledge and potential modelling approaches[J]. Environ Pollut, 2008, 154(3): 390-403.
[34] TANG Y, YE Z, WEI Y, et al. Vertebrate paralogous CRMPs in nervous system: evolutionary, structural, and functional interplay[J]. J Mol Neurosci, 2015, 55(2): 324-34. doi: 10.1007/s12031-014-0327-2
[35] 顾志良, 耿拓宇. 鸡重要性状主效基因和QTL 的研究进展[J]. 中国家禽, 2003(S1): 130-134. [36] AWADA H, THAPA B, VISCONTE V. The genomics of myelodysplastic syndromes: origins of disease evolution, biological pathways, and prognostic implications[J]. Cells, 2020, 9(11): 2512. doi: 10.3390/cells9112512
[37] 丁忠涛, 张锐, 郭三堆. 棉花抗逆相关基因GhDr1的克隆及生物信息学分析[J]. 生物技术通报, 2011(1): 99-106. -
期刊类型引用(1)
1. 程思忠,谢岩黎,孙淑敏,马卫宾,李倩,杨玉辉. 黄曲霉毒素B_1降解菌的筛选鉴定及降解酶挖掘. 食品安全质量检测学报. 2023(04): 1-7 .  百度学术
百度学术
其他类型引用(3)



 下载:
下载:









 粤公网安备 44010502001741号
粤公网安备 44010502001741号
