Detection of antibiotics resistance and distribution of resistance genes in Vibrio parahaemolyticus from cultured shrimp
-
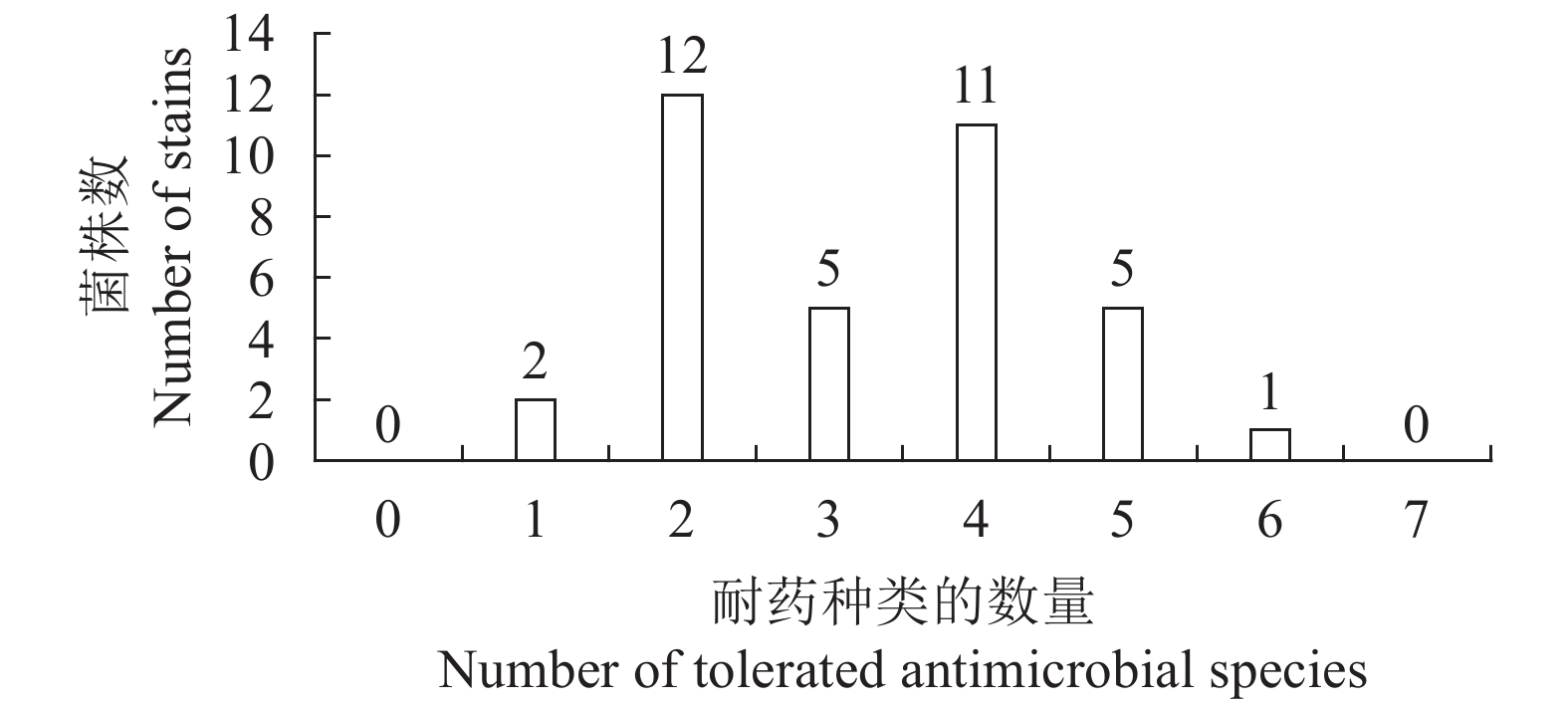
摘要: 文章采用琼脂稀释法检测从养殖患病对虾中分离的36株副溶血弧菌(Vibrio parahaemolyticus)对16种药物的耐药性,并用PCR法检测喹诺酮类耐药基因qnrA、qnrB、qnrS和qnrVC,酰胺醇类耐药基因cat、optrA、floR和cfr,四环霉素类耐药基因tetA、tetB和tetM,磺胺类耐药基因sul1、sul2和sul3,氨基糖苷类耐药基因strA、strB、aadA和aacA,利福霉素类耐药基因arr,β-内酰胺类耐药基因blaCARB和大环内酯类耐药基因erm的携带状况,分析其耐药表型和基因型之间的相关性。结果显示,36株副溶血弧菌对β-内酰胺类药物氨苄西林耐药率最高(88.9%),其次为磺胺类药物磺胺甲噁唑(66.7%),硫酸新霉素、庆大霉素、头孢曲松和美罗培南呈现100%敏感。多重耐药副溶血弧菌比例高达61.1% (22/36),其中1株对6类抗菌药耐药。喹诺酮类耐药基因qnrVC检出率最高达72.2% (26/36);其次为氨基糖苷类耐药基因srtB,检出率58.3% (21/36);大环内酯类、利福霉素类耐药基因均未检测到。耐药基因检出率与耐药表型没有表现出一一对应的关系,提示副溶血弧菌耐药的复杂性。Abstract: We used agar dilution method to detect the resistance of 36 strains of Vibrio parahaemolyticus to 16 drugs. And used PCR amplication and DNA sequencing to detect the antimicrobial resistance to quinolones (qnrA, qnrB, qnrS, qnrVC), phenicols (cat, optrA, floR, cfr), tetracyclines (tetA, tetB, tetM), sulfonamides (sul1, sul2, sul3), aminoglycosides (strA, strB, aadA, aacA), rifampicin (arr), β-lactams (carB) and macrolides (erm). The results indicate that the isolates exhibited high resistance to ampicillin (88.9%) and sulfamethoxazole (SMZ, 66.7%), all susceptible to neomycin sulfate, gentamicin, ceftriaxone and meropenem. In general, multi-drug resistance (MDR) was highly prevalent (61.1%), and one isolate was resistant to six antimicrobials. Furthermore, 72.2% and 58.3% of the isolates were primarily mediated by qnrVC and strB, respectively; and the macrolides and rifamycin resistant genes were not detected in all the isolates. Obvious mismatch was found between the antimicrobial resistance phenotypes and genotypes, revealing the complexity of resistance to V. parahaemolyticus.
-
刀额新对虾(Metapenaeus ensis)隶属于节肢动物门、甲壳纲、十足目、对虾科、新对虾属[1-2]。新对虾属是南海海域经济虾类中最重要的属,既是捕捞种类,也是养殖品种[3]。刀额新对虾是南海海域新对虾属中重要的经济种,资源量丰富、分布广、繁殖期长[2],是南海海域虾拖网的主要捕捞对象,渔汛旺期其产量占虾拖网虾类总产量的50%以上[4-5]。由于虾拖网网囊网目尺寸小、选择性差,导致捕获的刀额新对虾幼虾比例较高,有时甚至高达90%[5],严重影响虾资源的合理利用。因此,有必要对南海海域虾拖网的网囊进行优化设计,以降低对刀额新对虾幼虾的捕捞比例,减少捕捞作业对渔业资源补充群体的不良影响。
明确拖网网囊对主要捕捞种类的选择性是渔具管理、渔具优化和资源利用的基础[6-7]。目前,已对南海海域刀额新虾进行过食性、营养级和群体形态学等方面的专题研究[8-9];关于网囊对刀额新对虾的选择性有过少量报道[10-12],但是关于虾拖网网囊网目对刀额新对虾选择性的系统、专题研究尚未见报道。本文在整理和总结南海海域虾拖网网囊系列选择性试验数据的基础上,专题开展网囊对刀额新对虾的选择性研究,为南海海域刀额新对虾资源的合理利用和虾拖网渔具的设计优化提供科学依据。
1. 材料与方法
1.1 试验渔船、时间和区域
试验于2014—2017年分4个航次进行(表1),船名分别为“粤阳东渔12057”(船长16 m、主机功率79 kW)和“粤阳东渔12081”(船长21 m、主机功率98 kW)。试验区域为南海区近海渔场,经纬度范围为112°03'E~112°22′E、21°37'N~21°50′N,水深6~13 m,底质为沙泥。
表 1 网囊选择性试验基本信息Table 1. Basic information of tested codends航次
trial时间
time网囊
codend试验渔船
vessel方法
method网目内径/mm
mesh opening网囊规格 (圆周×纵向)
codend specification
(circumferernce×vertical)有效网次
valid haul1 2014.08 D25 粤阳东渔 12057YYDY12057 套网法 covered 21.10±0.93 58×60 6 1 2014.08 D30 粤阳东渔 12057YYDY12507 套网法 covered 26.64±0.73 48×50 8 2 2015.08 S35+D18 粤阳东渔 12081YYDY12081 套网法 covered S: 32.70±0.63
D: 15.07±0.36S: 29×43,D: 80×42 8 3 2016.08 S25+D25 粤阳东渔 12081YYDY12081 套网法 covered S: 21.80±0.42
D: 21.80±0.42S: 40×60,D: 58×30 11 3 2016.08 S30+D25 粤阳东渔 12081YYDY12081 套网法 covered S: 27.14±0.45
D: 21.80±0.42S: 35×50,D: 58×30 12 3 2016.08 S35+D25 粤阳东渔 12081YYDY12081 套网法 covered S: 32.42±0.18
D: 21.80±0.42S: 29×43,D: 58×30 10 4 2017.09 S35+D25 粤阳东渔 12081YYDY12081 裤网法 trouser S: 32.42±0.18
D: 21.80±0.42S: 23×29,D: 81×20 11 4 2017.09 S35+D30 粤阳东渔 12081YYDY12081 裤网法 trouser S: 32.42±0.18
D: 27.14±0.45S: 23×29,D: 67×17 10 4 2017.09 S35+D35 粤阳东渔 12081YYDY12081 裤网法 trouser S: 32.42±0.18
D: 32.42±0.18S: 23×29,D: 58×14 8 合计 total 84 注:S. 方形网目;D. 菱形网目
Note: S. square mesh; D. diamond mesh1.2 试验网具与网囊
试验网具为南海区最常见的双桁杆虾拖网(以下简称虾拖网),其原主尺度为10.50 m×7.15 m(2.2 m)。关于虾拖网的结构特征、渔法特点和渔获组成等信息,可参阅相应的参考文献[5,13-14]。
2014—2017年,采用套网法和裤式拖网法(简称裤网法)依次对2组菱目网囊和6组菱目与方目混合网囊(简称混目网囊)进行了84个有效网次作业试验(表1)。套网法的试验网囊拉直长度约为1.5 m;裤网法试验网囊和对照网囊的拉直长度约为1.2 m。
试验网囊的网目尺寸分4档:18 mm、25 mm、30 mm和35 mm。其中18 mm是南海海域虾拖网普遍使用的网囊网目尺寸;25 mm为现行虾拖网管理规定中的网囊最小网目尺寸;30 mm和35 mm为试验网网囊尺寸。按照网目尺寸对网囊进行编号,例如D30表示网目尺寸为30 mm的菱目网囊;S30+D25表示方目段网目尺寸为30 mm,菱目段网目尺寸为25 mm的混目网囊。套网法试验中套网的网目尺寸为15 mm,内径为(12.55±0.28) mm,圆周286目,纵向136目。裤网法中对照网囊第一段网目尺寸为24 mm,第二段为22 mm;对照网囊第二段网目内径为(19.49±0.63) mm。试验网具和网囊的具体参数详见表1和图1、图2。
1.3 试验与取样方法
为了方便操作,将试验网设置于渔船船首方向右舷第一顶网处。试验中不设置站点,也不规定渔船的拖曳速度、时间等作业参数,由船长根据实际情况控制,使试验尽量与传统虾拖网渔船作业保持一致。试验期间渔船拖速2.2~2.7 kn,网次作业时间1.5~3.5 h,平均1.75 h,网次平均拖曳距离9.01 km。
起网后,分别称量试验网囊和套网(或对照网囊)的渔获总质量,然后进行分类和鉴定,统计每个种类的渔获数量,并进行生物学测量。当刀额新对虾渔获数量≤50尾时,全部测量其长度;当渔获数量>50尾时,随机抽取50尾进行长度测量。在统计渔获种类的体长分布时,对于取样种类按取样比例进行加权处理。为了保证试验数据的准确性,尽量避免渔获物的2次取样。
1.4 数据处理的方法
1.4.1 基础数据的统计
以航次为单位统计虾拖网网囊和套网(或对照网囊)的渔获总数和总质量,分别计算各网囊刀额新对虾的数量和质量比例。将每个网次中网囊和套网(或对照网囊)的刀额新对虾体长数据进行排序,以5 mm为单位进行分组,分别统计各体长组刀额新对虾的数量。
1.4.2 选择性参数的估算
网囊对刀额新对虾的选择性估算按如下2个步骤进行:1)以网次为单位,使用Logistic曲线进行选择性模型拟合,估算网次选择性参数;2)将选择性模型拟合度较好的网次体长数据进行叠加,再次使用Logistic曲线进行选择性模型拟合,估算网囊对刀额新对虾的平均选择性参数,同时考虑网囊网次间差异对选择性的影响。
选择性参数的估算是在SELECT模型[6,15-18]的框架下,通过比较试验网囊和套网(或对照网囊)的刀额新对虾渔获数量,得到估算结果。在第i网次中,用NTij表示体长为lj的刀额新对虾被试验网囊捕获的数量,用NCij表示套网(或对照网囊)中刀额新对虾(体长亦为lj)的渔获数量,则体长为lj的刀额新对虾被试验网囊捕获的概率(φij)可表示为:
$${{\textit{φ}} _{ij}} = \frac{{{N_{Tij}}}}{{{N_{Tij}} + {N_{Cij}}}}$$ (1) 对于套网法试验,使用logistic方程拟合试验网囊对体长为l的刀额新对虾的选择率r(l),其计算公式为:
$$r(l) = \frac{{\exp(a + bl)}}{{1 + \exp(a + bl)}}$$ (2) 式中a、b为待估参数。
对于裤网法试验,引入相对作业强度(p),表示1尾刀额新对虾进入试验网囊的概率,则进入对照网囊的概率为1−p。体长为l的刀额新对虾被试验网囊捕获的概率可表示为:
$$\varphi(l) = \frac{{p\exp(a + bl)}}{{(1 - p) + \exp(a + bl)}}$$ (3) 根据选择性参数a和b,计算试验网囊对刀额新对虾的选择性指标50%选择体长(L50)和选择范围(SR),相应的计算公式为:
$${L_{50}} = - a/b$$ (4) $${\rm SR} = {L_{75}} - {L_{25}} = 2\ln 3/b$$ (5) 通过极大似然法估算选择性参数a、b和p,其似然方程[7]为:
$$L(a,b,p) = \sum\limits_i {\sum\limits_j {[{N_{Tij}}\ln \varphi({l_j}) + {N_{Cij}}\ln(1 - \varphi({l_j}))]} } $$ (6) 式(6)的求解通过MS-Excel的“规划求解”功能实现[19-20]。参数a、b、p和选择性指标L50、SR的标准差通过Delta方法估算[6,17,21]。
选择性模型拟合度的优劣可根据残差值(deviance)和自由度(degree of freedom, df )的大小关系判断。通常当残差值小于自由度(P>0.05),则认为模型拟合度为优。但当试验数据呈离散分布时,即使拟合度为优也会出现残差值远大于自由度(P<0.05)的情况。这时需要根据各体长组的实际残差值分布图作进一步判断[6,12,22]。
通过比较联合网次模型拟合的皮尔逊卡方统计量(Q)和自由度(d)的大小来判断网囊选择性是否存在网次间差异。当不存在网次间差异时,Q服从自由度为d的卡方分布(P>0.05),反之则说明存在网次间差异[6,17,21-25]。如果存在网次间差异,计算过度离散叠加估算值(replication estimate of dispersion, REP),并对联合网次估算的选择性参数标准差进行修正,即乘以(REP)1/2。Q和REP的计算公式为:
$$Q = \sum\limits_l {\sum\limits_h {\frac{{{{(n_{lt}^h - n_{l + }^h{y_l})}^2}}}{{n_{l + }^h{y_l}{{(1 - {y_l})}^{}}}}} } $$ (7) 式中
$n_{lt}^h$ 表示第h网次中试验网囊捕获的体长为l的刀额新对虾数量,$n_{l + }^h$ 表示第h网次中试验网囊与套网(或对照网囊)捕获的体长为l的刀额新对虾总数量,yl表示h网次中试验网囊捕获的体长为l的刀额新对虾的数量比例。$${\rm REP} = \frac{Q}{d}$$ (8) 式中d=(H−1)×体长组数,H为联网次的总网次数。
2. 结果
2.1 渔获概述
试验渔获质量为294.68 kg,刀额新对虾总渔获质量为100.71 kg,占渔获总质量的34.18%。各试验网囊中的总渔获质量及刀额新对虾所占比例详见表2。
表 2 试验渔获产量基本信息Table 2. Basic information of fish catch方法
method网囊
codend质量/kg mass 质量百分比/%
ratio of mass刀额新对虾
M. ensis总渔获
total catch套网法 cover D25 8.80 27.92 31.50 D30 13.43 45.01 29.83 S35+D18 9.50 28.53 33.28 S25+D25 15.36 23.36 65.77 S30+D25 4.94 33.61 14.70 S35+D25 20.13 37.60 53.53 裤网法 trouser S35+D25 18.47 32.47 56.89 S35+D30 7.18 42.91 16.73 S35+D35 2.92 23.27 12.53 合计 total 100.73 294.68 34.18 2.2 刀额新对虾的渔获数量及体长分布
试验中刀额新对虾的渔获总数量为14 138尾,各网囊和套网(或对照网囊)的数量、体长范围、众数体长及其比例等数据详见表3,体长分布见图3和图4。
表 3 刀额新对虾的渔获数量及体长分布Table 3. Number of M. ensis caught by each codend网囊
codend渔获尾数
number of catch体长范围/mm
length range众数体长/mm
modal length网囊
codend套网/对照网
cover/control网囊
codend套网/对照网
cover/control网囊
codend套网/对照网
cover/controlD25 964 2 53~123 33~58 88~93 (38.38%) D30 1 698 3 53~113 53~63 83~88 (51.94%) S35+D18 1 810 28 63~128 63~98 78~83 (37.07%) 73~78 (67.86%) S25+D25 2 307 384 58~103 28~93 78~83 (48.63%) 43~48 (39.84%) S30+D25 499 33 63~103 63~98 83~88 (46.49%) 73~78 (42.42%) [S35+D25]1 3 008 117 58~118 28~98 78~83 (41.42%) 73~78 (42.74%) [S35+D25]2 1 121 1 366 63~133 63~133 83~88 (58.25%) 83~93 (65.37%) S35+D30 283 270 63~133 73~133 93~103 (55.83%) 93~103 (60.00%) S35+D35 113 132 63~128 68~133 93~98 (47.79%) 93~98 (46.97%) 合计 total 11 803 2 335 注:[S35+D25]1. 套网法中的S35+D25网囊;[S35+D25]2. 裤网法中的S35+D25网囊
Note: [S35+D25]1. the S35+D25 codend for the covered codend method; [S35+D25]2. the S35+D25 codend for the trouser trawl method2.3 网囊对刀额新对虾的选择性
2.3.1 套网法
D25和D30网囊由于套网的刀额新对虾样本量过少,无法进行选择性分析;S35+D18网囊仅1个单网次数据获得有效解,模型的拟合度较好(P>0.05),刀额新对虾的L50和SR分别为51.25 mm和16.48 mm;S25+D25网囊共5个单网次数据获得有效解,网次数据拟合度均较好(P>0.05),联合网次拟合度P<0.05,但模型残差值分布正常,所以认为是由于体长数据的过度离散分布造成,联合网次估算的刀额新对虾平均L50和SR分别为60.84 mm和14.31 mm,存在网次间差异,对估算参数的标准差进行了修正;S30+D25网囊仅2个单网次数据获得有效解,单网次和联合网次拟合度较好(P>0.05),联合网次估算的刀额新对虾平均L50和SR分别为63.21 mm和12.84 mm,不存在网次间差异;S35+D25网囊共5个单网次数据获得有效解,1个单网次和联合网次拟合的P<0.05,但模型残差值分布正常,联合网次估算的刀额新对虾平均L50和SR分别为64.53 mm和9.75 mm,存在网次间差异,对估算参数的标准差进行了修正(表4)。各网囊对刀额新对虾的平均选择性曲线详见图5。
表 4 套网法选择性试验参数估算Table 4. Selective parameters of tested codends with covered codend method时间
time网囊
codend网次
NH选择性指标
selective index选择性参数
selective parameter拟合度
goodness of fit网次间差异
estimate of REP尾数
numberL50/
mmSD SR/
mmSD a SD b SD D df P Q d P REP 网囊
codend套网
cover2014 D25 c-b 964 2 2014 D30 c-b 1 698 3 2015 S35+D18 9 51.25 8.94 16.48 7.20 –6.83 4.14 0.13 0.06 4.295 8 0.83 203 11 2016 S25+D25 2 59.65 5.84 21.51 7.41 –6.09 2.66 0.10 0.04 5.919 7 0.55 137 24 4 53.89 12.68 50.44 26.94 –2.35 1.79 0.04 0.02 10.608 7 0.16 120 44 6 53.31 7.79 30.96 11.92 –3.78 1.98 0.07 0.03 6.283 9 0.71 127 31 9 60.86 1.19 6.05 1.07 –22.12 4.21 0.36 0.06 14.094 14 0.44 367 64 10 60.94 1.70 5.18 1.21 –25.84 6.39 0.42 0.10 2.288 14 1.00 278 75 c-b 60.84 1.74 14.31 1.90 –9.34 1.44 0.15 0.02 51.617 14 <0.05 155.94 39 <0.05 4.00 1 029 238 S30+D25 4 61.48 7.89 11.78 6.01 –11.47 7.25 0.19 0.10 9.811 10 0.46 106 4 5 63.18 3.47 13.93 3.23 –9.97 2.80 0.16 0.04 8.822 6 0.18 233 17 c-b 63.21 2.97 12.84 2.66 –10.82 2.70 0.17 0.04 3.657 9 0.93 20.22 21 0.51 0.96 342 21 S35+D25 1 66.16 1.22 3.56 0.89 –40.86 10.77 0.62 0.15 7.000 11 0.80 443 16 5 68.58 1.61 7.76 1.83 –19.42 4.92 0.28 0.07 9.946 6 0.13 188 17 7 62.43 3.45 9.00 2.31 –15.25 4.70 0.24 0.06 22.166 4 <0.05 356 15 8 65.19 3.10 10.64 2.78 –13.46 4.11 0.21 0.05 14.343 7 0.05 279 18 9 71.90 3.55 14.86 5.32 –10.63 4.20 0.15 0.05 5.303 6 0.51 40 12 c-b 64.53 1.15 9.75 1.00 –14.54 1.72 0.23 0.02 37.441 6 <0.05 72.31 71 0.43 1.02 1 306 78 注:NH. 网次;c-b. 联合网次;L50. 50%选择体长;SR. 选择范围;a、b. 选择性参数;SD. 标准差;D. 残差值;dof和d. 自由度;Q. 皮尔逊卡方统计量;REP. 过度离散叠加估算值. Note: NH. number of hauls; c-b. combined hauls; L50. 50% retention length; SR. selection range; a and b are selective parameters; SD. standard error; D. value of model deviance; dof and d indicate the degree of freedom; Q. Pearson chi-square statistic; REP. replication estimation of dispersion 2.3.2 裤网法
S35+D25网囊共8个单网次数据获得有效解,4个单网次和联合网次拟合的P<0.05,但模型残差值分布正常,联合网次估算的刀额新对虾平均L50、SR和相对作业强度(p)分别为75.43 mm、6.93 mm和0.51,存在网次间差异,对选择性参数的标准差进行了修正;S35+D30网囊共5个单网次数据获得有效解,单网次和联合网次的拟合度较好(P>0.05),联合网次估算的刀额新对虾平均L50、SR和p分别为82.38 mm、6.39 mm和0.52,不存在网次间差异;S35+D35网囊共4个网次获得有效解,所有单网次和联合网次估算的拟合度均较好(P>0.05),联合网次估算的刀额新对虾平均L50、SR和p分别为95.39 mm、20.44 mm和0.64,不存在网次间差异(表5)。各网囊对刀额新对虾的平均选择性曲线详见图6。
表 5 裤网法选择性试验参数估算Table 5. Selective parameters of tested codends with trouser trawl method时间
time网囊
codend网次
NH选择性指标
selective index选择性参数
selective parameter拟合度
goodness of fit网次间差异
estimate of REP尾数
numberL50/mm SD SR/mm SD a SD b SD P SD D df P Q d P REP 网囊
codend对照
control2017 S35+D25 2 78.16 3.15 3.74 4.45 –45.88 53.61 0.59 0.70 0.41 0.07 8.262 9 0.51 24 43 3 75.35 3.03 7.17 4.69 –23.08 14.44 0.31 0.20 0.46 0.07 8.968 6 0.18 55 101 4 73.52 1.40 3.45 2.58 –46.77 34.53 0.64 0.48 0.41 0.03 39.625 10 <0.05 149 244 6 78.46 7.90 7.46 6.76 –23.12 19.45 0.29 0.27 0.77 0.10 10.982 10 0.36 28 12 7 72.79 5.32 6.92 7.07 –23.12 22.96 0.32 0.32 0.51 0.06 9.766 8 0.28 54 57 8 75.98 2.38 8.55 3.66 –19.52 7.89 0.26 0.11 0.54 0.04 47.531 6 <0.05 244 288 9 73.52 1.47 4.22 3.61 –38.32 32.46 0.52 0.45 0.51 0.03 29.720 7 <0.05 182 193 11 85.59 4.74 10.74 3.67 –17.51 5.22 0.20 0.07 0.73 0.08 25.517 10 <0.05 116 108 c-b 75.43 1.36 6.93 2.14 –23.93 7.09 0.32 1.00 0.51 0.03 53.559 12 <0.05 152.00 81 <0.05 1.88 852 1046 S35+D30 1 78.84 6.40 7.68 8.90 –22.57 25.18 0.29 0.33 0.65 0.08 15.361 8 0.05 30 20 5 86.79 4.96 6.92 6.25 –27.56 24.11 0.32 0.29 0.50 0.07 9.579 11 0.57 39 48 7 81.18 3.61 5.01 4.56 –35.60 31.57 0.44 0.40 0.44 0.05 12.484 11 0.33 62 90 8 88.47 5.70 11.32 7.00 –17.17 9.82 0.19 0.12 0.60 0.08 14.706 11 0.20 64 65 9 92.09 8.49 9.80 7.41 –20.64 14.18 0.22 0.17 0.71 0.14 5.986 8 0.65 23 19 c-b 82.38 2.01 6.39 2.72 –28.32 11.68 0.34 0.15 0.52 0.03 14.308 8 0.07 44.21 50 0.70 0.88 218 242 S35+D35 1 83.59 7.52 8.72 9.18 –21.06 21.32 0.25 0.27 0.55 0.10 10.743 10 0.38 23 24 2 92.13 8.37 9.62 9.67 –21.03 19.67 0.23 0.23 0.65 0.14 6.200 7 0.52 24 22 3 110.78 31.97 20.43 19.45 –11.91 8.42 0.11 0.10 0.77 0.29 6.397 9 0.70 17 19 5 100.52 37.58 29.78 31.29 –7.42 5.58 0.07 0.08 0.65 0.30 10.246 11 0.51 36 47 c-b 95.39 10.43 20.44 10.51 –10.25 4.44 0.11 0.06 0.64 0.11 11.599 11 0.39 22.73 34 0.93 0.67 100 112 3. 讨论
3.1 菱目网囊与混目网囊的选择性比较
目前,南海海域传统虾拖网均使用菱目网囊作业,且其网目尺寸较小(一般<20 mm)。据相关调查,当虾拖网网囊网目尺寸为18 mm时,刀额新对虾的幼虾比例>70%[5]。本研究2014年对菱目网囊D25和D30进行试验,结果表明对于刀额新对虾而言,网目尺寸增大,网囊选择性未见显著提高。由于套网的样本量太少,上述2种菱目网囊对刀额新对虾的选择性参数也无法估算。
鉴于菱目网囊选择性差的结论,2015年研究团队首次尝试使用混目网囊(S35+D18)进行试验;该混目网囊菱目段网目尺寸与渔船所用网囊一致(18 mm),方目段网目尺寸为35 mm。结果表明,副渔获物(鱼类)的逃逸率显著提高,但目标种类的逃逸率仍然较低;刀额新对虾的L50太小(53.85 mm),远小于南海区刀额新对虾的首次性成熟体长(80 mm)[26],网囊网目尺寸需要进一步优化[11]。本研究采用的是单网次数据处理方法,得出S35+D18网囊对刀额新对虾的L50仅为51.52 mm,小于联合网次数据处理得到的值。此结论也进一步说明S35+D18网囊的网目尺寸需要进一步放大。
3.2 混目网囊之间的选择性比较
2016年使用3组混目网囊(S25+D25、S30+D25和S35+D25)进行试验,网囊菱目段网目尺寸均为25 mm,方目段网目尺寸分别为25 mm、30 mm和35 mm。根据前期联合网次分析法的结果,S25+D25和S35+D25网囊对刀额新对虾的L50分别为63.93 mm和67.96 mm; S30+D25网囊的刀额新对虾体长组数据不收敛,无法取得有效解[12]。本研究得出的S25+D25、S30+D25和S35+D25网囊对刀额新对虾的平均L50分别为60.84 mm、63.21 mm和64.53 mm。使用本研究的数据处理方法能够获得S30+D25网囊对刀额新对虾的选择性。这从侧面说明了如果在数据处理时完全忽略网囊的网次间差异,简单地将所有网次的体长组数据进行无条件叠加处理可能会得到错误的结论。本试验采用“2部曲”(two-step procedure)法:先以网次为单位进行数据处理,然后根据模型的拟合结果将一些网次数据剔除,在考虑网囊网次间差异的基础上叠加网次数据,求得平均选择性。这种数据处理方法得出的结论更加准确、客观和科学,国外一些相关研究也普遍采用这种方法进行数据处理[27-29]。
2017年,笔者课题组对3组混网囊(S35+D25、S35+D30和S35+D35)进行试验。此次网目尺寸的选择是以2014—2016年试验结论为基础,将方目段网目尺寸保持为35 mm,菱目段网目尺寸分别为25 mm、30 mm和35 mm。同时,为了避免套网法的“覆盖效应”,在试验方法上首次尝试采用目前国际上普遍使用的裤网法。根据试验结果,S35+D25、S35+D30和S35+D35网囊对刀额新对虾的平均L50随菱目段网目尺寸的增大而不断增大,SR则是先减少后增大,p不断增大。根据陈丕茂[26]的研究,南海区刀额新对虾的首次性成熟体长为80 mm。假设将刀额新对虾的可捕规格定为80 mm,所有试验网囊中仅S35+D30和S35+D35网囊的L50大于该可捕标准。但是,S35+D30网囊对刀额新对虾的SR远小于S35+D35的(6.39 mm vs 20.44 mm),说明S35+D30网囊的选择性尖锐度较好。另外,考虑到网目尺寸过大会影响到渔船的生产效益,同时兼顾渔民的心理接受程度,笔者认为将S35+D30网囊作为南海区虾拖网网囊最小网目尺寸标准较为理想。
3.3 存在的问题及今后的研究方向
本研究中的试验数据均在渔船上即时收集。由于网次间间隔短,测量时间有限,对于渔获数量较多的种类(>50尾),先统计该种类的总数量后,随机抽取50尾渔获进行体长测量。选择性数据处理时,以随机测量的50尾体长数据为基础,按照相应的取样比例进行加权处理,最后得出网次渔获种类的体长分布数据。但有些前期的研究则直接使用实测体长组数据进行选择性参数估算[12,15-16]。使用实测体长组数据和加权处理后的数据进行估算的结果是否存在显著差异?哪个方法计算的结果更加准确和科学?这将是未来的一个研究方向。
本研究存在的不足之处在于4个航次试验存在一定的时空差异。虽然网囊对刀额新对虾的选择性主要由网囊结构、网目尺寸和刀额新对虾的体型特征决定,但是为了减少时空差异对选择性研究的影响,今后可尝试将所有试验网囊在同一渔场、同一渔船和同一网次进行试验。南海区虾拖网通常是一船拖曳多顶网具。比如,本研究中租用的“粤阳东渔12081”作业中拖曳12顶网,一些大功率渔船甚至最多可拖曳32顶网。利用南海区虾拖网这种独特的作业方式,可将本研究中的所有试验网囊共同进行试验。同时,在试验方法上也可以将套网法、裤网法等进行有机结合,优势互补,相互印证。这样获得的试验数据会更加全面,得出的结论能够更好地为渔具管理和渔业资源的合理利用提供科学依据。
-
表 1 抗菌药物稀释范围、质控菌株质控范围及药敏试验结果判定标准
Table 1 Dilution range of antibacterial drugs, quality control range of quality control strains and criteria for determination of drug susceptibility test results
抗菌药物Antimicrobial 稀释范围Dilution range/(μg·mL−1) MIC判定标准Judgment standard/(μg·mL−1) ATCC25922质控范围Quality control range/(μg·mL−1) S I R 环丙沙星 Ciprofloxacin 0.12~64 ≤1 2 ≥4 0.004~0.015 诺氟沙星 Norfloxacin 0.06~32 ≤4 8 ≥16 0.03~0.12 恩诺沙星 Enrofloxacin 0.06~32 ≤0.5 1~2 ≥4 0.03~0.12 氟苯尼考 Florfenicol 0.25~128 ≤2 4 ≥8 2~8 氯霉素 Chloramphenicol 0.25~128 ≤8 16 ≥32 2~8 土霉素 Oxytetracycline 0.12~64 ≤4 8 ≥16 0.5~4 多西环素 Doxycycline 0.12~64 ≤4 8 ≥16 0.5~2 磺胺甲噁唑 Sulfamethoxazole 5~2 560 ≤256 − ≥512 4~32 复方新诺明 Compound sulfamethoxazole 0.125/2.375~64/1 216 ≤2/38 − ≥4/76 ≤0.5/9.5 硫酸新霉素 Neomycin sulfate 0.06~32 ≤2 4 ≥8 0.5~4 庆大霉素 Gentamicin 0.06~32 ≤4 8 ≥16 0.25~1 红霉素 Ergomycin 0.5~512 ≤0.5 1~4 ≥8 2~8 头孢曲松 Ceftriaxone 0.12~64 ≤1 2 ≥4 0.03~0.12 氨苄西林 Ampicillin 0.25~128 ≤8 16 ≥32 2~8 美罗培南 Meropenem 0.125~64 ≤4 8 ≥16 0.008~0.06 利福平 Rifampin 1~512 ≤1 2 ≥4 4~16 注:−. 缺少相应的参考数据 Note: −. Lack of corresponding reference data 表 2 耐药基因引物序列
Table 2 Primers sequences of drug resistance genes
基因名称Gene name 引物序列 (5'–3')Primer sequence 退火温度Annealing temperature/℃ 产物长度Length of product/bp 参考文献Reference qnrVC136 TTCTCACATCAGGACTTGCGGAACAATGATTACCCCT 55 615 [23] qnrVC457 ATAAAACAGACCAGTTATATGTACTATTAAACVCTAATTGCTCTA 55 630 [23] qnrA ATTTCTCACGCCAGGATTTGGATCGGCAAAGGTTAGGTCA 55 516 [24] qnrB GATCGTGAAACGCAGAAAGGACGATGCCTGGTAGTTGTCC 50 469 [25] qnrS ACGACATTCGTCAACTGCAATAAATTGGCACCCTGTAGGC 52 417 [26] tetA TTTCGGGTTCGGGATGGTCAGGCAGAGCAAGTAGAGG 55 696 [23] tetM GTGGACAAAGGTACAACGAGCGGTAAAGTTCGTCACACAC 55 405 [23] tetB GTCGCGGCATCGGTCATTTTTTCGCCCCATTTAGTG 55 489 [23] Sul1 GTGACGGTGTTCGGCATTCTTCCGAGAAGGTGATTGCGCT 63 779 [23] Sul2 GCGCAGGCGCGTAAGCTGATCGAAGCGCAGCCGCAATTC 65 793 [27] Sul3 GCAACAGTTGGTGCTAAACGAGAAGCAGATGTGATTGATTTGGGAG 56 578 [23] strA TGGCAGGAGGAACAGGAGGAGGTCGATCAGACCCGTGC 54 546 [27] strB ATCGTCAAGGGATTGAAACCGGATCGTAGAACATATTGGC 52 509 [23] aadA GGAGAATGGCAGCGCAATGTTACTGCGCTGTACCAAT 55 269 [23] aacA CTTGGRTGACCTCGGGATCCGATGCTCTATGAGTGGCTAA 54 344 [23] erm CCCGAAAAATACGCAAAATTTCATCCCTGTTTACCCATTTATAAACG 55 589 [23] cat ACAACAGCAACGGTACTAGCCAACTTTCACCGATGCCAC 52 550 [28] optrA TTCTCACCCAGATATGCCCGGGATCCCGGCAAACT 60 1395 [29] floR CTGCTGATGGCTCCTTTCGCCGTGGCGTAACAGAT 57 650 [30] cfr GTGAAGCTCTAGCCAACCGTCGCAGCGTCAATATCAATCCC 55 746 [31] blaCARB GCTGAGAGCTCATGAAAAAGTTACGTAGGATCCTTAACTTTATTTGTAGTGC 55 852 [32] 表 3 36株副溶血弧菌耐药状况
Table 3 Antibiotics resistance status of 36 strains of V. parahaemolyticus
抗菌药物Antimicrobial 耐药菌株数Number of resistant strains 抗菌药物Antimicrobial 耐药菌株数Number of resistant strains 恩诺沙星 Enrofloxacin 2 (5.6%) 复方新诺明 Compound sulfamethoxazole 8 (22.2%) 诺氟沙星 Norfloxacin 6 (16.7%) 硫酸新霉素 Neomycin sulfate 0 (0) 环丙沙星 Ciprofloxacin 6 (16.7%) 庆大霉素 Gentamicin 0 (0) 氟苯尼考 Florfenicol 4 (11.1%) 红霉素 Erythromycin 23 (63.9%) 氯霉素 Chloramphenicol 2 (5.6%) 头孢曲松 Ceftriaxone 0 (0) 土霉素 Oxytetracycline 17 (47.2%) 氨苄西林 Ampicillin 32 (88.9%) 多西环素 Doxycycline 4 (11.1%) 美罗培南 Meropenem 0 (0) 磺胺甲噁唑 Sulfamethoxazole 24 (66.7%) 利福平 Rifampin 9 (25%) 表 4 副溶血弧菌耐药基因携带率
Table 4 Drug resistance gene carrier rate of V. parahaemolyticus
耐药基因Resistance gene 检出率Detection rate/% 耐药基因Resistance gene 检出率Detection rate/% 耐药基因Resistance gene 检出率Detection rate/% qnrVC 72.2 cfr 22.2 strA 44.4 qnrA 41.7 tetA 16.7 strB 58.3 qnrB 0 terB 47.2 aadA 33.3 qnrS 36.1 tetM 13.9 aacA 16.7 cat 0 sul1 0 erm 0 optrA 0 sul2 13.9 blaCARB 47.2 floR 11.1 sul3 0 arr 0 表 5 36株副溶血弧菌耐药表型与其携带基因型关系
Table 5 Relationship between resistant phenotype of 36 V. parahaemolyticus strains and their carrying genotypes
菌株Strain 耐药谱型 Resistance profile 携带耐药基因 Resistance gene 301 NOR-CIP-SXT-CFR-MER-SMZ-E-AMP blaCARB 302 NOR-CIP-SMZ-E-AMP qnrVC-blaCARB 331 NOR-CIP-SMZ-E-AMP blaCARB 333 SMZ-E-AMP blaCARB 335 SMZ-E-AMP qnrVC-tetM-blaCARB 337 OTC-SMZ-E-AMP qnrVC-blaCARB 338 OTC-SMZ-E-AMP qnrVC-blaCARB 340 OTC-DOX-SMZ-AMP-RA qnrVC-blaCARB 361 ENR-NOR-CIP-SMZ-AMP qnrVC-aadA-blaCARB 362 ENR-NOR-CIP-SMZ-E-AMP aadA-blaCARB 365 NOR-CIP-OTC-SMZ-E-AMP blaCARB 371 OTC-SMZ-SXT-E-AMP qnrVC-sul2-strA-strB-aadA-blaCARB 372 OTC-SMZ-SXT-E-AMP qnrVC-sul2-strA-strB-aadA-blaCARB 383 OTC-SMZ-SXT-E-AMP floR-sul2-strA-strB-blaCARB 384 OTC-SMZ-SXT-E-AMP sul2-strA-strB-blaCARB 388 FFC-OTC-DOX-SMZ-SXT-AMP-RA qnrVC-tetB-tetM-sul2-blaCARB 389 SMZ-AMP aadA-aacA-blaCARB 391 SMZ-E qnrS-strB 392 SMZ-E-AMP qnrVC-tetB-strB 393 OTC-SMZ-SXT-AMP qnrVC-tetB-tetM-strB 394 OTC qnrVC-cfr-tetB-strA-strB 395 AMP qnrVC-qnrA-qnrS-cfr-tetB-strB-aadA 396 OTC-E-AMP-RA tetB-strA-strB 397 AMP-RA qnrVC-qnrA-tetB-strA 398 E-AMP qnrVC-qnrA-qnrS-tetA-strA 399 E qnrVC-qnrA-qnrS-cfr-strB-aadA-aacA 400 E-AMP qnrA-qnrS-tetA-tetB-strA-strB-aadA-aacA 401 E-AMP qnrVC-qnrA-qnrS-cfr-tetA-tetB-strA-strB-aadA 402 AMP qnrVC-qnrA-qnrS-cfr-tetB-strA-strB-aadA-aacA 403 OTC-AMP qnrVCqnrA-qnrS-cfr-tetA-tetB-strA-strB-aadA-aacA 404 AMP-RA qnrVC-qnrA-qnrS-cfr-tetA-tetB-strA-strB-aadA 405 AMP qnrVC-qnrA-qnrS-cfr-tetA-tetB-strA-strB-aacA 406 OTC-SMZ-SXT-E-AMP-RA qnrVC-qnrA-floR-tetB-strA 407 FFC-C-OTC-DOX-SMZ-SXT-E-AMP-RA qnrVC-qnrA-qnrS-floR-tetB-tetM-strB 408 FFC-OTC-SMZ-RA qnrVC-qnrA-qnrS-tetB-strA-strB 409 FFC-C-OTC-DOX-SMZ-E-RA qnrVC-qnrA-qnrS-floR-tetB-tetM-strB 注:ENR. 恩诺沙星;NOR. 诺氟沙星;CIP. 环丙沙星;FFC. 氟苯尼考;C. 氯霉素;OTC. 土霉素;DOX. 多西环素;SMZ. 磺胺甲噁唑;SXT. 复方新诺明;E. 红霉素;AMP. 氨苄西林;RA. 利福平 Note: ENR. Enrofloxacin;NOR. Norfloxacin;CIP. Ciprofloxacin;FFC. Florfenicol;C. Chloramphenicol;OTC. Oxytetracycline;DOX. Doxycycline;SMZ. Sulfamethoxazole;SXT. Compound Sulfoxime;E. Erythromycin;AMP. Ampicillin;RA. Rifampicin -
[1] DARAMOLA B A, WILLIAMS R, DIXON R A. In vitro antibiotic susceptibility of Vibrio parahaemolyticus from environmental sources in northern England[J]. Int J Antimicrob Ag, 2009, 34(5): 499-500. doi: 10.1016/j.ijantimicag.2009.06.015
[2] 张德福, 付绪磊, 张明, 等. 副溶血弧菌毒力因子及致病机理的研究进展[J]. 食品科学, 2015, 36(7): 216-222. doi: 10.7506/spkx1002-6630-201507040 [3] HU Q, CHEN L. Virulence and antibiotic and heavy metal resistance of Vibrio parahaemolyticus isolated from crustaceans and shellfish in Shanghai, China[J]. J Food Protect, 2016, 79(8): 1371-1377. doi: 10.4315/0362-028X.JFP-16-031
[4] CHEN Y, CHEN X, YU F, et al. Serology, virulence, antimicrobial susceptibility and molecular characteristics of clinical Vibrio parahaemolyticus strains circulating in southeastern China from 2009 to 2013[J]. Clin Microbiol Infec, 2016, 22(3): 258.e9-258.16. doi: 10.1016/j.cmi.2015.11.003
[5] 陈旭凌, 黄志坚. 水生细菌耐药性的遗传机制[J]. 中国抗生素杂志, 2013, 38(8): 573-578. doi: 10.3969/j.issn.1001-8689.2013.08.003 [6] 曾德乾, 冯娟, 徐力文, 等. 海水养殖鱼哈维弧菌分离株的耐药谱型分析[J]. 中国水产科学, 2015, 22(1): 129-138. [7] RODRIGUEZ-BLANCO A, LEMOS M L, OSORIO C R. Integrating conjugative elements as vectors of antibiotic, mercury, and quaternary ammonium compound resistance in marine aquaculture environments[J]. Antimicrob Agents Ch, 2012, 56(5): 2619-2626. doi: 10.1128/AAC.05997-11
[8] 张德福, 安慧, 张健, 等. 副溶血弧菌耐药及其机制的研究进展[J]. 食品工业科技, 2018, 39(9): 311-317, 324. [9] SILVESTER R, ALEXANDER D, AMMANAMVEETIL M A. Prevalence, antibiotic resistance, virulence and plasmid profiles of Vibrio parahaemolyticus from a tropical estuary and adjoining traditional prawn farm along the southwest coast of India[J]. Annal Microbiol, 2015, 65(4): 2141-2149. doi: 10.1007/s13213-015-1053-x
[10] YOU K G, BONG C W, LEE C W. Antibiotic resistance and plasmid profiling of Vibrio spp. in tropical waters of Peninsular Malaysia[J]. Environ Monit Assess, 2016, 188(3): 171-171. doi: 10.1007/s10661-016-5163-0
[11] KANG C H, SHIN Y, KIM W, et al. Prevalence and antimicrobial susceptibility of Vibrio parahaemolyticus isolated from oysters in Korea[J]. Environ Sci Poll Res Int, 2016, 23(1): 918-926. doi: 10.1007/s11356-015-5650-9
[12] SHAH S Q, CABELLO F C, L'ABEE-LUND T M, et al. Antimicrobial resistance and antimicrobial resistance genes in marine bacteria from salmon aquaculture and non-aquaculture sites[J]. Environ Microbiol, 2014, 16(5): 1310-1320. doi: 10.1111/1462-2920.12421
[13] LETCHUMANAN V, YIN W F, LEE L H, et al. Prevalence and antimicrobial susceptibility of Vibrio parahaemolyticus isolated from retail shrimps in Malaysia[J]. Front Microbiol, 2015, 6(1): 33-33.
[14] 黄伟德, 肖双燕, 黎姗梅, 等. 广西凡纳滨对虾源副溶血弧菌耐药性和耐药基因的检测[J]. 西南农业学报, 2018, 31(9): 1979-1988. [15] XIE T F, XU X K, WU Q P, et al. Prevalence, molecular characterization, and antibiotic susceptibility of Vibrio parahaemolyticus from ready-to-eat foods in China[J]. Front Microbiol, 2016, 7(97): 549.
[16] 刘英超, 孙静, 钟继红. 急性炎症性肠炎的病原菌分布与耐药性分析[J]. 中华医院感染学杂志, 2015(9): 1987-1988, 1996. [17] 谢红意, 叶鸿雁, 周方满, 等. 宁波地区腹泻患者副溶血弧菌毒力基因及耐药性分析[J]. 检验医学, 2019, 34(2): 136-139. doi: 10.3969/j.issn.1673-8640.2019.02.009 [18] 罗鹏, 何香燕, 胡超群. 细菌整合性接合元件SXT/R391研究进展[J]. 微生物学报, 2014, 54(5): 471-479. [19] AEDO S, IVANOVA L, TOMOVA A, et al. Plasmid-related quinolone resistance determinants in epidemic Vibrio parahaemolyticus, uropathogenic Escherichia coli, and marine bacteria from an aquaculture area in Chile[J]. Microb Ecol, 2014, 68(2): 324-328. doi: 10.1007/s00248-014-0409-2
[20] MATSUO T, NAKAMURA K, KODAMA T, et al. Characterization of all RND-type multidrug efflux transporters in Vibrio parahaemolyticus[J]. Microbiol Open, 2013, 2(5): 725-742.
[21] MORITA Y, KODAMA K, SHIOTA S, et al. Nor M, a putative multidrug efflux protein of Vibrio parahaemolyticus and its homolog in Escherichia coli[J]. Antimicrob Agents Ch, 1998, 42(7): 1778-1782. doi: 10.1128/AAC.42.7.1778
[22] CLSI. Methods for antimicrobial dilution and disk susceptibility testing of infrequently isolated or fastidious bacteria: approved guideline[M]. CLSI, 2010: 70-96.
[23] 刘旭. 海水养殖源弧菌耐药性调查及qnrVC基因在弧菌中的流行情况研究[D]. 上海: 上海海洋大学, 2016: 14-16. [24] ROBICSEK A, STRAHILEVITZ J, SAHM D F, et al. Qnr prevalence in ceftazidime-resistant Enterobacteriaceae isolates from the United States[J]. Antimicrob Agents Ch, 2006, 50(8): 2872-2874. doi: 10.1128/AAC.01647-05
[25] JACOBY G A, WALSH K E, MILLS D M, et al. QnrB, another plasmid-mediated gene for quinolone resistance[J]. Antimicrob Agents Ch, 2006, 50(4): 1178-1182. doi: 10.1128/AAC.50.4.1178-1182.2006
[26] HATA M, SUZUKI M, MATSUMOTO M, et al. Cloning of a novel gene for quinolone resistance from a transferable plasmid in Shigella flexneri 2b[J]. Antimicrob Agents Ch, 2005, 49(2): 801-803. doi: 10.1128/AAC.49.2.801-803.2005
[27] KITIYODOM S, KHEMTONG S, WONGTAVATCHAI J, et al. Characterization of antibiotic resistance in Vibrio spp. isolated from farmed marine shrimps (Penaeus monodon)[J]. FEMS Microbiol Ecol, 2010, 72(2): 219-227. doi: 10.1111/j.1574-6941.2010.00846.x
[28] YOO M H, HUH M D, KIM E H. Characterization of chloramphenicol acetyltransferase gene by multiplex polymerase chain reaction in multidrug-resistant strains isolated from aquatic environments[J]. Aquaculture, 2003, 217(1/2/3/4): 1-21.
[29] WANGY, LU Y. A novel gene, optr A, that confers transferable resistance to oxazolidinones and phenicols and its presence in entero-coccus faecalis and enterococcus faecium of human and animal origin[J]. J Antimicrob Ch, 2015, 70(8): 2182-2190. doi: 10.1093/jac/dkv116
[30] KIM E H, AOKI T. Sequence analysis of the florfenicol resistance gene encoded in the transferable R-plasmid of a fish pathogen, Pasteurella piscicida[J]. Microbiol Immunol, 1996, 40(9): 665-669. doi: 10.1111/j.1348-0421.1996.tb01125.x
[31] KEBRENBERG C, SCHWARZ S. Distribution of florfenicol resistance genes fex A and cfr among chloramphenicol-resistant Staph-yloccus isolates[J]. Antimicrob Agents Ch, 2006, 50(4): 1156-1163. doi: 10.1128/AAC.50.4.1156-1163.2006
[32] CHIOU J C, LI R C, CHEN S. CARB-17 family of β-lactamases mediates intrinsic resistance to penicillins in Vibrio parahaemolyticus[J]. Antimicrob Agents Ch, 2015, 59(6): 3593-3595. doi: 10.1128/AAC.00047-15
[33] 汪建国. 我国渔药研究与应用综述(14)[J]. 渔业致富指南, 2013(14): 68-72. [34] 汪涛, 杨再福, 陈勇航, 等. 磺胺类抗性基因的产生及演变研究进展[J]. 环境污染与防治, 2017, 39(11): 1251-1255. [35] JOSEPH S W, DEBELL R M, BROWN W P. In vitro response to chloramphenicol, tetracycline, ampicillin, gentamicin, and beta-lactamase production by halophilic vibrios from human and environmental sources[J]. Antimicrob Agents Ch, 1978, 13(2): 244-248. doi: 10.1128/AAC.13.2.244
[36] CHARMAINE N G, CHEN H, GOH S G, et al. Microbial water quality and the detection of multidrug resistant E. coli and antibiotic resistance genes in aquaculture sites of Singapore[J]. Mar Pollut Bull, 2018, 135(7): 475-480.
[37] JIANG Y, YAO L, LI F, et al. Characterization of antimicrobial resistance of Vibrio parahaemolyticus from cultured sea cucumbers (Apostichopus japonicas)[J]. Lett Appl Microbiol, 2014, 59(2): 147-154. doi: 10.1111/lam.12258
[38] 蒋魁, 徐力文, 苏友禄, 等. 2012年~2014年南海海水养殖鱼类病原菌哈维弧菌分离株的耐药性分析[J]. 南方水产科学, 2016, 12(6): 99-107. doi: 10.3969/j.issn.2095-0780.2016.06.013 -
期刊类型引用(3)
1. 李进京,刘颖,李祥付,刘好真,徐鹏,孙元,焦海峰,王一农,尤仲杰. 韭山列岛海洋生态自然保护区及其附近海域底拖网生物资源的分布及群落结构分析. 海洋与湖沼. 2020(01): 176-185 .  百度学术
百度学术
2. 杨咏文,黄汉英,冯婉娴,李路,熊善柏,赵思明. 基于被动水声信号的淡水鱼混合数量预测. 华中农业大学学报. 2020(05): 147-152 .  百度学术
百度学术
3. 马孟磊,陈作志,许友伟,张魁,袁伟,徐姗楠. 基于Ecopath模型的胶州湾生态系统结构和能量流动分析. 生态学杂志. 2018(02): 462-470 .  百度学术
百度学术
其他类型引用(4)



 下载:
下载:
















 粤公网安备 44010502001741号
粤公网安备 44010502001741号
