Size selectivity of codend mesh size of shrimp beam trawl for Metapenaeus ensis in South China Sea
-
摘要:
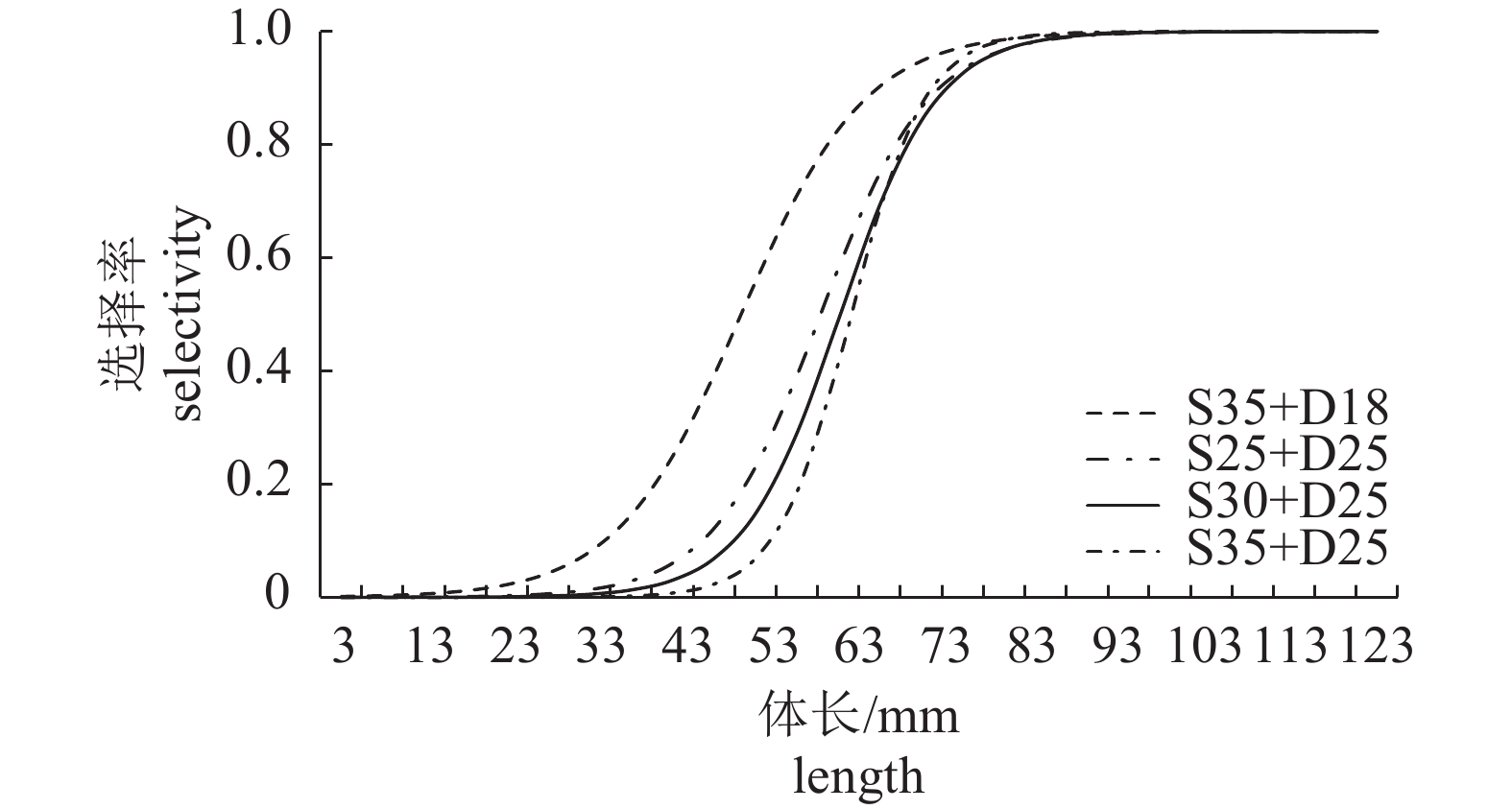
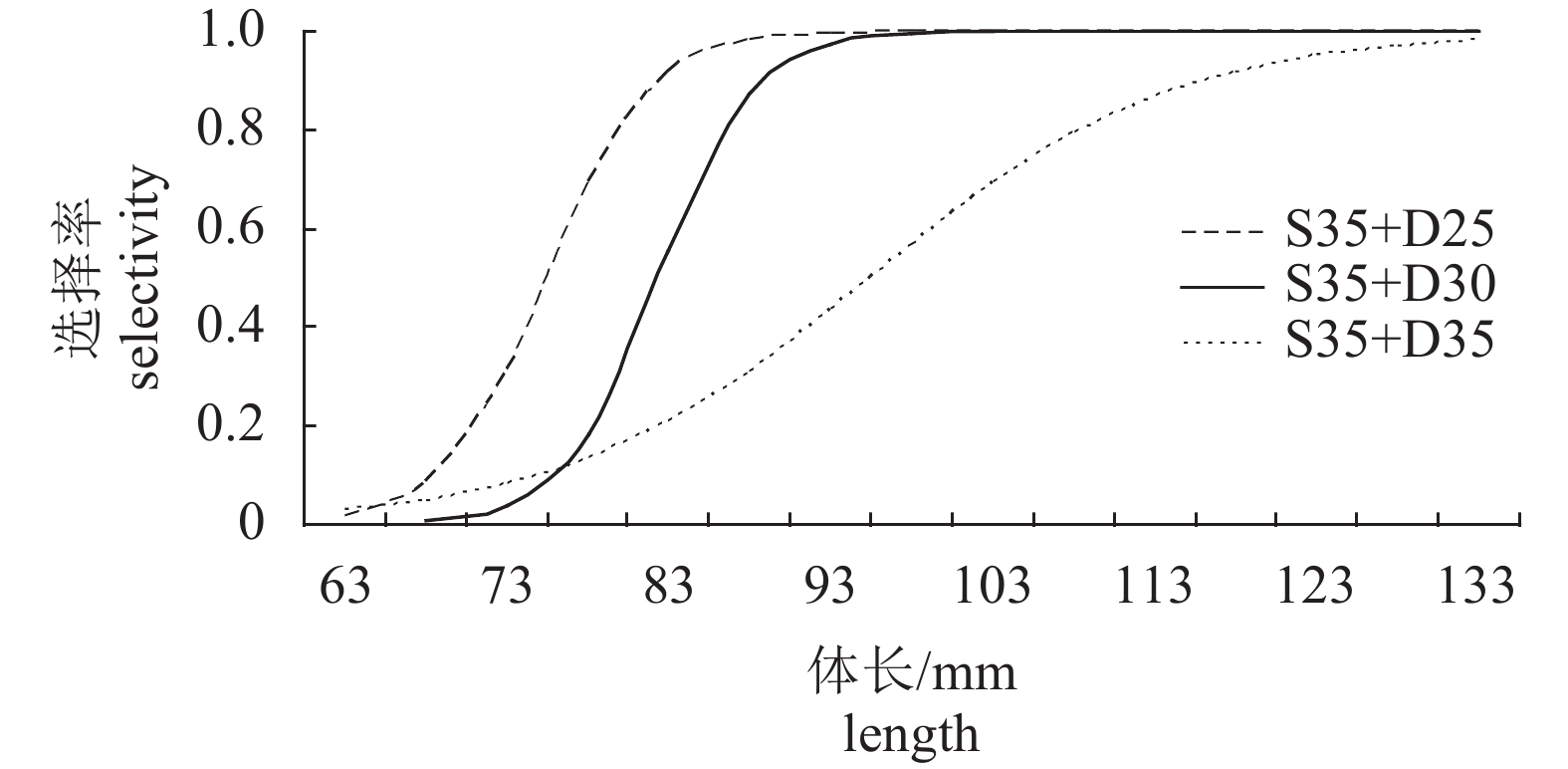
为了研究虾拖网网囊对刀额新对虾(Metapenaeus ensis)的选择性,2014—2017年于南海海域使用套网法和裤网法对2组菱目网囊(D25和D30)和6组混目网囊(S35+D18、S25+D25、S30+D25、S35+D25、S35+D30和S35+D35)进行选择性试验。以SELECT模型为基础框架,以logsitic曲线为模型,使用极大似然估算法估算单网次和联合网次下网囊对刀额新对虾的选择性。结果表明,菱目网囊D25和D30对刀额新对虾的选择性很差,逃逸数量很少;套网法试验中,混目网囊S35+D18、S25+D25、S30+D25和S35+D25对刀额新对虾的平均50%选择体长(L50)分别为51.52 mm、60.84 mm、63.21 mm和64.53 mm,平均选择范围(SR)分别为16.48 mm、14.31 mm、12.84 mm和9.75 mm;裤网法试验中,混目网囊S35+D25、S35+D30和S35+D35对刀额新对虾的平均L50分别为75.43 mm、82.38 mm和95.39 mm,平均SR分别为6.93 mm、6.39 mm和20.44 mm,平均相对作业强度(p)分别为0.51、0.52和0.64。综合南海区刀额新对虾的首次性成熟体长(80 mm)和网囊选择性等多方面因素,认为S35+D30网囊的选择性较好,研究结果可为南海海域虾拖网的渔具管理和刀额新对虾资源的合理利用提供参考。
Abstract:In order to investigate the size selectivity of codend of shrimp beam trawl for Metapenaeus ensis, with covered codend method and trouser trawl method, we carried out fishing experiments on two traditional diamond-mesh codends (D25 and D30), and six novel combined square-mesh and diamond-mesh codends (S35+D18, S25+D25, S30+D25, S35+D25, S35+D30 and S35+D35) in the South China Sea during 2014−2017. We used maximum likelihood method to estimate the selective parameters at haul by haul level and combined haul level with the logistic equation. The results indicate that the D25 and D30 codend were nearly nonselective for M. ensis. In experiments of covered codend method, the mean 50% retention lengths (L50) of M. ensis for the S35+D18, S25+D25, S30+D25 and S35+D25 codends were 51.52 mm, 60.84 mm, 63.21 mm and 64.53 mm, respectively, and the mean selection ranges (SR) were 16.48 mm, 14.31 mm, 12.84 mm and 9.75 mm, respectively. In the experiments of trouser trawl method, the mean L50s for the S35+D25, S35+D30 and S35+D35 were 75.43 mm, 82.38 mm and 95.39 mm, respectively, and the mean SRs were 6.93 mm, 6.39 mm and 20.44 mm, respectively, and the mean relative fishing intensities (p) were 0.51, 0.52 and 0.64, respectively. Considering the first mature length of M. ensis (80 mm) in the South China Sea as well as economical efficiency of fishing and selective properties of codends, it is suggested that the S35+D30 has the best selectivity. The results are benefit for the management of shrimp beam trawl and sustainable harvest of the fishery resources of M. ensis in the South China Sea.
-
Keywords:
- shrimp beam trawl /
- codend /
- Metapenaeus ensis /
- selectivity
-
三角帆蚌 (Hyriopsis cumingii) 是中国南方一种重要的经济淡水蚌类。根据《2024中国渔业统计年鉴》,2023年中国淡水珍珠养殖产量为754 920 kg[1],而三角帆蚌珍珠产量在人工养殖淡水育珠蚌中占据重要地位。此外,三角帆蚌可通过高强度的滤食增加水体透明度,直接或间接地改变浮游植物和沉水植物的群落结构。因此,养殖三角帆蚌具有重要的经济价值和生态意义[2-4]。然而,近年来细菌和病毒感染导致贝类大规模死亡的现象时有发生,给珍珠养殖产业造成重大损失[5]。此外,养殖环境的恶化导致水生动物的发病率不断上升,污染物通过诱导免疫抑制,严重破坏了宿主先天免疫对病原体的防御[6]。因此,在集约化养殖条件下如何有效地防治贝类病害已成为产业发展面临的新一轮挑战。了解贝类先天性免疫机制和抗菌作用将有助于制定贝类病害治理方法,有利于推动贝类健康养殖和珍珠产业的可持续发展。
先天免疫系统是脊椎动物和无脊椎动物抵抗微生物入侵的第一道防线[7]。其中,Toll样受体 (Toll-like receptors, TLR) 作为先天免疫中的关键组分,在识别和应对多种病原体中发挥着至关重要的作用[8]。TLR1通过直接招募并结合髓样分化因子88 (Myeloid differentiation factor 88, MyD88) 蛋白,引发自身活化并募集下游信号转导相关蛋白白细胞介素-1受体相关激酶 (Interleukin-1 receptor-associated kinases, IRAKs),活化的IRAKs驱动自体磷酸化并募集肿瘤坏死因子受体相关因子6 (Tumour-necrosis factor receptor-associated factor 6, TRAF6)[9-11],TRAF6进而激活转化生长因子-β激活激酶1 (Transforming growth factor-β-activated kinase 1, TAK1),TAK1进一步刺激IkB激酶 (Inhibitor of kappa B kinase, IKK) 介导的核因子κB (Nuclear factor kappa-B, NF-κB) 和丝裂原活化蛋白激酶 (Mitogen-activated protein kinase, MAPK) 介导的激活蛋白1 (Activating protein-1, AP-1) 的转录反应,从而诱导炎性细胞因子的活化和表达[12-13]。目前已在三角帆蚌中鉴定了11个TLRs。在病原菌刺激下,三角帆蚌肝胰腺中Toll1基因的表达量升高,且过表达可以激活S2细胞中的Toll信号通路,从而上调抗真菌肽 (Drosomycin, DRS) 的表达[14]。然而并无研究证明在受到病原菌刺激时,TLR1是否直接激活MyD88信号通路,并调节下游相关免疫基因发挥抗菌应答作用。HcToll2参与诱导抗真菌肽表达以进行抗菌免疫[15];HcToll3介导调节乳清酸蛋白 (Whey acidic protein, WAP) 和溶菌酶 (Lysozyme, Lys) 的表达,参与了三角帆蚌对弧菌的防御[16];HcToll4和HcToll5则能够识别金黄色葡萄球菌 (Staphyloccocus aureus)、副溶血弧菌 (Vibrio parahemolyticus)、白斑综合征病毒 (White Spot Syndrome Virus) 和聚肌胞苷酸 (Polyinosinic acid-polycytidylic acid, poly I:C),并参与调节三角帆蚌抗菌肽 (Theromacin, Ther) 和乳清酸蛋白的表达[17];此外,HcToll6和HcToll7介导鳃中溶菌酶和防御素 (Defensins, Def) 的表达,参与了副溶血弧菌刺激后机体的抗菌应答过程[18];HcTLRn[19]、HcToll9和HcToll10可通过NF-κB信号通路参与三角帆蚌的抗菌免疫应答[20]。由此可见,在三角帆蚌体内存在多种不同功能的TLRs,它们在对抗不同病原微生物感染中发挥着重要作用。
为探讨三角帆蚌HcTLR1基因在先天性免疫中的作用机制,本研究从感染维氏气单胞菌 (Aeromonas veronii)的三角帆蚌肝胰腺转录组数据库[21]中筛选并克隆了三角帆蚌HcTLR1基因的全长cDNA。采用实时荧光定量分析技术和dsRNA干扰技术研究了HcTLR1基因在不同组织中的表达情况,以及HcTLR1-MyD88信号通路对不同免疫刺激物和维氏气单胞菌的免疫响应情况。为进一步了解TLRs在软体动物先天免疫防御和抗菌应答中的作用奠定了基础。
1. 材料与方法
1.1 实验材料
选用2龄三角帆蚌250只,体质量约为 (75±10) g。实验前,将蚌置于含过滤淡水和空气泵的15 L水循环系统中养殖1周左右,养殖水温 (23.0±1.0) ℃。每天喂食1次小球藻液 (5.6×105 个·mL−1)。暂养期间剔除不健康个体,选取健康个体用于后续实验。
1.2 HcTLR1基因克隆
从构建的三角帆蚌肝胰腺转录组文库中,筛选出TLR1基因片段 (数据未发表)[22]。使用SMARTer® RACE 5'/3' Kit试剂盒 (TaKaRa,日本),结合特异性和通用引物 (表1),以肝胰腺为模板,进行cDNA末端快速克隆 (RACE),扩增两端序列。PCR反应体系为:12 μL LA taq (TaKaRa,日本),1 μL 特异性引物 (Gene specific primer, GSP, 10 μmol·L−1),1 μL 通用引物 (Universal primer, UPM, 10 μmol·L−1),3 μL cDNA,8 μL ddH2O。PCR扩增程序为:98 ℃ 10 s,68 ℃ 30 s,72 ℃ 3 min,30个循环。将回收的PCR扩增产物连接到pMD19T质粒 (TaKaRa,日本) 载体中,并转化至大肠杆菌感受态DH5α细胞 (擎科生物,中国) 中进行菌液PCR和测序。最后,通过拼接HcTLR1的两端序列,获得基因全长序列。
表 1 引物序列Table 1 Sequences of primers引物名称
Primer name引物序列 (5'—3')
Primers sequence (5'−3')用途
Application来源
SourceHcTLR1-3'race-F TGCCATGCTGTACAGACTTGCTACCGAA 基因克隆 本研究 HcTLR1-5'race-R CGTGCACTTCTCGGGACATCTGTGT 本研究 UPM Long CTAATACGACTCACTATAGGGCAAGCAGTGGTATCAACGCAGAGT 本研究 UPM Short CTAATACGACTCACTATAGGGC 本研究 TLR1-iF+T7 TAATACGACTCACTATAGGGTAAAGTTCGGATGGCGAATC RNA干扰 本研究 TLR1-iR TTTTCCCAAAACCAAGCATC 本研究 GFP-iF+T7 TAATACGACTCACTATAGGGAAGGGCGAGGAGCTGTTCACCG 本研究 GFP-iR CAGCAGGACCATGTGATCGCGC 本研究 HcTLR1-qPCR-F TGCCATGCTGTACAGACTTGCT qPCR 本研究 HcTLR1-qPCR-R CGTGCACTTCTCGGGACATCTGT 本研究 MyD88-qPCR-F GAACGCCACCCTTGGGAAAC 本研究 MyD88-qPCR-R TCCACGACAGCACCCTCAAG 本研究 TRAF6-qPCR-F CGCAGTGTGGTCATCGCTTC 本研究 TRAF6-qPCR-R TGGCACACGAGTTAGGGCAT 本研究 IRAK4-qPCR-F TCGCAAGTGGTACAGGCCAT 本研究 IRAK4-qPCR-R TGACGTGCCAATTACGAGCG 本研究 WAP-qPCR-F TGTAATGTTGACGGGAGTG [22] WAP-qPCR-R CTGTTTTGTTTTGATGGCT LBP/BPI1-qPCR-F TGGAGAACAGAGTCAGAAAGA [23] LBP/BPI1-qPCR-R CGATAGTCAAGCAGGAAATG LBP/BPI2-qPCR-F TATCAGTGTCAGCGGTAGTG [23] LBP/BPI2-qPCR-R CATCAGCGTAAAGAGGGA p65-qPCR-F TGGCAAGGACTGTAAGAAAGG [23] p65-qPCR-R CTTGTGCTTAAATCCAGTCTGA P105-qPCR-F TGTTGTATCCACTCCCATCT [23] P105-qPCR-R TCTTGCTCAACGAACTTCAC Lys-qPCR-F CACAGTTGGTGGTTCAGTAA 本研究 Lys-qPCR-R CGAATCCTTCAGTAGATGGT 本研究 IL17-qPCR-F CCATCCACGATCCTCAACG 本研究 IL17-qPCR-R CGCAAGTGTATCCAACAGCAA 本研究 Def-qPCR-F GGTGTCGTCTATCTTGCTTC [19] Def-qPCR-R AGGTTATTTGGTCATCTATTTTG Ther-qPCR-F CACAGTTGGTGGTTCAGTAA [18] Ther-qPCR-R CGAATCCTTCAGTAGATGGT EF1α-qPCR-F GGAACTTCCCAGGCAGACTGTGC [3] EF1α-qPCR-R TCAAAACGGGCCGCAGAGAAT 注:下划线部分引物序列为T7启动子序列。 Note: The underlined primer sequence is T7 promoter sequence. 1.3 生物信息学分析
使用NCBI (https://www.ncbi.nlm.nih.gov/) 中ORF Finder对基因序列开放阅读框 (Open reading frame, ORF) 与编码氨基酸序列进行预测;用Smart Blast对氨基酸序列进行同源性分析;通过Simple Modular Architecture Research Tool SMART (http://smart.embl-heidelberg.de/) 预测氨基酸所包含的结构域;利用Protparam (http://www.expasy.org/tools/protparam.html) 线上工具获取氨基酸序列组成、分子质量大小、等电点等物理参数信息;利用MEGA 11的邻接法 (Neighbor-Joining, NJ) 构建系统进化树,Bootstrap分析其可靠性,重复1 000次。
1.4 HcTLR1基因的组织表达
分别取三角帆蚌的肝胰腺、斧足、闭壳肌、外套膜、鳃、性腺、血细胞、肾脏和肠组织,并采用心脏取血的方式收集血淋巴,1 000×g离心5 min,收集血细胞。共9只蚌,每3只的组织混合作为1个样品,根据RNAiso plus提取试剂盒 (TaKaRa,日本) 说明书提取组织RNA。参照Hifair III 1st Strand cDNA Synthesis SuperMix (Yeasen,中国) 说明书合成cDNA。以EF-1α基因为内参,以不同组织的cDNA为模板,结合特定的荧光定量引物 (表1),通过BIORAD CFX ConnectTM荧光定量仪 (Bio-Rad Laboratories,美国),检测了2龄三角帆蚌不同组织中HcTLR1基因的相对表达量。反应体系为:10 μL SYBR Green Mix (Yeasen,中国)、0.4 μL q-F (10 μmol·L−1)、0.4 μL q-R (10 μmol·L−1)、2 μL cDNA和7.2 μL ddH2O。反应程序为:95 ℃ 2 min;95 ℃ 10 s,60 ℃ 30 s,72 ℃ 10 s,39个循环。使用2−ΔΔCt法计算基因的相对表达量,每组3个重复,计算平均值。
1.5 免疫刺激
脂多糖 (Lipopolysaccharides, LPS)、肽聚糖 (Peptidoglycan, PGN) 和poly I:C 3种刺激物代表不同的病原体相关分子模式 (Pathogen-associated molecular pattern, PAMP),均购自麦克林公司;维氏气单胞菌GL1由本实验室前期从患病三角帆蚌中分离[23]。将健康三角帆蚌随机分为5组,其中4组向闭壳肌中分别注射200 μL维氏气单胞菌GL1 (107 CFU·mL−1)、LPS、PGN和poly I:C (0.5 mg·mL−1,溶于pH 7.4的PBS),另一组注射相同体积的磷酸盐缓冲溶液 (PBS, pH 7.4) 作为阴性对照。未经处理的三角帆蚌作为空白对照组。在注射后的第6、第12、第24、第48小时,分别收集9只三角帆蚌的血细胞、肝胰腺和鳃组织。每3只蚌的组织混合作为1个样品,用于RNA提取。
1.6 血细胞内RNA干扰实验
使用Primer 5.0软件设计1对含有T7启动子的基因特异性引物:TLR1-iF+T7、TLR1-iR (表1)。以血细胞cDNA为模板,采用rTaq DNA聚合酶 (TaKaRa,日本) 进行特异性DNA片段的扩增。同时,绿色荧光蛋白 (Green fluorescent protein, GFP) 片段用于阴性对照dsRNA的合成。根据T7 High Yield RNA Transcription Kit (Beyotime Biotechnology,中国) 试剂盒操作,合成并纯化相应的dsRNA。
挑选健康的三角帆蚌个体暂养后收集血细胞。血细胞培养于含10% (φ) FBS (Gibico,美国)、2% (φ) 青链霉素 (Beyotime Biotechnology,中国) 的DMEM培养基 (Gibico,美国) 中,培养条件为27 ℃、5% (φ) CO2,并定期观察细胞状态。按照每毫升106个细胞的密度,向细胞中同时加入40 μg的dsRNA及灭活后的维氏气单胞菌GL1。每个时间点的不同处理组各设置3个重复。将上述处理的细胞置于27 ℃、5% (φ) CO2培养箱中静置培养。在培养后的第6、第12和第24小时,分别收集不同处理组的血细胞,以便进行后续的RNA提取和qPCR分析。
1.7 数据分析
所有数据均以“平均值±标准差 ($ \overline x \pm s $)”表示。采用SPSS 26.0软件进行单因素方差 (One-Way ANOVA) 分析及Tukey氏多重检验,显著性水平α为0.05 (n=3)。
2. 结果
2.1 三角帆蚌HcTLR1基因的克隆与序列分析
通过测序、拼接获得HcTLR1基因全长序列 (GenBank登录号:PP051478) 3 969 bp,包含45 bp的5'非翻译区 (Untranslated region, UTR)、237 bp的3'UTR和3 687 bp的ORF,共编码1 228个氨基酸 (图1),预测分子质量为139 kD,理论等电点为5.54。SMART结构域分析预测HcTLR1蛋白包含14个LRR结构域、3个LRR亚家族结构域、2个C端LRR结构域、1个LRR-NT结构域、1个跨膜结构域和1个胞内TIR结构域 (图2)。
![]() 图 1 HcTLR1核苷酸序列和预测的氨基酸序列注:红色碱基代表起始密码子 (ATG) 和终止密码子 (TAA);富含亮氨酸的重复序列 (LRR) 结构域用下划线标出;富含亮氨酸的重复序列典型的亚家族 (LRR TYP) 用双下划线标出;富含亮氨酸重复C-末端结构域 (LRR CT) 用波浪线标出;富含亮氨酸重复N-末端结构域 (LRR NT) 用虚线标出;跨膜结构域 (TM) 用灰色标出;白细胞介素-1受体 (TIR) 结构域用蓝色标出。Fig. 1 Nucleotide sequences and predicted amino acid sequences of HcTLR1Note: Red bases represent the initiation codon (ATG) and stop codon (TAA); the Leucine-rich repeats (LRR) domains were underlined; the Leucine-rich repeats typical subfamily (LRR TYP) was double underlined; the Leucine rich repeat C-terminal domain (LRR CT) was marked with wavy lines; the Leucine rich repeat N-terminal domain (LRR NT) was marked with dotted line; the transmembrane domain (TM) was marked with gray and the Toll/interleukin-1 receptor (TIR) domain was marked with blue.
图 1 HcTLR1核苷酸序列和预测的氨基酸序列注:红色碱基代表起始密码子 (ATG) 和终止密码子 (TAA);富含亮氨酸的重复序列 (LRR) 结构域用下划线标出;富含亮氨酸的重复序列典型的亚家族 (LRR TYP) 用双下划线标出;富含亮氨酸重复C-末端结构域 (LRR CT) 用波浪线标出;富含亮氨酸重复N-末端结构域 (LRR NT) 用虚线标出;跨膜结构域 (TM) 用灰色标出;白细胞介素-1受体 (TIR) 结构域用蓝色标出。Fig. 1 Nucleotide sequences and predicted amino acid sequences of HcTLR1Note: Red bases represent the initiation codon (ATG) and stop codon (TAA); the Leucine-rich repeats (LRR) domains were underlined; the Leucine-rich repeats typical subfamily (LRR TYP) was double underlined; the Leucine rich repeat C-terminal domain (LRR CT) was marked with wavy lines; the Leucine rich repeat N-terminal domain (LRR NT) was marked with dotted line; the transmembrane domain (TM) was marked with gray and the Toll/interleukin-1 receptor (TIR) domain was marked with blue.将三角帆蚌HcTLR1蛋白的功能结构域序列与其他物种功能结构域序列进行多重序列比对,发现不同物种 (包括脊椎动物和无脊椎动物) 的TIR结构域序列具有较高的同源性 (图3)。基于三角帆蚌氨基酸全长序列的系统进化树分析显示,HcTLR1与三角帆蚌Toll 1及栉孔扇贝 (Azumapecten farreri) Toll、长牡蛎 (Crassostrea gigas) TLR1聚为一支,其中与三角帆蚌Toll 1的亲缘关系最近 (图4)。
![]() 图 3 三角帆蚌与其他物种TIR结构域的氨基酸多序列比对注:黑色阴影表示氨基酸相同,其他阴影表示氨基酸部分相同;用于多重序列比对的其他物种的氨基酸序列分别是智人 (CAG38593.1),小鼠 (AAG35062.1),大鼠 (XP_038947854.1),原鸡 (BAD67422.1),斑马鱼 (NP_001124065.1),三角帆蚌 (Toll 2: AIA66467.1, Toll 4: AVR52705.1, Toll 5: AVR52704.1, Toll 6: QCR63936.1, Toll 7: QCR63937.1),紫贻贝 (AFU48614.1),虾夷扇贝 (XP_021346851.1),海湾扇贝 (AVP74315.1),巨海扇蛤 (XP_033755286.1)。Fig. 3 Multiple sequence alignment of TIR domain between H. cumingii and other speciesNote: Black shading represents amino acid identity, and other shading represents partial amino acid identity. The amino acid sequences used for multiple alignment include Homo sapians (CAG38593.1), Mus musculus (AAG35062.1), Rattus norvegicus (XP_038947854.1), Gallus gallus (BAD67422.1), Danio rerio (NP_001124065.1), Hyriopsis cumingii (Toll 2: AIA66467.1, Toll 4: AVR52705.1, Toll 5: AVR52704.1, Toll 6: QCR63936.1, Toll 7: QCR63937.1), Mytilus galloprovincialis (AFU48614.1), Mizuhopecten yessoensis (XP_021346851.1), Argopecten irradians (AVP74315.1) and Pecten maximus (XP_033755286.1).
图 3 三角帆蚌与其他物种TIR结构域的氨基酸多序列比对注:黑色阴影表示氨基酸相同,其他阴影表示氨基酸部分相同;用于多重序列比对的其他物种的氨基酸序列分别是智人 (CAG38593.1),小鼠 (AAG35062.1),大鼠 (XP_038947854.1),原鸡 (BAD67422.1),斑马鱼 (NP_001124065.1),三角帆蚌 (Toll 2: AIA66467.1, Toll 4: AVR52705.1, Toll 5: AVR52704.1, Toll 6: QCR63936.1, Toll 7: QCR63937.1),紫贻贝 (AFU48614.1),虾夷扇贝 (XP_021346851.1),海湾扇贝 (AVP74315.1),巨海扇蛤 (XP_033755286.1)。Fig. 3 Multiple sequence alignment of TIR domain between H. cumingii and other speciesNote: Black shading represents amino acid identity, and other shading represents partial amino acid identity. The amino acid sequences used for multiple alignment include Homo sapians (CAG38593.1), Mus musculus (AAG35062.1), Rattus norvegicus (XP_038947854.1), Gallus gallus (BAD67422.1), Danio rerio (NP_001124065.1), Hyriopsis cumingii (Toll 2: AIA66467.1, Toll 4: AVR52705.1, Toll 5: AVR52704.1, Toll 6: QCR63936.1, Toll 7: QCR63937.1), Mytilus galloprovincialis (AFU48614.1), Mizuhopecten yessoensis (XP_021346851.1), Argopecten irradians (AVP74315.1) and Pecten maximus (XP_033755286.1).2.2 HcTLR1基因组织特异性表达分析
实时荧光定量PCR结果显示,HcTLR1基因在所有检测的组织中均有表达,且表达程度各异 (图5)。其中,血细胞中HcTLR1的表达量最高,并显著高于其他组织 (p<0.05)。其在血细胞中的表达量分别为肝胰腺的13.67倍、斧足的23.27倍、闭壳肌的19.06倍、外套膜的7.1倍、鳃的11.45倍、性腺的18.63倍、肾脏的2.5倍和肠的2.07倍。
![]() 图 5 HcTLR1 mRNA在三角帆蚌组织中的相对表达量注:1. 肝胰腺;2. 斧足;3. 闭壳肌;4. 外套膜;5. 鳃;6. 性腺;7. 血细胞;8. 肾脏;9. 肠;所标字母不同表示差异显著 (p<0.05)。Fig. 5 Relative mRNA expression of HcTLR1 in different tissues from H. cumingiiNote: 1. Hepatopancreas; 2. Foot; 3. Adductor; 4. Mantle; 5. Gill; 6. Gonad; 7. Hemocyte; 8. Kidney; 9. Intestine. Different letters represent significant differences (p<0.05).
图 5 HcTLR1 mRNA在三角帆蚌组织中的相对表达量注:1. 肝胰腺;2. 斧足;3. 闭壳肌;4. 外套膜;5. 鳃;6. 性腺;7. 血细胞;8. 肾脏;9. 肠;所标字母不同表示差异显著 (p<0.05)。Fig. 5 Relative mRNA expression of HcTLR1 in different tissues from H. cumingiiNote: 1. Hepatopancreas; 2. Foot; 3. Adductor; 4. Mantle; 5. Gill; 6. Gonad; 7. Hemocyte; 8. Kidney; 9. Intestine. Different letters represent significant differences (p<0.05).2.3 免疫刺激后不同组织中HcTLR1的表达
在2 龄三角帆蚌受到维氏气单胞菌和不同PAMPs刺激后,采用qPCR技术检测了不同时间点三角帆蚌各组织中HcTLR1基因的相对表达量。结果显示,在鳃组织中,经维氏气单胞菌GL1和poly I:C刺激后,HcTLR1基因表达量呈先上升后下降的趋势。刺激后第12 和第24小时,其表达量显著增加 (p<0.05),分别为PBS组的5.2和4.5倍。而LPS、PNG刺激后,其表达量呈下降趋势,但在刺激后第6小时,与PBS组相比,其表达量显著增加 (p<0.05),分别为4.2和5.4倍 (图6-a)。在肝胰腺中,经维氏气单胞菌GL1、LPS、PNG、poly I:C刺激后,HcTLR1基因的表达量也呈先上升后下降的趋势,且在刺激后第24小时表达量显著上调 (p<0.05),分别为PBS组的14、5.8、8和4.9倍 (图6-b)。在血细胞中,维氏气单胞菌GL1刺激后第6、第12和第24小时,HcTLR1基因的表达量显著上调 (p<0.05),分别为PBS组的2.1、2和2.4倍 (图6-c)。然而,在LPS、PNG、poly I:C刺激后,其表达量先下降后上升,但无显著性差异 (p>0.05)。由此可见,当三角帆蚌经过维氏气单胞菌GL1、LPS、PNG和poly I:C刺激后,HcTLR1基因在不同组织和时间点的表达水平存在显著差异。
![]() 图 6 免疫刺激后三角帆蚌鳃 (a)、肝胰腺 (b) 和血细胞 (c) 中HcTLR1 mRNA在不同时间点的相对表达量注:同一处理时间下不同字母表示差异显著 (p<0.05),相同字母表示差异不显著 (p>0.05)。Fig. 6 Relative mRNA expression of HcTLR1 after immune challenges in gill (a), hepatopancreas (b) and hemocyte (c) of H. cumingii at different time pointNote: Different letters at the same treatment time represent significant differences (p<0.05), while the same letters represent insignificant differences (p>0.05).
图 6 免疫刺激后三角帆蚌鳃 (a)、肝胰腺 (b) 和血细胞 (c) 中HcTLR1 mRNA在不同时间点的相对表达量注:同一处理时间下不同字母表示差异显著 (p<0.05),相同字母表示差异不显著 (p>0.05)。Fig. 6 Relative mRNA expression of HcTLR1 after immune challenges in gill (a), hepatopancreas (b) and hemocyte (c) of H. cumingii at different time pointNote: Different letters at the same treatment time represent significant differences (p<0.05), while the same letters represent insignificant differences (p>0.05).2.4 HcTLR1基因干扰后MyD88依懒性通路及免疫相关基因的表达
为了探究三角帆蚌HcTLR1基因对TLR信号通路的激活作用,采用dsRNA干扰方法来抑制血细胞中HcTLR1基因的表达。通过qPCR检测HcTLR1及其介导的MyD88依赖性信号通路中多个关键基因的表达变化,包括MyD88、IRAK4、TRAF6、核因子-κB/Rel转录因子p65 (NF-κB/RELA p65, p65)、核因子-κB/Rel 蛋白1 p105 (NF-κB/RELA 1 p105, p105) 和下游免疫相关基因Ther、Lys、Def、Wap、脂多糖结合蛋白/杀菌通透性增加蛋白 (LPS binding protein/bactericidal permeability increasing protein, LBP/BPI) 1、2和白细胞介素17 (Interleukin-17, IL-17)。结果显示,与未处理组和阴性对照dsGFP组相比,dsTLR1组在干扰后的第6、第12和第24小时,HcTLR1 基因的表达量显著下降,干扰效果显著 (p<0.05,图7-a)。在此基础上,进一步在干扰后使用维氏气单胞菌GL1刺激血细胞发现,dsTLR1组的HcTLR1基因表达量在第6、第12和第24小时均显著降低 (p<0.05,图7-b)。此外,与未处理组相比,维氏气单胞菌GL1刺激血细胞12 h后检测发现,dsTLR1组中MyD88依赖性通路相关基因MyD88、IRAK4、TRAF6、p65和p105的表达量分别下降87.8%、97.7%、94.6%、93%和96.3% (p<0.05,图8-a—e)。同时,通路下游抗菌免疫相关基因Ther、Def、Lys、WAP、LBP/BPI 2和IL-17的表达量分别下降97.4%、98.3%、99.7%、91.7%、91.2%和94.4% (p<0.05,图8-f—i,8-k—l)。然而,LBP/BPI 1基因的表达量与对照组相比无显著性差异 (p>0.05,图8-j)。上述结果表明,HcTLR1基因参与三角帆蚌TLR1-MyD88-NF-κB信号通路的激活,并通过激活下游免疫相关基因发挥抗菌应答作用。
![]() 图 7 HcTLR1干扰及免疫刺激后血细胞中HcTLR1 mRNA的相对表达量注:a. dsRNA干扰后血细胞中HcTLR1 mRNA的表达水平;b. dsRNA干扰后并经维氏气单胞菌GL1刺激血细胞中HcTLR1 mRNA的表达水平。同一处理时间下不同字母表示差异显著 (p<0.05),相同字母表示差异不显著 (p>0.05)。Fig. 7 mRNA expression levels in hemocytes of H. cumingii after HcTLR1 interference and immune challengesNote: a. HcTLR1 mRNA expression levels in hemocytes after dsRNA interference; b. HcTLR1 mRNA expression levels in hemocytes after dsRNA interference in A. veronii GL1 stimulation. Different letters at the same treatment time represent significant differences (p<0.05), while the same letters represent insignificant differences (p>0.05).
图 7 HcTLR1干扰及免疫刺激后血细胞中HcTLR1 mRNA的相对表达量注:a. dsRNA干扰后血细胞中HcTLR1 mRNA的表达水平;b. dsRNA干扰后并经维氏气单胞菌GL1刺激血细胞中HcTLR1 mRNA的表达水平。同一处理时间下不同字母表示差异显著 (p<0.05),相同字母表示差异不显著 (p>0.05)。Fig. 7 mRNA expression levels in hemocytes of H. cumingii after HcTLR1 interference and immune challengesNote: a. HcTLR1 mRNA expression levels in hemocytes after dsRNA interference; b. HcTLR1 mRNA expression levels in hemocytes after dsRNA interference in A. veronii GL1 stimulation. Different letters at the same treatment time represent significant differences (p<0.05), while the same letters represent insignificant differences (p>0.05).![]() 图 8 HcTLR1 基因干扰及免疫刺激后血细胞中TLR1-MyD88-NF-κB信号通路及下游抗菌相关基因的相对表达量注:不同字母表示差异显著 (p<0.05),相同字母表示差异不显著 (p>0.05)。Fig. 8 Relative expression of TLR1-MyD88-NF-κB signaling pathway and downstream antimicrobial-associated gene in hemocytes after HcTLR1 gene interference and immune challengesNote: Different letters represent significant differences (p<0.05), while the same letters represent insignificant differences (p>0.05).
图 8 HcTLR1 基因干扰及免疫刺激后血细胞中TLR1-MyD88-NF-κB信号通路及下游抗菌相关基因的相对表达量注:不同字母表示差异显著 (p<0.05),相同字母表示差异不显著 (p>0.05)。Fig. 8 Relative expression of TLR1-MyD88-NF-κB signaling pathway and downstream antimicrobial-associated gene in hemocytes after HcTLR1 gene interference and immune challengesNote: Different letters represent significant differences (p<0.05), while the same letters represent insignificant differences (p>0.05).3. 讨论
本研究克隆了三角帆蚌HcTLR1基因的cDNA序列,编码一个长为3 687 bp的ORF。蛋白质结构预测分析显示,HcTLR1蛋白中含有多个LRR结构域、1个跨膜结构域和1个胞内Toll/白细胞介素-1受体 (TIR),表现出典型的Toll样受体结构。其中,Toll样受体胞外结构域由LRR组成,每个LRR被疏水性的氨基酸间隔开,在识别多种病原体相关的分子模式中发挥重要作用[24-25];而胞内结构域由TIR构成,具有3个保守的氨基酸序列框,负责C端信号传导,并能与含有TIR结构域的接头蛋白MyD88发生相互作用以激活下游信号通路[26-28]。这表明HcTLR1能够识别多种病原体相关分子模式并启动天然免疫反应,参与三角帆蚌的抗菌应答过程。在维氏气单胞菌GL1、LPS、PNG和poly I:C刺激下,三角帆蚌鳃、肝胰腺和血细胞中HcTLR1基因的表达量在不同时间点呈现显著性差异。同时,不同刺激诱导下,免疫持续时效也存在差异。这一发现与已报道的贝类研究结果相符。例如,在副溶血弧菌刺激后,香港牡蛎 (C. hongkongensis) 鳃和血细胞中TLR4基因表达量显著性升高[29];在溶藻弧菌 (V. alginolyticus)、LPS、PGN和poly I:C刺激后,合浦珠母贝 (Pinctada fucata) 肝胰腺中PfTLR3和PfTLR13基因的表达量在不同时间点出现显著性差异[30-31];在LPS、PGN和poly I:C刺激后,三角帆蚌肝胰腺和鳃中TLRn基因的表达水平在不同时间点均显著性升高[20]。以上研究结果均证实了贝类TLRs在应对病原体和PAMPs的刺激时扮演着重要的免疫识别作用[32]。因此,推测HcTLR1分子具有广泛的识别谱,能够识别多种病原体及不同类型的PAMPs,并可能引起一系列级联反应,参与生物体防御功能。
三角帆蚌各组织HcTLR1基因定量结果显示,其在肝胰腺、斧足、闭壳肌、外套膜、鳃、性腺、血细胞、肾脏和肠等9种组织中均有不同程度的表达,特别是在血细胞中的表达量显著高于其他组织。而在贝类抵御外来病原微生物的过程中,血细胞起到了至关重要的作用,既是细胞免疫的主体,又是体液免疫的提供者[33]。已有研究发现,长牡蛎的TLR2和TLR3基因[34-35]及厚壳贻贝 (Mytilus coruscus) 的TLR-like1基因[36]均在血细胞中高表达,并在贝类的免疫应答中发挥关键作用。这与本研究的结果相似,提示HcTLR1在三角帆蚌抗菌应答中同样扮演着重要角色。
鉴于血细胞在贝类免疫系统中的中心地位[37-38],本研究在血细胞内进行RNA干扰实验。结果显示,HcTLR1的干扰导致多个基因 (如MyD88、IRAK4、TRAF6、p65、p105等) 的表达量明显下调,同时下游抗菌免疫基因 (如Ther、Lys、Def、WAP、LBP/BPI 2和IL-17) 的表达量也显著降低。这一结果与厚壳贻贝中干扰McTLRw导致McMyD88、McIRAK4和McTRAF6基因的表达受到显著抑制[39],青蛤 (Cyclina sinensis) 中干扰CsTLR13导致CsMyD88、CsIRAK4、CsTRAF6、CsIKKa、CsIkB、CsNF-kB、CsC-LYZ和CsAMP基因[40]表达显著下调,以及三角帆蚌中干扰HcTLRn导致NF-κB (p65、p105) 及Lys、Ther、WAP、LBP/BPI 1和LBP/BPI 2的表达显著下降[20]等研究结果相一致,这提示HcTLR1可能参与TLR信号通路的激活,且TLR1-MyD88-NF-κB信号通路在三角帆蚌先天免疫应答中起重要作用。Toll信号通路是宿主抗菌应答重要的信号转导通路之一[17],TLR1属于跨膜蛋白,位于信号通路最上游,当受到病原体刺激时,其胞内TIR结构域与包含TIR结构域的接头蛋白MyD88直接相互作用,并经过刺激将IRAK4吸引到TLRs,进而活化IKK复合体、MAPKs和NF-κB等级联反应,最终调节免疫相关基因的表达以发挥多种生物学功能[41]。因此,在三角帆蚌血细胞中,干扰HcTLR1 基因从而导致MyD88依赖性信号通路基因的表达受到抑制,表明HcTLR1可能通过激活MyD88-NF-κB依赖性信号通路来调节下游免疫相关基因的表达,从而发挥抗菌应答作用,但下游MAPK信号通路是否参与三角帆蚌的抗菌应答仍未可知。本研究结果对于深入了解Toll信号通路及HcTLR1在生物体内的抗菌应答机制具有积极意义。
-
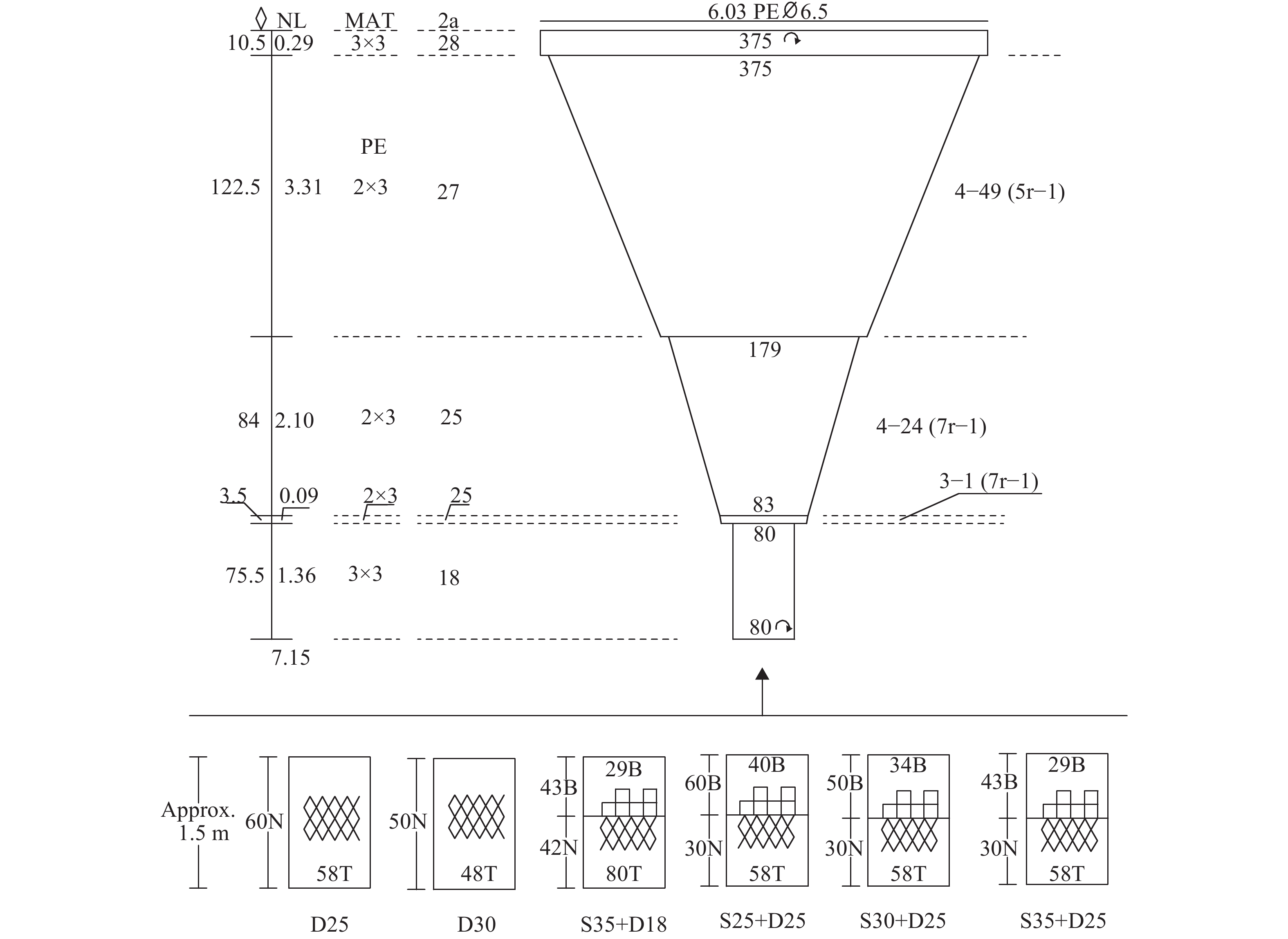
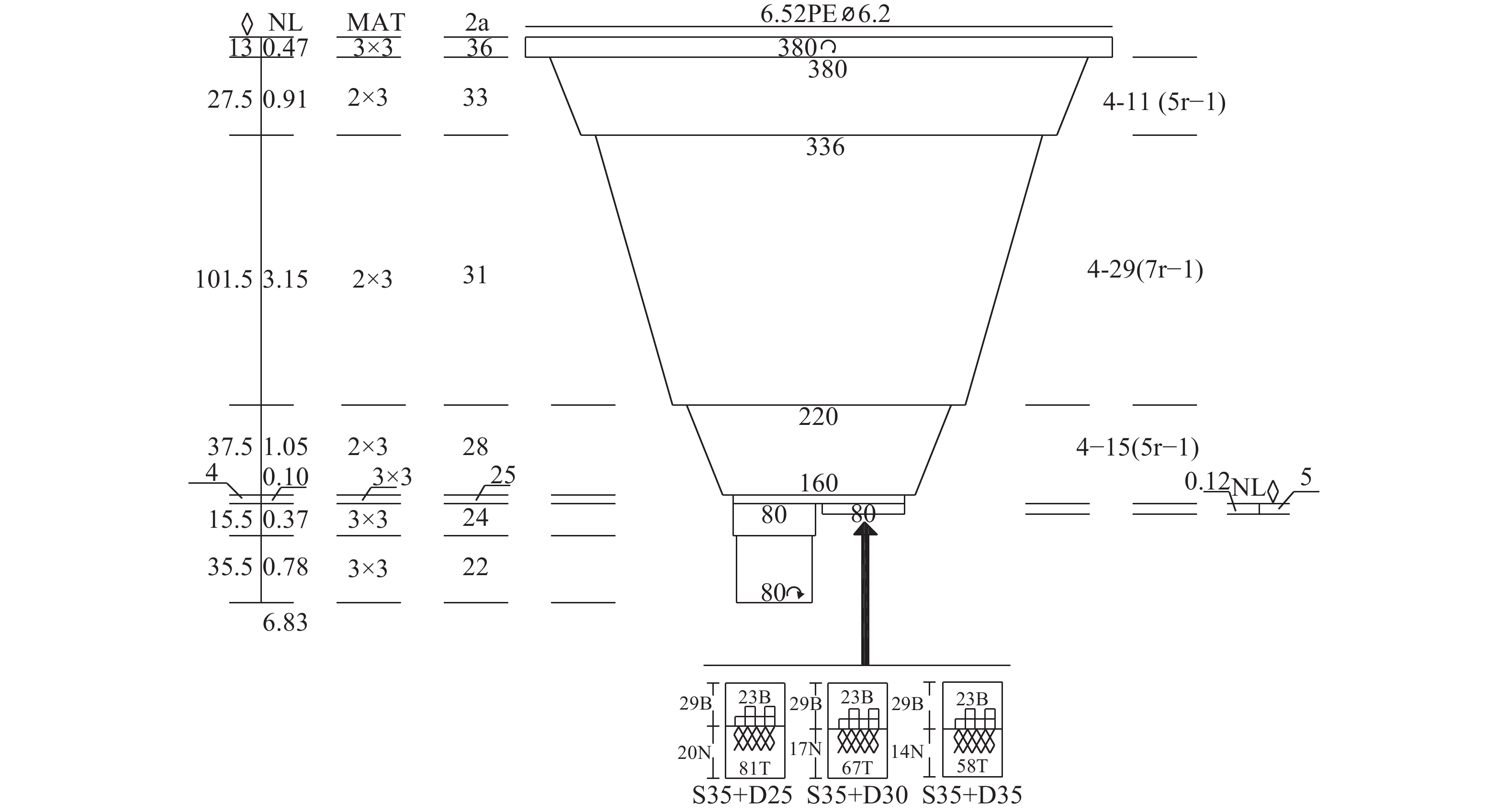
表 1 网囊选择性试验基本信息
Table 1 Basic information of tested codends
航次
trial时间
time网囊
codend试验渔船
vessel方法
method网目内径/mm
mesh opening网囊规格 (圆周×纵向)
codend specification
(circumferernce×vertical)有效网次
valid haul1 2014.08 D25 粤阳东渔 12057YYDY12057 套网法 covered 21.10±0.93 58×60 6 1 2014.08 D30 粤阳东渔 12057YYDY12507 套网法 covered 26.64±0.73 48×50 8 2 2015.08 S35+D18 粤阳东渔 12081YYDY12081 套网法 covered S: 32.70±0.63
D: 15.07±0.36S: 29×43,D: 80×42 8 3 2016.08 S25+D25 粤阳东渔 12081YYDY12081 套网法 covered S: 21.80±0.42
D: 21.80±0.42S: 40×60,D: 58×30 11 3 2016.08 S30+D25 粤阳东渔 12081YYDY12081 套网法 covered S: 27.14±0.45
D: 21.80±0.42S: 35×50,D: 58×30 12 3 2016.08 S35+D25 粤阳东渔 12081YYDY12081 套网法 covered S: 32.42±0.18
D: 21.80±0.42S: 29×43,D: 58×30 10 4 2017.09 S35+D25 粤阳东渔 12081YYDY12081 裤网法 trouser S: 32.42±0.18
D: 21.80±0.42S: 23×29,D: 81×20 11 4 2017.09 S35+D30 粤阳东渔 12081YYDY12081 裤网法 trouser S: 32.42±0.18
D: 27.14±0.45S: 23×29,D: 67×17 10 4 2017.09 S35+D35 粤阳东渔 12081YYDY12081 裤网法 trouser S: 32.42±0.18
D: 32.42±0.18S: 23×29,D: 58×14 8 合计 total 84 注:S. 方形网目;D. 菱形网目
Note: S. square mesh; D. diamond mesh表 2 试验渔获产量基本信息
Table 2 Basic information of fish catch
方法
method网囊
codend质量/kg mass 质量百分比/%
ratio of mass刀额新对虾
M. ensis总渔获
total catch套网法 cover D25 8.80 27.92 31.50 D30 13.43 45.01 29.83 S35+D18 9.50 28.53 33.28 S25+D25 15.36 23.36 65.77 S30+D25 4.94 33.61 14.70 S35+D25 20.13 37.60 53.53 裤网法 trouser S35+D25 18.47 32.47 56.89 S35+D30 7.18 42.91 16.73 S35+D35 2.92 23.27 12.53 合计 total 100.73 294.68 34.18 表 3 刀额新对虾的渔获数量及体长分布
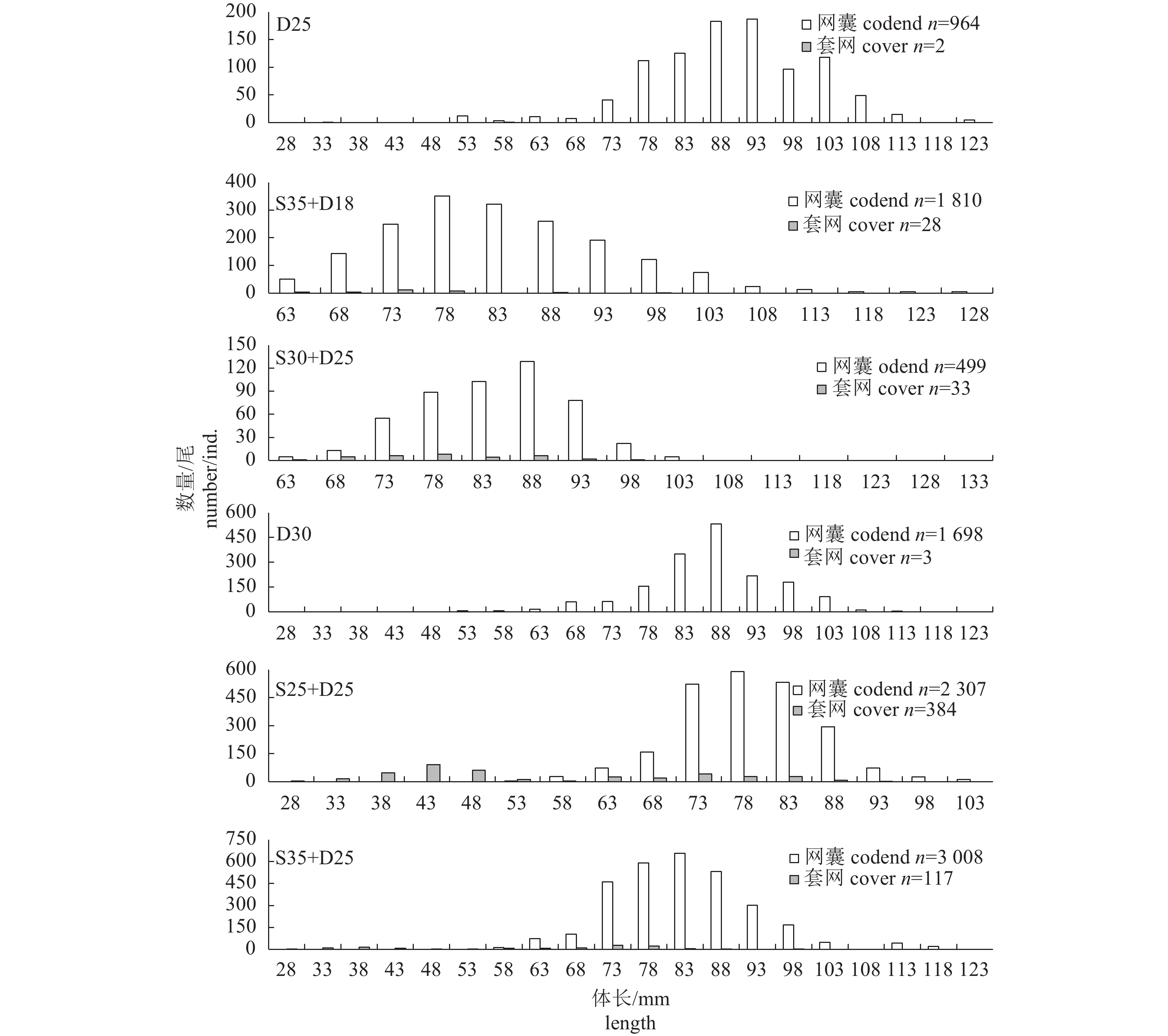
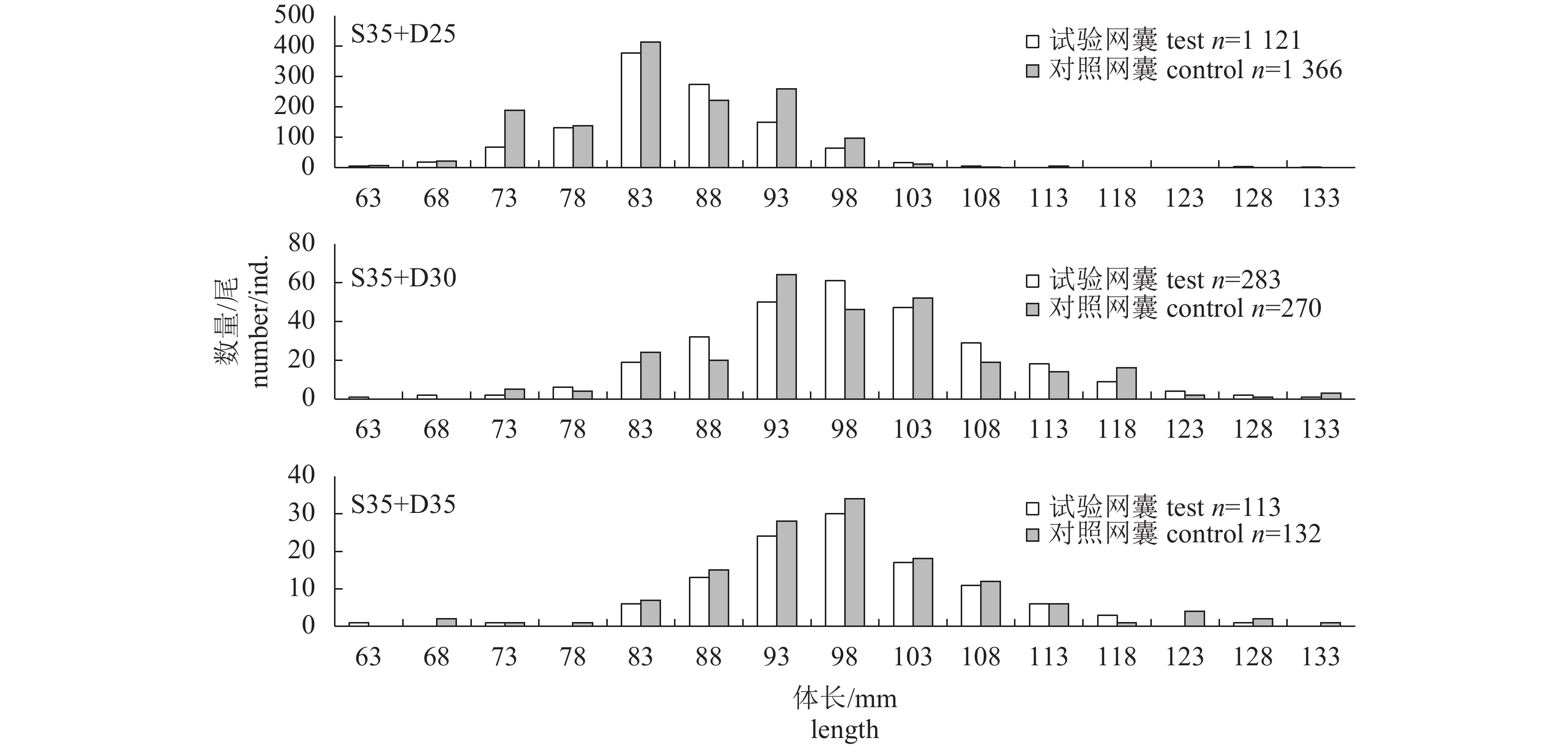
Table 3 Number of M. ensis caught by each codend
网囊
codend渔获尾数
number of catch体长范围/mm
length range众数体长/mm
modal length网囊
codend套网/对照网
cover/control网囊
codend套网/对照网
cover/control网囊
codend套网/对照网
cover/controlD25 964 2 53~123 33~58 88~93 (38.38%) D30 1 698 3 53~113 53~63 83~88 (51.94%) S35+D18 1 810 28 63~128 63~98 78~83 (37.07%) 73~78 (67.86%) S25+D25 2 307 384 58~103 28~93 78~83 (48.63%) 43~48 (39.84%) S30+D25 499 33 63~103 63~98 83~88 (46.49%) 73~78 (42.42%) [S35+D25]1 3 008 117 58~118 28~98 78~83 (41.42%) 73~78 (42.74%) [S35+D25]2 1 121 1 366 63~133 63~133 83~88 (58.25%) 83~93 (65.37%) S35+D30 283 270 63~133 73~133 93~103 (55.83%) 93~103 (60.00%) S35+D35 113 132 63~128 68~133 93~98 (47.79%) 93~98 (46.97%) 合计 total 11 803 2 335 注:[S35+D25]1. 套网法中的S35+D25网囊;[S35+D25]2. 裤网法中的S35+D25网囊
Note: [S35+D25]1. the S35+D25 codend for the covered codend method; [S35+D25]2. the S35+D25 codend for the trouser trawl method表 4 套网法选择性试验参数估算
Table 4 Selective parameters of tested codends with covered codend method
时间
time网囊
codend网次
NH选择性指标
selective index选择性参数
selective parameter拟合度
goodness of fit网次间差异
estimate of REP尾数
numberL50/
mmSD SR/
mmSD a SD b SD D df P Q d P REP 网囊
codend套网
cover2014 D25 c-b 964 2 2014 D30 c-b 1 698 3 2015 S35+D18 9 51.25 8.94 16.48 7.20 –6.83 4.14 0.13 0.06 4.295 8 0.83 203 11 2016 S25+D25 2 59.65 5.84 21.51 7.41 –6.09 2.66 0.10 0.04 5.919 7 0.55 137 24 4 53.89 12.68 50.44 26.94 –2.35 1.79 0.04 0.02 10.608 7 0.16 120 44 6 53.31 7.79 30.96 11.92 –3.78 1.98 0.07 0.03 6.283 9 0.71 127 31 9 60.86 1.19 6.05 1.07 –22.12 4.21 0.36 0.06 14.094 14 0.44 367 64 10 60.94 1.70 5.18 1.21 –25.84 6.39 0.42 0.10 2.288 14 1.00 278 75 c-b 60.84 1.74 14.31 1.90 –9.34 1.44 0.15 0.02 51.617 14 <0.05 155.94 39 <0.05 4.00 1 029 238 S30+D25 4 61.48 7.89 11.78 6.01 –11.47 7.25 0.19 0.10 9.811 10 0.46 106 4 5 63.18 3.47 13.93 3.23 –9.97 2.80 0.16 0.04 8.822 6 0.18 233 17 c-b 63.21 2.97 12.84 2.66 –10.82 2.70 0.17 0.04 3.657 9 0.93 20.22 21 0.51 0.96 342 21 S35+D25 1 66.16 1.22 3.56 0.89 –40.86 10.77 0.62 0.15 7.000 11 0.80 443 16 5 68.58 1.61 7.76 1.83 –19.42 4.92 0.28 0.07 9.946 6 0.13 188 17 7 62.43 3.45 9.00 2.31 –15.25 4.70 0.24 0.06 22.166 4 <0.05 356 15 8 65.19 3.10 10.64 2.78 –13.46 4.11 0.21 0.05 14.343 7 0.05 279 18 9 71.90 3.55 14.86 5.32 –10.63 4.20 0.15 0.05 5.303 6 0.51 40 12 c-b 64.53 1.15 9.75 1.00 –14.54 1.72 0.23 0.02 37.441 6 <0.05 72.31 71 0.43 1.02 1 306 78 注:NH. 网次;c-b. 联合网次;L50. 50%选择体长;SR. 选择范围;a、b. 选择性参数;SD. 标准差;D. 残差值;dof和d. 自由度;Q. 皮尔逊卡方统计量;REP. 过度离散叠加估算值. Note: NH. number of hauls; c-b. combined hauls; L50. 50% retention length; SR. selection range; a and b are selective parameters; SD. standard error; D. value of model deviance; dof and d indicate the degree of freedom; Q. Pearson chi-square statistic; REP. replication estimation of dispersion 表 5 裤网法选择性试验参数估算
Table 5 Selective parameters of tested codends with trouser trawl method
时间
time网囊
codend网次
NH选择性指标
selective index选择性参数
selective parameter拟合度
goodness of fit网次间差异
estimate of REP尾数
numberL50/mm SD SR/mm SD a SD b SD P SD D df P Q d P REP 网囊
codend对照
control2017 S35+D25 2 78.16 3.15 3.74 4.45 –45.88 53.61 0.59 0.70 0.41 0.07 8.262 9 0.51 24 43 3 75.35 3.03 7.17 4.69 –23.08 14.44 0.31 0.20 0.46 0.07 8.968 6 0.18 55 101 4 73.52 1.40 3.45 2.58 –46.77 34.53 0.64 0.48 0.41 0.03 39.625 10 <0.05 149 244 6 78.46 7.90 7.46 6.76 –23.12 19.45 0.29 0.27 0.77 0.10 10.982 10 0.36 28 12 7 72.79 5.32 6.92 7.07 –23.12 22.96 0.32 0.32 0.51 0.06 9.766 8 0.28 54 57 8 75.98 2.38 8.55 3.66 –19.52 7.89 0.26 0.11 0.54 0.04 47.531 6 <0.05 244 288 9 73.52 1.47 4.22 3.61 –38.32 32.46 0.52 0.45 0.51 0.03 29.720 7 <0.05 182 193 11 85.59 4.74 10.74 3.67 –17.51 5.22 0.20 0.07 0.73 0.08 25.517 10 <0.05 116 108 c-b 75.43 1.36 6.93 2.14 –23.93 7.09 0.32 1.00 0.51 0.03 53.559 12 <0.05 152.00 81 <0.05 1.88 852 1046 S35+D30 1 78.84 6.40 7.68 8.90 –22.57 25.18 0.29 0.33 0.65 0.08 15.361 8 0.05 30 20 5 86.79 4.96 6.92 6.25 –27.56 24.11 0.32 0.29 0.50 0.07 9.579 11 0.57 39 48 7 81.18 3.61 5.01 4.56 –35.60 31.57 0.44 0.40 0.44 0.05 12.484 11 0.33 62 90 8 88.47 5.70 11.32 7.00 –17.17 9.82 0.19 0.12 0.60 0.08 14.706 11 0.20 64 65 9 92.09 8.49 9.80 7.41 –20.64 14.18 0.22 0.17 0.71 0.14 5.986 8 0.65 23 19 c-b 82.38 2.01 6.39 2.72 –28.32 11.68 0.34 0.15 0.52 0.03 14.308 8 0.07 44.21 50 0.70 0.88 218 242 S35+D35 1 83.59 7.52 8.72 9.18 –21.06 21.32 0.25 0.27 0.55 0.10 10.743 10 0.38 23 24 2 92.13 8.37 9.62 9.67 –21.03 19.67 0.23 0.23 0.65 0.14 6.200 7 0.52 24 22 3 110.78 31.97 20.43 19.45 –11.91 8.42 0.11 0.10 0.77 0.29 6.397 9 0.70 17 19 5 100.52 37.58 29.78 31.29 –7.42 5.58 0.07 0.08 0.65 0.30 10.246 11 0.51 36 47 c-b 95.39 10.43 20.44 10.51 –10.25 4.44 0.11 0.06 0.64 0.11 11.599 11 0.39 22.73 34 0.93 0.67 100 112 -
[1] 闵信爱. 南海北部的新对虾[J]. 海洋渔业, 1987(1): 6-9. [2] 刘瑞玉, 钟振如.南海对虾类[M]. 北京: 农业出版社, 1988: 1-278. [3] 纪成林, 刘必亮. 世界新对虾属的资源现状及养殖发展前景[J]. 水产学报, 1996, 20(3): 72-77. [4] 杨炳忠, 杨吝, 谭永光, 等. 湛江近海虾拖网虾蟹类渔获组成[J]. 广东农业科学, 2014, 41(4): 152-155. doi: 10.3969/j.issn.1004-874X.2014.04.038 [5] 杨炳忠, 杨吝, 谭永光, 等. 南海区2种桁杆虾拖网渔获组成调查与分析[J]. 南方水产科学, 2017, 13(6): 115-122. doi: 10.3969/j.issn.2095-0780.2017.06.014 [6] WILEMAN D, FERRO R S T, FONTEYNE R, et al. Manual of methods of measuring the selectivity of towed fishing gear[R]. ICES Coop Res Rep, 1996, 215: 1-126.
[7] MAHJOUB M S, TAKEDA S, HAYASHI T, et al. Codend selectivity for jack mackerel and whitefin jack and unequal split parameter estimates observed in trouser trawl experiments[J]. Fish Sci, 2011, 77(2): 169-181. doi: 10.1007/s12562-011-0331-1
[8] 巩法慧, 张殿昌, 刘田田, 等. 中国东南沿海刀额新对虾群体形态学比较研究[J]. 南方水产科学, 2016, 12(6): 76-82. doi: 10.3969/j.issn.2095-0780.2016.06.010 [9] 余景, 陈丕茂, 冯雪. 珠江口浅海4种经济虾类的食性和营养级研究[J]. 南方农业学报, 2016, 47(5): 736-741. doi: 10.3969/j:issn.2095-1191.2016.05.736 [10] 张健, 孙满昌, 罗炎标. 广东大鹏湾虾拖网网囊网目选择性研究[J]. 海洋渔业, 2007, 29(2): 166-173. doi: 10.3969/j.issn.1004-2490.2007.02.012 [11] 杨炳忠, 杨吝, 谭永光, 等. 南海区虾拖网方目与菱目混合网囊渔获性能初步研究[J]. 海洋科学, 2017, 41(1): 57-64. [12] 杨炳忠, 杨吝, 谭永光, 等. 南海区桁杆虾拖网方目与菱目混合网囊网目选择性研究[J]. 南方水产科学, 2018, 14(1): 105-113. doi: 10.3969/j.issn.20950780.2018.01.014 [13] 杨吝. 南海区海洋渔具渔法[M]. 广州: 广东科技出版社, 2002: 1-237. [14] 杨吝, 张旭丰, 张鹏, 等. 南海区海洋小型渔具渔法[M]. 广州: 广东科学技术出版社, 2007: 1-310. [15] MILLAR R B. Sampling from trawl gears used in sized selectivity experiments[J]. ICES J Mar Sci, 1994, 51(3): 293-298. doi: 10.1006/jmsc.1994.1030
[16] MILLAR R B. Reliability of size-selectivity estimates from paired-trawl and covered-codend experiments[J]. ICES J Mar Sci, 2010, 67(3): 530-536. doi: 10.1093/icesjms/fsp266
[17] MILLAR R B, FRYER R J. Estimating the size-selection curves of towed gears, traps, and hooks[J]. Rev Fish Biol Fish, 1999, 9(1): 89-116. doi: 10.1023/A:1008838220001
[18] 杨炳忠, 张鹏, 谭永光, 等. 基于SELECT模型的南海金线鱼刺网选择性研究[J]. 南方水产科学, 2016, 12(5): 8-15. doi: 10.3969/j.issn.2095-0780.2016.05.002 [19] 東海正. MS-Excelのソルバーによる曳網の網目選択性Logistic式パラメータの最尤推定[J]. 水産海洋研究, 1997, 61(3): 288-298. [20] 東海正, 三橋廷央. 比較操業実験から選択性曲線を求めるSELECTモデルについて[J]. 水産海洋研究, 1998, 62(3): 235-247. [21] 孙满昌. 渔具渔法选择性[M]. 北京: 中国农业出版社, 2004: 1-305. [22] MADSEN N, SKEIDE R, BREEN M, et al. Selectivity in a trawl codend during haul-back operation: an overlooked phenomenon[J]. Fish Res, 2008, 91(2/3): 168-174.
[23] MILLAR R B, BROADHURST M K, MACBETH W G. Modelling between-haul variability in the size selectivity of trawls[J]. Fish Res, 2004, 67(2): 171-181. doi: 10.1016/j.fishres.2003.09.040
[24] 张健, 孙满昌, 钱卫国. 桁拖网不同网目结构网囊对主要鱼类的选择性研究[J]. 上海水产大学学报, 2008, 17(4): 457-463. [25] 张健, 孙满昌. 桁拖网不同网目结构的网囊对虾类的选择性[J]. 中国水产科学, 2008, 15(4): 667-676. doi: 10.3321/j.issn:1005-8737.2008.04.019 [26] 陈丕茂. 南海北部主要捕捞种类最适开捕规格研究[J]. 水产学报, 2004, 28(4): 393-400. [27] SISTIAGA M, HERRMANN B, GRIMALDO E, et al. Assessment of dual selection in grid based selectivity systems[J]. Fish Res, 2010, 105(3): 187-199. doi: 10.1016/j.fishres.2010.05.006
[28] FRANDSEN R P, HERRMANN B, MADSEN N, et al. Development of a codend concept to improve size selectivity of Nephrops (Nephrops norvegicus) in a multi-species fishery[J]. Fish Res, 2011, 111(1/2): 116-126.
[29] POL M V, HERRMANN B, RILLAHAN C, et al. Impact of codend mesh sizes on selectivity and retention of Acadian redfish Sebastes fasciatus in the Gulf of maine trawl fishery[J]. Fish Res, 2016, 184: 54-63. doi: 10.1016/j.fishres.2016.06.013
-
期刊类型引用(10)
1. 衣帆,王娇,刘航,陈静,陈琳琳,李晓静,李学鹏,李宝泉. 烟台长岛秋季海洋牧场内外大型底栖动物群落特征分析. 海洋学报. 2024(05): 57-67 .  百度学术
百度学术
2. 袁华荣,章守宇,陈丕茂. 海洋牧场建设效益评价研究进展与展望. 南方水产科学. 2024(05): 1-13 .  本站查看
本站查看
3. 张启宇,张文博,郑奕,陈英义,何锦辉. 基于CiteSpace分析的国内海洋牧场研究进展. 农业工程. 2024(11): 65-73 .  百度学术
百度学术
4. 马文刚,夏景全,魏一凡,尹洪洋,覃乐政,刘相波,胡雪晴,许强,李秀保,王爱民. 三亚蜈支洲岛海洋牧场近岛区底表大型底栖动物群落结构及评价. 热带海洋学报. 2022(03): 135-146 .  百度学术
百度学术
5. 张亚洲,蒋日进,梁君. 嵊泗马鞍列岛海域国家级海洋牧场渔业资源增殖养护效果评析. 浙江海洋大学学报(自然科学版). 2022(05): 466-472 .  百度学术
百度学术
6. 许双杰,高源. 中国海洋牧场发展潜力的时空差异分析. 海洋经济. 2022(06): 64-73 .  百度学术
百度学术
7. 刘伟峰,刘大海,管松,姜伟. 海洋牧场生态效益的内涵与提升路径. 中国环境管理. 2021(02): 33-38+54 .  百度学术
百度学术
8. 王书献,张胜茂,戴阳,王永进,隋江华,朱文斌. 利用声呐数据提取磷虾捕捞深度方法研究. 南方水产科学. 2021(04): 91-97 .  本站查看
本站查看
9. 裴琨,吴一桂,杨润琼. 中国最早的人工鱼礁试验地——防城港市白龙珍珠湾海洋牧场人工鱼礁建设概述. 河北渔业. 2020(06): 22-27+63 .  百度学术
百度学术
10. 吴程宏,张羽翔,赵海龙,陈敏,刘维,林国尧. 海南文昌冯家湾人工鱼礁区渔业资源养护效果初步评估. 海洋湖沼通报. 2020(06): 158-167 .  百度学术
百度学术
其他类型引用(8)



 下载:
下载:








 粤公网安备 44010502001741号
粤公网安备 44010502001741号
