Molecular cloning, expression of alpha-amylase gene of Haliotis diversicolor supertexta and correlation with growth traits
-
摘要:
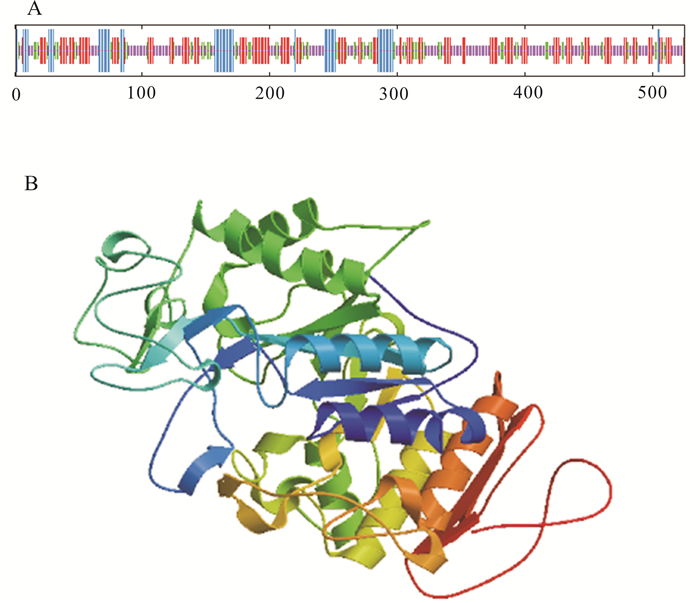
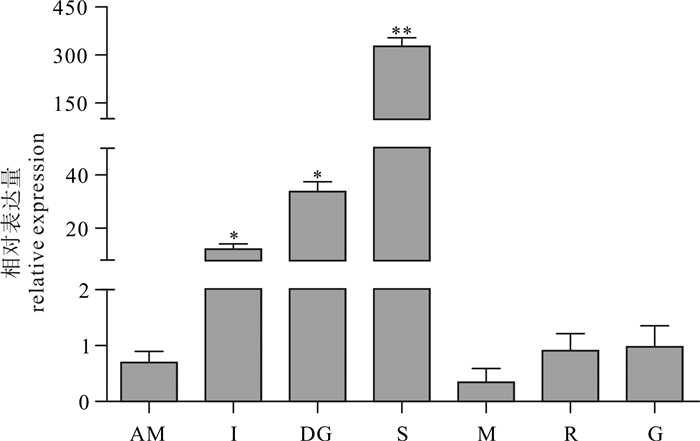
实验采用cDNA末端快速扩增(RACE)和反转录聚合酶链式反应(RT-PCR)技术对九孔鲍(Haliotis diversicolor supertexta)α-淀粉酶基因cDNA进行克隆,并分析该基因的组织表达及其与生长性状的相关性。结果表明,九孔鲍α-淀粉酶基因cDNA全长为2 260 bp,其中开放阅读框长度为2 088 bp,共编码695个氨基酸。经SingalP分析,α-淀粉酶多肽链中含有信号肽结构,且长度为18个氨基酸(MWAQYGIVSALLVLSASA),由该多肽链折叠成的三级结构中含有domain A、domain C 2个结构域,并且在domain A第28~第396氨基酸处存在催化中心。该基因在九孔鲍右侧壳肌、肠、肝脏、胃、外套膜、吻、腹足7种组织中均有不同程度表达。其中在胃中表达量最高,表达量高达326.803;而在外套膜中表达量最低,表达量仅为0.35。经ANOVA分析,消化组织与非消化组织间的α-淀粉酶基因表达量存在显著差异(P < 0.05)。α-淀粉酶基因mRNA表达量与生长性状呈显著正相关(P < 0.05)。
Abstract:We cloned the alpha-amylase gene cDNA of Haliotis diversicolor supertexta by rapid amplification of cDNA ends method (RACE) and reverse transcription polymerase chain reaction (RT-PCR) to analyze its tissue expression and correlation with growth traits. The results show that the full length of alpha-amylase gene cDNA was 2 260 bp with an open reading frame (ORF) of 2 088 bp encoding 695 amino acids. The alpha-amylase contained a signal peptide with 18 amino acid residues (MWAQYGIVSALLVLSASA). Two functional structure domains (domain A, domain C) were found in the protein of alpha-amylase. Domain A contained one catalytic center between 28th and 396th amino acids. The alpha-amylase mRNA expression in seven tissues (adductor muscle, intestine, digestive gland, stomach, mantle, rostral, foot) was observed, highest in stomach (326.803) and lowest in mantle (0.35). Significant difference was found in the expression of alpha-amylase between digestive and non-digestible tissues (P < 0.05). Significant positive correlation existed between alpha-amylase gene expression and growth traits (P < 0.05).
-
Keywords:
- Haliotis diversicolor supertexta /
- alpha-amylase /
- gene clone /
- gene expression /
- growth traits
-
剩余产量模型因其简单的形式和所需数据较少的特点为渔业资源评估广泛采用[1]。当渔获物的年龄组成无法精确获得时,年龄结构模型(age-structured model)便不能应用,而剩余产量模型只需多年的渔获量和捕捞努力量或单位捕捞努力量渔获量的渔业统计数据。剩余产量模型也可作为年龄结构模型的辅助工具,为渔业动态研究提供不同的思路。这类模型的优点在于其形式简单便于评估和评估结果比较易于理解[2]。通常较难获得现实渔业的相关参数真值,文章主要利用蒙特卡罗(Monte Carlo)模拟方法来检验连续Fox产量模型在几种不同渔业史条件下的模拟渔业中评估结果的有效性,借此来探索能使连续Fox产量模型产生最佳评估效果所需要的渔业条件,为渔业管理部门和渔业种群数量变动的研究人员使用该种模型对真实渔业进行管理和研究时提供参考。
1. 材料与方法
1.1 模型
Fox模型[3]是一个连续的微分方程,假定生长率与生物量的关系为Gompertz分布,方程如下:
$$ d B_t/d t=r B_t \ln \left(K/B_t\right)-C_t $$ (1) 式中Bt表示t时间的生物量,dBt/dt为种群增长率,r为种群内禀生长率,K为种群最大生物量或称环境容纳量,Ct为t时间的捕捞量。
设时间间隔为一年对方程(1)进行积分,得到一个连续的Fox形生物量动态方程:
$$ \begin{aligned} & \ln B_{t+1}=\left(1-\mathrm{e}^{-r}\right) \ln (K)+\mathrm{e}^{-r} \ln \left(B_t\right)-(1- \\ & \left.\mathrm{e}^{-1}\right) r^{-1} F_t \end{aligned} $$ (2) $$ \begin{aligned} &\text { 渔获量: } C_t=\int_t^{t+1} F_t B_t d t=F_t \int_{B_t}^{B_{t+1}} d B_t/[rln (K)\\ &\left.-r \ln \left(B_t\right)-F_t\right] \end{aligned} $$ (3) $$ \begin{aligned} & \quad C_t=F_t B_t\left[\left(1+\mathrm{e}^{-r}/r-r^{-1}\right) \times\left(\ln (K)-\ln \left(B_t\right)\right.\right. \\ & \left.\left.-r^{-1} F_t\right)+1\right] \end{aligned} $$ (4) Ft为t时刻的捕捞死亡系数。Ft=qft,q为可捕系数,ft为t年中的捕捞努力量。
1.2 评估方法
文章所用的评估方法是假设生物量方程(2) 没有误差,误差仅出现在渔获量方程(4)中。因此,此模型估计参数的方法为观测误差估计法,假设渔获量方程存在相乘的误差结构,目标函数表示为:
$$ \min \sum\limits_{t=1}^{\mathrm{T}}\left[\ln \left(C_t\right)-\ln \left(\hat{C}_t\right)\right]^2 $$ 其中t表示年份,Ct表示t年中渔获量的观测值,Ĉt表示t年中渔获量的估计值。应用Excel中的规划求解计算出参数的估计值。直接计算的参数包括B1 (第一年的生物量)、r (种群内禀增长率)、K (环境容纳量)和q (可捕系数)。具体算法如下:
(1) 给出4个参数(B1、r、K、q)的起始估计值。
(2) 利用方程(2)计算出逐年的生物量,再利用方程(4)计算逐年的渔获量估计值。
(3) 最小化目标函数。
(4) 监测目标函数收敛性,如果收敛, 计算结束。否则,调整各个参数起始值后,从步骤2重新做,直至目标函数收敛。
当种群的参数(B1、r、K和q)通过非线性回归方法得出后,相应重要的管理参数可以计算得出fMSY=r/(2q),MSY=rKe-1,其中MSY表示最大可持续产量(maximal stay yield),fMSY表示获得最大可持续产量时的捕捞努力量。
文章通过以下各项指标来比较模型在各个渔业中的评估效果,参数估计值与真值之间的偏差(relative bias of median,RBM%)=|真实值-估计值|×100%/真实值,各个参数的四分位数间距(interquartile range,IQR),相对四分位数间距(relative interquartile range,RIQR%)=四分位数间距×100%/中值。预测误差百分数(percentage prediction error,PPE%)=|CPUE真实值-CPUE估计值|×100%/CPUE真实值[3]。为了计算真实渔业各参数的各项指标,文章采用了非参数化Bootstrap分析法[4],Bootstrap分析过程重复了1 000次,在随机抽取数据过程中,采用的是成对抽取数据。
1.3 模拟渔业
文章采用蒙特卡罗模拟方法产生数据,该方法需要一个操作模型和一个评估模型,采用的操作模型和评估模型都是连续形Fox产量模型。假设不同的渔业史或渔业过程,即给出不同的捕捞努力量变化过程,根据连续形Fox产量模型方程(2)、(4),计算出相应渔业的模拟生物量和渔获量,在此基础上叠加正态分布的随机数[5]作为该渔业的观测值。文章设置了不同的白色噪音水平(变异系数为1%、10%、30%和50%)。种群参数的真值都设为r=0.4,K=1 000,q=0.01,B1的真值随不同的渔业过程而不同。根据相应的计算公式主要的管理参数真值为fMSY=20,MSY=147.15。图 1-a显示的是一种过度捕捞型渔业,以下称为渔业一;该渔业从开始就经历了高的捕捞努力量,并且捕捞努力量逐年增加,生物量逐渐减少。图 1-b显示的是一种恢复型渔业,以下称为渔业二;该渔业由于经历了过度捕捞的缘故,初始阶段生物量较低, 随后采取了保护措施,捕捞努力量一直被限制在较低的水平,生物量随着时间不断增加。图 1-c显示的是一种波动型渔业,也可称为管理良好的渔业,以下称为渔业三;该渔业开始于资源未开发阶段,加入高捕捞努力量生物量下降,然后限制捕捞努力量,生物量逐渐增加,生物量和捕捞努力量随时间上下波动。
1.4 真实渔业
北大西洋剑鱼渔业评估所用数据是ICCAT对该渔业产量和生物量丰度指标——单位捕捞努力渔获量(catch per unit of effort,CPUE)的统计[6],共有1975~1998年24年间的数据,该渔业1988年以前产量逐渐升高,但是CPUE却不断降低,随后产量逐渐降低,CPUE降低趋势趋于平缓(图 2)。
2. 结果
文章对每种渔业在不同白色噪音水平下分别进行了1 000次模拟分析计算,去除部分无效结果(包括参数值< 0、q>=1、B1>K 3种情况)按白色噪音0.01、0.1、0.3、0.5的顺序,各渔业的有效结果次数,渔业一为524、577、639、172;渔业二为496、355、114、22;渔业三为485、445、425、90。取各估计参数的中值为参数的估计值。RBM%、IQR、RIQR%的最终计算结果列于表 1~表 3。
表 1 参数估计值与真实值之间偏差RBM%Table 1. Bias between the estimated and true parameter values白色噪音 white noise 0.01 0.1 0.3 0.5 B1 渔业一 7.1×10-10 2.2×10-4 10.2 21.4 渔业二 1.1×10-6 2.61 0.3 0.3 渔业三 1.9×10-6 0.64 0.9 2.9 r 渔业一 0.001 0.73 2.7 1.8 渔业二 0.03 0.02 1.4 7.8 渔业三 0.01 0.57 0.9 11.5 K 渔业一 1.1×10-8 0.3 2.6×10-4 4.3 渔业二 6.11×0-7 9.3×10-4 0.6 6.0 渔业三 6.3×10-10 0.9 7.5 17.2 q 渔业一 0.2 1.31 0 7.5 渔业二 0.2 0.26 3.4 32.8 渔业三 0.3 2.8 16.7 2.4 最大可持续产量
MSY渔业一 0.01 0.5 3.8 2.0 渔业二 0.02 1.07 0.1 6.9 渔业三 6.5×10-4 0.08 5.8 1.9 最大可持续产量时的捕捞努力量
fMSY渔业一 0.2 1.4 0.4 3.8 渔业二 0.2 0.04 8.6 36.7 渔业三 0.3 3.3 12.0 15.2 表 2 参数估计值的四分位数间距IQRTable 2. Interquartile range of the estimated parameters白色噪音 white noise 0.01 0.1 0.3 0.5 B1 渔业一 2 222 976 1 306 渔业二 3 139 268 325 渔业三 13 136 411 799 r 渔业一 0.001 0.04 0.20 0.26 渔业二 0.002 0.10 0.20 0.20 渔业三 0.002 0.04 0.14 0.20 K 渔业一 0.2 192 878 1 172 渔业二 0.7 565 1 036 828 渔业三 9.0 209 621 1 287 q 渔业一 6.6×10-5 0.002 0.01 0.013 渔业二 9.9×10-5 0.006 0.01 0.012 渔业三 6.4×10-5 0.002 0.006 0.007 最大可持续产量
MSY渔业一 0.5 14 56 83 渔业二 0.6 40 81 66 渔业三 0.6 14 44 81 最大可持续产量时的捕捞努力量
fMSY渔业一 0.1 2.6 11.7 14.5 渔业二 0.3 6.5 12.7 8.6 渔业三 0.09 1.3 3.3 4.8 表 3 参数估计值的相对四分位数间距RIQR%Table 3. Relative IQR(RIQR%)of the estimated parameters白色噪音 white noise 0.01 0.1 0.3 0.5 B1 渔业一 0.3 4.9 110.8 134.5 渔业二 1.4 67.7 133.6 162.2 渔业三 1.6 17.1 51.9 97.1 r 渔业一 0.3 9.8 52.3 65.3 渔业二 0.4 24.2 48.9 44.8 渔业三 0.5 11.2 34.5 51.9 K 渔业一 0.2 19.2 87.8 112.4 渔业二 0.07 56.5 104.2 88.1 渔业三 0.9 20.7 67.1 109.9 q 渔业一 0.7 18.3 101.7 125.3 渔业二 0.9 57.0 137.9 92.6 渔业三 0.6 16.7 53.6 72.1 最大可持续产量
MSY渔业一 0.4 9.6 39.5 55.4 渔业二 0.3 26.8 55.1 48.5 渔业三 0.4 9.6 31.6 54.4 最大可持续产量时的捕捞努力量
fMSY渔业一 0.6 13.2 58.1 75.1 渔业二 1.3 32.5 69.3 68.3 渔业三 0.5 6.9 18.5 28.2 图 3、图 4体现了在白色噪音为10%的情况下3种模拟渔业的观测产量和估计产量的对比关系。
文章还计算了各渔业的预测误差百分数PPE%(图 5)。从总体上看,当白色噪音小于10%的情况下,所有模拟渔业的各个参数估计值接近于真实值。当白色噪音达到30%时,评估结果偏差较大,模型表现不好。从各参数的评估结果来看,渔业起始生物量B1和最大环境容纳量K的评估结果偏差较小,但是RIQR%较大,参数r和q的评估结果比较接近真值。MSY和fMSY的评估结果最为准确,总体RIQR%小于75.1%。从不同渔业来看,用RBM%来衡量,模型在渔业一中的表现为最好,其次为渔业三,各个渔业24个被估参数的偏差平均值,渔业一为2.6%、渔业为二4.6%、渔业三为4.3%;用RIQR%来衡量,模型在渔业三中表现最好,各渔业RIQR%的平均值,渔业一为46.6%、渔业二为55.1%、渔业三为31.6%。
对北大西洋剑鱼渔业的1 000次Bootstrap分析结果列于表 4中。为证明可靠渔业信息对评估结果准确性的影响,文章在白色噪音为0.1的情况下对参数加以约束,假设B1=0.2K,将评估参数减少为3个,对渔业2进行了分析计算,并将计算结果与参数未加约束时作了比较,比较结果见表 5。
表 4 北大西洋剑鱼渔业参数估计值和参数四份位数间距及相对四份位数间距Table 4. Median and IQR, IQR% for the North Atlantic swordfish fishery参数名称
parameter参数中值
median四分位数间距
IQR相对四分位数间距
RIQR%B1 30 773 6 021 19.6 r 0.94 0.05 5.6 K 44 733 2 697 6.0 q 0.013 0.001 9.2 最大可持续产量 MSY 15 678 621 3.9 最大可持续产量时的捕捞努力量 fMSY 37 4.5 12.3 表 5 约束参数与不约束参数评估结果比较Table 5. Comparison of the results with constrained and unconstrained parametersr K q 最大可持续产量
MSY最大可持续产量时的捕捞努力量
fMSY四分位数间距
IQR无约束 0.10 565 0.006 40.0 6.5 约束 0.03 116 0.001 8.9 2.1 相对四分位数间距
RIQR%无约束 24.2 56.5 57.0 26.8 32.5 约束 8.2 11.9 11.2 6.1 10.7 3. 讨论
采用连续Fox模型对渔业初始生物量B1和环境容纳量K的评估结果表明,虽然估计值与真实值之间的偏差较小,但是RIQR%较大,这是因为将中值作为最终参数估计值时个别较大值和较小值对中值影响很小,因此,建议将中值作为最终估计值来平衡个别较大值和较小值对结果的影响。B1和K的RIQR%较大,证明模型对这2个参数的评估结果不够理想,个别渔业甚至出现B1估计值高于环境容纳量K真实值的情况,在最终计算参数时将这样的结果归为无效结果删除,以防止影响结果的准确性,产生这种情况主要是由于白色噪音的影响及该模型对数据较为敏感。但是当B1估计值较大时,K估计值也偏高,B1估计值准确性较差可能主要是由于模型为非线性的,且同时要估计4个参数所导致的,在模拟分析计算时假设B1值与K值呈线性关系;如渔业二中假设B1=0.2K,这样评估参数减少为3个,如表 5所示,添加约束时各参数的IQR和RIQR%仅为未添加约束时的1/3左右,所得结果准确性明显好于同时评估4个参数时的结果。
具体的渔业种群评估中,如果能合理利用渔业信息以确定某个参数,则可以简化计算,并且能提高评估结果的准确性。
模型估计管理参数MSY和fMSY的准确性高于其它参数,这可能由于被估各参数间存在一定的相关性[7],MSY和fMSY的估计值是参数r、K、q以相乘或相除形式结合在一起,这样降低了单个参数估计偏差的影响程度。因此,管理参数MSY和fMSY的估计值更为准确[8]。
当白色噪音大于30%时,参数评估偏差较大,模型表现不好,这可能主要因为较高的白色噪音干扰了种群动态变动的内在规律。例如,噪音较大时曾出现观测产量为负值的不合理情况,使得模型不能捕捉渔业在没有噪音情况下的真实关系。所以,高质量的渔业统计数据对资源评估结果十分重要。
渔业三评估结果好于其它渔业可能因为该渔业经历了过度捕捞和恢复2个渔业阶段,渔业过程较为完整,所含数据信息丰富。而其它2种渔业的信息相对缺乏,导致模型对这2种渔业评估表现较渔业三差。
文章评估北大西洋剑鱼(Xiphias gladius)渔业的MSY为15 678 t,fMSY为37×106钩次·a-1;PRAGER[6]评估该渔业的MSY为14 070和14 510 t;孙利东[8]评估该渔业的MSY为12 493 t,fMSY为53×106钩次·a-1。文章评估的MSY与其它不同模型评估的MSY结果相差不大,证明连续性Fox模型对北大西洋剑鱼渔业的评估结果具有参考价值。该渔业的捕捞努力量从20世纪80年代开始就超过了文章评估的fMSY,这种情况造成了北大西洋剑鱼渔业的衰退,评估结果与图 2反映的情况吻合。从20世纪80年代开始该渔业的CPUE不断降低,为了保护和合理利用该渔业资源,应该将捕捞努力量严格控制到合理水平。
-
表 1 九孔鲍α-淀粉酶基因全长克隆及实时荧光定量表达中所有引物序列
Table 1 Full length cloning of alpha-amylase gene of H.diversicolor supertexta and sequence of all primers in real time qPCR
引物primer 序列sequence AMY(s) GTTTGTCCGAGTGATGAGTAGTTATTT AMY(a) ACTATTGGTTCCTCGTTGTTGCT 5′Race GATTACGCCAAGCTTAGCCATTTCCGCACGAACCATCTCC 3′Race GATTACGCCAAGCTTACAGTGGATGGCTCAGGAAATGCTC UPM TAATACGACTCACTATAGGGCAAGCAGTGGTATCAACGCAGAGT qRT-AMY(s) ATGGTAGCGTTCAGGAATGCCGTAG qRT-AMY(a) GGGAGCCCTGTTTGGAATGTTTGAT β-actin(s) CCATCCAGGCTGTTCTGTCTC β-actin(a) GCGGTGGTGGTGAATGAGTAA 表 2 九孔鲍α-淀粉酶同源性分析
Table 2 Alpha-amylase homology analysis of H.diversicolor supertexta
物种species 同源性/% homology 九孔鲍(H.diversicolor supertexta) 100 皱纹盘鲍(H.discus hannai) 87.9 100 盘鲍(H.discus discus) 87.9 100 100 合浦珠母贝(Pinctada fucata) 46.7 46.9 46.9 100 长牡蛎(Crassostrea gigas) 64.7 67.6 67.6 50.5 100 海蜗牛(Janthina janthina) 45.7 45.9 45.9 63.1 47.6 100 草鱼(Ctenopharyngodon idellus) 44.5 44.5 44.5 56.0 45.8 53.1 100 黄斑篮子(Siganus canaliculatus) 45.4 45.8 45.8 57.8 47.5 55.5 76.0 100 小鼠(Mus musculus) 48.1 47.9 47.9 58.4 50.2 56.1 70.3 72.0 100 山羊(Capra hircus) 49.4 49.2 49.2 59.2 51.0 54.1 72.4 73.4 82.2 100 人(Homo sapiens) 48.7 48.7 48.7 58.0 50.4 55.1 69.5 72.2 84.3 87.1 100 表 3 九孔鲍2个群体生长性状测定
Table 3 Results of growth traits in two groups of H.diversicolor supertexta
壳长/mm
shell length壳宽/mm
shell width体质量/g
body mass基因表达量
gene expression大个体群体big group 30.73±1.82a 22.88±1.24a 4.23±0.30a 27.40±0.83a 小个体群体small group 20.30±2.28b 15.67±1.37b 3.23±0.17b 19.92±1.22b 注:同列不同上标字母表示差异显著(P<0.05)
Note:Values with different superscripts in the same column indicate significant difference(P<0.05).表 4 九孔鲍α-淀粉酶基因表达量与生长性状的相关性
Table 4 Correlation between alpha-amylase gene expression and growth traits for H.diversicolor supertexta
生长性状
growth trait相关系数
correlation coefficient壳长shell length 0.888** 壳宽shell width 0.846** 体质量body mass 0.838** 注:**代表差异极显著(P<0.01)
Note:** indicates very significant difference(P<0.01). -
[1] 蔡英亚, 谢绍河.广东的海贝[M].汕头:汕头大学出版社, 2006:59-60. [2] NIE Z Q, WANG S P.The status of abalone culture in China[J].J Shellfish Res, 2004, 23(4):941-945. https://www.researchgate.net/publication/287772058_The_status_of...
[3] MGAYA Y D, MERCER J P.The effects of size grading and stocking density on growth performance of juvenile abalone, Haliotis tuberculata Linnaeus[J].Aquaculture, 1995, 136(3/4):297-312. https://es.scribd.com/document/83280314/OIE-Manual-Diagnostic-ENGLISH
[4] 蒋湘. 九孔鲍群体选育与杂交育种[D]. 湛江: 广东海洋大学, 2013: 1-2. http://cdmd.cnki.com.cn/Article/CDMD-10566-1013225131.htm [5] 蒋湘, 刘建勇, 赖志服.九孔鲍幼鲍生长性状的遗传参数估计[J].中国水产科学, 2013, 20(3):544-550. http://www.wenkuxiazai.com/doc/a4edf3a15022aaea998f0fdf.html [6] 胡志国, 刘建勇, 包秀凤, 等.九孔鲍双列杂交家系子代的杂种优势与配合力分析[J].南方水产科学, 2014, 10(1):43-49. http://www.schinafish.cn/CN/abstract/abstract9106.shtml [7] HUANG X, GUO F, KE C, et al.Responses of abalone Haliotis diversicolor to sublethal exposure of waterborne and dietary silver and cadmium[J].Ecotoxicol Environ Saf, 2010, 73(6):1130-1137. doi: 10.1016/j.ecoenv.2010.05.018
[8] ZHOU J, ZHU X S, CAI Z H.The impacts of bisphenol A (BPA) on abalone (Haliotis diversicolor supertexta) embryonic development[J].Chemosphere, 2011, 82(3):443-450. doi: 10.1016/j.chemosphere.2010.09.056
[9] SELLOS D Y, van WORMHOUDT A.Structure of the of α-amylase genes in crustaceans and molluscs:evolution of the exon/intron organization[J].Biologia (Bratisl), 2002, 57(11):191-196. https://ar.scribd.com/document/164097540/Gene-Regulation-Taylor...
[10] DA LAGE J L, MACZKOWIAK F, CARIOU M L.Phylogenetic distribution of intron positions in alpha-amylase genes of bilateria suggests numerous gains and losses[J].PLoS One, 2011, 6(5):e19673. doi: 10.1371/journal.pone.0019673
[11] NIKAPITIYA C, OH C, WHANG I, et al.Molecular characterization, gene expression analysis and biochemical properties of α-amylase from the disk abalone, Haliotis discus discus[J].Comp Biochem Physiol B, 2009, 152(3):271-281. doi: 10.1016/j.cbpb.2008.12.007
[12] SABAPATHY U, TEO L H. Some kinetic properties of amylase from the intestine of the rabbitfish, Siganus canaliculatus (Park)[J].Comp Biochem Physiol B, 1994, 109(1):139-144. doi: 10.1016/0305-0491(94)90150-3
[13] MA P, SIVALOGANATHAN B, REDDY K P, et al.Hormonal influence on amylase gene expression during seabass (Lates calcarifer) larval development[J].Gen Comp Endocrinol, 2004, 138(1):14-19. doi: 10.1016/j.ygcen.2004.04.007
[14] SELLOS D, MOAL J, DEGREMONT L, et al.Structure of amylase genes in populations of Pacific Cupped oyster (Crassostrea gigas):tissue expression and allelic polymorphism[J].Mar Biotechnol (NY), 2004, 5(4):360-372. https://www.researchgate.net/publication/271951447_Analysis_of_the...
[15] PRUDENCE M, MOAL J, BOUDRY P, et al.An amylase gene polymorphism is associated with growth differences in the Pacific cupped oyster Crassostrea gigas[J].Anim Genet, 2006, 37(4):348-351. doi: 10.1111/age.2006.37.issue-4
[16] 辛静静, 刘小林, 李喜莲, 等.凡纳滨对虾α-淀粉酶基因PCR-RFLP多态性与生长性状的相关分析[J].海洋学报, 2011, 33(3):124-130. http://bianke.cnki.net/Web/Article/SEAC201103014.html [17] 李璐. 合浦珠母贝α-淀粉酶基因结构、SNP筛选、生长关联与生态响应研究[D]. 上海: 上海海洋大学, 2013: 54-55. http://cdmd.cnki.com.cn/Article/CDMD-10264-1014013536.htm [18] HIXSON S M, PARRISH C C, XUE X, et al.Growth performance, tissue composition, and gene expression responses in Atlantic salmon (Salmo salar) fed varying levels of different lipid sources[J].Aquaculture, 2017, 467:76-88. doi: 10.1016/j.aquaculture.2016.04.011
[19] AKOLKAR D B, ASADUZZAMAN M, KINOSHITA S, et al.Characterization of Pax3 and Pax7 genes and their expression patterns during different development and growth stages of Japanese pufferfish Takifugu rubripes[J].Gene, 2016, 575(1):21-28. doi: 10.1016/j.gene.2015.08.031
[20] 魏洁书, 杨锦芬.应用荧光定量比较Ct值法测定基因相对表达量[J].中国科技论文, 2013, 6(5):390-395. http://www.docin.com/p-598715002.html [21] HUANG G, GUO Y, LI L, et al.Genomic structure of the α-amylase gene in the pearl oyster Pinctada fucata and its expression in response to salinity and food concentration[J].Gene, 2016, 587(1):98-105. doi: 10.1016/j.gene.2016.04.044
[22] XIE D Z, XU S D, WANG S Q, et al.Cloning, tissue expression, and nutritional regulation of the α-amylase gene in the herbivorous marine teleost Siganus canaliculatus[J].Aquaculture, 2016, 454:229-236. doi: 10.1016/j.aquaculture.2015.12.031
[23] DOUGLAS S E, MANDLA S, GALLANT J W.Molecular analysis of the amylase gene and its expression during development in the winter flounder, Pleuronectes americanus[J].Aquaculture, 2000, 190(3/4):247-260. https://www.researchgate.net/publication/12121899_Evolution_of_the...
[24] 潘俐玲. 大珠母贝和企鹅珍珠贝组织蛋白酶D基因及α-淀粉酶基因的克隆与表达分析[D]. 上海: 上海海洋大学, 2011: 34-51. http://cdmd.cnki.com.cn/Article/CDMD-10264-1011304956.htm [25] 王永刚, 马建忠, 马雪青, 等.马铃薯α-淀粉酶基因的克隆及生物信息学分析[J].食品科学, 2010, 31(19):216-220. http://www.cqvip.com/QK/95574X/201019/35890065.html [26] 陈亮, 梁旭方, 王琳, 等.鳜鱼胰蛋白酶和淀粉酶与胃蛋白酶原基因的克隆与序列分析[J].中国生物化学与分子生物学报, 2009, 25(12):1115-1123. http://www.cqvip.com/QK/92593A/200912/32576836.html [27] NAKAMURA Y, OGAWA M, NISHIDE T, et al.Sequences of cDNAs for human salivary and pancreatic α-amylases[J].Gene, 1984, 28(2):263-270. doi: 10.1016/0378-1119(84)90265-8
[28] HUVET A, FABIOUX C, McCOMBIES H, et al.Natural hybridization between genetically differentiated populations of Crassostrea gigas and C.angulata highlighted by sequence variation in flanking regions of a microsatellite locus[J].Mar Ecol Prog Ser, 2004, 272:141-152. doi: 10.3354/meps272141
[29] 唐小红. 草鱼3种丙酮酸激酶和α-淀粉酶基因的结构、表达分析和生长相关标记筛选[D]. 上海: 上海海洋大学, 2015: 37-38. http://cdmd.cnki.com.cn/Article/CDMD-10264-1015974960.htm [30] 陈春娜. 胭脂鱼α-淀粉酶的cDNA克隆与组织表达研究[D]. 重庆: 西南大学, 2007: 31-32. [31] 黄瑾, 熊邦喜, 陈洁, 等.鱼类消化酶活性与体长、体重和水质的相关性研究[J].水生态学杂志, 2012, 33(2):121-126. http://www.wenkuxiazai.com/doc/e190f02aaf45b307e9719701-2.html [32] 赵艳民, 赵云龙, 王群, 等.日本沼虾胚胎发育时期消化酶活力[J].华东师范大学学报(自然科学版), 2006(2):75-80. http://www.wenkuxiazai.com/doc/177ca1f45ef7ba0d4b733b0f.html -
期刊类型引用(1)
1. 姜建萍,杨学明,袁翔,邱庆庆,杨秀荣,蒋钦杨,黄光华,蒋和生. 罗氏沼虾淀粉酶基因克隆及其表达规律分析. 南方农业学报. 2021(05): 1362-1369 .  百度学术
百度学术
其他类型引用(0)




 下载:
下载:





 粤公网安备 44010502001741号
粤公网安备 44010502001741号
