Analysis of genetic diversity of nine families of Pinctada fucata
-
摘要:
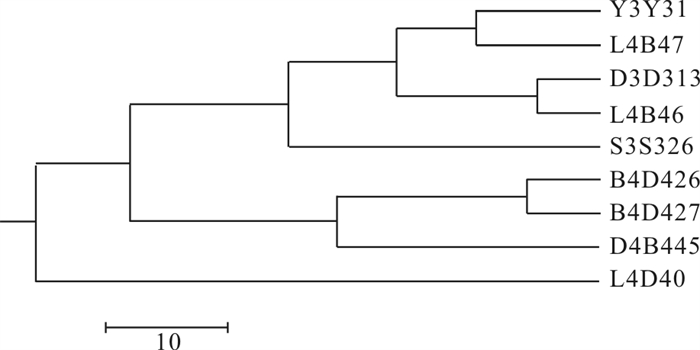
该研究选取具有多态性的6对微卫星引物对构建的2批合浦珠母贝(Pinctada fucata)完全双列杂交家系的遗传多样性进行了分析。6个微卫星标记在9个家系360个个体中共检测到32个等位基因,有效等位基因(Ne)为1.758 7~3.586 5,观测杂合度(Ho)为0.144 4~0.488 9,期望杂合度(He)为0.432 0~0.722 2,Shannon指数(I)为0.691 9~1.507 4。9个家系都有单态位点,平均Ho为0.129 2~0.466 7,平均He为0.155 0~0.439 6,平均I为0.248 5~0.712 2。有19个位点(占35.19%)极显著地偏离Hardy-Weinberg平衡。各家系之间的遗传距离为0.109 0~1.137 2,遗传相似性系数为0.320 7~0.896 8。家系L4B46与L4B48的遗传距离最大,与D3D313的遗传距离最小。UPGMA法聚类分析显示,9个家系分为3支,L4B48单独成一支,B4D426、B4D427与D4B445聚成一支,其余家系聚成一支。该研究结果为合浦珠母贝家系选择育种的亲本选择与交配设计提供了科学依据。
Abstract:We studied the genetic diversity of breeding family of Pinctada fucata. Six microsatellite markers were chosen for genetic analysis of 360 individuals of nine families. A total of 32 alleles were detected and the number of allele at each locus was 3~8. The effective allele was 1.758 7~3.586 5. The observed heterozygosity (Ho) was 0.144 4~0.488 9. The expected heterozygosity (He) was 0.432 0~0.722 2 and the Shannon index was 0.691 9~1.507 4. Monomorphism loci were found in each family. The average Ho (0.129 2~0.466 7), average He (0.155 0~0.439 6) and Shannon index (0.248 5~0.712 2) were detected in family population. Nineteen loci (35.19%) were deviated from Hardy-Weinberg equilibrium test significantly. The genetic distance was 0.109 0~1.137 2 and the genetic identify index was 0.320 7~0.896 8. L4B46 was the furthest with L4B48 and the nearest with D3D313 in terms of genetic distance. Nei′s genetic distance was used to construct the dendrogram by using the unweighted pair group method with arithmetic (UPGMA). Nine families were divided into three clusters: family L4B48 clustered alone and layed outside, while three families (B4D426, B4D427 and D4B445) clustered together, and the other families clustered into one clade. The study provides references for construction of next generation of nuclear populations in P. fucata selective breeding.
-
Keywords:
- Pinctada fucata /
- family selection /
- microsatellite /
- genetic diversity /
- mating design
-
鳞片是由鱼类皮肤原始干细胞定向分化产生[1],是鱼体与外界接触的边缘组织。其基本功能除减少游泳产生的摩擦力、维持体型骨架之外,还起到保护鱼体免遭病原损害的作用。同时,鳞片的形态结构也是研究鱼类分类学特征、资源分布、栖息环境以及生长规律的重要依据[2]。根据鳞片的外形、构造和发生特点,可将鳞片划分为3种基本类型:盾鳞、硬鳞和骨鳞。骨鳞是最常见的一种鳞片,为真骨鱼类所有,呈覆瓦状排列。根据鳞片露出部的表面边缘的构造不同可将骨鳞分为两类:圆鳞和栉鳞。
条石鲷(Oplegnathus fasciatus)隶属于辐鳍鱼纲、鲈形目、鲈亚目、石鲷科、石鲷属,自然分布范围在东海、黄海、台湾海峡和太平洋[3]。俗称日本鹦鹉鱼,栖息于温热带近海碎石、岩礁或珊瑚礁底质的底层水域。条石鲷形态优美,色泽艳丽,肉质细嫩,营养价值高,是一种极具养殖前景和观赏价值的海产名贵鱼类[4]。近年来对条石鲷的早期发育已有许多研究[5-9],但关于鳞片的形成在中国未见有报道,同时由于南北方水域环境的差异,鳞片的发育在时间和速度上也可能会存在一些差异。该文采用茜素红染色法初步分析了条石鲷鳞片最初出现的时间和位置,以及鳞片形成的时间及各个发育阶段的特点,旨在丰富条石鲷早期发育的生物学文库,同时通过对鳞片发育过程的观察,判断鱼类种苗的培育效果,及时调整种苗生产管理措施。
1. 材料与方法
1.1 材料
观察样本为采自广东省饶平县中国水产科学研究院南海水产研究所海水鱼类试验基地人工孵化和培育的0~40日龄的条石鲷仔稚鱼。
1.2 方法
将人工催产的受精卵置于容积为0.5 m3的玻璃纤维孵化桶内进行孵化,连续充气,仔鱼孵出后在面积为1/15~2/15 hm2,水深为1.5~2 m的池塘内进行人工育苗,育苗期间水温为24~27 ℃(其中前期培育水温为22~23 ℃,后期为24~27 ℃),盐度29~30,pH 7.4~8.2。
实验方法采用茜素红染色法[10]。从2008年5月2日到6月11日育苗期间每天用手抄网随机采集10~20尾样本,用5%甲醛溶液保存,将要观察鳞片的鱼苗样本放入清水中浸泡3 h,然后放入2%茜素红溶液中染色,染色后的样本用滤纸吸干体表水分,在体视显微镜下观察鳞片出现的部位及其分布情况、鳞片上的鳞嵴数、鳞焦的中心以及水平鳞列数。用解剖针小心将鱼体的鳞片掀起,用解剖刀轻轻刮下,在显微镜下观察、拍照。
1.3 实验数据处理
采用SPSS 18.0软件,对鳞嵴数与发育阶段的相关关系分别用直线方程、幂函数方程以及二次方程进行拟合,对相关系数做显著性检验,依最小剩余平方和来确定最适回归方程。
2. 结果
2.1 早期鳞被的形成
2.1.1 条石鲷鳞片起始中心及鳞被形成
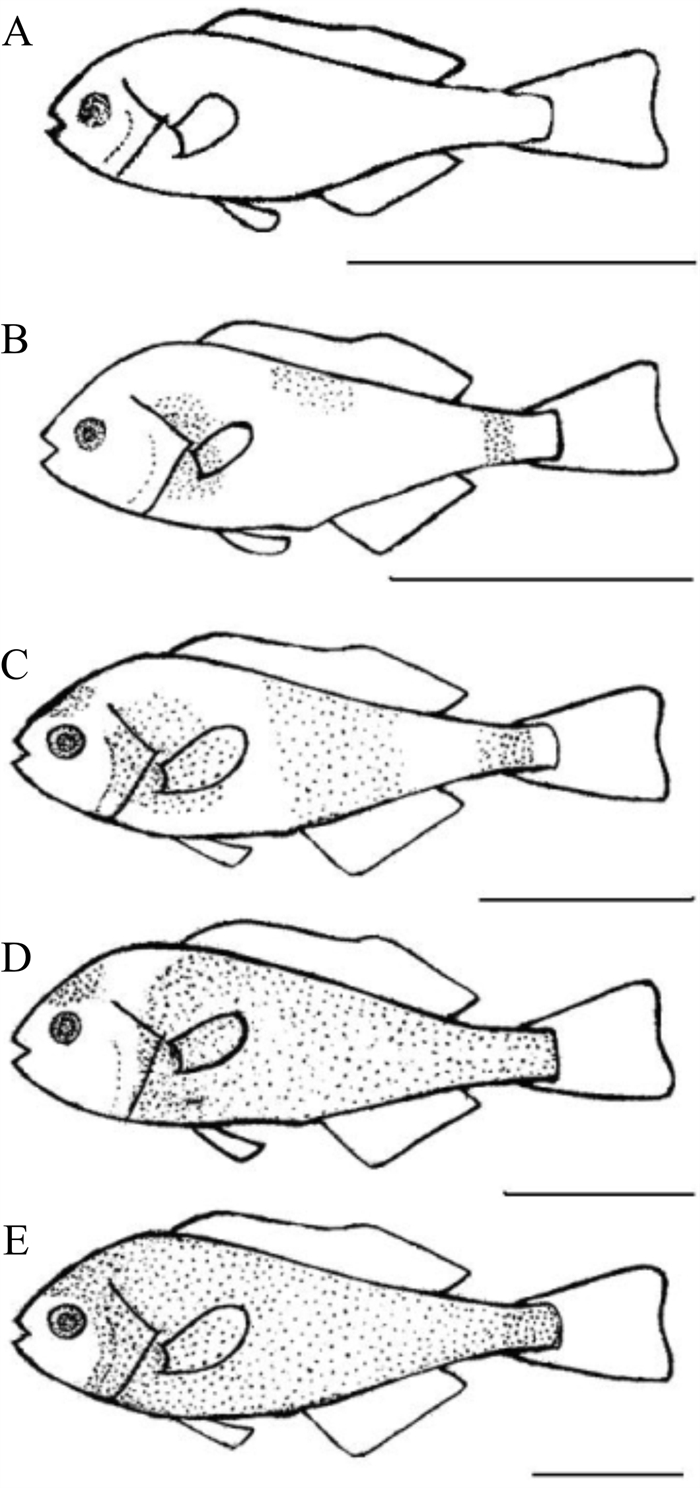
条石鲷鳞片最初出现的部位在鳃盖后缘至胸鳍基部、胸鳍后缘的侧线上方和尾柄处,共有3个鳞被起始中心。鳃盖后缘至胸鳍基部的鳞被向上下前后扩展,胸鳍后缘的侧线上方鳞被向下和向前后扩展,尾柄处的鳞被前后扩展,头顶部的鳞被向四周扩展,最后连成一片,鳞被覆盖全身。根据条石鲷鳞被在鱼体体表的形成过程将其划分为5个阶段(图 1)。
阶段A:鳞片未出现,全身无鳞。
阶段B:鳃盖后缘至胸鳍基部、胸鳍后缘的侧线上方和尾柄处开始出现鳞片,水平鳞列数1~2。
阶段C:鳞被向各方扩展,在侧线的上下方均出现鳞被,尾柄处的鳞被发育较快,水平鳞列数5~11。头部开始出现鳞片。
阶段D:鳞被向全身扩展,鳃盖后缘至胸鳍基部、胸鳍后缘的侧线上方和尾柄处的鳞被连成一片,并覆盖大部分鱼体。鱼体腹部出现鳞被,鳞被发育较均匀,尾柄全部覆盖鳞被,鳃盖上也出现鳞片,鳞被发育较晚。
阶段E:体表鳞被与头部鳞被连合,鳞被发育完全,鱼体全身具鳞。
2.1.2 鳞被形成特点
条石鲷鳞被向下方扩展的速度快于上方,上下扩展速度快于前后扩展,向前扩展到鳃盖后暂停,向后发育到达尾柄末端,最后出现鳞片的部位是鳃盖。同一个部位鳞被的发育速度并不完全相同。
2.1.3 条石鲷鳞被形成的体长和日龄
条石鲷仔鱼最初出现鳞片的最小个体为全长19 mm、体长15 mm,最大个体为全长26 mm、体长20 mm,为孵化后18~20日龄(表 1);鳞被发育完全时的最小个体为全长36 mm、体长29.9 mm,最大个体为全长66 mm、体长51 mm,为孵化后35~40日龄。最初出现鳞片和鳞被发育完全时个体的大小差异最大为全长47 mm、体长36 mm,22日龄。
表 1 条石鲷鳞被发育阶段与个体生长的关系(n=220)Table 1 Relationship between early developmental stage of squamation and body length and total length of O. fasciatus鳞被发育阶段
developmental stage全长/mm total length 体长/mm body length 日龄
day after hatching最大maximum 最小minimum 最大maximum 最小minimum 阶段A Stage A 22 15 18 11 15~20 阶段B Stage B 26 19 20 15 18~20 阶段C Stage C 39 26 31 20 25~32 阶段D Stage D 35 32 27 25 32~35 阶段E Stage E 66 36 51 29.9 35~40 2.2 鳞片形态结构和初生鳞的发育
2.2.1 鳞片的形态结构
条石鲷鳞片为细栉鳞,长方形,前区较平截,后区边缘和鳞嵴具细栉齿(图 2);具放射状辐射沟2~6条,向基区(前区)辐射,起始处不一致,有的始于鳞焦,有的始于中途,成鱼辐射沟界限不太清晰;鳞焦偏于后区。
2.2.2 初生鳞的发育
条石鲷初生鳞为圆形或椭圆形,表面光滑、透明,鳞嵴(环片)、栉齿、鳞焦和辐射沟等均无。鳞片在发育过程中,其外形、鳞嵴、栉齿、鳞焦和辐射沟均随个体大小和日龄而发生变化。条石鲷初生鳞的发育特征与鳞被形成阶段的关系为:
阶段A:鳞片未出现。
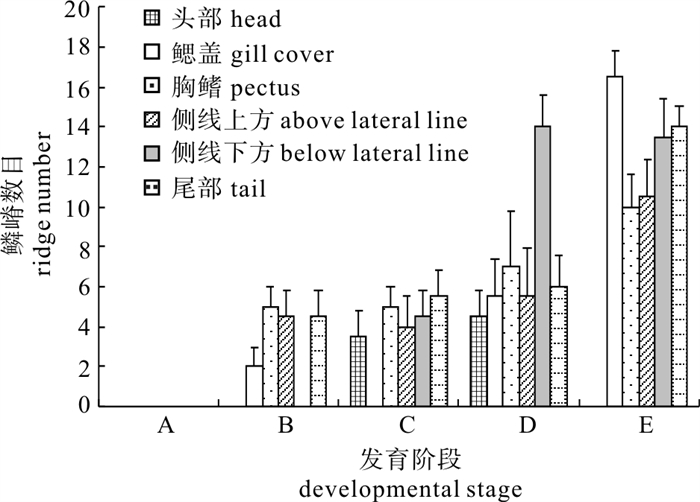
阶段B:鳃盖后缘初生鳞的形态为较原始的圆鳞,鳞嵴为1~3,胸鳍基部鳞嵴为4~6;侧线上方鳞片的鳞嵴为3~6,鳞片极薄易碎;尾部鳞片鳞嵴为3~6(图 3-B-a,b,c)。
![]() 图 3 条石鲷各个鳞片形成阶段的鳞片B-a. 侧线上鳞片;B-b. 尾柄鳞片;B-c. 鳃盖后缘鳞片;C-a. 侧线上鳞片;C-b. 胸鳍基鳞片;C-c. 头部鳞片;C-d. 鳃盖鳞片;C-e. 尾柄鳞片;C-f. 腹部鳞片;D-a. 侧线上鳞片;D-b. 侧线下鳞片;D-c. 胸鳍基鳞片;D-d. 头部鳞片;D-e. 鳃盖鳞片;D-f. 尾柄鳞片;E-a. 侧线上鳞片;E-b. 鳃盖鳞片;E-c. 尾柄鳞片;E-d. 胸鳍基鳞片;标尺=0.25 mmFig. 3 Scales at different developmental stages in O. fasciatusB-a. scale up lateral line; B-b. scale of caudal peduncle; B-c. scale of posterior margin of gill cover; C-a. scale up lateral line; C-b. scale of pectoral fin base; C-c. scale of head; C-d. scale of gill cover; C-e. scale of caudal peduncle; C-f. scale of ventral; D-a. scale up lateral line; D-b. scale down ateral line; D-c. scale of pectoral fin base; D-d. scale of head; D-e. scale of gill cover; D-f. scale of caudal peduncle; E-a. scale up the lateral line; E-b. scale of gill cover; E-c. scale of caudal peduncle; E-d. scale of pectoral fin base; scale=0.25 mm
图 3 条石鲷各个鳞片形成阶段的鳞片B-a. 侧线上鳞片;B-b. 尾柄鳞片;B-c. 鳃盖后缘鳞片;C-a. 侧线上鳞片;C-b. 胸鳍基鳞片;C-c. 头部鳞片;C-d. 鳃盖鳞片;C-e. 尾柄鳞片;C-f. 腹部鳞片;D-a. 侧线上鳞片;D-b. 侧线下鳞片;D-c. 胸鳍基鳞片;D-d. 头部鳞片;D-e. 鳃盖鳞片;D-f. 尾柄鳞片;E-a. 侧线上鳞片;E-b. 鳃盖鳞片;E-c. 尾柄鳞片;E-d. 胸鳍基鳞片;标尺=0.25 mmFig. 3 Scales at different developmental stages in O. fasciatusB-a. scale up lateral line; B-b. scale of caudal peduncle; B-c. scale of posterior margin of gill cover; C-a. scale up lateral line; C-b. scale of pectoral fin base; C-c. scale of head; C-d. scale of gill cover; C-e. scale of caudal peduncle; C-f. scale of ventral; D-a. scale up lateral line; D-b. scale down ateral line; D-c. scale of pectoral fin base; D-d. scale of head; D-e. scale of gill cover; D-f. scale of caudal peduncle; E-a. scale up the lateral line; E-b. scale of gill cover; E-c. scale of caudal peduncle; E-d. scale of pectoral fin base; scale=0.25 mm阶段C:圆鳞逐渐发育成栉鳞,胸鳍基鳞嵴为4~6,侧线上方鳞片的鳞嵴为2~6,侧线下方鳞片的鳞嵴为3~8,尾柄处鳞片的鳞嵴为4~7,头部鳞片鳞嵴为2~5(图 3-C-a,b,c,d,e,f)。
阶段D:侧线上方鳞片鳞嵴为2~9,侧线下方鳞片的鳞嵴为12~16,腹部鳞片鳞嵴为5~9,胸鳍基鳞片鳞嵴为3~11,尾柄部鳞嵴为4~8,头部的鳞片鳞嵴为3~6,鳃盖上的鳞片鳞嵴为3~8(图 3-D-a,b,c,d,e,f)。
阶段E:侧线上方鳞片鳞嵴为8~13,下方为11~16,尾柄处鳞嵴为13~15,胸鳍基鳞片鳞嵴为8~12,鳃盖后缘鳞片鳞嵴为15~18(图 3-E-a,b,c,d)。
条石鲷初生鳞鳞嵴(环片)数目与发育阶段的关系为条石鲷初生鳞在发育过程中,鱼体不同部位鳞嵴(环片)数目随个体大小和日龄而发生变化(图 4)。从总体趋势看,随着发育进程,鱼体不同部位鳞片的鳞嵴数逐渐增加。经统计学拟合,鳞嵴数与发育阶段的相关关系可分别用直线方程y=2.88x-2.96(n=220,R2=0.918 1)、幂函数方程y=6.802 4ln(x)-0.833 3(n=220,R2=0.827 4)以及二次方程y=0.414 3x2 +0.394 3x-0.06(n=220,R2=0.944 6)表示。式中y为鳞嵴数,x为发育阶段。统计结果显示3种方程的适合度均较高,表现出较好的拟合性,因而三者都可以较好地描述条石鲷初生鳞鳞嵴(环片)数目与发育阶段的关系,依最小剩余平方和来选择回归方程,在3种相关方程中,以二次方程的描述更为吻合。
3. 讨论
3.1 鱼类鳞片最先出现部位和起始位置数量
此研究观察显示,条石鲷具有3个鳞片起始中心。徐恭昭等[11]把鱼类的鳞片起始中心分成两大类型:1)鳞片发育只具有1个起始中心,2)具有2个或2个以上起始中心。鲷科的黑鲷(Sparus macroeephlus)[12]、真鲷(Pagrus major)[13],科的黑边石斑鱼(Epinephelus fasciatus)[14],鳗鲡科的奥克兰鳗鲡(Anguilla australis)和大鳗鲡(A.dieffenbachii)[15]等,只具有1个起始中心。具有2个或2个以上起始中心的种类,如鲻科的梭鱼(Mugil soiuy)[11]、溪鳉科的花斑溪鳉(Rivulus marmoratus)[16]、鲱科的大鳞油鲱(Brevooritia patronus)[17]有2个;齿鳉科杂色鳉(Cyprinodon variegatus variegatus)[18]、太阳鱼科的暗斑棘盖太阳鱼(Pomoxis nigromaeulatus)[19]有3个;鳢科的乌鳢(Channa agrus)[20]、底鳉科的沼泽底鳉(Fundulus confluentus)、底鳉(F.heteroclitus)等种类[18]有4个。而鲤科的齐口裂腹鱼(Schizothorax prenanti)[21]和重口裂腹鱼(S.davidi)[22]稚鱼鳞片发生起始位置有6个,以此为中心向四周扩展延伸直到覆盖全鱼体。据SIRE和ARNULF[23]报道,4种硬骨鱼类的鳞片发生起始位置有7个。从现有这些文献来看,不同鱼类物种之间鳞片发生起始点的数目及其覆盖方式存在着差异,是否与物种进化以及栖息环境具有一定的相关性尚待深入探讨。
3.2 鱼类鳞被覆盖过程
此研究显示,条石鲷稚鱼体表开始长出形态较为原始的圆鳞,而后圆鳞逐渐发育成栉鳞,其发生过程与圆鳞鱼类大致类似[24]。在鳞片开始形成的时间以及鳞片发育完全的时间,条石鲷和黑鲷等种类相似,而且鳞片发育的过程也很相近。关于鱼类鳞片最初出现的时间和位置的问题,KUBO和YASHIWARA[25]把鱼体最早出现鳞片的区域分为两大类型:1)在躯体的前部,如鲤(Cyprinus carpio)和鳟(Salmo trutta)等。草鱼(Ctenopharyngodon idella)、鲢(Hypophthalmichthys molitrix)、鳙(Aristrichthys nobilis)以及重口裂腹鱼等种类,其鳞片最早均出现在躯干前部的侧线处,鲤和草鱼的鳞片以1个起始点为中心,等速纵向和横向伸展,最后覆盖腹部中线;鳙和鲢鳞片向腹部和尾柄部扩展的速度快于向背部方向,当腹部与尾柄部基本覆盖时鳞片才从侧线上方向背部和由尾柄上部向前伸展,头后背部最后覆盖[26]。2)在鱼体后部尾柄末端的侧线上,如白刺盖太阳鱼(P. annuaris)及淡水鼓鱼(Aptodiotus grunnieus)等种类。根据PENAZ[27]、ABLE和LAMOMACA[28]的报道,许多淡水鱼类和鲆鲽鱼类的鳞片发育起始于尾柄。软骨鱼类澳大利亚虎鲨(Heterodontus portusjacksoni)和眶嵴虎鲨(H. galeatus) 的鳞片发育起始于尾鳍[29];斯氏罗非鱼(Tilapia sparrmani)的鳞片最先出现在尾柄中部,沿侧线处向前覆盖的速度快于向后,并以等速向腹部和背部方向扩展,最后覆盖头后背部[25]。从鳞片形成的整个过程来看,条石鲷早期鳞片的形成在整个鱼群中呈不同步性,鳞片出现的最初时间,鳞片发育的速度,鳞片发育完全的时间,在鱼体大小以及日龄方面均有很大变化。同样是20日龄的仔鱼有的没有一片鳞,有的已开始形成鳞片。所以笔者认为,决定鱼类鳞片发育的因素是多方面的,它并不仅受到水温或是饵料的影响,还可能受到其他一些因素的影响。这有待于以后进一步的研究。
-
表 1 微卫星引物序列及扩增情况
Table 1 Sequence and amplification of microsatellite loci
位点
locus引物序列(5′→3′)
primer sequence重复序列
repeated sequence退火温度/℃
annealing temperature文献
ReferencePf02 S:GCTTGCCAGTGACTTCCTC
A:GGCCTGAGCTATCACAAGTC(AT)8 55 [17] Pf08 S:AACCCTGGTGGACACTCTT
A:GTAGCTTCGGCTTCTTGC(TAA)5 53 [17] pf11 S:TGCTGTACCCAAAGGGCTTATT
A:TGGCGATTTTCTTGTATGTGAC(AT)6 53 [17] scd1.1-12 S:TCAGGCAATATCCATATCCACC
A:TGGAGTTCAGTCCCCTTCTTGT(AT)9 55 [13] scd2.1-10 S:GTATCTGAAAGAAACGATCTG
A:CACTAGAATAAATGTGGAAGC(GAT)14 49 [13] scd3.1-3 S:TCTGTACCTCCACATGACTG
A:CAGCCATTTGGAGACATAGA(ATGA)8 53 [13] 表 2 6个微卫星位点在9个合浦珠母贝家系的多样性指数
Table 2 Genetic diversity index analysis of six microsatellite loci in nine P. fucata families
位点
locus等位基因数
Na有效等位基因数
Ne观测杂合度
Ho期望杂合度
HeShannon指数
Ipf02 3 1.902 1 0.144 4 0.474 9 0.691 9 pf08 4 2.279 8 0.488 9 0.562 1 0.920 7 pf11 6 1.758 7 0.258 3 0.432 0 0.875 1 scd1.1-12 5 3.586 5 0.155 6 0.722 2 1.368 6 scd2.1-10 6 3.036 7 0.366 7 0.671 6 1.338 7 scd3.1-3 8 3.410 2 0.211 1 0.707 7 1.507 4 平均mean 5.333 3 2.662 3 0.270 8 0.595 1 1.117 1 表 3 9个合浦珠母贝家系的遗传多样性分析
Table 3 Genetic diversity of nine families of P. fucata
家系
family参数
parameter位点locus 平均值
meanpf02 pf08 pf11 scd1.1-12 scd2.1-10 scd3.1-3 Y3Y31 Na 1 2 1 3 1 1 1.500 0 Ne 1 1.940 6 1 2.228 4 1 1 1.361 5 I 0 0.677 8 0 0.890 9 0 0 0.261 4 Ho 0 0.575 0 0 0.200 0 0 0 0.129 2 He 0 0.490 8 0 0.558 2 0 0 0.174 8 P 单态 0.271 6 单态 0** 单态 单态 D3D313 Na 1 2 3 3 2 3 2.333 3 Ne 1 1.600 0 1.353 6 1.524 5 1.956 0 1.894 6 1.554 8 I 0 0.562 3 0.490 3 0.594 2 0.681 9 0.817 0 0.524 3 Ho 0 0.500 0 0.250 0 0.125 0 0 0.250 0 0.150 0 He 0 0.379 7 0.264 6 0.348 4 0.494 9 0.478 2 0.327 6 P 单态 0.040 7 0** 0** 0** 0** S3S326 Na 2 2 1 1 3 1 1.666 7 Ne 1.249 5 1.249 5 1 1 2.079 3 1 1.263 0 I 0.351 7 1.351 7 0 0 0.787 6 0 0.248 5 Ho 0.225 0 0.225 0 0 0 0.475 0 0 0.154 2 He 0.202 2 0.202 2 0 0 0.525 6 0 0.155 0 P 0.452 3 0.452 3 单态 单态 0** 单态 L4B46 Na 1 3 3 3 3 3 2.666 7 Ne 1 1.615 3 1.615 3 2.034 3 2.067 2 1.838 0 1.695 0 I 0 0.670 4 0.670 4 0.816 6 0.784 7 0.758 5 0.616 8 Ho 0 0.375 0 0.375 0 0.225 0 0.500 0 0.375 0 0.308 3 He 0 0.385 8 0.385 8 0.514 9 0.522 8 0.461 7 0.373 8 P 单态 0** 0** 0** 0** 0** L4B47 Na 1 2 2 2 1 2 1.666 7 Ne 1 1.567 9 1.724 1 1.834 9 1 1.198 0 1.517 8 I 0 0.548 2 0.610 9 0.647 4 0 0.688 1 0.415 8 Ho 0 0.475 0 0.600 0 0.300 0 0 0.050 0 0.237 5 He 0 0.366 8 0.425 3 0.460 8 0 0.501 3 0.292 4 P 单态 0.056 3 0.008 4 0.025 0 单态 0** L4B48 Na 1 2 1 2 2 3 1.833 3 Ne 1 1.342 3 1 1.663 2 1.503 1 1.830 7 1.389 9 I 0 0.422 7 0 0.588 2 0.517 3 0.718 1 0.374 5 Ho 0 0.300 0 0 0.550 0 0.325 0 0.100 0 0.212 5 He 0 0.258 2 0 0.403 8 0.338 9 0.459 5 0.243 4 P 单态 0.287 9 单态 0.019 6 0.789 8 0** B4D426 Na 3 4 3 1 3 3 2.833 3 Ne 1.698 5 2.318 8 1.891 3 1 2.705 0 2.095 6 1.951 5 I 0.671 9 1.025 6 0.737 5 0 1.046 7 0.791 4 0.712 2 Ho 0.500 0 0.600 0 0.350 0 0 0.800 0 0.475 0 0.454 2 He 0.416 5 0.575 9 0.477 2 0 0.638 3 0.529 4 0.439 6 P 0** 0** 0** 单态 0.039 2 0** B4D427 Na 2 3 2 1 3 2 2.166 7 Ne 1.470 6 2.320 5 1.470 6 1 2.474 9 1.995 0 1.788 6 I 0.500 4 0.962 0 0.500 4 0 0.999 4 0.691 9 0.609 0 Ho 0.400 0 0.675 0 0.400 0 0 0.775 0 0.550 0 0.466 7 He 0.324 1 0.576 3 0.324 1 0 0.603 5 0.505 1 0.388 8 P 0.127 6 0.144 3 0.127 6 单态 0.085 0 0.568 7 D4B445 Na 3 3 2 1 3 2 2.333 3 Ne 1.732 5 2.061 9 1.406 0 1 2.034 3 1.903 6 1.689 7 I 0.684 8 0.886 5 0.463 7 0 0.816 6 0.667 6 0.586 5 Ho 0.175 0 0.675 0 0.350 0 0 0.425 0 0.325 0 0.325 0 He 0.428 2 0.521 5 0.292 4 0 0.514 9 0.490 7 0.372 9 P 0** 0.066 9 0.198 3 单态 0** 0.037 8 注:Na. 等位基因数;Ne. 有效等位基因数;Ho. 观测杂合度;He. 期望杂合度;I. Shannon多样性指数;P. Hardy-Weinberg平衡的卡方检验;*. P < 0.05/6=0.008 3;* *. P < 0.01/6=0.001 7
Note:Na. allele number;Ne. effective allele;Ho. observed heterozygosity;He. expected heterozygosity;I. Shannon diversity index;P. Hardy-Weinberg equilibrium test表 4 9个家系间的遗传相似系数(上三角)和遗传距离(下三角)
Table 4 Genetic identity (above diagonal) and genetic distances (below diagonal) of nine families of P. fucata
Pop ID Y3Y31 D3D313 S3S326 L4B46 L4B47 L4B48 B4D426 B4D427 D4B445 Y3Y31 0.697 7 0.568 9 0.725 2 0.811 4 0.363 3 0.325 2 0.387 5 0.444 2 D3D313 0.360 0 0.641 0 0.896 8 0.669 5 0.345 7 0.383 5 0.493 4 0.594 1 S3S326 0.564 1 0.444 7 0.542 8 0.638 1 0.431 2 0.411 9 0.506 1 0.456 3 L4B46 0.321 4 0.109 0 0.611 0 0.753 8 0.320 7 0.437 4 0.552 9 0.619 4 L4B47 0.209 0 0.401 3 0.449 3 0.282 7 0.463 1 0.391 9 0.447 8 0.514 7 L4B48 1.012 5 1.062 2 0.841 3 1.137 2 0.769 8 0.338 5 0.464 3 0.443 5 B4D426 1.123 4 0.958 5 0.887 0 0.826 9 0.936 8 1.083 3 0.880 3 0.685 7 B4D427 0.948 0 0.706 3 0.681 1 0.592 5 0.803 3 0.767 2 0.127 5 0.608 1 D4B445 0.811 4 0.520 7 0.784 6 0.479 0 0.664 2 0.813 0 0.377 4 0.497 4 -
[1] 蒙钊美, 李有宁, 邢孔武, 等. 珍珠养殖理论与技术[M]. 北京: 科学出版社, 1996: 10-35. [2] HWANG J J, YAMAKAWA T, AOKI I. Growth of wild pearl oysters Pinctada fucata, Pinctada margaritifera and Pinctada sugillata (Bivalvia: Pteriidae) in Taiwan[J]. Fish Sci, 2007, 73(1): 132-141. doi: 10.1111/j.1444-2906.2007.01311.x
[3] 闫玉科. 广东南珠产业可持续发展路径研究[J]. 农业经济问题, 2011, 32(3): 97-102. https://www.cnki.com.cn/Article/CJFDTotal-NJWT201103018.htm [4] 喻达辉, 黄桂菊, 张立楠. 关于振兴南珠的思考[J]. 中国宝石, 2010(4): 240-241. [5] 王清印, 等. 水产生物育种理论与实践[M]. 北京: 科学出版社, 2013: 22-23. [6] REMEDIOS B B, GARY F N. Response to within family selection for body weight in Nile tilapia using a single-trait animal model[J]. Aquaculture, 2002, 204(3): 371-381. https://www.sciencedirect.com/science/article/abs/pii/S0044848601008249
[7] 马爱军, 郭建丽, 王新安, 等. 大菱鲆选育家系抗鳗弧菌性能[J]. 中国水产科学, 2014, 21(3): 484-493. [8] 梁健, 闫喜武, 霍忠明, 等. 不同近交系数蛤仔橙色品系的生长和成活研究[J]. 南方水产科学, 2013, 9(4): 87-93. doi: 10.3969/j.issn.2095-0780.2013.04.015 [9] 王好锋, 刘萍, 高保全, 等. 近交对三疣梭子蟹若干经济性状衰退的影响[J]. 中国水产科学, 2013, 20(6): 1157-1165. doi: 10.3724/SP.J.1118.2013.01157 [10] 刘伟, 苏胜彦, 董在杰, 等. 3个鲤群体的微卫星标记与生长性状相关性分析[J]. 南方水产科学, 2012, 8(3): 17-24. doi: 10.3969/j.issn.2095-0780.2012.03.003 [11] 罗明坤, 郭金涛, 赵金良, 等. 尼罗罗非鱼(♀)×萨罗罗非鱼(♂)F1家系亲权关系微卫星分析[J]. 南方水产科学, 2014, 10(1): 9-15. doi: 10.3969/j.issn.2095-0780.2014.01.002 [12] KUMAR R, TANYA C. Molecular markers and their applications in fisheries and aquaculture[J]. Adv Biosci Biotechnol, 2010, 1(4): 281-291. doi: 10.4236/abb.2010.14037
[13] TAKEUCHI T, KAWASHIMA T, KOYANAGI R, et al. Draft genome of the pearl oyster Pinctada fucata: a platform for understanding bivalve biology[J]. DNA Res, 2012, 19(2): 117-130. doi: 10.1093/dnares/dss005
[14] HUANG X D, ZHAO M, LIU W G, et al. Gigabase-scale transcriptome analysis on four species of pearl oysters[J]. Mar Biotechnol, 2013, 15(3): 253-264. doi: 10.1007/s10126-012-9484-x
[15] ZHAO X, WANG Q, JIAO Y, et al. Identification of genes potentially related to biomineralization and immunity by transcriptome analysis of pearl sac in pearl oyster Pinctada martensii[J]. Mar Biotechnol, 2012, 14(6): 730-739. doi: 10.1007/s10126-012-9438-3
[16] 许成帅, 范嗣刚, 黄桂菊, 等. 合浦珠母贝家系遗传多样性与性状相关性[J]. 广东农业科学, 2013, 40(12): 156-158. doi: 10.3969/j.issn.1004-874X.2013.12.048 [17] FAN S, WANG J, HUANG G, et al. Identification of twenty novel polymorphic microsatellite DNA markers from transcripts of the pearl oyster Pinctada fucata using next-generation sequencing approach[J]. J Gene, 2014, 93: e82-e85. doi: 10.1007/s12041-012-0171-5
[18] YEH F C, YANG R C, BOYLE T, et al. POPGENE, version 1.32: the user friendly software for population genetic analysis[Z]. Edmonton, Canada: Molecular Biology and Biotechnology Centre, University of Alberta, 1999.
[19] TAMURA K, DUDLEY J, NEI M, et al. MEGA4: molecular evolutionary genetics analysis (MEGA) software version 4.0[J]. Mol Biol Evol, 2007, 24(8): 1596-1599. doi: 10.1093/molbev/msm092
[20] 曲妮妮, 龚世园, 黄桂菊, 等. 合浦珠母贝3个养殖群体遗传多样性的微卫星分析[J]. 水生态学杂志, 2009, 2(4): 89-93. [21] YU D H, CHU K H. Genetic variation in wild and cultured populations of the pearl oyster Pinctada fucata from southern China[J]. Aquaculture, 2006, 258(1/2/3/4): 220-227. https://www.sciencedirect.com/science/article/pii/S0044848606002213
[22] 汤健, 管云雁, 刘文广, 等. 马氏珠母贝家系遗传结构的微卫星分析[J]. 海洋科学, 2013, 37(8): 35-41. https://www.cnki.com.cn/Article/CJFDTotal-HYKX201308007.htm [23] 赵晓霞, 邓岳文, 杜晓东, 等. 利用微卫星标记分析马氏珠母贝4个养殖群体遗传结构[J]. 基因组学与应用生物学, 2010, 29(5): 879-884. https://www.cnki.com.cn/Article/CJFDTotal-GXNB201005010.htm [24] 闫学春, 佟广香, 匡友谊, 等. 两个马氏珠母贝养殖群体遗传多样性微卫星分析[J]. 水产学杂志, 2009, 22(1): 5-9. doi: 10.3969/j.issn.1005-3832.2009.01.002 [25] LI Q, WANG Q, QI M, et al. Development, characterization, and inheritance of 113 novel EST-SSR markers in the Pacific oyster (Crassostrea gigas)[J]. Genes Genom, 2011, 33(3): 313-316. doi: 10.1007/s13258-011-0021-5
[26] HERBINGER C M, SMITH C A, LANGY S. Development and characterization of novel tetra- and dinucleotide microsatellite markers for the French Polynesia black-lipped pearl oyster, Pinctada margaritifera[J]. Mol Ecol Notes, 2006, 6(1): 107-109. doi: 10.1111/j.1471-8286.2005.01156.x
[27] ANDREA A E, NATALIA V A, RODOLFO F, et al. Development of novel polymorphic EST-SSR markers in Californian abalone Haliotis rufescens and genetic analysis in wild and hatchery-bred populations[J]. Aquac Res, 2014, 45: 1942-1952. doi: 10.1111/are.12141
[28] WANG Y, GUO X. Development and characterization of EST-SSR markers in the eastern oyster Crassostrea virginica[J]. Mar Biotechnol, 2007, 9(4): 500-511. doi: 10.1007/s10126-007-9011-7
[29] WANG Y, WANG A, GUO X. Development and characterization of 30 polymorphic microsatellite markers for the Atlantic surfclam, Spisula solidissima (Dillwyn, 1817)[J]. Mol Ecol Res, 2009, 9(4): 1264-1267. doi: 10.1111/j.1755-0998.2009.02660.x
[30] WANG Y, WANG A, GUO X. Development and characterization of polymorphic microsatellite markers for the northern quahog Mercenaria mercenaria (Linnaeus, 1758)[J]. J Shellfish Res, 2010, 29(1): 77-82. doi: 10.2983/035.029.0130
[31] WATTIER R, ENGEL C R, SAUMITOU-LAPRADE P, et al. Short allele dominance as a source of heterozygote deficiency at microsatellite loci: experimental evidence at the dinucleotide locus Gv1CT in Gracilaria gracilis (Rhodophyta)[J]. Mol Ecol, 1998, 7(11): 1569-1573. doi: 10.1046/j.1365-294x.1998.00477.x
[32] PEMBERTON J M, SLATE J, BANCROFT D R, et al. Non amplifying alleles at microsatellite loci: a caution for parentage and population studies[J]. Mol Ecol, 1995, 4(2): 249-252. doi: 10.1111/j.1365-294X.1995.tb00214.x
[33] SMITH O S, SMITH J S C, BOWEN S L, et al. Similarities among a group of elite maize inbreeds as measured by pedigree, F1 grain yield, grain yield heterosis and RFLPs[J]. Theor Appl Genet, 1990, 80(6): 833-840. doi: 10.1007/BF00224201
[34] 张一, 储德裕, 金国庆, 等. 马尾松亲本遗传距离与子代生长性状相关性分析[J]. 林业科学研究, 2010, 23(2): 215-220. [35] ZHANG T, NI X L, JIANG K F, et al. Relationship between heterosis and parental genetic distance based on molecular markers for functional genes related to yield traits in rice[J]. Rice Sci, 2010, 17(4): 288-295. doi: 10.1016/S1672-6308(09)60029-9
[36] 鲁翠云, 曹顶臣, 孙效文, 等. 微卫星分子标记辅助镜鲤家系构建[J]. 中国水产科学, 2008, 15(6): 893-901. doi: 10.3321/j.issn:1005-8737.2008.06.001 [37] 毕金贞, 陈松林. 牙鲆亲本间遗传距离与其后代生长速度的相关性分析[J]. 中国农学通报, 2010, 26(15): 395-401. https://www.cnki.com.cn/Article/CJFDTotal-ZNTB201015084.htm [38] 许成帅. 合浦珠母贝选育群体与家系的遗传多样性及其与生长表现的相关性分析[D]. 上海: 上海海洋大学, 2013: 54-57. -
期刊类型引用(9)
1. 邹杰,彭慧婧,郑德斌,张守都. 广西合浦珠母贝3个不同群体遗传关系分析. 水产科学. 2023(03): 466-473 .  百度学术
百度学术
2. 苏晓盈,代永仙,谭杰,张元,刘付少梅,刘志刚. 马氏珠母贝(Pinctada martensii)黑壳色养殖群体SSR遗传多样性分析. 基因组学与应用生物学. 2021(02): 615-621 .  百度学术
百度学术
3. 苏钰玲,李敏,杨永春,刘佳豪,李振海,阮惠婷,戴嘉格,刘丽,邹柯姝. 南方拟微卫星标记筛选及遗传多样性分析. 水产科学. 2020(02): 224-233 .  百度学术
百度学术
4. 魏海军,邓正华,陈明强,邓林琪,谭春明,李有宁,王雨,王爱民. 不同壳色合浦珠母贝生产性能评估及其雌雄群体间的差异. 南方农业学报. 2020(02): 412-420 .  百度学术
百度学术
5. 陈健,白丽蓉,罗会,喻达辉. 2种贝龄合浦珠母贝数量性状的相关与通径分析. 南方农业学报. 2020(10): 2557-2564 .  百度学术
百度学术
6. 朱华平,苏换换,马冬梅,黄樟翰. 华南鲤选育品种与地方品种的遗传多样性比较分析. 农业生物技术学报. 2018(08): 1371-1381 .  百度学术
百度学术
7. 张磊,王锦旭,杨贤庆,魏涯,岑剑伟. 响应面法优化合浦珠母贝糖胺聚糖咀嚼片的制备工艺. 食品工业科技. 2018(17): 159-166 .  百度学术
百度学术
8. 马冬梅,苏换换,朱华平,黄樟翰. 华南鲤选育群体不同世代遗传多样性与遗传结构的微卫星分析. 水生生物学报. 2018(05): 887-895 .  百度学术
百度学术
9. 张磊,王锦旭,杨贤庆,魏涯,杨少玲. 合浦珠母贝糖胺聚糖泡腾片制备工艺优化及其质量分析. 大连海洋大学学报. 2018(05): 644-650 .  百度学术
百度学术
其他类型引用(0)



 下载:
下载:


 粤公网安备 44010502001741号
粤公网安备 44010502001741号
