Genetic structure of Priacanthus macracanthus population from the South China Sea
-
摘要:
在南海北部陆架和南海南沙西南陆架6个地理位点采集224尾短尾大眼鲷(Priacanthus macracanthus)样品,采用线粒体控制区(mtDNA D-loop区)序列分析了其种群遗传结构,探讨了6个采样位点之间短尾大眼鲷种群归属关系。所得224条同源序列D-loop片段(729 bp)中检测到101个突变位点和94个核苷酸多态位点,定义了172种单倍型;遗传多样性表现出高单倍型多样性(0.980 5~0.997 1)和低核苷酸多样性(0.048 2~0.060 9)的特点;系统发育分析、分子方差分析和成对的Fst分析显示南海各群体之间有较高的遗传同质性,遗传分化不显著,也没有出现明显的地理分支或聚簇;中性检测和不对称分布分析发现南海的短尾大眼鲷群体发生过种群扩张。结果表明,南海海域的6个短尾大眼鲷群体属于同一种群,该结果为今后中国与南海周边国家渔业资源共享和争取捕捞配额提供了依据。
Abstract:We investigated the genetic structure of red bigeye (Priacanthus macracanthus) population in six sampling sites off the South China Sea including five coastal waters of Beibu Gulf, Sanya, Maoming, Zhuhai, Shantou and the Southwest Nansha. Sequences of the mitochondrial control region (D-loop) were amplified by polymerase chain reaction. Fragments of 729 bp of the mitochondrial D-loop were sequenced from 224 individuals, and 101 mutated sites, 94 nucleotide polymorphism sites as well as 172 haplotypes were identified. All the six groups were characterized with high haplotype diversity (0.980 5~ 0.997 1) and low nucleotide diversity (0.048 2~0.060 9). Analyses of phylogeny, molecular variances and pairwise Fst showed no significant genealogical clades or clusters of samples corresponding to sampling localities. Both neutrality tests and mismatch distribution analyses revealed a recent population expansion in P.macracanthus. Thus, the six P.macracanthus groups in the South China Sea belong to one population. The results provide references for the fishery resources sharing and quotas winning between China and the South China Sea surrounding countries.
-
短尾大眼鲷(Priacanthus macracanthus),隶属鲈形目、大眼鲷科、大眼鲷属,广泛分布于日本南部到印度尼西亚西部、阿拉弗拉海与澳大利亚等西太平洋区域[1],主要栖息于200 m以内的大陆架海区底层。短尾大眼鲷在中国主要分布于南海、台湾海峡、东海和黄海南部,是南海重要经济种类,为底拖网和流刺网渔业的重要捕捞对象之一[2]。
目前短尾大眼鲷生物学方面的研究有东海区短尾大眼鲷的食性分析[1]、卵及仔稚鱼的发育[3]、鱼体的寄生虫大眼鲷匹里虫[4]等;其种群研究主要在生态学方面,如生长和死亡参数分析[5]、虚拟种群分析[6]、繁殖特征分析[7]、肌肉营养状况[8]等。鱼类种群遗传结构的研究是鱼类资源评估及其数量变动规律研究的基础和首要解决的基本问题[9],目前对短尾大眼鲷种群遗传结构资源状况的系统调查与评估[10]和渔业管理所掌握的资料还不够全面。该研究对南海北部陆架和南沙西南陆架采集的短尾大眼鲷样品,采用线粒体mtDNA D-loop区序列扩增及测序分析方法,从遗传结构多样性、系统进化发育以及分子系统地理学角度探讨遗传资源现状,并确定种群归属。分析结果可为短尾大眼鲷种群遗传特性和遗传分化提供基础数据,也可为其种群遗传资源现状的准确评价与合理开发利用提供理论指导和技术支持;还对维护南海渔业权益,为今后争取渔业资源共享和捕捞配额提供参考依据。
1. 材料与方法
1.1 试验材料
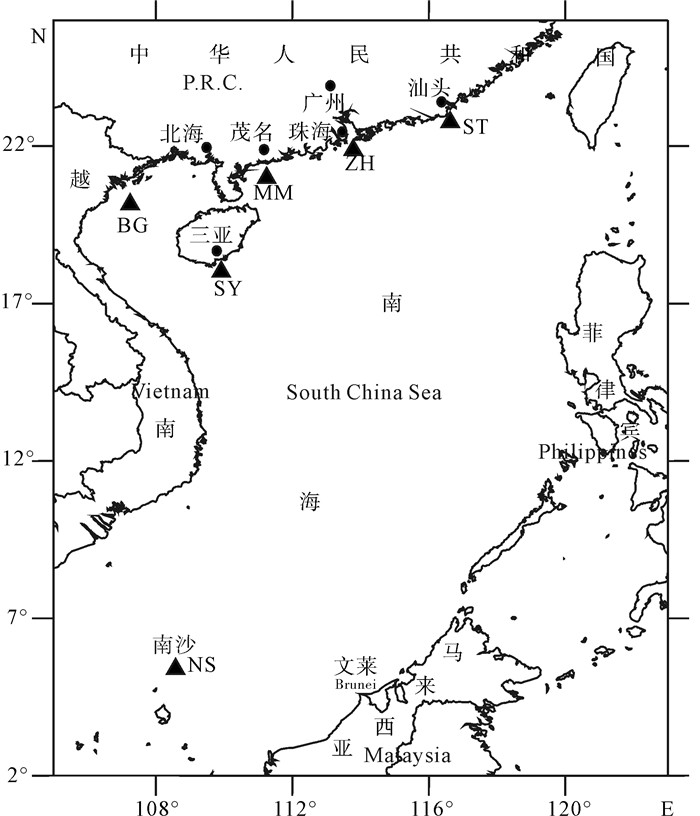
224个短尾大眼鲷群体收集于2013年7月至2014年3月,分别采自南沙(NS)40尾、北部湾(BG)46尾、三亚(SY)37尾、茂名(MM)34尾、珠海(ZH)37尾、汕头(ST)30尾,采样位点见图 1。样品是在不同地点不同船上采集以保证其随机性。根据相关分类标准对样本进行形态鉴定,并冷冻保存,带回实验室。
1.2 基因组的提取、PCR扩增和测序
每个样品取背部肌肉约25 mg,置于1.5 mL离心管内,使用上海生工生物工程股份有限公司的海洋动物组织DNA提取试剂盒提取总基因组DNA。根据已设计引物Pma-Dloop-F(5′-GCTTAGAGCACCAGTCTTGTAAGCTG-3′)和Pma-Dloop-R(5′-GCGGATACTTGCATGTGTAAGCTTAG-3′)扩增线粒体控制区序列。PCR反应体积为50 μL,包括约1 μL模版DNA、1 μL引物F(10 μmol·L-1)、1 μL引物R(10 μmol·L-1)、5 μL 10× ExTaq buffer、4 μL dNTP、0.5 μL ExTaq polymerase (TaKaRa, Japan)、和37.5 μL超纯水。PCR反应条件为94 ℃预变性3 min,94 ℃变性30 s,55 ℃退火25 s,72 ℃延伸1 min 30 s,共30个循环,最后72 ℃延伸5 min。用1% 的TBE琼脂糖胶电泳分离产物,GelRed染色,观察凝胶成像,选取阳性产物送交测序公司进行序列测定,测序引物为扩增正向引物。
1.3 数据处理
1.3.1 遗传多样性与种群结构
所有测得序列比对GenBank中短尾大眼鲷的线粒体D-loop基因全序列,由于线粒体控制区变异速度比较大,控制区序列也存在复杂结构,该研究样品数量较大,因此只选取了比较可信的部分序列,用MEGA 6[11]软件进行同源序列拼接和手工校正,得出序列碱基组成、变异位点数及遗传距离。利用DnaSP 5 [12]和Arlequin 3.11[13]统计单倍型数量(H)、多态位点(S)、单倍型多样性(h)[14]、核苷酸多样性(π)[14]、平均核苷酸差异数(k)[15]。
用分子方差分析(AMOVA)方法评估遗传变异,采用1 000次重抽样检验不同遗传结构水平上协方差的显著性。若群体Fst为0 ~ 0.05,则表明其各亚群间不存在分化;若Fst为0.05 ~ 0.15,为中度分化;若Fst为0.15 ~ 0.25,为高度分化[16-18]。AMOVA法和分化固定指数Fst均应用Arlequin 3.11执行。
1.3.2 种群系统发育关系
应用MEGA 6的邻近距离法(NJ)构建短尾大眼鲷mtDNA D-loop序列亲缘关系树,自展法(Bootstrap)检验重复次数1 000估计系统中结点的置信度。群体单倍型间的进化关系,使用Network 6[19]构建短尾大眼鲷mtDNA D-loop序列单倍型的MJ网络图。
1.3.3 种群历史动态
应用Arlequin 3.11中的不对称分布[20]分析短尾大眼鲷的种群历史动态。历史动态参数包括3个参数:种群扩张后的单位突变时间τ[21]、种群增长前后的θ值(分别为θ0和θ1)[22]。以年数为单位的种群扩张近似时间τ的求值方程:τ=2ut[21], 其中t为世代数,u为每一代单位序列的突变率,可由方程u=2μk求得,其中μ为每个核苷酸的突变率,控制区的分化速率采用(3%~10%)/MY(million year, MY)[23], k为所分析片段的核苷酸数目。
据报道不对称分布非常保守[24],该研究还通过2个突变漂移平衡的中性检验辅助检测短尾大眼鲷群体的历史动态。利用无限突变位点模型的Tajima′s D[25]和Fu′s Fs[26]方法分析推测物种进化中的主导因素。
2. 结果与分析
2.1 基因多样性分析
对224个样品mtDNA D-loop序列进行比对后,通过校正得到729 bp的线粒体D-loop的5′端序列,碱基T、C、A、G含量分别为34.1%、19.1%、31.8%、15.0%(表 1),其中A+T(65.9%)含量显著高于G+C(34.1%),表现出AT偏好和反G偏倚。基因序列中,保守位点是634个,变异位点有94个(包括57个简约信息位点,37个单一变异位点),转换位点79个,颠换位点22个,4个插入/缺失位点。
表 1 不同群体短尾大眼鲷的碱基频率、中性检验和不对称分布参数Table 1. Statistics of Tajima′s D, Fu′s Fs and mismatch distribution parameter of P.macracanthus群体group 编号ID T/% C/% A/% G/% Tajima′s D Fu′s Fs mismatch distribution D P Fs P τ θ0 θ1 南沙Nansha NS 34.2 19.1 31.8 15.0 -1.798 0 0.014 2 -25.346 8 0.000 0 3.675 7 1.453 7 99 999.000 0 北部湾Beibu Gulf BG 34.1 19.1 31.8 15.0 -1.696 4 0.022 7 -25.345 2 0.000 0 5.718 7 0.000 0 99 999.000 0 三亚Sanya SY 34.1 19.1 31.8 15.0 -1.801 9 0.015 0 -25.642 2 0.000 0 4.500 0 0.000 0 99 999.000 0 茂名Maoming MM 34.1 19.1 31.8 15.0 -1.641 3 0.031 0 -25.779 2 0.000 0 4.343 7 0.001 7 99 999.000 0 珠海Zhuhai ZH 34.1 19.1 31.8 15.0 -1.730 1 0.020 9 -25.597 2 0.000 0 4.679 6 0.014 0 143.359 3 汕头Shantou ST 34.1 19.1 31.7 15.0 -1.704 2 0.024 5 -25.104 1 0.000 0 4.187 5 1.221 4 260.625 0 平均average 34.1 19.1 31.8 15.0 -2.062 6 0.000 3 -25.101 2 0.000 9 4.931 6 0.008 7 99 999.000 0 此试验在短尾大眼鲷D-loop序列中共定义172个单倍型(表 2),其中有158个独享单倍型,占91.86%,14个共享单倍型,Hap 10频率最高,为9.88%(17/172),在试验6个采样点都有分布。
表 2 短尾大眼鲷样本信息及遗传多样性参数Table 2. Sampling information and descriptive statistics of genetic diversity of P.macracanthus编号ID 群体group 经度longitude 纬度latitude 采样数量quantity of sample 单倍型数(私有单倍型数) No.of haplotypes (private) 多态位点(S) segregating site 单倍型多样性(h) haplotype diversity 核苷酸多样性(π) nucleotide diverity 平均核苷酸差异(k) mean number of nucleotide differences NS 南沙Nansha 109.650E 6.037N 40 37(28) 49 0.994 9±0.007 5 0.060 6±0.032 9 5.698 7±2.788 8 BG 北部湾Beibu Gulf 107.483E 20.427N 46 42(37) 49 0.997 1±0.005 7 0.060 9±0.032 9 5.725 6±2.792 8 SY 三亚Sanya 110.135E 18.075N 37 30(26) 39 0.989 5±0.009 2 0.048 9±0.027 3 4.603 6±2.311 9 MM 茂名Maoming 111.548E 21.132N 34 31(23) 32 0.992 9±0.009 9 0.044 7±0.025 3 4.210 3±2.142 9 ZH 珠海Zhuhai 113.774E 22.353N 37 29(23) 37 0.980 5±0.013 5 0.048 2±0.026 9 4.531 5±2.280 2 ST 汕头Shantou 116.932E 23.062N 30 28(21) 42 0.993 1±0.011 8 0.060 7±0.033 3 5.710 3±2.813 9 平均average 224 172(158) 94 0.990 3±0.002 3 0.094 2±0.048 1 9.140 7±4.218 3 6个群体的遗传多样性参数见表 2。南海北部与西南陆架6个采样位点的短尾大眼鲷群体呈现高的单倍型多样性和较低的核苷酸多样性的特点,总体单倍型多样性h为0.990 3,核苷酸多样性π为0.094 2,平均核苷酸多样性k为9.140 7。6个群体的单倍型多样性指数均达到0.980 0以上的较高水平,其中以BG群体最高为0.997 1。MM群体具有最低的核苷酸多样性数(0.043 4)和最低的平均核苷酸多样性指数(4.210 3),BG群体具有最高的核苷酸多样性数(0.060 9)和最高平均核苷酸多样性数(5.725 6)。
2.2 种群系统地理学分析
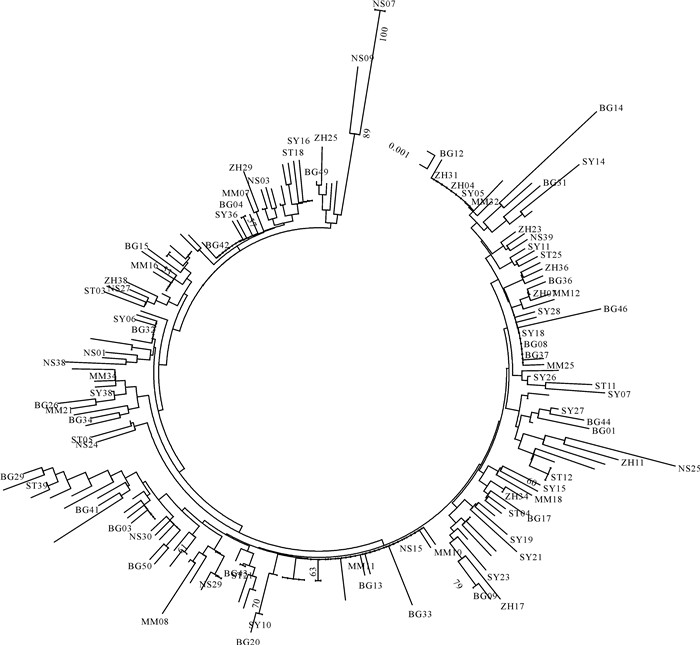
利用224尾短尾大眼鲷mtDNA控制区5′端高变区序列构建邻接法(NJ)系统发育树(图 2)发现,南海海域内短尾大眼鲷群体不存在显著的遗传分化,没有出现明显的地理分枝和集群。
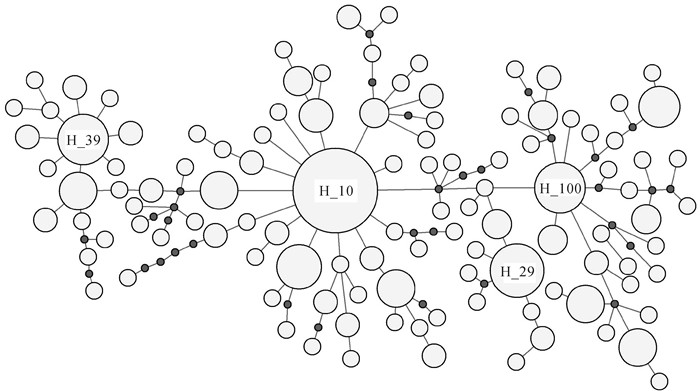
Network软件分析构建的南海海域短尾大眼鲷群体的单倍型网络(MJ)图(图 3)与其NJ系统发育树揭示了相似的结果,谱系发现大量的单倍型显示为明显的星状分布,Hap10、Hap29、Hap39是高频分布的优势单倍型。特有单倍型分散地分布在网络图中,未形成特有的进化枝。短尾大眼鲷单倍型没有表现出明显的地理聚群分布特征。
2.3 种群遗传结构分析
6个群体的群体遗传分化指数Fst显示南海海域各群体之间Fst均小于0.05,各群体之间不存在显著的遗传结构(表 3)。
表 3 短尾大眼鲷的遗传分化系数Fst(对角线下)和P值(对角线上)Table 3. Pairwise Fst (below diagonal) and P value of population differentiation (above diagonal) of P.macracanthus南沙NS 北部湾BG 三亚SY 茂名MM 珠海ZH 汕头ST 南沙NS 0.040 0 0.135 7 0.036 1 0.324 2 0.958 9 北部湾BG 0.013 2 0.607 4 0.669 9 0.652 3 0.044 9 三亚SY 0.008 1 -0.002 9 0.793 9 0.976 5 0.338 8 茂名MM 0.017 1 -0.003 4 -0.006 9 0.832 0 0.015 6 珠海ZH 0.002 4 -0.003 7 -0.011 4 -0.008 0 0.238 2 汕头ST -0.012 6 0.014 6 0.001 8 0.024 0 0.004 9 将南海海域的6个群体作为一组,采用1 000次单倍型重复随机抽样进行分子方差分析(表 4)。南海海域各群体内存在较高的遗传变异(99.72%),Fst为0.002 77。因此分子遗传变异主要来南海海域各群体内,南海海域各群体间短尾大眼鲷是随机交配的。
表 4 不同群体短尾大眼鲷遗传差异的AMOVA多样性分析Table 4. Analysis of molecular variance (AMOVA) among different groups of P.macracanthus变异来源source of variation 自由度degrees of freedom 平方和sum of squares 变异组成variance components 变异百分比percentage of variation F P 群体间among populations 5 14.087 0.007 1 0.28 群体内within populations 218 556.654 2.553 5 99.72 总计total 223 570.741 2.560 6 100 Fst=0.002 8 0.207 9 2.4 历史动态分析
中性检验的结果(表 1)显示南海海域各群体各自的Tajima′s D均为负,为-1.641 3~-1.801 9,且所有群体都达到显著性差异(P<0.05);南海海域整体的Tajima′s D为-2.062 6,其P为0.000 3,达到极显著水平(P<0.01)。南海海域各群体各自的Fu′s Fs为-25.779 2~-25.104 1,所有群体都达到显著性差异(P<0.01);南海海域整体的Fu′s Fs为-25.101 2,其P为0.000 9,达到极显著水平(P<0.01)。南海海域各群体各自的Tajima′s D和Fu′s Fs均为负值,可推测其很有可能在过去的某个时间段发生过种群扩张。南海海域短尾大眼鲷整个群体的扩张参数τ为4.931 6,南沙西南陆架NS群体的τ相对较小(3.675 7);BG群体的τ最大(5.718 7)。
3. 讨论
3.1 遗传多态性
南海北部陆架和南沙西南陆架6个群体224尾短尾大眼鲷样本的mtDNA D-loop 5′端高变序列有高的单倍型多样性(0.980 5~0.997 1)和较低的核苷酸多样性(0.048 2~0.060 9),同蓝圆鲹(Decapterus maruadsi)、竹筴鱼(Trachurus japonicus)、梭鱼(Liza haematocheila)、二长棘鲷(Parargyrops edita)[27-30]等鱼类线粒体控制区遗传多样性比较,发现该研究短尾大眼鲷各群体具有相对丰富的遗传多样性。遗传多样性是物种或种群长期进化的产物,影响着生物的生存和进化能力[31],研究显示,短尾大眼鲷遗传多样性高,对栖息地的生存适应能力较强,种群延续发展情况良好。
3.2 群体分子系统地理学
根据以往历史资料记载,短尾大眼鲷通常生长较慢,性成熟也较迟,寿命短,抗捕能力相当弱。虽有休渔季和禁捕区的保护,其资源量仍然遭到严重破坏。判别南海短尾大眼鲷种群可以为渔业资源管理过程中了解生物进化和制定长期可持续利用的管理政策提供重要信息[32]。要准确了解短尾大眼鲷种群结构,除了依据短尾大眼鲷本身的生物学特性如繁殖期时间、幼体扩散等,还可以综合依据历史地理进化关系、海流模式等因素来预测群体间相互关系。
1) 南海短尾大眼鲷群体的历史地理进化。短尾大眼鲷群体历史动态中,Tajima′s D检测到南海海域内群体值为-2.062 6,并达到显著差异(P<0.01)。Fu′s Fs中性检验中检测到南海海域内群体呈现较大的负(-25.101 2)和差异显著(P<0.01)。说明南海海域短尾大眼鲷群体在历史上可能发生过种群扩张,或者说经历过种群瓶颈,种群在致危因素作用时,数量快速减少成为一个小群体,而后增加[33]。根据南海海域整体的τ(4.931 6),估计其群体扩张时间在17 000 ~ 56 000年前,该时期正是晚更新世,这个时期,南海海域群体内缺乏系统地理结构主要是因为其群体栖息地扩张时间相对比较晚,南海海域各个群体之间还没有在迁移和漂变两者之间取得平衡。“更新世冰期剧烈的气候变迁会对生物的分布范围和资源量产生巨大影响,在生物的遗传信息上,也会留下明显印记”[34-37]。南海自新生代晚期形成以来,其面积和结构都发生过巨大变化。在更新世冰期,南海是一个内陆海,伴随每次盛冰期,海平面下降约为120~140 m[38],南海的短尾大眼鲷群体可能发生隔离导致分化。冰消期海平面上升,被隔离的群体间还未有重新获得交流,由于底层鱼类短尾大眼鲷的扩散能力非常有限,或者群体间虽存在交流,但迁移个体充分融入到新群体中的机会非常低。
2) 南海海流模式对短尾大眼鲷群体的影响。在系统发育树和进化网络中均可发现南海海域短尾大眼鲷群体不存在显著的遗传分化,属于同一个种群,但是在不对称分布表中南沙西南陆架的τ为3.675 7比南海北部陆架各群体的τ小(4.187 5~5.718 7),南沙西南陆架群体与茂名群体和北部湾群体的Fst达到研究群体中的最大和第二大, 分别为0.017 1和0.013 2,因此南海北部陆架各群体与南沙西南陆架的基因交流没有南海北部陆架的短尾大眼鲷群体间那么广泛,这可能与南海内复杂的环流模式[39]有关。南海北部有强的气旋式环流,导致南海北部陆架的短尾大眼鲷群体存在广泛的基因交流。越南沿岸强流可能导致南沙西南陆架的短尾大眼鲷群体向北洄游与南海北部陆架的短尾大眼鲷群体发生基因交流,但是由于越南沿岸的大陆坡很小,越南以东存在着静止的中尺度涡[40-41],南沙西南陆架与南海北部陆架的短尾大眼鲷群体并没有像南海北部陆架各群体间那样非常充分地发生交流。基于以上洋流模式,南海海域内不存在显著的遗传结构差异,这与南海北部陆架多齿蛇鲻(Saurida tumbi)[42]、中沙和南沙群岛密点胡椒鲷(Plectorhynchus pumctatissimus)[43]、南海北部金线鱼(Nemipterus virgatus)[44]、南海北部和南沙西南陆架的黄鳍马面鲀(Thamnaconus hypargyreus)[45]等的南海鱼类种群分析研究结果一致。
-
表 1 不同群体短尾大眼鲷的碱基频率、中性检验和不对称分布参数
Table 1 Statistics of Tajima′s D, Fu′s Fs and mismatch distribution parameter of P.macracanthus
群体group 编号ID T/% C/% A/% G/% Tajima′s D Fu′s Fs mismatch distribution D P Fs P τ θ0 θ1 南沙Nansha NS 34.2 19.1 31.8 15.0 -1.798 0 0.014 2 -25.346 8 0.000 0 3.675 7 1.453 7 99 999.000 0 北部湾Beibu Gulf BG 34.1 19.1 31.8 15.0 -1.696 4 0.022 7 -25.345 2 0.000 0 5.718 7 0.000 0 99 999.000 0 三亚Sanya SY 34.1 19.1 31.8 15.0 -1.801 9 0.015 0 -25.642 2 0.000 0 4.500 0 0.000 0 99 999.000 0 茂名Maoming MM 34.1 19.1 31.8 15.0 -1.641 3 0.031 0 -25.779 2 0.000 0 4.343 7 0.001 7 99 999.000 0 珠海Zhuhai ZH 34.1 19.1 31.8 15.0 -1.730 1 0.020 9 -25.597 2 0.000 0 4.679 6 0.014 0 143.359 3 汕头Shantou ST 34.1 19.1 31.7 15.0 -1.704 2 0.024 5 -25.104 1 0.000 0 4.187 5 1.221 4 260.625 0 平均average 34.1 19.1 31.8 15.0 -2.062 6 0.000 3 -25.101 2 0.000 9 4.931 6 0.008 7 99 999.000 0 表 2 短尾大眼鲷样本信息及遗传多样性参数
Table 2 Sampling information and descriptive statistics of genetic diversity of P.macracanthus
编号ID 群体group 经度longitude 纬度latitude 采样数量quantity of sample 单倍型数(私有单倍型数) No.of haplotypes (private) 多态位点(S) segregating site 单倍型多样性(h) haplotype diversity 核苷酸多样性(π) nucleotide diverity 平均核苷酸差异(k) mean number of nucleotide differences NS 南沙Nansha 109.650E 6.037N 40 37(28) 49 0.994 9±0.007 5 0.060 6±0.032 9 5.698 7±2.788 8 BG 北部湾Beibu Gulf 107.483E 20.427N 46 42(37) 49 0.997 1±0.005 7 0.060 9±0.032 9 5.725 6±2.792 8 SY 三亚Sanya 110.135E 18.075N 37 30(26) 39 0.989 5±0.009 2 0.048 9±0.027 3 4.603 6±2.311 9 MM 茂名Maoming 111.548E 21.132N 34 31(23) 32 0.992 9±0.009 9 0.044 7±0.025 3 4.210 3±2.142 9 ZH 珠海Zhuhai 113.774E 22.353N 37 29(23) 37 0.980 5±0.013 5 0.048 2±0.026 9 4.531 5±2.280 2 ST 汕头Shantou 116.932E 23.062N 30 28(21) 42 0.993 1±0.011 8 0.060 7±0.033 3 5.710 3±2.813 9 平均average 224 172(158) 94 0.990 3±0.002 3 0.094 2±0.048 1 9.140 7±4.218 3 表 3 短尾大眼鲷的遗传分化系数Fst(对角线下)和P值(对角线上)
Table 3 Pairwise Fst (below diagonal) and P value of population differentiation (above diagonal) of P.macracanthus
南沙NS 北部湾BG 三亚SY 茂名MM 珠海ZH 汕头ST 南沙NS 0.040 0 0.135 7 0.036 1 0.324 2 0.958 9 北部湾BG 0.013 2 0.607 4 0.669 9 0.652 3 0.044 9 三亚SY 0.008 1 -0.002 9 0.793 9 0.976 5 0.338 8 茂名MM 0.017 1 -0.003 4 -0.006 9 0.832 0 0.015 6 珠海ZH 0.002 4 -0.003 7 -0.011 4 -0.008 0 0.238 2 汕头ST -0.012 6 0.014 6 0.001 8 0.024 0 0.004 9 表 4 不同群体短尾大眼鲷遗传差异的AMOVA多样性分析
Table 4 Analysis of molecular variance (AMOVA) among different groups of P.macracanthus
变异来源source of variation 自由度degrees of freedom 平方和sum of squares 变异组成variance components 变异百分比percentage of variation F P 群体间among populations 5 14.087 0.007 1 0.28 群体内within populations 218 556.654 2.553 5 99.72 总计total 223 570.741 2.560 6 100 Fst=0.002 8 0.207 9 -
[1] SOKOLOVSKAYA T G, SOKOLOVSKII A S, SOBOLEVSKII E I. A list of fishes of Peter the Great Bay (the Sea of Japan)[J]. J Ichthyol, 1998, 38(1): 1-11. https://www.semanticscholar.org/paper/A-LIST-OF-FISHES-OF-PETER-THE-GREAT-BAY-(THE-SEA-OF-Sokolovskaya-Sokolovskii/c65b189aad74d9abcdf1cd8052fe67600840f0b1
[2] 卢占晖, 朱增军, 林显鹏. 东海区短尾大眼鲷食性分析[J]. 海洋渔业, 2010, 32(2): 199-203. doi: 10.3969/j.issn.1004-2490.2010.02.014 [3] 张仁斋, 陆穗芬. 短尾大眼鲷卵及仔、稚鱼的发育[J]. 水产学报, 1982(3): 243-251. http://scxuebao.ijournals.cn/scxuebao/article/abstract/19820306 [4] 何筱洁. 大眼鲷匹里虫病及其病原体的研究——包括匹里虫属一新种的描述[J]. 水产学报, 1982(2): 97-106. http://scxuebao.ijournals.cn/scxuebao/article/abstract/19820202 [5] 孙典荣, 邱永松. 北部湾长尾大眼鲷生长和死亡参数估计[J]. 海洋湖沼通报, 2004(3): 27-34. doi: 10.3969/j.issn.1003-6482.2004.03.005 [6] LIU K M, CHENG Y L. Virtual population analysis of the big eye Priacanthus macracanthus in the waters off northeastern Taiwan[J]. Fish Res, 1999, 41(3): 243-254. doi: 10.1016/S0165-7836(99)00025-9
[7] LIU K M, HUNG K Y, CHEN C T. Reproductive biology of the big eye Priacanthus macracanthus in the north-eastern waters off Taiwan[J]. Fish Sci, 2001, 67(6): 835-838. doi: 10.1046/j.1444-2906.2001.00355.x
[8] BENJAKUL S, OUNGBHO K, VISESSANGUAN W, et al. Characteristics of gelatin from the skins of bigeye snapper, Priacanthus tayenus and Priacanthus macracanthus[J]. Food Chem, 2009, 116(2): 307-325. doi: 10.1016/j.foodchem.2009.02.063
[9] 孙冬芳, 李永振, 董丽娜. AFLP技术在鱼类种群分析中的应用[J]. 南方水产, 2009, 5(6): 71-75. doi: 10.3969/j.issn.1673-2227.2009.06.013 [10] 宁平, 俞存根, 虞聪达, 等. 浙江南部外海渔场鱼类区系特征研究[J]. 浙江海洋学院学报, 2008, 27(3): 267-270. https://www.zhangqiaokeyan.com/academic-journal-cn_journal-zhejiang-ocean-university-natural-science_thesis/0201248287722.html [11] KOICHIRO T, GLEN S, DANIEL P, et. al. MEGA6: Molecular evolutionary genetics analysis version 6.0[J]. Mol Biol Evol, 2013, 30(12): 2725-2729. doi: 10.1093/molbev/mst197
[12] LIBRADO P, ROZAS J. DnaSP v5: a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 2009, 25(11): 1451-1452. doi: 10.1093/bioinformatics/btp187
[13] EXCOFFIER L, LAVAL G, SCHNEIDER S. Arlequin ver. 3.0: an integrated software package for population genetics data analysis[J]. Evol Bioinformatics, 2005, 1: 47-50. https://www.researchgate.net/publication/24238444_ARLEQUIN_Ver_30_An_integrated_software_package_for_population_genetic_data_analysis
[14] NEI M. Molecular evolutionary genetics[M]. New York: Columbia University Press, 1987: 153-187. doi: 10.7312/nei-92038
[15] TAJIMA F. Evolutionary relationship of DNA sequences in finite populations[J]. Genetics, 1983, 105(2): 437-467. doi: 10.1093/genetics/105.2.437
[16] WRIGHT S. The genetical structure of populations[J]. Annals of Eugenics, 1951, 15(1): 323-354. doi: 10.1111/j.1469-1809.1949.tb02451.x
[17] WRIGHT S. Evolution and the genetics of populations[M]//Variability within and among natural populations Vol. 4, Chicago: University of Chicago Press, 1978: 179-182. https://www.semanticscholar.org/paper/Evolution-and-the-Genetics-of-Populations%2C-Volume-Curnow-Wright/7ffa4dd7ecfb20e985c073866956be97764d86a1
[18] FREELAND J R. Molecular ecology[M]. Chichester: John Wiley & Sons Ltd, 2005: 112-116. https://onlinelibrary.wiley.com/toc/1365294x/2005/14/1
[19] BANDELT H J, FORSTER P, ROHL A. Median joining networks for inferring intraspecific phylogenics[J]. Mol Biol Evol, 1999, 16(1) : 37-48. doi: 10.1093/oxfordjournals.molbev.a026036
[20] ROGERS A R, HARPENDING H. Population growth makes waves in the distribution of pairwise genetic differences[J]. Mol Biol Evol, 1992, 9(3): 552-569. doi: 10.1093/oxfordjournals.molbev.a040727
[21] LI W H. Molecular evolution[M]. Sunderland, Massachusetts, USA: Sinauer Associates, Inc. Publishers, 1997: 59-63.
[22] HARPENDING H C. Signature of ancient population growth in a low-resolution mitochondrial DNA mismatch distribution[J]. Hum Biol, 1994, 66(4): 591-600. https://pubmed.ncbi.nlm.nih.gov/8088750/
[23] LEE W J, CONROY J, HOWELL W H, et al. Structure and evolution of teleost mitochondrial control region[J]. Mol Evol, 1995, 41(4): 54-66. doi: 10.1007/BF00174041
[24] RAMOS-ONSINS S E, ROZAS J. Statistical properties of new neutrality tests against population growth[J]. Mol Biol Evol, 2002, 19(12): 2092-2100. doi: 10.1093/oxfordjournals.molbev.a004034
[25] TAJIMA F. Statistical-method for testing the neutral mutation hypothesis by DNA polymorphism[J]. Genetics, 1989, 123(3): 585-595. doi: 10.1093/genetics/123.3.585
[26] FU Y X. Statistical tests of neutrality of mutations against population growth, hitchhiking and background selection[J]. Genetics, 1997, 147(2): 915-925. doi: 10.1093/genetics/147.2.915
[27] 牛素芳, 苏永全, 王军, 等. 福建近海蓝圆鲹群体遗传结构分析[J]. 厦门大学学报: 自然科学版, 2012, 51(4): 759-766. https://www.cnki.com.cn/Article/CJFDTotal-XDZK201204024.htm [28] 牛素芳, 苏永全, 王军, 等. 福建近海竹筴鱼线粒体DNA控制区和细胞色素b遗传多态性[J]. 中国水产科学, 2011, 19(1): 66-74. https://www.doc88.com/p-9734747774803.html [29] LIU J X, GAO T X, WU S F, et al. Pleistocene isolation in the Northwestern Pacific marginal seas and limited dispersal in a marine fish, Chelon haematocheilus (Temminck & Schlegel, 1845)[J]. Mol Ecol, 2007, 16(2): 275-288. doi: 10.1111/j.1365-294X.2006.03140.x
[30] 苏天凤, 江世贵, 马之明, 等. 二长棘犁齿鲷线粒体DNA控制区结构和进化[J]. 南方水产科学, 2012, 8(6): 1-8. doi: 10.3969/j.issn.2095-0780.2012.06.001 [31] 罗旭光, 齐景伟, 安晓萍, 等. 内蒙古达里湖及岗更湖东北雅罗鱼D-loop基因序列及多样性研究[J]. 南方水产科学, 2014, 10(2): 92-96. doi: 10.3969/j.issn.2095-0780.2014.02.014 [32] TUDELA S, GARCÍA-MARÍN J L, PLA C. Genetic structure of the European anchovy, Engraulis encrasicolus, in the north-west Mediterranean [J]. J Exp Mar Biol Ecol, 1999, 234(1): 95-109. doi: 10.1016/S0022-0981(98)00142-7
[33] 张东亚, 汪登强, 刘绍平, 等. 怒江濒危鱼类缺须盆唇鱼基于线粒体Cyt b序列的群体遗传结构分析[J]. 中国水产科学, 2009, 17(4): 477-486. doi: 10.3321/j.issn:1005-8737.2009.04.002 [34] 刘进贤, 高天翔, 吴世芳, 等. 梭鱼的分子系统地理学研究——晚更新世西北太平洋边缘海隔离分化及其有限的扩散能力[J]. 中国海洋大学学报: 自然科学版, 2007, 37(6): 931-938. doi: 10.16441/j.cnki.hdxb.2007.06.014 [35] AVISE J C. Phylogeography: the history and formation of species[M]. Cambridge, Massachusets: Harvard Unversity Press, 2000. https://www.nature.com/articles/6887654
[36] HEWITT G M. The genetic legacy of the Quaternary ice age[J]. Narture, 2000, 405(6789): 907-913. https://www.nature.com/articles/35016000
[37] DYNESIUS M, JANSSON R. Evolutionary consequences of changes in species′ geographical distribution driven by Milankovitch climate oscillations[J]. Proc Natl Acad Sci USA, 2000, 97(16): 9115-9120. doi: 10.1073/pnas.97.16.9115
[38] LAMBECK K, HONZA E. Global tectonics, and formation of marginal basins: role of the westerm Pacific[J]. Episodes, 1991, 14: 224-230. doi: 10.18814/epiiugs/1991/v14i3/005
[39] 杨海军, 刘秦玉. 南海海洋环流研究综述[J]. 地球科学进展, 1998(4): 47-51. doi: 10.3321/j.issn:1001-8166.1998.04.007 [40] 苏纪兰. 南海环流动力机制研究综述[J]. 海洋学报: 中文版, 2005, 27(6): 3-10. http://www.hyxb.org.cn/aos/ch/reader/view_abstract.aspx?file_no=20050601&st=alljournals [41] 管秉贤, 袁耀初. 中国近海及其附近海域若干涡旋研究综述Ⅰ. 南海和台湾以东海域[J]. 海洋学报: 中文版, 2006, 28(3): 1-16. https://qikan.cqvip.com/Qikan/Article/Detail?id=21970714 [42] 孙冬芳, 董丽娜, 李永振, 等. 南海北部海域多齿蛇鲻的种群分析[J]. 水产学报, 2010, 34(9): 1387-1394. doi: 10.3724/SP.J.1231.2010.06975 [43] 孙冬芳, 卢伟华, 艾红, 等. 中沙和南沙群岛密点胡椒鲷的种群分析[J]. 广东农业科学, 2010(5): 4-7. doi: 10.3969/j.issn.1004-874X.2010.05.002 [44] 董丽娜. 南海北部陆架区金线鱼(Nemipterus virgatus)种群分子分析[D]. 上海: 上海海洋大学, 2011. https://cdmd.cnki.com.cn/Article/CDMD-10264-1011304986.htm [45] 李玉芳, 李永振, 陈国宝, 等. 基于线粒体Cyt b基因的黄鳍马面鲀种群分析[J]. 水产学报, 2014, 38(1): 23-32. doi: 10.3724/SP.J.1231.2014.48864 -
期刊类型引用(10)
1. 孙良娟,李红权,蔡润斌,庄姜云,唐庆强,唐媛媛,钟键,黄武. 高效液相色谱-串联质谱法测定水产品中9种禁用染料类药物残留. 分析测试学报. 2023(01): 89-95 .  百度学术
百度学术
2. 李涛,贾立平,李锋刚,王绿洲. 亚甲基蓝在鲤体内消除规律研究. 水产科技情报. 2022(02): 99-105 .  百度学术
百度学术
3. 黄宣运,杨光昕,孔聪,黄冬梅,蔡友琼. 间接竞争酶联免疫法测定鱼体中的亚甲基蓝. 食品安全质量检测学报. 2020(22): 8235-8239 .  百度学术
百度学术
4. 余玮玥,黄冬梅,史永富,孔聪,田良良,韩峰,张政权. 液相色谱-串联质谱法测定水产品中亮绿、亚甲基蓝及其代谢物残留量. 分析测试学报. 2019(01): 58-63 .  百度学术
百度学术
5. 施雅. 淡水鱼中亚甲基蓝、孔雀石绿类药物残留检测. 广州化工. 2019(14): 131-133 .  百度学术
百度学术
6. 高平,杨曦,莫彩娜,陈日檬,曾丹丹,刘唤明,洪鹏志,周凯,陈营寿. 通过式固相萃取净化/高效液相色谱-串联质谱法快速测定水产品中6种麻醉剂残留. 分析测试学报. 2019(09): 1059-1065 .  百度学术
百度学术
7. 杨卫军,张静余,严敏鸣. 亚甲基蓝检测方法及代谢动力学研究进展. 食品安全质量检测学报. 2018(10): 2419-2425 .  百度学术
百度学术
8. Jin Fan,Zhi-Hai Xie,Xiao-Xiao Teng,Yu Zhang. Determination of methylene blue by resonance light scattering method using silica nanoparticles as probe. Chinese Chemical Letters. 2017(05): 1104-1110 .  必应学术
必应学术
9. 梁楠,胡鲲,刘腾飞,吴映捷,杨先乐. 亚甲基蓝及其代谢物在异育银鲫体内分布及消除规律的研究. 南方水产科学. 2017(01): 19-25 .  本站查看
本站查看
10. 龙举,李子孟,喻亮,李佩佩,严忠雍,张小军. 高效液相色谱法测定养殖水体中的亚甲基蓝. 安徽农业科学. 2015(24): 5-6+16 .  百度学术
百度学术
其他类型引用(5)



 下载:
下载:
 粤公网安备 44010502001741号
粤公网安备 44010502001741号
