Population genetic structure of Sthenoteuthis oualaniensis in the northwestern Indian Ocean by RAPD analysis
-
摘要:
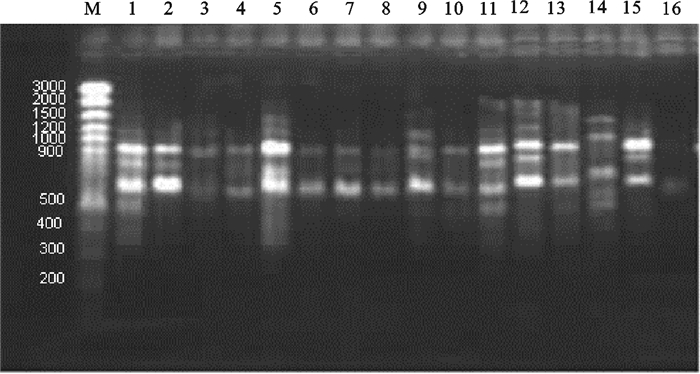
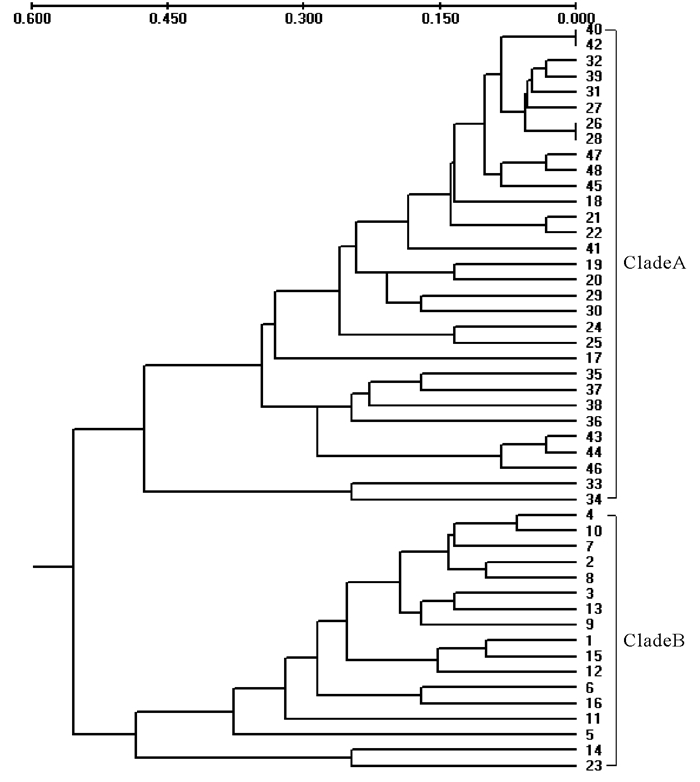
根据2004年9~12月和2005年3~5月印度洋西北部公海海域(13°N~20°N、59°E~64°E)鸢乌贼(Sthenoteuthis oualaniensis)资源调查中所采集的样本,选取了12个站点48尾鸢乌贼肌肉样本进行随机扩增多态性DNA分析。根据UPGMA聚类分析,样本中存在2个种群,13°N~18°N海域和18°N~20°N海域。分析得知,2种群间遗传分化指数为0.2150,表明不同种群间在遗传背景上存在较大的差异,且种群内的遗传变异水平较高。Shannon多样性指数为0.3676±0.1801。2种群间的遗传距离为0.1338,遗传相似性指数为0.8748。
Abstract:During the exploitation of purpleback flying squid Sthenoteuthis oualaniensis resources in the northwestern Indian Ocean (13°N~20°N, 59°E~64°E) from September to December in 2004 and from March to May in 2005, 48 individuals muscle samples collected from 12 stations were used for population genetic structure identification by Random Amplified Polymorphic DNA (RAPD) analysis. The results showed that the purpleback flying squid in the northwestern Indian Ocean could be divided into two populations by UPGMA clustering analysis.One population distributed in the area of 13°N~18°N, while another population distributed in the area of 18°N~20°N. The results showed that high genetic variations were found within population and the coefficient of genetic differentiation between the two populations was 0.2150, which indicated that quite different genetic background existed in the two populations. Shannon′s diversity index was 0.3676±0.1801. Genetic distance between the two populations was 0.1338, and genetic similarity was 0.8748.
-
鱼类的生长与发育和哺乳动物相似,主要受生长激素/胰岛素样生长因子(growth hormone/insulin-like growth factor,GH/IGF)轴调节[1-4]。近年来,已有多种鱼类的GH和IGF基因被克隆,并且有关其重组蛋白的促生长活性也有一定的研究。研究表明,重组GH和IGF都能促进多数鱼类的生长,但是重组GH对虹鳟(Oncorhynchus mykiss)[5]并无显著促生长活性。最近研究表明,重组鲮(Cirrhinus molitorella)GH和IGF-Ⅰ均有促进罗非鱼(Oreochromis niloticus)生长活性,且重组鲮GH对罗非鱼稚鱼的促生长活性较重组鲮IGF-Ⅰ弱[6],但是重组鲮GH和IGF-Ⅰ促生长作用的分子机制以及它们在鲮体内的相互作用方式并不清楚。
笔者在研究了重组鲮IGF-Ⅰ对鲮GH表达的影响后[7],为更深一步了解重组鲮GH和IGF-Ⅰ促生长作用的分子机制以及它们在鲮体内的相互作用方式,该试验以我国南方主要养殖鱼类之一鲮为研究对象,在鲮GH和IGF-ⅠcDNA被克隆和体外重组表达的基础上[3, 8],对鲮IGF-ⅠmRNA进行组织表达分析,并分析重组鲮GH处理后鲮IGF-Ⅰ表达变化情况。
1. 材料与方法
1.1 重组鲮GH
重组鲮GH的表达和纯化参照ZHANG等[8]的方法进行,纯化后的重组鲮GH充分透析,冷冻干燥,溶解在0.7%的生理盐水中待用(pH 7.0)。
1.2 实验动物的处理以及组织的提取
将鲮(500±10 g)从广州市番禺区鱼窝头镇养殖户的池塘中捕捞起后暂养于广东省鲮鱼原种场,待其状态稳定后,按鱼体重120 μg·g-1注射重组鲮GH或0.7%的生理盐水,12 h后取鲮鱼脑、肝和肌肉保存于液氮中,对照组取脑、肝、肌肉、肠、性腺、鳃、皮肤、脾脏、心脏和肾10种组织保存于液氮;并从每条鱼中抽取血液(大约3 mL)4℃冷凝30 min,4℃ 1 500 g离心15 min,取上清保存于-20℃,待检测GH和IGF-Ⅰ含量[9]。
1.3 总RNA的提取与RT-PCR
按Trizol Reagent(Invitrogen)操作说明提取各组织总RNA,用0.5×TBE配置1%琼脂糖凝胶,取2 μL总RNA与2 μL 2×DEPC处理后加样缓冲液混合,65℃加热15 min后,立即于冰上放置2 min,加样电泳,鉴定所提取总RNA的质量,取2 μL总RNA测定其光密度值OD260和OD280,计算总RNA浓度。
进行RT-PCR前必须测RNA浓度,逆转录体系对RNA量有一定要求,该研究按照逆转录试剂盒TakaRa RNA LA PCRTM Kit (AMV) Ver. 1.1说明书要求,所用总RNA量为500 ng。
按逆转录试剂盒TakaRa RNA LA PCRTM Kit (AMV) Ver. 1.1介绍方法进行逆转录,MgCl2 2μL,10×RNA PCR Buffer 1μL,RNase Free H2O 4.25 μL,dNTP Mixture(各10 mM) 1μL,Rnase Inhibitor 0.25 μL,AMV Reverse Transcriptase 0.5 μL, Oligo(dT)-Adaptor Primer 0.5 μL,Total RNA 0.5 μL(500 ng)。50℃45 min,99℃5 min,5℃5 min,1个循环。
1.4 PCR循环数确定及鲮IGF-ⅠmRNA组织表达
首先确定PCR扩增反应条件,即确定PCR扩增反应循环数[10]。用对照组肝cDNA作模板,引物为Actin-F,Actin-R(表 1),94℃变性30 s,54℃退火30 s,72℃延伸45 s,每PCR循环数为15、20、25、30、35、40、45取4 μL,PCR产物在1.5%的琼脂糖凝胶电泳EB染色分析。循环数确定后利用看家基因Actin做内标,PCR检测鲮IGF-ⅠmRNA各组织表达情况以及重组鲮GH处理后IGF-ⅠmRNA在脑、肝和肌肉的表达,所有PCR反应体系包括1 μL(0.15 μg)cDNA,0.25 μL rTaq酶(TaKaRa公司),正反向引物各1 μL,4 μL dNTP,5 μL 10×PCR缓冲液,双蒸水37.75 μL,总体积50 μL。PCR反应均94℃变性5 min,35个循环(94℃变性30 s,54℃退火30 s,72℃延伸45 s),最后72℃延伸10 min。IGF-Ⅰ mRNA检测引物序列如表 1。
表 1 扩增IGF-Ⅰ、Actin的引物序列Table 1. Oligonucleotide primers used to amplify cDNA for mud carp IGF-Ⅰand Actin引物 primer 序列 sequence Actin-F 5′-GTGTTGGCG/ATACAGGTCCTTACG-3′ Actin-R 5′-CAGACTACCTC/GATGAAGATCCTGAC-3′ IGF-Ⅰ-F 5′-ATGGAAAACCAGCGCCTCTTC-3′ IGF-Ⅰ-R 5′-TGCATGTCCTTCTTGAAGCAAG-3′ 1.5 各组织mRNA表达检测结果分析
利用GeneTools软件分析,计算出步骤1.4中所有IGF-Ⅰ和Actin条带的相对质量,再将对应的IGF-Ⅰ与Actin质量相比,得出GH的相对表达量。
1.6 IGF-Ⅰ含量检测
利用GeneTools软件分析,计算出步骤1.4中所有IGF-Ⅰ和Actin条带的相对质量,再将对应的IGF-Ⅰ与Actin质量相比,得出IGF-Ⅰ的相对表达量。
2. 结果
2.1 总RNA提取与RT-PCR
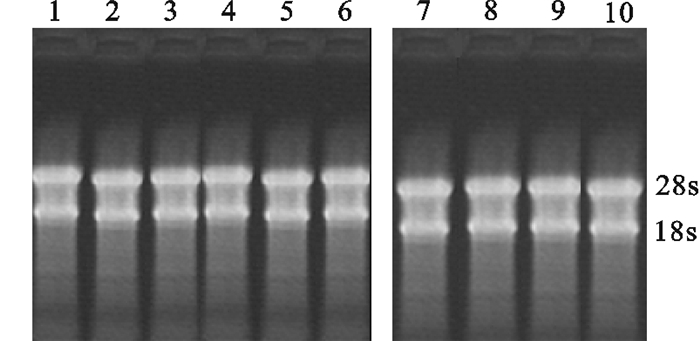
从100 mg鲮组织中提取RNA,经1%琼脂糖凝胶电泳检测,可见清晰的28 s、18 s条带(图 1),表明总RNA完整性很好,根据测定RNA的OD260和OD280值,计算其比值,比值均在1.9~2.0之间,也说明RNA纯度较高,可用于后续反应。
2.2 PCR循环数的确定
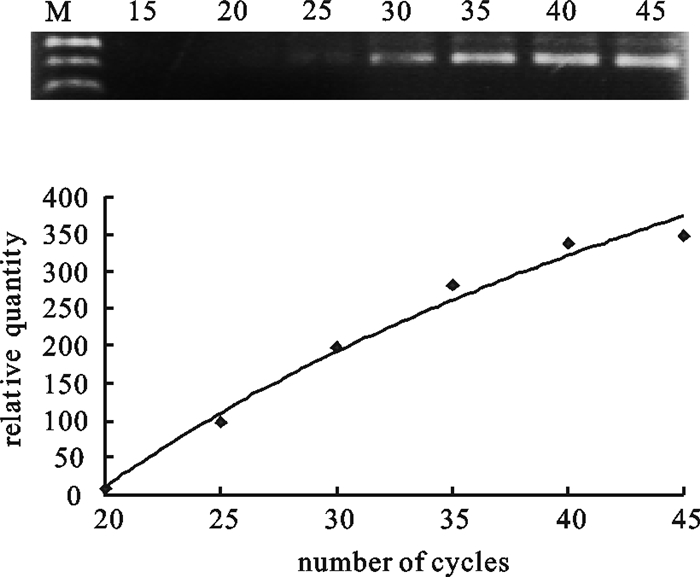
首先确定PCR反应条件,即确定PCR扩增反应循环数。通过鲮的分子内标Actin基因的相对表达量确定PCR扩增反应循环数,用于检测鲮IGF-ⅠmRNA组织表达。由图 2结果可知,循环数在25以下时,Actin扩增量较低,电泳条带不够清晰;循环数在30以上时结果较好,循环数为35最为理想(图 2)。
2.3 鲮IGF-ⅠmRNA组织表达与重组鲮GH对鲮IGF-ⅠmRNA组织表达的影响
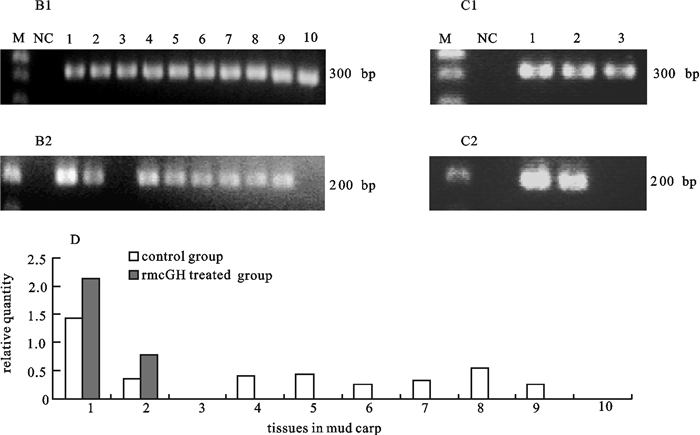
通过半定量RT-PCR方法对经重组鲮GH处理前后鲮IGF-ⅠmRNA在各组织中表达检测结果显示,鲮IGF-ⅠmRNA在肝组织中表达最高,其次是肾和脑,另外在肠、鳃脾、性腺和心脏组织中也有表达,而在肌肉、皮肤中没有检测到鲮IGF-ⅠmRNA的表达;研究了经腹腔注射重组鲮GH(120 μg·g-1鱼体重) 12 h后,鲮IGF-Ⅰ表达变化情况,结果表明,经重组鲮GH处理后,鲮肝组织IGF-ⅠmRNA水平显著升高,脑组织IGF-ⅠmRNA水平也稍有升高,而在肌肉中仍未检测到IGF-ⅠmRNA的表达(图 3)。
![]() 图 3 半定量RT-PCR检测重组鲮GH处理前后鲮actin和IGF-ⅠmRNA的组织表达B1. 半定量RT-PCR检测对照组中鲮各组织中actin mRNA的表达;B2. 半定量RT-PCR检测对照组中鲮各组织中IGF-ⅠmRNA的表达;C1. 半定量RT-PCR检测处理组中鲮各组织中actin mRNA的表达;C2. 半定量RT-PCR检测处理组中鲮各组织中IGF-ⅠmRNA的表达;D. 处理前后鲮组织IGF-ⅠmRNA的相对表达;M. 100 bp DNA分子量标准;NC. 负对照;1~10. 肝、脑、肌肉、肾、肠、鳃、脾、性腺、心脏、皮肤Figure 3. Semi-quantitative RT-PCR analysis of expression of Actin and IGF-Ⅰ mRNA in various tissues in mud carpB1. Actin mRNA was detected in various tissues by semi-quantitative RT-PCR in control group; B2. IGF-ⅠmRNA was detected in various tissues by semi-quantitative RT-PCR in control group; C1. Actin mRNA was detected in various tissues by semi-quantitative RT-PCR in rmcGH in treated group; C2. IGF-Ⅰ mRNA was detected in various tissues by semi-quantitative RT-PCR in rmcGH in treated group; D. relative quantity of IGF-ⅠmRNA in various tissues in mud carp; M.100 bp DNA molecular weight marker; NC. a negative control (no template); 1~10. brain, liver, muscle, kidney, intestine, gill, spleen, gonad, heart, skin
图 3 半定量RT-PCR检测重组鲮GH处理前后鲮actin和IGF-ⅠmRNA的组织表达B1. 半定量RT-PCR检测对照组中鲮各组织中actin mRNA的表达;B2. 半定量RT-PCR检测对照组中鲮各组织中IGF-ⅠmRNA的表达;C1. 半定量RT-PCR检测处理组中鲮各组织中actin mRNA的表达;C2. 半定量RT-PCR检测处理组中鲮各组织中IGF-ⅠmRNA的表达;D. 处理前后鲮组织IGF-ⅠmRNA的相对表达;M. 100 bp DNA分子量标准;NC. 负对照;1~10. 肝、脑、肌肉、肾、肠、鳃、脾、性腺、心脏、皮肤Figure 3. Semi-quantitative RT-PCR analysis of expression of Actin and IGF-Ⅰ mRNA in various tissues in mud carpB1. Actin mRNA was detected in various tissues by semi-quantitative RT-PCR in control group; B2. IGF-ⅠmRNA was detected in various tissues by semi-quantitative RT-PCR in control group; C1. Actin mRNA was detected in various tissues by semi-quantitative RT-PCR in rmcGH in treated group; C2. IGF-Ⅰ mRNA was detected in various tissues by semi-quantitative RT-PCR in rmcGH in treated group; D. relative quantity of IGF-ⅠmRNA in various tissues in mud carp; M.100 bp DNA molecular weight marker; NC. a negative control (no template); 1~10. brain, liver, muscle, kidney, intestine, gill, spleen, gonad, heart, skin2.4 血清中IGF-Ⅰ的含量
血清中IGF-Ⅰ的含量,对照组为145.59±21.84 ng·mL-1,重组鲮GH处理组为247.71±2.83 ng·mL-1,经t检验,P=0.043 < 0.05,存在显著性差异;血清中GH的含量,对照组为0.037±0.0067 ng·mL-1,重组鲮IGF-Ⅰ处理组为0.04±0.0058 ng·mL-1,经t检验,P=0.508>0.05,故不存在显著性差异(表 2)。
表 2 对照组和重组鲮IGF-Ⅰ处理组鲮血清中GH和IGF-Ⅰ浓度Table 2. The GH and IGF-Ⅰconcentration in sera in control and rmc IGF-Ⅰtreated fish浓度±S.E.(n=3)/ng·mL-1 concentration±S.E. 对照组
control重组鲮IGF-Ⅰ处理组
rmcIGF-Ⅰtreated groupP值 胰岛素样生长因子-Ⅰ浓度
IGF-Ⅰconcentration145.59±21.84 247.71±2.83 P=0.043<0.05 生长激素浓度
GH concentration0.037±0.0067 0.053±0.0067 P=0.508>0.05 3. 讨论
采用半定量RT-PCR方法,检测到鲮肝、肾、脑、肠、鳃、脾、性腺、心脏组织都有IGF-ⅠmRNA表达,且在鲮肝组织中表达量最高,在鲮肾、脑、肠、鳃、脾、性腺、心脏组织中表达量较低;而在鲮皮肤和肌肉组织中未检测到有IGF-ⅠmRNA的表达。鲮IGF-ⅠmRNA组织表达的特异性,表明了鲮IGF-Ⅰ主要由肝组织合成并进入血液(内分泌功能),其它组织也能合成少量的IGF-Ⅰ作用于自身细胞(自分泌或旁分泌功能)。这一结果与草鱼(Cirrhinus molitorella)、金鲷(Sparus aurata)以及其它一些脊椎动物中的结果一致[10-15]。
与哺乳类相似,生长激素是鱼类IGF-Ⅰ表达调控的主要影响因子之一。生长激素能诱导肝脏中IGF-Ⅰ和IGF-Ⅱ的合成[10, 16-17]。用放射免疫分析法分析经重组大麻哈鱼GH投喂或注射后,虹鳟的细胞内GH在较短时间内(12 h)升到最高值,并很快降低至正常水平,而IGF-Ⅰ逐渐升高并维持在较高水平至少96 h[18]。生长激素能提高鱼类IGF-ⅠmRNA表达水平,但生长激素对IGF-Ⅱ基因的转录调控可能在不同的物种具有不同的作用模式,例如,生长激素能使金鲷肝脏IGF-ⅠmRNA表达水平提高2.2倍,但IGF-ⅡmRNA表达水平不受生长激素的影响;而虹鳟IGF-Ⅰ和IGF-Ⅱ表达水平都受生长激素的影响。CAO[19]研究生长激素GH对鱼IGF-Ⅰ表达的调节作用,发现切除垂体后,肝组织IGF-ⅠmRNA丰度下降,而注射GH后IGF-ⅠmRNA水平又得到恢复。后来对GH家族对IGF-Ⅰ表达的调节作用进行的在体和离体试验均证明,GH能显著促进肝组织IGF-ⅠmRNA的表达,且具有剂量依赖性。该研究也证实了重组mcGH对鲮IGF-Ⅰ的表达和分泌都有促进作用。
在银大麻哈鱼(Oncorhynchus kisutch)和罗非鱼(O.mossambicus)中重组GH对肝组织IGF-ⅠmRNA表达有促进作用,但对肝外组织的IGF-ⅠmRNA表达水平没有影响[20-21]。该试验中,重组mcGH不仅促进了肝组织IGF-ⅠmRNA表达,而且促进肝外组织(脑组织)IGF-ⅠmRNA表达。BIGA等[10]利用重组牛生长激素(recombinant bovine somatotropin,rbST)处理虹鳟,发现不仅肝脏组织中IGF-ⅠmRNA水平升高,在鳃、性腺、脑、肠等一些肝外组织IGF-ⅠmRNA水平也有升高。TSE等[22]研究了用不同剂量的猪生长激素处理鲤(Cyprinus carpio)之后脑和肝脏中IGFs的表达情况,结果表明,生长激素能提高鲤脑和肝脏组织IGF-Ⅰ和IGF-ⅡmRNA的表达水平,且具有剂量依赖效应,脑中IGF-ⅡmRNA水平对生长激素反应更灵敏。GH能诱导肝细胞和其它细胞合成IGF-Ⅰ,并且能调节机体内IGF-Ⅰ浓度。PEREZ-SANCHEZ等[23]对虹鳟研究也表明重组GH刺激了肝和肝外组织IGF-ⅠmRNA表达。该试验中经重组mcGH处理,鲮脑组织中大于肝组织中IGF-ⅠmRNA增加量,这可能是器官特异性引起的不同的调节。
该试验中发现, 经外源GH处理,鲮血清中GH浓度变化不显著(表 2)。这可能由于重组mcGH不影响鲮内源GH的分泌。这一观点与LEEDOM等[24]利用重组牛生长激素刺激罗非鱼发现其内源GH变化并不明显的结果一致。
尽管重组mcGH刺激对鲮内源GH影响不大,但鲮内源IGF-Ⅰ显著升高(表 2)。这与对哺乳动物和其它一些脊椎动物中GH和IGF的研究结果一致[5, 25-26]。这也说明了在鲮中,GH对IGF-Ⅰ的分泌起到很重要的作用。
-
表 1 印度洋西北部海域鸢乌贼肌肉样品取样时间、地点、样本尾数以及分析的样本尾数
Table 1 Sampling localities, sampling dates, total numbers and the numbers used for RAPD analysis ofS.oualaniensis in the northwestern Indian Ocean
取样时间
sampling date经度/°N
longitude纬度/°E
latitude尾数/ind
numberRAPD分析尾数/ind
numbers used for RAPD analysis2004-10-11 65.25 12.78 5 0 2004-10-12 63.37 13.45 5 0 2004-10-14 62.55 14.55 5 0 2004-10-15 62.35 16.38 5 4 2004-10-16 62.33 18.93 5 4 2004-10-18 63.00 18.88 5 4 2004-10-21 63.93 18.95 5 4 2004-10-22 63.48 18.47 5 0 2004-10-24 62.83 18.12 5 4 2004-10-25 61.45 17.17 5 0 2004-10-26 61.45 17.72 5 0 2004-10-27 61.50 17.78 10 0 2004-10-31 60.93 16.32 10 4 2004-11-01 60.92 15.57 10 0 2004-11-02 59.42 15.08 10 0 2004-11-06 59.67 13.17 10 4 2004-11-07 60.10 13.33 10 4 2004-11-09 60.92 14.13 10 0 2004-11-10 61.02 14.50 10 4 2004-11-12 60.72 14.42 10 4 2004-11-14 60.78 16.97 10 4 2004-11-15 60.53 16.87 10 0 2004-11-17 60.82 15.87 10 4 2004-11-20 60.45 15.40 10 0 2005-03-27 60.43 13.00 4 0 2005-03-31 60.00 15.00 5 0 2005-04-03 61.05 16.95 6 0 合计 total 200 48 表 2 所用的随机引物及其序列
Table 2 Primers and their sequence used for RAPD analysis
引物
primers序列
sequence引物
primers序列
sequenceR1 5′-ccatcctacc R5 5′-ccatggtgtc R2 5′-acagtaccgcc R6 5′-aaccgcgtcc R3 5′-gatggctgtg R7 5′-ctcaccgtcc R4 5′-ctccccaact R8 5′-acggcgtatg 表 3 印度洋西北部海域鸢乌贼种群多态位点比例与遗传多态性
Table 3 Proportion of polymorphic loci and genetic diversity of S.oualaniensis populations in the northwest Indian Ocean
内容
content18°N~20°N种群
population located in 18°N~20°N13°N~18°N种群
population located in 13°N~18°N多态位点比例/%
proportion of polymorphic loci68.75 93.75 遗传多态性(平均值±标准差)
genetic diversity (Mean±SE)0.2072±0.1928 0.1656±0.1441 -
[1] ZUEV G V, NIGMATULLIN C M, NIKOVLSKY V N. Nektonic oceanic squid[M]. Moscow: Agropromizdat, 1985.
[2] NESIS K N. Population structure of oceanic ommestrephids, with particular reference to Symplectoteuthis oualaniensis: a review[M]//OKUTANI T, O′DOR R K, KU-BODERA T. Recent advances in cephalopod fisheries biology. Tokyo: Tokai University Press, 1993: 375-383. https://www.semanticscholar.org/paper/Population-structure-of-oceanic-Ommastrephids%2C-with-Nesis/510610895740d037d8d13fbc2fe86ea5a60150f8
[3] 谷津明彦. ィンド洋におけゐトビイカの生物学ならびに新资源としての可能性[J]. 远洋, 1997, 101: 6-9. https://cir.nii.ac.jp/crid/1573950400218005376 [4] SNYDER R. Aspects of the biology of the giant form of Sthenoteuthis oualaniensis (Cephalopoda: Ommastrephidae) from the Arabian Sea[J]. J Mollus Stud, 1998, 64(1): 21-34. doi: 10.1093/mollus/64.1.21
[5] 杨德康. 两种鱿鱼资源和其开发利用[J]. 上海水产大学学报, 2002, 11(2): 176-179. doi: 10.3969/j.issn.1004-7271.2002.02.017 [6] 陈新军, 钱卫国, 田思泉. 阿拉伯海北部公海海域鸢乌贼资源密度及其分布[J]. 海洋科学进展, 2006(3): 360-364. doi: 10.3969/j.issn.1671-6647.2006.03.014 [7] 田思泉, 陈新军, 杨晓明. 阿拉伯北部公海海域鸢乌贼渔场分布及其与海洋环境因子关系[J]. 海洋湖沼通报, 2006(1): 51-57. doi: 10.3969/j.issn.1003-6482.2006.01.007 [8] 陈新军, 赵小虎, 刘金立. 印度洋西北海域鸢乌贼钓捕技术试验[J]. 海洋渔业, 2005, 27(3): 200-205. doi: 10.3969/j.issn.1004-2490.2005.03.005 [9] 叶旭昌, 陈新军. 印度洋西北海域鸢乌贼生物学特性初步研究[J]. 上海水产大学学报, 2004, 13(4): 316-322. doi: 10.3969/j.issn.1004-7271.2004.04.006 [10] 恽锐, 钟敏, 王洪新, 等. 北京东灵山辽东栎种群DNA多样性的研究[J]. 植物学报, 1998, 40(2): 69-175. doi: 10.3321/j.issn:1672-9072.1998.02.013 [11] NEI M. Genetic distance between populations[J]. Am Nat, 1972, 106(949): 283-292. doi: 10.1086/282771
[12] 陈新军, 刘金立. 利用形态学方法分析印度洋西北部海域鸢乌贼种群结构[J]. 上海水产大学学报, 2007, 16(2): 174-179. doi: 10.3969/j.issn.1004-7271.2007.02.015 [13] NEI M, LI W H. Mathematical model for studying genetic variation in terms of restriction endonucleases[C]//Proceedings of the National Academy of Science USA, 1979, 75(10): 5 269-5 273. 10.1073/pnas.76.10.5269
[14] WELSH J, MCCELLAND M. Fingerprinting genomes using PCR with arbitrary primers[J]. Nucleic Acids Res, 1990, 18(24): 7 213-7 218. doi: 10.1093/nar/18.24.7213
[15] WELSH J, PETERSON C, MCCELLAND M. Polymorphism generated by arbitrarily primed PCR in the mouse: application to strain identification and genetic mapping[J]. Nucleic Acids Res, 1991, 19(2): 303-306. doi: 10.1093/nar/19.2.303



 下载:
下载:
 粤公网安备 44010502001741号
粤公网安备 44010502001741号
