RAPD analysis of genetic difference among five species of grouper
-
摘要:
运用随机扩增多态性DNA(random amplified polymorphic DNA,RAPD)技术对鞍带石斑鱼(Epinephelus lanceolatus)、驼背鲈(Cromileptes altivelis)、棕点石斑鱼(E.fuscoguttatus)、斜带石斑鱼(E.coioides)和鲑点石斑鱼(E.fario)的遗传多样性及系统发生进行了研究。从120个RAPD随机引物中筛选出扩增效果好的引物19个,分别用于5种石斑鱼基因组DNA的扩增。结果显示,5种石斑鱼平均多态性位点比率(P)分别为64.23%,72.61%,60.34%,69.97%,73.94%;个体间平均遗传相似系数(S)分别为0.8238,0.8110,0.8345,0.8277,0.8064;平均遗传距离(D)分别为0.1762,0.1890,0.1655,0.1723,0.1936;平均Nei基因多样性指数(H)分别为0.1189,0.1364,0.1028,0.1439,0.1648;平均Shannon信息指数(Hi)分别为0.1801,0.1992,0.1530,0.2100,0.2434。5个种间的遗传距离(Dxy)在0.3964~0.6085之间。
Abstract:Genetic diversity and molecular evolution of five species of grouper, viz.Epinephelus lanceolatus, Cromileptes altivelis, E.fuscoguttatus, E.coioides and E.fario, were detected using RAPD technique. A total 120 random primers were evaluated for the usefulness. And thereinto, 19 random primers were selected according to the effect of PCR of genomic DNA of 5 species of grouper. The results showed as the followings: the polymorphic loci ratio in E.lanceolatus, C.altivelis, E.fuscoguttatus, E.coioides and E.fario (P) were 64.23%, 72.61%, 60.34%, 69.97% and 73.94%, respectively. The mean genetic similarity index of intra-species (S) were 0.8238, 0.8110, 0.8345, 0.8277, 0.8064, respectively, the mean genetic distances of intra-species (D) were 0.1762, 0.1890, 0.1655, 0.1723 and 0.1936, respectively, Nei′s gene diversity (H) were 0.1189, 0.1364, 0.1028, 0.1439 and 0.1648, respectively, and Shannon′s information index (Hi) were 0.1801, 0.1992, 0.1530, 0.2100, 0.2434, respectively. The mean genetic distances of inter-species (Dxy) were between 0.3964~0.6085.
-
Keywords:
- RAPD /
- grouper /
- Epinephelus lanceolatus /
- Cromileptes altivelis /
- E.fuscoguttatus /
- E.coioides /
- E.fario
-
鸢乌贼(Sthenoteuthis oualaniensis)隶属于柔鱼科鸢乌贼属,广泛分布在印度洋、太平洋的赤道和亚热带等海域,以印度洋西北部海域的资源量为最大[1]。前苏联和日本学者[2-3]曾多次对印度洋鸢乌贼资源进行调查,同时根据种类的发光器、形态特征、肥满度等初步分为大型、中型和小型3个群体,主要是侧重于对其资源量的研究;SNYDER[4]曾对阿拉伯海鸢乌贼大型群体进行了生物学的初步研究;杨德康[5]根据中国拖网渔船在亚丁湾海域兼捕的鸢乌贼,从捕捞时间和渔获物的性腺成熟度来分析,认为鸢乌贼由春生群、夏生群和秋生群3个群体组成;我国于2003~2005年对印度洋西北部海域鸢乌贼资源进行调查研究,对其资源密度及其分布、钓捕技术、渔场形成机制与海洋环境因子之间的关系等作了较全面的分析,对其生物学特性也作了初步分析[6-9],但是对该海域鸢乌贼的种群及其遗传结构没有作出进一步研究。文章是根据2004~2005年2次对印度洋西北部公海海域(13°N~20°N、59°E~64°E)鸢乌贼资源调查中所采集的鸢乌贼肌肉样本,利用随机扩增多态性DNA(random amplified polymorphic DNA,RAPD)检测方法,对该海域鸢乌贼种群及其遗传结构进行研究,为其资源评估、群体数量变动分析提供最基础的资料。
1. 材料和方法
1.1 材料来源
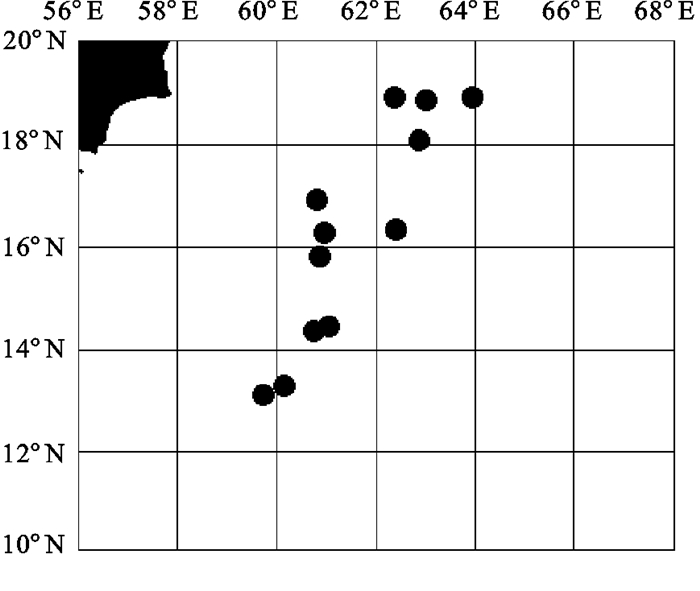
根据2004年9~12月和2005年3~5月2次印度洋西北部公海海域鸢乌贼资源调查结果,在27个站点中共采集鸢乌贼肌肉样本200尾(表 1),其胴长范围为20.3~53.0 cm,平均胴长为36.7 cm。肌肉样本用75%的酒精固定并保存于4℃的冰箱中备用。根据形态学特征及其空间分布,选取了12个站点48尾鸢乌贼的肌肉样本进行RAPD分析(图 1)。
表 1 印度洋西北部海域鸢乌贼肌肉样品取样时间、地点、样本尾数以及分析的样本尾数Table 1. Sampling localities, sampling dates, total numbers and the numbers used for RAPD analysis ofS.oualaniensis in the northwestern Indian Ocean取样时间
sampling date经度/°N
longitude纬度/°E
latitude尾数/ind
numberRAPD分析尾数/ind
numbers used for RAPD analysis2004-10-11 65.25 12.78 5 0 2004-10-12 63.37 13.45 5 0 2004-10-14 62.55 14.55 5 0 2004-10-15 62.35 16.38 5 4 2004-10-16 62.33 18.93 5 4 2004-10-18 63.00 18.88 5 4 2004-10-21 63.93 18.95 5 4 2004-10-22 63.48 18.47 5 0 2004-10-24 62.83 18.12 5 4 2004-10-25 61.45 17.17 5 0 2004-10-26 61.45 17.72 5 0 2004-10-27 61.50 17.78 10 0 2004-10-31 60.93 16.32 10 4 2004-11-01 60.92 15.57 10 0 2004-11-02 59.42 15.08 10 0 2004-11-06 59.67 13.17 10 4 2004-11-07 60.10 13.33 10 4 2004-11-09 60.92 14.13 10 0 2004-11-10 61.02 14.50 10 4 2004-11-12 60.72 14.42 10 4 2004-11-14 60.78 16.97 10 4 2004-11-15 60.53 16.87 10 0 2004-11-17 60.82 15.87 10 4 2004-11-20 60.45 15.40 10 0 2005-03-27 60.43 13.00 4 0 2005-03-31 60.00 15.00 5 0 2005-04-03 61.05 16.95 6 0 合计 total 200 48 1.2 试验方法和数据处理
1.2.1 基因组DNA的提取和检测
取肌肉样本25~30 mg加液氮后碾碎,-70℃保存备用。采用基因组DNA纯化试剂盒(SK1252,Sangon公司生产)提取基因组DNA。用Beckman DU-650紫外分光光度计检测DNA的含量,并用1%的琼脂糖凝胶电泳检测基因组DNA的质量。检测后的基因组DNA放置于-20℃冰箱中备用。
1.2.2 PCR-RAPD扩增反应及电泳
PCR-RAPD所采用的随机引物由上海Sangon公司合成。扩增反应中体积为25 μL,其中包括10×Taq buffer 2.5 μL,dNTPs(Fermentas公司生产,25 mol·L-1)0.5 μL,MgCl2(Fermentas公司生产,25 mmol·L-1)2.5 μL,Taq DNA Polymerase(Fermentas公司生产,5 μ·μL-1)0.2 μL,随机引物(Sangon公司生产,50 μmol·μL-1)0.5 μL,基因组DNA 1μL(50~100 ng·μL-1),ddH2O 17.8 μL。
PCR扩增在GeneAmp PCR System 9700 PCR仪上进行,所有样本对每一个引物都进行1~2次扩增反应。反应条件为经94℃预变性2 min后,接着40个循环,每个循环包括94℃变性15 s,35℃复性60 s,72℃延伸90 s,最后是72℃终延伸10 min,4℃保温。扩增产物用1.5%琼脂糖凝胶电泳分离,EB染色,凝胶成像系统(genius bio imaging system,GENE公司生产)观察、拍照并记录。
1.2.3 数据处理
根据电泳后记录下清晰的扩增条带进行数据统计,在RAPD图谱中相对位置无条带的用“0”表示,在相对位置有条带的用“1”表示,将RAPD图谱转化成0、1矩阵,利用Popgene 1.31软件计算不同站点鸢乌贼样本的遗传相似度(S)和遗传距离(D)。计算公式为:
$$ S=\frac{2 N_\mathtt{x y}}{N_\mathtt{x}+N_\mathtt{y}} ; D=1-S $$ 式中Nxy为X、Y 2个样本共有的扩增条带,Nx、Ny分别为X、Y样本各自拥有的扩增带。
采用PHYLIP(phylogeny inference package,Ver.3.5)软件包中的NEIGHBOR程进行UPGMA(unweighted pair-group method with arithmetic average)聚类分析。参照恽锐等[10]的方法,用Shannon多样性指数计算种群的遗传多样性,其平均值即为种群的遗传多样性。计算公式为:
$$ h=-\sum p_i \log _2 p_i $$ 式中pi为某位点的表型频率,包括有带样本的频率和无带样本的频率,h为该位点的表型多样性,即样本在该位点出现“有带”或“无带”的不确定性。
利用Arlequin 2.0软件进行分子方差分析(analysis of molecular variance,AMOVA),计算其遗传分化指数(GST),GST即为种群间的遗传多样性占种群多样性的比例,以检测鸢乌贼种群内和种群间的遗传变异情况的显著性。计算公式为:
$$ G_{\mathrm{ST}}=\frac{H_{\mathrm{T}}-H_{\mathrm{S}}}{H_{\mathrm{T}}} $$ 式中HT为种群的总遗传多样性,HS为种群内平均遗传多样性。
2. 结果
2.1 RAPD扩增结果
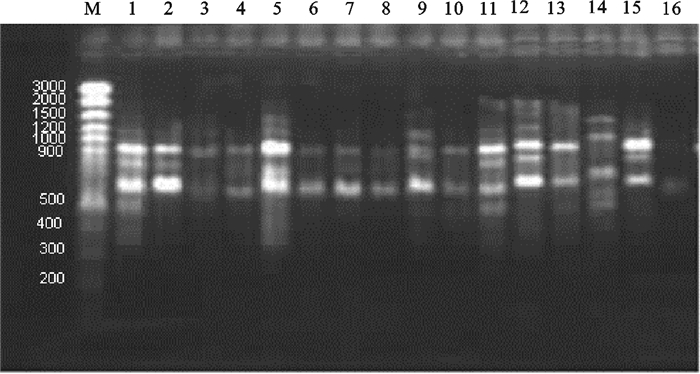
PCR-RAPD试验所使用的16个随机引物中,经过筛选,选取扩增条带丰富且稳定性好的8个引物进行分析,引物序列见表 2。每个引物均可得到条带清晰且重复性好的扩增图谱,扩增条带为3~8,其分子量大小为200~1 500 bp。图 2为引物R8的扩增图谱。
表 2 所用的随机引物及其序列Table 2. Primers and their sequence used for RAPD analysis引物
primers序列
sequence引物
primers序列
sequenceR1 5′-ccatcctacc R5 5′-ccatggtgtc R2 5′-acagtaccgcc R6 5′-aaccgcgtcc R3 5′-gatggctgtg R7 5′-ctcaccgtcc R4 5′-ctccccaact R8 5′-acggcgtatg 2.2 UPGMA分析
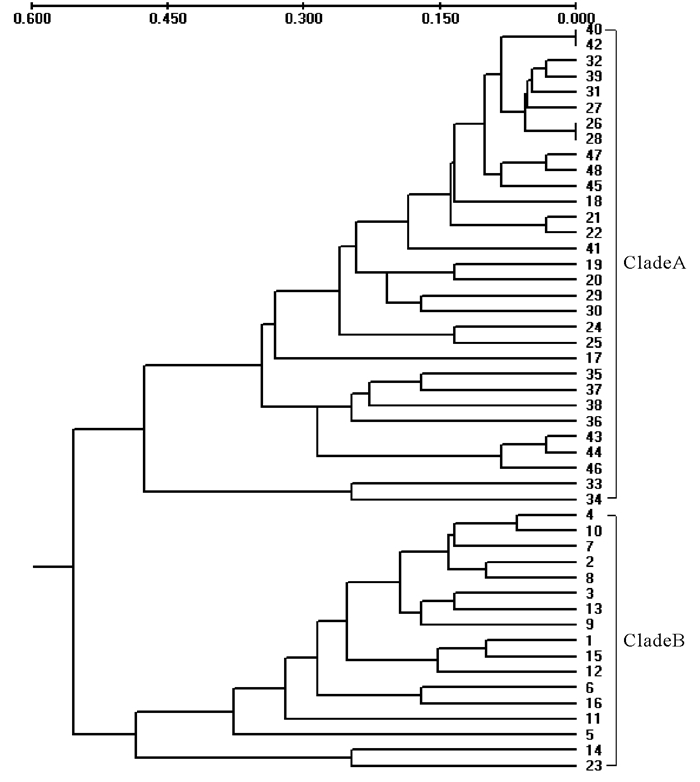
将RAPD图谱转化成0、1矩阵,经过POPGENE 1.31处理,根据NEI[11]的方法可得出各个样本间的遗传相似性指数(S)和遗传距离(D)。根据遗传距离,利用PHYLIP软件包中的NEIGHBOR程序进行UPGMA聚类分析,得出48尾鸢乌贼样本的聚类图(图 3)。
由图 3可知,18°N~20°N海域4个站点的16尾样本聚集在一起,且样本间的最大遗传距离为0.4858,可以推测认为,该海域的鸢乌贼形成一个种群。而在13°N~18°N海域的8个站点的32尾样本中,除第23号样本外,其余样本都聚集在一起,且样本间的最大遗传距离为0.4767,该海域的鸢乌贼也同样形成一个种群。因此,根据UPGMA聚类分析,可以得出在13°N以北的印度洋西北部海域鸢乌贼存在2个不同种群,且2个种群之间的遗传距离为0.1338,遗传相似性指数为0.8748。
2.3 遗传多样性
利用Shannon多样性指数计算印度洋西北部海域鸢乌贼种群的遗传多样性,其平均值即为种群的遗传多样性。计算结果表明,印度洋西北部海域鸢乌贼种群平均每个位点的多样性指数为0.3676±0.1801,由此可以看出其种群的遗传多样性较高,种群分化较大。
2.4 DNA多态性与遗传分化
根据获得的RAPD扩增带,计算种群间的多态位点比例(表 3),2个种群多态位点比例分别为68.75%和93.75%,这说明印度洋西北部海域鸢乌贼2个种群均保持较高的遗传多样性。以种群内不同扩增图谱类型之间的遗传差异值为基础,计算种群的遗传多样性,18°N~20°N海域鸢乌贼种群的遗传多样性为0.2072,13°N~18°N海域鸢乌贼种群为0.1656,其平均值为0.1864。
表 3 印度洋西北部海域鸢乌贼种群多态位点比例与遗传多态性Table 3. Proportion of polymorphic loci and genetic diversity of S.oualaniensis populations in the northwest Indian Ocean内容
content18°N~20°N种群
population located in 18°N~20°N13°N~18°N种群
population located in 13°N~18°N多态位点比例/%
proportion of polymorphic loci68.75 93.75 遗传多态性(平均值±标准差)
genetic diversity (Mean±SE)0.2072±0.1928 0.1656±0.1441 GST是用来判断种群间的遗传分化情况,当GST < 0.05时,种群间没有遗传分化;当0.05 < GST < 0.15时,种群间的遗传分化程度为中等;当0.15 < GST < 0.25时,种群间有高度的分化;当GST>0.25时,种群间的分化程度非常高。印度洋西北部12°N以北海域鸢乌贼种群总遗传多样性为0.2375,种群内平均遗传多样性为0.1864,可以得出其种群间遗传分化指数为0.2150,即21.5%的遗传变异来自于种群间,而78.5%来自于种群内。该结果表明,不同种群间在遗传背景上存在较大的差异,且种群内的遗传变异水平较高。
3. 讨论
3.1 关于印度洋西北部海域鸢乌贼种群结构的探讨
通过对印度洋西北部海域鸢乌贼样本的RAPD分析,并根据遗传距离对其进行UPGMA聚类,发现18°N~20°N海域4个站点的16尾鸢乌贼样本聚集在一起,形成了一个种群,而13°N~18°N海域8个站点的32尾鸢乌贼样本聚集在一起,形成了另一个种群。对这2个不同种群的形态学参数进行统计,18°N~20°N海域鸢乌贼胴长为45.4~53.0 cm,平均胴长为48.9±2.81 cm,而13°N~18°N海域胴长为20.3~51.2 cm,平均胴长为36.3±8.03 cm,优势胴长为32.0~42.0 cm。经单因素方差(ANOVA)分析2个种群间的胴长的P=0.00002 < 0.05,差异性显著。陈新军等[12]认为印度洋西北部海域鸢乌贼分为形态特征存在一定差异性的3个种群:大型种群、中型种群和小型种群,其中大型种群主要分布在18°N以北海域,中型种群主要分布在12°N~18°N海域,小型种群主要分布在12°N以南及赤道附近海域,且这3个种群重叠分布;谷津明彦[3]也认为该海域的鸢乌贼存在3个不同体型的种群,此文所得出的种群结构与陈新军、谷津明彦等研究的结果基本一致。因此,印度洋西北部13°以北海域鸢乌贼种群在形态学与遗传上都可以被区分为18°N~20°N、13°N~18°N 2个不同的种群。
3.2 印度洋西北部海域鸢乌贼的遗传多样性
Shannon多样性指数表示种群间的多样性占总多样性的比例,可以用来估测遗传多样性在种群内和种群间的分布,即估测种群的遗传分化程度。利用Shannon多样性指数计算出的印度洋西北部海域鸢乌贼的遗传多样性指数为0.3676±0.1801,为较高的水平。由于其遗传多样性水平较高,种群分化较大,从侧面可以说明印度洋西北部海域鸢乌贼2个种群在形态上差别很大的原因。
另外,此研究结果还揭示,与18°N~20°N海域鸢乌贼种群相比,13°N~18°N海域鸢乌贼种群拥有较高的多态性位点比例,而遗传多态性却相对较低(表 3),这一结果可能与所选用8条RAPD引物有关;笔者因此推测出13°N~18°N海域鸢乌贼可能所受的捕捞压力相对较大,生长速度较快。基于此研究的分析结果,该海域鸢乌贼2个种群间在遗传背景上存在较大的差异,且种群内的遗传变异水平较高,笔者认为,对该海域鸢乌贼资源的规模性开发还处于较合理水平。
3.3 RAPD结果的分析方法
种以下类群包括亚种、品种及地理种群等,遗传分析的目的在于了解遗传多样性、鉴别种群、分析种群间的差异大小和微进化等,多数学者采用2种方法对RAPD结果进行处理并对上述问题进行探讨[13-15]:(1)寻找种群的特有遗传标记,据此可以鉴别不同的种群;(2)基于遗传相似率的分析,包括相似率比较、遗传距离分析、聚类分析等。此研究在进行RAPD实验过程中未能寻找到用于区分印度洋西北部海域鸢乌贼2个种群的RAPD分子标记,这可能是由于在此次实验中使用引物较少的原因所造成的。因而,此研究选用了第2种分析方法。
RAPD技术能够快速、简便地检测大量基因组DNA的遗传变异,只要采用适当的分析方法,不仅可以用于鉴定头足类资源的品系、种群结构并探讨其进化关系,还可以在探讨头足类种群分化等方面发挥重要作用。
-
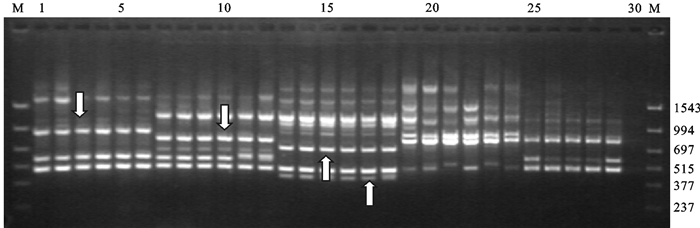
图 3 引物S61在5种石斑鱼中的RAPD带谱电泳图
M. PCR markers;1~6. 鞍带石斑鱼的6个个体;7~12. 驼背鲈的6个个体;13~18. 棕点石斑鱼的6个个体;19~24. 斜带石斑鱼的6个个体;25~30. 鲑点石斑鱼的6个个体;箭头所指谱带分别为鞍带石斑鱼、驼背鲈和棕点石斑鱼(2个)的特异性谱带,从左到右依次为966,867,697,434 bp
Figure 3. RAPD bands generated by primer S61 in five species of grouper
M. molecular size marker (DL1543);1~6. E.lanceolatus; 7~12. C.altivelis; 13~18. E.fuscoguttatus; 19~24. E.coioides; 25~30. E.fario; Arrow species-specific markers in E.lanceolatus, C.altivelis and E.fuscoguttatus (two), respectively, molecular weight from left to right, 966, 867, 697, 434 bp.
表 1 筛选出的19个引物的序列
Table 1 The sequences of selected 19 primers
引物 primer 序列(5′~3′) sequence S61 TTCGAGCCAG S70 TGTCTGGGTG S121 ACGGATCCTG S287 AGAGCCGTCT S321 TCTGTGCCAC S325 GTGCCGTTCA S326 GTGCCGTTCA S327 CCAGGAGGAC S340 ACTTTGGCGG S462 TCGGCACGCA S464 GTGTCTCAGG S468 ACATCGCCCA S469 GTGGTCCGCA S471 AACGCGTCGG S474 CCAGCCGAAC S475 GGAAGCCAAC S477 TGACCCGCCT S478 GGCTTGGCCT S479 GGGAAGGACA 表 2 5种石斑鱼的分子遗传分析
Table 2 Molecular genetic analysis among 5 species of grouper
鞍带石斑鱼
E.lanceolatus驼背鲈
C.altivelis棕点石斑鱼
E.fuscoguttatus斜带石斑鱼
E.coioides鲑点石斑鱼
E.fario多态性位点比率(P)/%
polymorphic loci64.23 72.61 60.34 69.97 73.94 平均遗传相似系数(S)
mean genetic similarity index0.8238 0.8110 0.8345 0.8277 0.8064 平均遗传距离(D)
mean genetic distances0.1762 0.1890 0.1655 0.1723 0.1936 平均Nei基因多样性指数(H)
mean Nei′s gene diversity0.1189 0.1364 0.1028 0.1439 0.1648 平均Shannon信息指数(Hi)
Shannon′s information index0.1801 0.1992 0.1530 0.2100, 0.2434 表 3 5种石斑鱼的Nei′s遗传相似系数和遗传距离值
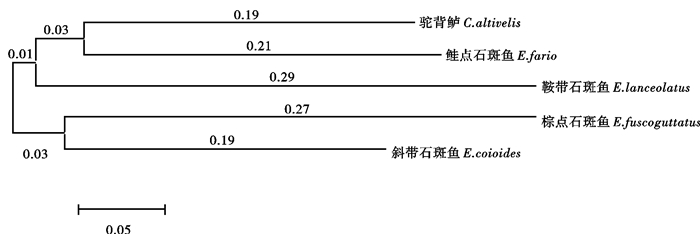
Table 3 Nei′s genetic identity and genetic distance among 5 species of grouper
鞍带石斑鱼
E.lanceolatus驼背鲈
C.altivelis棕点石斑鱼
E.fuscoguttatus斜带石斑鱼
E.coioides鲑点石斑鱼
E.fario鞍带石斑鱼 - 0.6184 0.5577 0.6188 0.5973 驼背鲈 0.4980 - 0.5930 0.6537 0.6781 棕点石斑鱼 0.6085 0.5517 - 0.6433 0.6045 斜带石斑鱼 0.5103 0.4446 0.4565 - 0.6439 鲑点石斑鱼 0.5315 0.3964 0.5228 0.4691 - 注:对角线以上为Nei′s遗传相似系数,对角线以下为遗传距离
Note:Nei′s genetic identity (above diagonal) and genetic distance (below diagonal). -
[1] 黄原. 分子系统学——原理、方法及应用[M]. 北京: 中国农业出版社, 1998: 186-193. https://book.douban.com/subject/4773037/ [2] LYNCH M. The similarity index and DNA fingerprinting[J]. Mol Biol Evol, 1990(7): 478-484. doi: 10.1093/oxfordjournals.molbev.a040620
[3] ARMSTRON G J S, GIBBS A J, PEAKALL R, et al. The RAPDistance Package[P/OL]. [1994]. http://www.anu.edu.au/BoZo/software/.
[4] NEI M, LI W H. Mathematical model for studying genetic variation in terms of restriction endonucleases[J]. Proc Nat Acad Sci USA, 1979, 76(10): 5 269-5 273. doi: 10.1073/pnas.76.10.5269
[5] NEI M. Estimation of average heterozygosity and genetic distance from a small number of individuals[J]. Genetics, 1978, 89: 583-590. doi: 10.1093/genetics/89.3.583
[6] CHALMERS K J, WAUGH R, SPRENT J I, et al. Detection of genetic variation between and within populations of Gliricidia sepium and G. maculata using RAPD markers[J]. Heredity, 1992, 69: 465-472. doi: 10.1038/hdy.1992.151
[7] NEI M. Genetic distance between populations[J]. Am Nat, 1972, 106: 283-292. doi: 10.1086/282771
[8] 郑莲, 刘楚吾. 4种石斑鱼亲缘关系的RAPD分析[J]. 水产科学, 2004, 23(11): 16-20. doi: 10.3969/j.issn.1003-1111.2004.11.006 [9] 尹绍武, 黄海, 廖经球, 等. 四种石斑鱼的分子遗传多样性和亲缘关系的RAPD分析[J]. 海洋学报, 2006, 28(6): 119-126. http://qikan.cqvip.com/Qikan/Article/Detail?id=23511686 [10] GOVINDARAJU G S, JAYASANKAR P. Taxonomic relationship among seven species of grouper (Genus Epinephelus; Family Serranidae) as revealed by RAPD fingerprinting[J]. Mar Bio, 2004, 6(3): 229-237. doi: 10.1007/s10126-003-0021-9
[11] 刘楚吾, 刘丽, 王中铎, 等. 军曹鱼的分子遗传特性研究[J]. 热带海洋学报, 2005, 24(1): 77-85. doi: 10.3969/j.issn.1009-5470.2005.01.011 [12] 丁少雄, 苏永全, 王军, 等. 闽粤沿海 
[13] 中国科学院动物研究所, 中国科学院海洋研究所, 上海水产学院. 南海鱼类志[M]. 北京: 科学出版社, 1962: 292-308. https://book.douban.com/subject/10512105/ [14] 孟庆闻, 苏锦祥, 缪学祖, 等. 鱼类分类学[M]. 北京: 中国农业出版社, 1995: 606-610;620-622. https://book.douban.com/subject/1059981/ [15] 成庆泰, 郑葆珊. 中国鱼类系统检索(上、下册)[M]. 北京: 科学出版社: 287-293;1 035-1 047. https://book.douban.com/subject/6872030/ [16] 夏德全, 曹莹, 吴婷婷, 等. 用RAPD方法分析太湖大银鱼、太湖新银鱼和寡齿新银鱼的亲缘关系[J]. 中国水产科学, 2000, 7(1): 12-15. doi: 10.3321/j.issn:1005-8737.2000.01.003 [17] 张四明, 邓怀, 汪登强, 等. 7种鲟形目鱼类亲缘关系的随机扩增多态性DNA研究[J]. 自然科学进展, 1999, 9(9): 818-823. doi: 10.1088/0256-307X/15/12/024 [18] 邹曙明, 楼允东. 用RAPD方法研究草鱼、柏氏鲤和3个地理种群鲤的亲缘关系[J]. 中国水产科学, 2000, 7(1): 6-11. doi: 10.3321/j.issn:1005-8737.2000.01.002



 下载:
下载:

 粤公网安备 44010502001741号
粤公网安备 44010502001741号
