Effect of lipid removal on carbon and nitrogen isotopes of Sthenoeuthis oualaniensis from South China Sea
-
摘要: 碳 (C)、氮(N)稳定同位素分析目前已成为研究水生生物摄食生态和洄游等方面的重要手段。作为主要分析材料的肌肉组织,常因含有碳稳定同位素比值 (δ13C)较低的脂类而对稳定同位素分析造成影响。为了探讨脂类去除对鸢乌贼(Sthenoeuthis oualaniensis)肌肉样品δ13C和氮稳定同位素比值 (δ15N)分析的影响,对2017年4月在南海采集的鸢乌贼样品进行了稳定同位素研究,并对不同的脂类校正模型进行对比以优选出最适用于鸢乌贼的δ13C校正模型。结果显示,脂类去除后鸢乌贼的δ13C和δ15N均发生了显著变化(P<0.01),分别平均升高0.85‰和0.71‰。脂类去除对鸢乌贼δ13C的影响与个体大小和性别无关(P>0.05)。根据最小赤池信息准则(AIC),模型3最适用于鸢乌贼δ13C校正。研究结果表明了脂类去除在鸢乌贼肌肉δ13C分析中的必要性,但δ15N同样受到了影响,今后进行δ15N分析时不需进行脂类去除。Abstract: The stable isotopes of carbon and nitrogen are important in evaluating feeding ecology and migration of organisms. Muscle tissues are commonly used as analytical samples, but lipids in muscles with low δ13C values can affect the stable isotopes analysis. To understand the effect of lipid removal on carbon and nitrogen stables isotopes in Sthenoeuthis oualaniensis muscle samples, we analyzed the stable isotopes of S.oualaniensis captured in the South China Sea in April, 2017. In addition, we compared different lipid removal models to get the best fitted model to revise δ13C values in S.oualaniensis muscles. The results show that after lipid removal, both δ13C and δ15N values changed significantly (P<0.01), increasing by 0.85‰ and 0.71‰, respectively. The lipid removal of δ13C values in S.oualaniensis muscles did not change with size and sex (P>0.05). Besides, Model 3 had the lowest Akaike information criterion value (AIC), indicating that it is the best fitted model for revising δ13C values. The results reveal that lipid removal is necessary in analyzing δ13C values in S.oualaniensis muscles, but δ15N values are affected when lipids are removed, so lipid removal is unnessary in δ15N values analysis.
-
Keywords:
- stable isotope /
- Sthenoeuthis oualaniensis /
- lipid removal /
- South China Sea
-
20世纪80年代初起,碳(C)、氮(N)稳定同位素技术逐渐被应用到生态系统研究中,该技术的兴起和发展对生态学领域的研究起着重要的作用。研究者可根据消费者与其潜在食物之间碳稳定同位素比值(δ13C)相近(差值为0~0.1%)的原理来判断该消费者的食物来源[1],δ13C的变化也可以反映该消费者栖息地的变化及洄游情况[2-3]。另外,氮稳定同位素比值(δ15N)在食物网营养级传递过程中会产生富集,故可以通过δ15N确定被研究物种在食物网中的营养位置[4-7]。头足类在海洋生态系统中扮演着捕食者和被捕食者的双重角色,学者对其营养动力学、洄游及种群结构等方面的研究非常关注。随着稳定同位素技术的推广,该方法已经成为研究头足类洄游、栖息地、食性转换和营养生态学等的重要手段。鸢乌贼(Sthenoeuthis oualaniensis)广泛分布于太平洋、印度洋的热带和亚热带海域[8],以中国南海和印度洋西北部海域的数量较大[9]。近年来近海渔业的枯竭推动了外海渔业的发展,资源量巨大的鸢乌贼具有广阔的开发前景。目前已有的鸢乌贼研究主要集中在种群结构[10-12]、资源评估[13-15]以及渔业生物学[16-17]等方面;而稳定同位素技术在鸢乌贼生态学方面的应用相对较少,国内仅张宇美[18]对其摄食生态学和营养级进行了初步研究。
在稳定同位素分析研究中,肌肉组织因取样简单、易于处理、能够反映近期的摄食状况[19]等优点而常被作为分析样品。但肌肉组织中因常含有δ13C较低的脂类,造成脂类与肌肉组织相比具有更低的δ13C,因而可能造成样品的δ13C分析出现偏差[1,20]。鉴于此,研究者们在脂类去除对肌肉样品δ13C和δ15N的影响方面进行了大量研究,结果表明脂类去除对不同种类动物个体的影响并不一致[19,21-24]。为此,本研究通过比较脂类去除前后南海鸢乌贼肌肉的δ13C、δ15N及碳氮比值[w(C)∶w(N)] 的差异性,确定脂类去除对δ13C和δ15N的影响,并分析它们之间的关系;根据结果利用已有的模型优选出最适用于鸢乌贼δ13C的脂类校正模型,为碳、氮稳定同位素技术今后应用于鸢乌贼的研究提供参考资料和理论依据。
1. 材料与方法
1.1 样品的采集与处理
鸢乌贼样品于2017年4月由“桂北渔68209”和“粤电渔42212”灯光罩网渔船在南海(110°E~117°E,10°N~19°N)采集,样品急速冷冻后带回实验室做进一步处理。样品在实验室解冻后首先进行生物学测定,测定指标包括胴长(ML)、性别等。随机选取75尾鸢乌贼样品,取其背部肌肉组织,去除表面杂质并剪碎,将每份样品分成两份。取其中一份样品剪碎后置于离心管中加入5 mL混合液 [V(三氯甲烷)∶V(甲醇)=2∶1],300 g离心5 min,离心结束后去除上清液,以上步骤重复3次[25-26],然后用蒸馏水清洗干净后将样品置于60 ℃烘箱至少48 h至恒质量。另一份样品直接烘至恒质量。最后将上述样品研磨成粉末后留待测定。
1.2 稳定同位素与元素含量测定
样品的碳、氮稳定同位素比值及C和N含量测定在中国农业科学院农业环境稳定同位素实验室进行。所用仪器为Vario PYRO Cube型元素分析仪和Isoprime-100型稳定同位素比值分析仪,得出待测样品脂类去除前的δ13C、δ15N和C、N含量并计算出C∶N值 [w(C)∶w(N)],以及脂类去除后的δ13C'、δ15N'和C、N含量并计算出C'∶N'值。另外,分别以Δδ13C、Δδ15N表示δ13C'与δ13C、δ15N'与δ15N的差值。稳定同位素比值δ以如下公式表示:
$$ \quad\quad\quad\quad\quad\delta_x = [\left( {{R_{{\rm{samples}}}}/{R_{{\rm{standard}}}}} \right) - 1] \times 1\;000 $$ (1) 式中x表示13C或者15N,R表示13C/12C或者15N/14N的比值。碳、氮稳定同位素标准样品分别采用国际通用的标准物质VPDB和大气中的氮气。为保证实验结果的准确性和仪器的稳定性,每测试12个样品穿插1个标样进行校正。样品δ13C和δ15N分析精度均为±0.08‰。
1.3 数据统计分析
本文利用R语言对脂类去除前后样品的δ13C、δ15N和C∶N值分别进行t检验(α=0.05)和回归分析,并比较脂类去除前后δ13C、δ15N和C∶N值的差异性和相关性。
1.4 模型优选
已有的研究表明,根据C∶N值可估算脂类的含量,而脂类含量可估算Δδ13C。故C∶N值可用于脂类δ13C的标准化,并对肌肉样品的δ13C进行校正[27]。而且对于水生生物来说,由于C∶N值和Δδ13C之间具有显著的相关关系,因此C∶N值和Δδ13C常用于校正模型的构建[27-28]。为此,本文根据上述化学脂类去除法得到的结果,并利用如下已有的脂类校正模型以优选出最适用于鸢乌贼的δ13C校正模型。
$$ \quad\quad\quad\quad\quad\quad \text{模型}\;1 \text{:} y = a{x_1} + {b^{[28]}} $$ (2) $$ \quad\quad\quad\quad\quad\quad \text{模型}\;2\text{:} y = a\ln {x_1} + {b^{[29]}} $$ (3) 式中y表示Δδ13C值,x1表示C∶N值。由于不同脂类含量的乌贼个体时常具有相似的C∶N值,根据C∶N值可能并不能准确估算出乌贼的脂类含量[30],故本文根据贡艺等[31]对北太平洋柔鱼 (Ommastrephes bartramii)的研究结果选取了如下模型:
$$ \quad\quad\quad\quad\quad\quad \text{模型}\;3\text{:} y = a{x_2} + {b^{[31]}} $$ (4) 式中y表示Δδ13C值,x2表示δ13C。为了比较不同校正模型对鸢乌贼肌肉δ13C校正的适用性,利用R语言计算3个模型的赤池信息准则(Akaike Information Criterion, AIC)值,并根据赤池信息准则,选定AIC值最小的模型为最佳模型。
2. 结果
2.1 脂类去除前后δ13C、δ15N及C∶N值变化分析
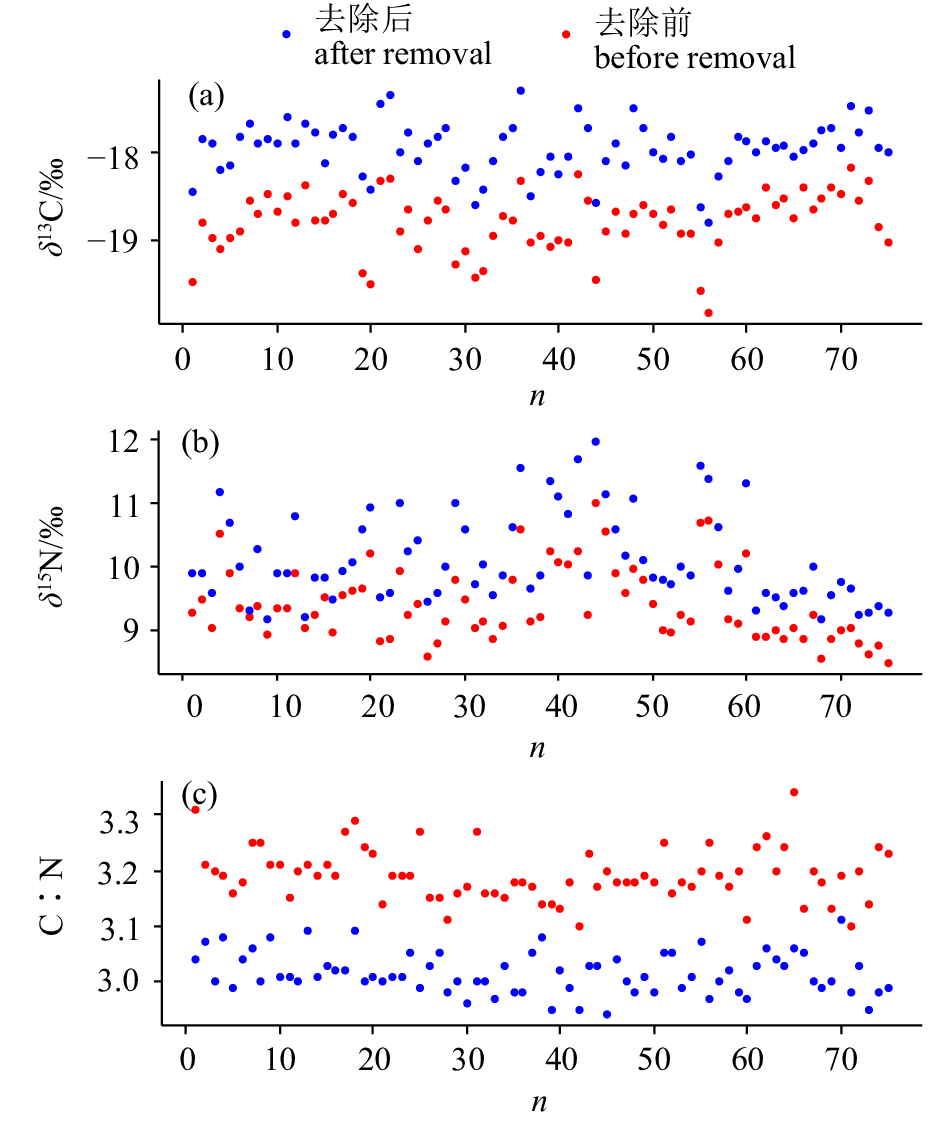
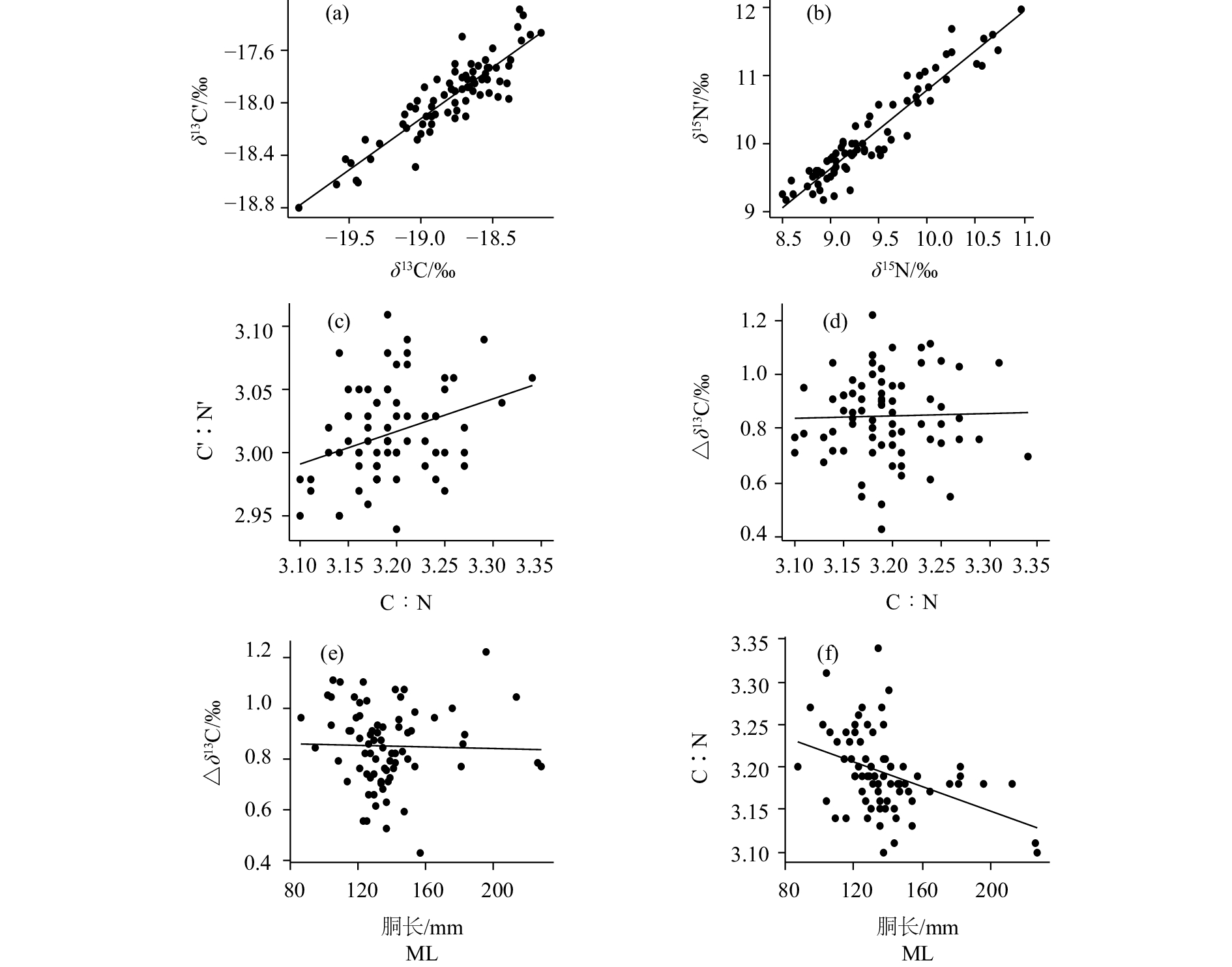
脂类去除前后δ13C发生了较大变化(图1-a)。脂类去除前的δ13C和去除后的δ13C'的变化范围分别为–19.84‰~ –18.17‰和 –18.79‰~ –17.28‰,δ13C'与δ13C相比,升高0.43‰~1.22‰,平均升高0.85‰。t检验结果表明,δ13C'显著高于δ13C (P<0.01)。线性回归分析表明,δ13C与δ13C'存在显著的正相关关系(δ13C'=0.788 1 δ13C –3.136 2,R2=0.796 8,P<0.01,图2-a)。
脂类去除前的δ15N和去除后的δ15N'的变化范围分别为8.50‰~10.98‰和9.16‰~11.17‰,δ15N'比δ15N升高了0.11‰~1.44‰,平均升高0.71‰ (图1-b)。δ15N'显著高于δ15N (P<0.01),且脂类去除前后的δ15N有显著的正相关关系(δ15N'=1.153 5 δ15N –0.738 8,R2=0.895 7,P<0.01,图2-b)。
脂类去除前的C∶N值和去除后的C'∶N'值的变化范围分别为3.10~3.34和2.94~3.11(图1-c),C'∶N'值比C∶N值低0.06~0.28,平均降低0.18,且两者存在正相关关系(图2-c)。另外C∶N值与Δδ13C没有显著相关关系(图2-d)。
选取的75尾鸢乌贼样品雌雄比例接近2∶3,ML变化范围为87~228 mm,Δδ13C和C∶N值与ML的相关关系均不显著(图2-e,图2-f)。t检验结果表明,在雌雄个体间,Δδ13C、Δδ15N和C∶N值均没有显著性差异(P>0.1)。
2.2 模型校正
根据表1,本文选取的3个脂类校正模型经本实验测得的δ13C和C∶N值拟合,得出函数:
表 1 拟合函数的AIC值Table 1. AIC value of fitted function编号
No.模型
model拟合函数
fitted function最小赤池信息准则 AIC 模型1 Model 1 y=ax1+b y=0.073x1+0.613 – 61.881 模型2 Model 2 y=alnx1+b y=0.245lnx1+0.562 – 61.884 模型3 Model 3 y=ax2+b y= – 0.212x2 –3.136 – 80.565 模型1:y=0.073x1+0.613 (R=0.02,P=0.85,n=75)
模型2:y=0.245lnx1+0.562 (R=0.02,P=0.84,n=75)
模型3:y= – 0.212x2–3.136 (R=0.47,P<0.01,n=75)
其中模型3的AIC值最小,模型1和模型2的AIC值相近,赤池信息准则表明,模型3的拟合效果最好。
3. 讨论
3.1 脂类去除对δ13C和δ15N的影响
本实验结果表明,脂类去除后鸢乌贼肌肉组织δ13C显著升高。Ruiz-Cooley等[30]通过脂类去除对茎柔鱼(Dosidicus gigas) δ13C影响的研究发现,脂类去除后δ13C升高0.8‰,并认为升高的范围可能与不同物种、个体和组织的脂类含量有关。本研究采用的样本为鸢乌贼背部肌肉组织,不存在不同组织或者物种带来的差异,由此可推测,鸢乌贼δ13C升高的可能原因是肌肉组织脂类含量较高。脂类合成过程中在丙酮酸转化成乙酰辅酶A期间发生的同位素动力学效应,常常造成脂类与肌肉组织相比具有更低的δ13C[20]。Kiljunen等[27]研究发现,14种鱼类的δ13C尽管在种间和空间水平上存在差异,但是脂类去除后的δ13C均高于去除前;Mateo等[32]研究显示,双壳纲和腹足纲动物肌肉脂类去除后δ13C显著升高;这些研究均表明了脂类去除在进行稳定同位素研究前的必要性。本实验中鸢乌贼肌肉组织的δ13C在脂类去除前后的变化接近1‰,Deniro和Epstein[1]的研究表明,消费者与其潜在食物之间δ13C相近,差值在0~1‰之间。若不进行脂类的去除,在分析鸢乌贼食物来源时结果将产生一定偏差,故在利用碳稳定同位素研究鸢乌贼生态学时需要进行脂类去除。但也有一些研究结果显示,脂类去除后δ13C变化不大。如Pinnegar和Polunin[23]对鱼肌肉稳定同位素的研究结果表明,由于鱼类脂肪含量较低,脂类去除前后δ13C和δ15N差异不明显。由于C∶N值与脂类含量间存在着一定的正相关关系[25],Post等[28]在总结了前人对热带河流、温带湖泊、北极以及近海的水生动物研究结果后认为,脂类质量分数<5%或C∶N<3.5时不需要进行脂类的去除。本实验结果显示,采自外海的鸢乌贼肌肉组织C∶N值虽低于3.5,但脂类去除后的δ13C仍显著升高。这与贡艺等[31]对同样来自外海的北太平洋柔鱼的研究结果一致,而且他们发现柔鱼的粗脂肪质量分数为5.30%。Ruiz-Cooley等[30]研究发现,虽然脂类去除后茎柔鱼δ13C升高范围较大(>0.65‰),但是C∶N值的变化却很小,并提出C∶N值可能不适合作为衡量乌贼脂类含量的标准,因为脂类含量存在差异的组织仍然可能含有相近的C∶N值。
雌、雄个体间的Δδ13C无显著差异,说明性别差异对脂类的去除没有显著影响。Ruiz-Cooley等[30]对茎柔鱼和圆鳍枪乌贼(Lolliguncula brevis)以及贡艺等[31]对北太平洋柔鱼的研究都得出类似的结论。另外,通过回归分析发现,鸢乌贼个体胴长与Δδ13C和C∶N值没有显著的相关关系,说明Δδ13C和C∶N值的变化与个体大小无关。这与Vinagre等[33]对鱼类和头足类的研究结果一致,并且Vinagre 等[33]认为个体大小、体质量、新陈代谢以及其他生理现象都会混淆δ13C与个体大小的关系。
脂类去除除了导致鸢乌贼肌肉组织δ13C显著升高外,δ15N也显著升高,这与Sotiropoulos等[34]、Pinnegar和Polunin[23]、Murry等[35]对鱼类以及Ruiz-Cooley等[30]对茎柔鱼的研究结果一致。Sweeting等[36]认为,δ15N发生变化可能与脂类去除过程中同时将肌肉组织中的某些相关的蛋白质去除有关。Peterson和Fry[4]的研究表明,δ15N随营养位置的递增逐级增加3‰~4‰;本实验中鸢乌贼肌肉组织在脂类去除后δ15N升高了0.11‰~1.44‰。若使用该值计算鸢乌贼的营养位置,得到的结果将会偏高,因而要尽量避免使用脂类去除后的δ15N分析鸢乌贼的营养位置。
3.2 模型优选
本研究选取了3种常见的修正模型对脂类去除前后13C稳定同位素值进行回归分析,结果表明,模型1和模型2拟合度较低。但Post等[28]研究表明,模型1对选取的16种水生生物的Δδ13C和C∶N值拟合度较好。本研究结果显示,鸢乌贼的C∶N值与Δδ13C并不显著相关,由于模型1和模型2的建立主要基于C∶N值和Δδ13C之间具有显著的相关关系[28-29],因而模型1和模型2不适用于鸢乌贼脂类去除的校正模型。模型3主要基于贡艺等[31]的研究结果,他们认为北太平洋柔鱼的C∶N值与其粗脂肪含量相关关系较差,不适合以C∶N值作为脂类的指示物与Δδ13C建立函数关系,而δ13C与Δδ13C的线性模型可能更适用于头足类。本研究中该模型与其他模型相比,拟合度较高,最适合作为鸢乌贼脂类去除的校正模型。而且只要测出未经脂类去除的鸢乌贼样品的δ13C,就可通过模型3得到脂类去除后的δ13C,从而达到节约时间、成本和简化实验操作的效果。
相比化学脂类去除方法,模型校正法摒除了化学方法繁琐的操作过程,但模型的建立需要大量的数据支持。而且从上述分析可知,相同模型的适用范围也有限,所以今后的研究需要对不同区域、不同物种样品进行脂类去除分析,以优选出适用的模型。
4. 结论
脂类是肌肉组织δ13C分析过程中重要的误差来源。本研究发现,脂类去除前后鸢乌贼的δ13C发生了显著变化,表明脂类去除在鸢乌贼肌肉δ13C分析中的必要性。δ15N同样发生了显著的增加,该值的增加在一定程度上会影响鸢乌贼营养位置的估算。因此,消除该影响的最好方法是将样品的碳和氮稳定同位素分开测定,以减少脂类去除对δ15N分析的干扰。通过未经脂类去除的样品δ13C的测定,结合适当的脂类校正模型,可以在节约时间和成本的前提下得到脂类去除后的δ13C。今后的研究可以进一步寻求更优化的校正模型,应用到鸢乌贼的稳定同位素研究中。
致谢:对中国水产科学研究院南海水产研究所钟智辉在实验样品获取过程中给予的帮助,柯兰香、陈盟基和王守信等在样品前处理和实验过程中的协助,谨致谢忱!
-
表 1 拟合函数的AIC值
Table 1 AIC value of fitted function
编号
No.模型
model拟合函数
fitted function最小赤池信息准则 AIC 模型1 Model 1 y=ax1+b y=0.073x1+0.613 – 61.881 模型2 Model 2 y=alnx1+b y=0.245lnx1+0.562 – 61.884 模型3 Model 3 y=ax2+b y= – 0.212x2 –3.136 – 80.565 -
[1] DENIRO M J, EPSTEIN S. Mechanism of carbon isotope fractionation associated with lipid synthesis[J]. Science, 1977, 197(4300): 261-263.
[2] FRY B. Stable isotope ecology[M]. New York: Springer, 2006: 69-72.
[3] KATO Y, SAKAI M, NISHIKAWA H, et al. Stable isotope analysis of the gladius to investigate migration and trophic patterns of the Neon flying squid (Ommastrephes bartramii)[J]. Fish Res, 2016, 173(2, SI): 169-174.
[4] PETERSON B J, FRY B. Stable isotopes in ecosystem studies[J]. Ann Rev Ecol Syst, 1987, 18(1): 293-320.
[5] POST D M. Using stable isotopes to estimate trophic position: models, methods, and assumptions[J]. Ecology, 2002, 83(3): 703-718.
[6] RUBENSTEIN D R, HOBSON K A. From birds to butterflies: animal movement patterns and stable isotopes[J]. Trends Ecol Evol, 2004, 19(5): 256-263.
[7] TAKAI N, HIROSE N, OSAWA T, et al. Carbon source and trophic position of pelagic fish in coastal waters of south-eastern Izu Peninsula, Japan, identified by stable isotope analysis[J]. Fish Sci, 2007, 73(3): 593-608.
[8] 董正之. 世界大洋经济头足类生物学[M]. 济南: 山东科学技术出版社, 1991: 91-94. [9] 王尧耕, 陈新军. 世界大洋性经济柔鱼类资源及其渔业[M]. 北京: 海洋出版社, 2005: 284-296. [10] 陈新军, 钱卫国. 印度洋西北部海域鸢乌贼资源密度分布的初步分析[J]. 上海水产大学学报, 2004, 13(3): 218-223. [11] 陈新军, 刘金立. 利用形态学方法分析印度洋西北部海域鸢乌贼种群结构[J]. 上海水产大学学报, 2007, 16(2): 174-179. [12] 张鹏, 晏磊, 杨炳忠, 等. 春季南沙海域鸢乌贼种群结构特征的研究[J]. 南方水产科学, 2015, 11(5): 11-19. [13] 张鹏, 杨吝, 张旭丰, 等. 南海金枪鱼和鸢乌贼资源开发现状及前景[J]. 南方水产, 2010, 6(1): 68-74. [14] 杨权, 李永振, 张鹏, 等. 基于灯光罩网法的南海鸢乌贼声学评估技术研究[J]. 水产学报, 2013, 37(7): 1032-1039. [15] 张俊, 陈国宝, 张鹏, 等. 基于渔业声学和灯光罩网的南海中南部鸢乌贼资源评估[J]. 中国水产科学, 2014, 21(4): 822-831. [16] 刘必林, 陈新军, 钟俊生. 采用耳石研究印度洋西北海域鸢乌贼的年龄、生长和种群结构[J]. 大连水产学院学报, 2009, 24(3): 206-212. [17] 颜云榕, 冯波, 卢伙胜, 等. 南沙群岛北部海域鸢乌贼(Sthenoteuthis oualaniensis)夏季渔业生物学研究[J]. 海洋与湖沼, 2012, 43(6): 1177-1186. [18] 张宇美. 基于碳氮稳定同位素的南海鸢乌贼摄食生态与营养级研究[D]. 湛江: 广东海洋大学, 2014: 24-31. [19] HOBSON K A, CLARK R G. Turnover of 13C in cellular and plasma fractions of blood: implications for nondestructive sampling in avian dietary studies[J]. Auk, 1993, 110(3): 638-641.
[20] FOCKEN U, BECKER K. Metabolic fractionation of stable carbon isotopes: implications of different proximate compositions for studies of the aquatic food webs using δ13C data[J]. Oecologia, 1998, 115(3): 337-343.
[21] MCCONNAUGHEY T, MCROY C P. Food-web structure and the fractionation of carbon isotopes in the Bering Sea[J]. Mar Biol, 1979, 53(3): 257-262.
[22] KLING G W, FRY B, O'BRIEN W J. Stable isotopes and planktonic trophic structure in arctic lakes[J]. Ecology, 1992, 73(2): 561-566.
[23] PINNEGAR J K, POLUNIN N V. Differential fractionation of δ13C and δ15N among fish tissues: implications for the study of trophic interactions[J]. Funct Ecol, 1999, 13(2): 225-231.
[24] CHALONER D T, MARTIN K M, WIPFLI M S, et al. Marine carbon and nitrogen in southeastern Alaska stream food webs: evidence from artificial and natural streams[J]. Can J Fish Aquat Sci, 2002, 59(8): 1257-1265.
[25] FOLCH J, LEES M, STANLEY G H S. A simple method for the isolation and purification of total lipids from animal tissues[J]. J Biol Chem, 1957, 226(1): 497-509.
[26] BLIGH E G, DYER W J. A rapid method of total lipid extraction and purification[J]. Can J Biochem Physiol, 1959, 37(8): 911-917.
[27] KILJUNEN M, GREY J, SINISALO T, et al. A revised model for lipid-normalizing δ13C values from aquatic organisms, with implications for isotope mixing models[J]. J Appl Ecol, 2006, 43(6): 1213-1222.
[28] POST D M, LAYMAN C A, ARRINGTON D A, et al. Getting to the fat of the matter: models, methods and assumptions for dealing with lipids in stable isotope analyses[J]. Oecologia, 2007, 152(1): 179-189.
[29] LOGAN J M, JARDINE T D, MILLER T J, et al. Lipid corrections in carbon and nitrogen stable isotope analyses: comparison of chemical extraction and modelling methods[J]. J Anim Ecol, 2008, 77(4): 838-846.
[30] RUIZ-COOLEY R I, GARCIA K Y, HETHERINGTON E D. Effects of lipid removal and preservatives on carbon and nitrogen stable isotope ratios of squid tissues: implications for ecological studies[J]. J Exp Mar Biol Ecol, 2011, 407(1): 101-107.
[31] 贡艺, 陈新军, 高春霞, 等. 脂类抽提对北太平洋柔鱼肌肉碳、氮稳定同位素测定结果的影响[J]. 应用生态学报, 2014, 25(11): 3349-3356. [32] MATEO M A, SERRANO O, SERRANO L, et al. Effects of sample preparation on stable isotope ratios of carbon and nitrogen in marine invertebrates: implications for food web studies using stable isotopes[J]. Oecologia, 2008, 157(1): 105-115.
[33] VINAGRE C, MÁGUAS C, CABRAL H N, et al. Effect of body size and body mass on δ13C and δ15N in coastal fishes and cephalopods[J]. Estuar Coast Shelf Sci, 2011, 95(1): 264-267.
[34] SOTIROPOULOS M A, TONN W M, WASSENAAR L I. Effects of lipid extraction on stable carbon and nitrogen isotope analyses of fish tissues: potential consequences for food web studies[J]. Ecol Freshw Fish, 2004, 13(3): 155-160.
[35] MURRY B A, FARRELL J M, TEECE M A, et al. Effect of lipid extraction on the interpretation of fish community trophic relationships determined by stable carbon and nitrogen isotopes[J]. Can J Fish Aquat Sci, 2006, 63(10): 2167-2172.
[36] SWEETING C J, POLUNIN N V, JENNINGS S. Effects of chemical lipid extraction and arithmetic lipid correction on stable isotope ratios of fish tissues[J]. Rapid Commun Mass Sp, 2006, 20(4): 595-601.
-
期刊类型引用(3)
1. 刘娜,刘必林. 脂类去除对赤道外海茎柔鱼软组织稳定同位素的影响. 大连海洋大学学报. 2021(01): 155-160 .  百度学术
百度学术
2. 杨蕊,田思泉,高春霞,戴黎斌,王士聪. 浙江南部近海前肛鳗肌肉脂质去除对其稳定同位素测定结果的影响. 中国水产科学. 2020(09): 1085-1094 .  百度学术
百度学术
3. 黄佳兴,龚玉艳,徐姗楠,陈作志,张俊,于文明. 南海中西部海域鸢乌贼中型群和微型群的营养生态位. 应用生态学报. 2019(08): 2822-2828 .  百度学术
百度学术
其他类型引用(3)



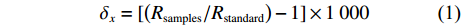
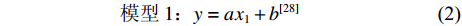
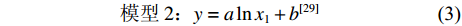
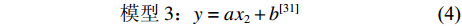
 下载:
下载:
 粤公网安备 44010502001741号
粤公网安备 44010502001741号
