Effect of nylon screen mesh as artificial substrate on variation of bacterial community in shrimp culture water
-
摘要:
采用基于16S rDNA的PCR-DGGE(变性梯度凝胶电泳)技术,比较分析了添加尼龙筛绢网基质对凡纳滨对虾(Litopenaeus vannamei)精养系统中水体细菌群落结构演替的影响。结果表明:1)基质组水体菌群结构丰富度稍低于对照组;2)基质组水体菌群结构相似性始终高于对照组;随着时间的推移,基质组的相似性系数不断升高,而对照组出现了先升高后下降的波动;3)对照组水体养殖前期优势菌主要为变形菌门和未培养细菌,在养殖后期则以厚壁菌门芽孢杆菌属(Bacillus spp.)细菌为主,基质组水体菌群则在整个养殖周期中均以厚壁菌门芽孢杆菌属细菌和未培养细菌为主。该研究结果可为进一步阐明人工基质养殖系统中微生物的生态调控功能和作用机制提供参考。
Abstract:The bacterial community composition in the substrate-biofilm shrimp culture system was monitored and investigated by pcr-denaturing gradient gel electrophoresis (DGGE). Results show that: 1) The water flora structure of substrate group was less diversified than that of the control. 2) The similarity coefficient of substrate group increased with time, while that of the control increased first then decreased. 3) Uncultured Firmicutes (Bacillus spp.), the dominant bacteria at the beginning of culture in the control, were uncultured Proteobacteria and uncultured bacterium, and the dominant bacteria at the end of culture was uncultured Firmicutes (Bacillus spp.). Uncultured Firmicutes (Bacillus spp.) and uncultured bacterium were the dominant bacteria throughout the culturing period. The results provide references for clarifying the function and mechanism of ecological control of microbes in artificial substrate system.
-
Keywords:
- shrimp /
- artificial substrate /
- bacterial community /
- DGGE
-
对虾养殖是世界水产业中最具有代表性的产业之一,中国是全球最大的对虾养殖国[1]。广东省作为中国对虾养殖第一大省,其养殖产量占到全国总产的1/3以上;对虾养殖业的发展带动了饲料、加工和出口的发展,也使广东成为世界对虾饲料生产基地、水产品加工基地和对虾出口基地[2]。然而随着对虾养殖集约化程度的不断提高,养殖水质恶化问题日益突出,养殖水环境稳定调控对于对虾健康生长显得益发重要。发挥养殖系统中微生物的功能,实现微生态环境的良好调控是集约化水产养殖走向生态健康之路的有效技术措施之一[3-4]。
研究表明,养殖水体中细菌群落结构组成与演替不仅关系着系统的物质循环,还在对虾的生长、虾体营养组分、机体免疫力等方面起到非常重要的作用[5-6]。在养殖系统中人为设置人工基质,可以促进微生物在其表面附着生长,形成生物膜,具有促进水体净化、维持良好水环境的效果,目前人工基质-生物膜技术已经发展成一种高效的养殖水体原位清洁修复技术[7-9]。研究证实,对虾养殖系统中设置人工基质可有效调控养殖水质,减少换水;提供额外饵料,降低饵料系数;增加栖附场所,降低对虾相对密度;提高有益菌数量,抑制病原菌生长;进而提高对虾成活率,促进生长,提高规格均匀度,降低养殖成本[9-14]。笔者在前期研究中亦发现添加人工基质可明显改善凡纳滨对虾养殖系统的水质,抑制弧菌丰度,显著提高对虾养殖性能[15]。
随着现代分子生物学技术的不断发展,不依赖于微生物培养的微生物分子生态学研究技术日益成熟,其中变性梯度凝胶电泳(denaturing gradient gel electrophoresis,DGGE)具有准确、灵敏、重复性好、易操作和分析速度快等优点,自1993年首次应用于土壤细菌微生物群落分析研究之后,便广泛应用于各种环境微生物群落多样性的研究[16]。
1. 材料与方法
1.1 实验材料
养殖实验在广东省中山市某水产养殖场6个室内方形圆角水泥池内进行,养殖池规格为5 m × 5 m × 1.2 m,基质组(S组)与对照组(C组)各设3个重复。人工基质选用40目尼龙筛绢网,悬空垂直悬挂于池中,基质面积与养殖池底面积相同(不含池壁面积,基质设置见图 1)。养殖水体盐度为5,为天然海水兑入河水,经消毒曝气后使用,水深0.8 m,整个养殖期间不换水,仅补充蒸发的水分。养殖为期73 d(2012年11月1日~2013年1月13日),实验期间水温为19.0~26.5 ℃,pH为7.8~8.2,自然光周期。凡纳滨对虾(Litopenaeus vannamei)[初始规格(0.13±0.02)g]养殖密度为250尾· m-2,日投喂“粤海”牌南美白对虾配合饲料3次,日投喂量为虾体质量的3%~7%,依摄食情况做调整;每5 d按0.1 g · m-3全池泼洒芽孢杆菌制剂(“利生菌王”,广州市欣海利生生物科技有限公司生产,活菌量为1010 CFU · g-1)1次。养殖水体中细菌DNA提取和纯化采用Water DNA Kit(OMEGA)试剂盒,PCR引物(表 1)由英潍捷基(上海)贸易有限公司合成,PCR反应体系购自TaKaRa(大连)。
表 1 特异性引物序列Table 1 Specific sequence of primers目的基因片段
target gene fragment引物名称
primer name引物序列(5′→3′)
primer sequence片段大小/ bp
product size16S rDNA[20] 8f AGAG TTTG ATCC TGGC TAAG 1 500 1492r GGTT ACCT TGTT ACGA CTT 16S rDNA-V3区[21] 341f-GC CGCCCGCCGCGCGCGGCGGGCGGGGCGGGGGCACGGGGGGCCTACGGGAGGCAG CAG 230 341f CCTACGGGAGGCAG CAG 534r ATTACCGCGGCTGCTGG 1.2 实验方法
1.2.1 样品采集
每隔14 d采集养殖水样1次,利用采水器在养殖池表、中和底部水层各采水1 L,混匀后作为一个样本。在实验室内取各池混合水样500 mL经0.2 μm的聚碳酸酯膜过滤以富集水样中细菌,滤膜密封保存于-80 ℃。
1.2.2 细菌总DNA提取与16S rDNA PCR扩增
根据试剂盒说明书提取细菌总DNA,并以之为模板,进行16S rDNA扩增。PCR反应体系为模板DNA 1 μL,4×dNTPs Mix(10 mmol · μL-1)4 μL,TaqDNA聚合酶(5 U · μL-1)1 μL,引物(10 pmol ·μL-1)各1 μL,10×buffer 5 μL,ddH2O补足至50 μL。PCR扩增条件为95 ℃ 3 min;95 ℃ 1 min,48 ℃ 1 min,35个循环;72 ℃延伸10 min。1.2%琼脂糖凝胶电泳(110 V,20 min)检测片段大小和浓度。
1.2.3 16S rDNA-V3区PCR扩增
以16S rDNA PCR扩增产物为模板,进行16S rDNA-V3区片段扩增。PCR反应体系为模板DNA 1 μL,4×dNTPs Mix 1.5 μL,Taq酶0.25 μL,引物(341f-GC与534r)各1.5 μL,10×buffer 5 μL,ddH2O补足至50 μL。PCR扩增条件为94 ℃ 4 min;94 ℃ 45 s,60 ℃ 45 s,72 ℃ 1 min,35个循环;72 ℃延伸10 min。1%琼脂糖凝胶电泳检测片段大小和浓度。PCR产物采用DNA Gel Extraction Kit(OMEGA)纯化回收,用于DGGE分析。
1.2.4 变性梯度凝胶电泳
DGGE在Bio-Rad Dcode上进行,采用变性梯度为35% ~55%、质量分数为8%的聚丙烯酰胺凝胶(100%的变性剂包含7 mol · L-1尿素和40%去离子甲酰胺)在1×TAE缓冲液中150 V、60 ℃下电泳4 h。凝胶经SYBR GreenⅠ染色20 min后Bio-Rad凝胶成像系统拍照,系统自带软件标记指纹图谱中优势条带。
1.2.5 DGGE条带的回收、克隆、测序及序列分析
用灭菌手术刀切下各标记条带,分别置于1.5 mL离心管中,无菌去离子水冲洗几次,枪头捣碎凝胶后,加入50 μL无菌去离子水,置于4 ℃过夜重悬。取2 μL上清液作为模板进行扩增,以534r和341f作为扩增引物,重新对16S rDNA-V3区进行扩增。PCR反应体系为10×Buffer 2.5 μL,dNTP Mixure 2 μL,引物各1 μL,TaqDNA聚合酶0.5 μL,模版2 μL,去离子水17 μL。PCR反应条件为94 ℃ 4 min;94 ℃ 45 s,60 ℃ 45 s,72 ℃ 45 s,30个循环;72 ℃ 10 min。扩增产物经回收纯化后,连接到pMD18-T载体(TaKaRa,大连),转化到感受态大肠杆菌DH5α中,挑选阳性克隆子送广州华大生物科技有限公司测序。
1.2.6 数据统计分析
PCR-DGGE指纹图谱分析采用BIO-RAD公司的Quantity One软件进行,计算条带相对丰度,通过UPGMA计算相似性矩阵并进行聚类分析。DICE法计算相似性系数[19],采用移动窗口分析法[22]分析2个处理组不同时间点菌群结构相似性变化,以每2次连续采样生物膜样品的相似系数为纵坐标,相对应的时间为横坐标,图线表示其菌群结构变化。t(连续采样的时间间隔)表示菌群的结构变化速率,其值为100%减去对应的2个连续样品间的相似性系数。
优势条带测序结果在NCBI中进行BLAST比对,选择最高同源性的序列作为参比序列,并同时记录其序列登录号。利用MEGA 4.0软件以邻接法(neighbor-joining)构建系统发生树,自举值Bootstrap重复1 000次。
2. 结果与分析
2.1 PCR-DGGE指纹图谱构建与菌群结构分析
各处理组对虾养殖水体不同时期菌群PCR-DGGE图谱见图 2。图谱共检测到24条特异性条带。分析发现,图谱中基质组菌群结构丰富度稍低于对照组,按照采样时间先后顺序,基质组检测到的条带数依次为10、7、11、13和11条,而对照组中则依次为11、11、13、13和11条。其中条带3、7、22和23为对照组所特有,条带12、19和20为基质组特有。该实验选取了18条不同位置上的特征条带进行后续克隆测序分析。
![]() 图 2 各处理组水体菌群随时间变化的变性梯度凝胶电泳分析示意图S1~S5,C1~C5分别代表基质组、对照组不同采样时间样品;带边框为测序条带,编号自上向下依次为S2-1、S4-2、S5-4、C2-7、C5-8、S4-9、C1-10、S2-11、C3-13、C5-15、C2-16、C4-17、C3-18、S3-20、S5-21、C3-22、C5-23、S3-24Fig. 2 Sketch map of DGGE analysis of bacteria in water with time in different groupsS1~S5 represent the experimental groups and C1~C5 represent the control groups at different sampling time. The sequencing bands are framed, and cutting band No. from top to bottom is S2-1, S4-2, S5-4, C2-7, C5-8, S4-9, C1-10, S2-11, C3-13, C5-15, C2-16, C4-17, C3-18, S3-20, S5-21, C3-22, C5-23 and S3-24.
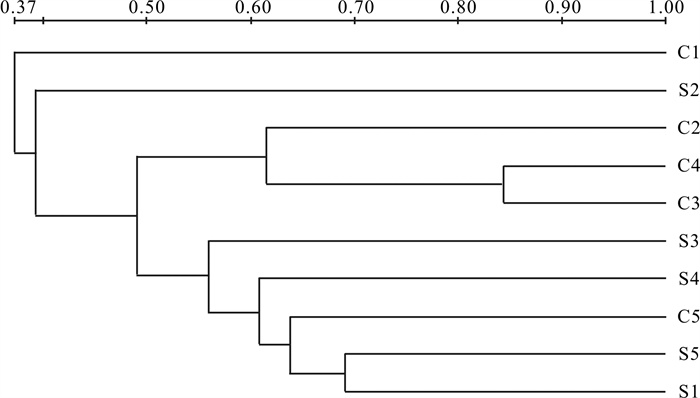
图 2 各处理组水体菌群随时间变化的变性梯度凝胶电泳分析示意图S1~S5,C1~C5分别代表基质组、对照组不同采样时间样品;带边框为测序条带,编号自上向下依次为S2-1、S4-2、S5-4、C2-7、C5-8、S4-9、C1-10、S2-11、C3-13、C5-15、C2-16、C4-17、C3-18、S3-20、S5-21、C3-22、C5-23、S3-24Fig. 2 Sketch map of DGGE analysis of bacteria in water with time in different groupsS1~S5 represent the experimental groups and C1~C5 represent the control groups at different sampling time. The sequencing bands are framed, and cutting band No. from top to bottom is S2-1, S4-2, S5-4, C2-7, C5-8, S4-9, C1-10, S2-11, C3-13, C5-15, C2-16, C4-17, C3-18, S3-20, S5-21, C3-22, C5-23 and S3-24.依据DGGE图谱中条带的位置、数量和亮度,用Quantity One软件对样品进行UPGMA聚类分析(图 3)。各个样品细菌群落按照其相似程度大致可以聚为3支,C1和S2一支,C2、C3和C4一支,而S1、S3、S4、S5和C5聚为另一支。
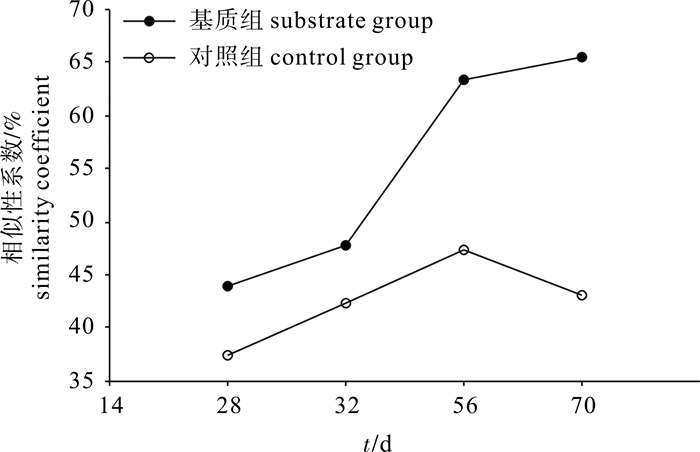
采用移动窗口分析法(图 4),开展了2个处理组菌群结构动态变化的定量分析。比较发现,随着时间的推移,基质组的相似性系数不断升高,而对照组出现了先升高后下降的波动;基质组菌群结构相似性波动范围为43.4%~66.3%,高于对照组的37.2%~46.8%,而其平均变化率t为(45.27±10.66)%,低于对照组的(61.02±10.68)%。
2.2 基因序列测定及系统发育树分析
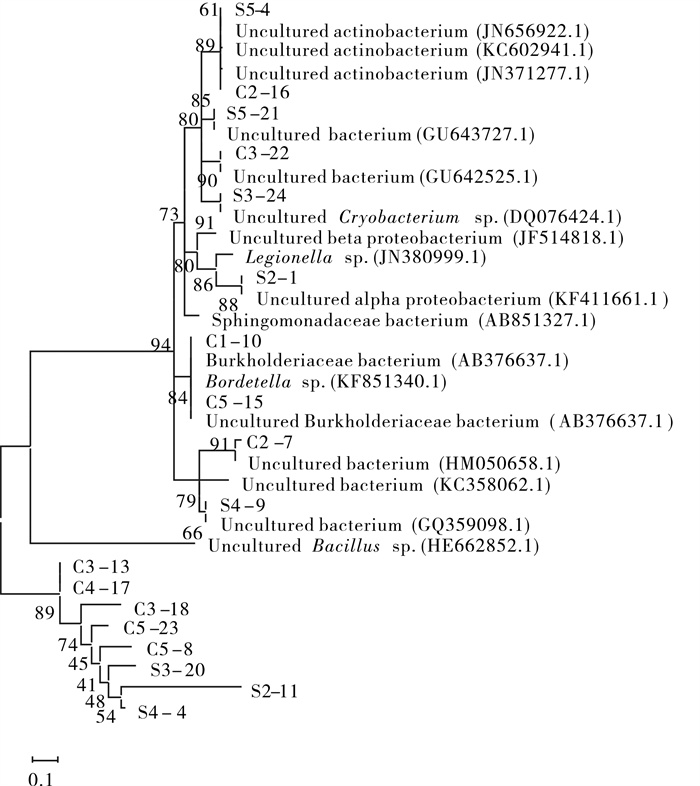
对所选取的18个特异条带的测序序列进行BLAST比对分析,其系统发生树见图 5。其中厚壁菌门未可培养芽孢杆菌属(Bacillus spp.)占44.4%;变形菌门占16.7%(其中α-变形菌纲占5.6%,β-变形菌纲占11.1%);未可培养的放线菌门占11.1%;其余为未可培养细菌,占22.2%。最优势菌群为未可培养厚壁菌门的芽孢杆菌属,在2个处理组各个采样时间点的养殖水样中均有检出。从优势菌的分布情况来看,基质组水体菌群在整个养殖周期中均以厚壁菌门芽孢杆菌属细菌和未培养细菌为主;对照组养殖水体前期优势菌主要为变形菌门和未培养细菌,而在养殖后期则以厚壁菌门芽孢杆菌属细菌为主。
3. 讨论
对虾集约化养殖因较高的养殖密度,单一的人工饲料投喂,相对简单的生态系统,以及过低的环境承载力极易造成水体环境的大幅变化,尤其是在养殖中后期,随着投喂量的加大,水体透明度、pH、营养盐等都发生较大改变,水体环境的变化亦影响了细菌群落组成。从PCR-DGGE图谱分析发现,2个处理组各个采样时期检测条带数波动不大,但其优势条带则出现较大变化。由此可得出,随着养殖的进行,养殖水体中菌群丰富度保持相对稳定,但其优势菌种类随着环境变化与时间推移形成一定程度的更替。该发现与罗鹏等[17]、王亭芳等[19]的研究结果一致。
生物膜系统由建立到稳定,通常为一个自然驯化成熟的过程,可人为干预调控的部分较少,因此这个过程较为缓慢,一般需要30 d才能够完成[11]。按照采样时间先后顺序,基质组水体中检测到的条带数依次为10、7、11、13和11条,对照组为11、11、13、13和11条。分析认为,在养殖前、中期基质组水体中优势菌群丰富度稍低于对照组,推测可能与生物膜生长期养殖系统中某类功能细菌生长依赖附着于特定的附着基质有关[11],而生物膜成熟后的周期性脱落在养殖后期补充了水体中菌群的丰富度。
人工基质上稳定的生物膜系统,通过吸附和脱落与水体中菌群紧密关联,进而维持着水体菌群结构的稳定。李敬美等[23]认为,人工基质系统中微生物以生物膜和悬浮菌团2种形式构成新的微生态系统,且在纵、横2个方向相互关联。结合系统论的观点可知,系统的结构越为复杂,其稳定性则越强,适应环境变化的能力也越强[24]。从该实验中的菌群结构相似性移动窗口分析亦可以发现,整个养殖期间,基质组不同采样时间水体菌群结构相似性均要高于对照组,且随着养殖时间的推进,前者结构相似性逐渐提高,而后者则在养殖中后期出现回落。水质环境的稳定造就了水体菌群结构的相对稳定,从前期实验结果看[15],添加人工基质组水体透明度始终维持在一定范围,且在养殖中后期氨氮与亚硝酸盐氮维持在较低水平,水质状况明显优于对照组,且波动范围较小,亦可解释该研究中基质组水体菌群结构的稳定性高于对照组的结论。
从优势菌的分布情况来看,与对照组养殖水体前期优势菌主要为变形菌门和未培养细菌,养殖后期则以厚壁菌门芽孢杆菌属细菌为主不同,基质组水体菌群在整个养殖周期中均以厚壁菌门芽孢杆菌属细菌和未培养细菌为主。这可能与养殖期间投放的芽孢杆菌制剂能被基质载体所固定化吸附,得以较好地生长有关,此推断有待进一步验证。但在2个处理组优势菌群中均存在芽孢杆菌属细菌,也反映了芽孢杆菌在对虾养殖微生态系统中所扮演的关键角色,其在改善与稳定养殖水体微环境中所发挥的重要功能已在相关研究中得以证实[25-28]。
人工基质-生物膜技术应用于对虾集约化养殖系统中,除了可以调控水质、稳定水体环境、减少换水之外,还兼具饵料补充、为对虾提供栖息场所、控制弧菌丰度等诸多作用,可有效实现对虾养殖的经济性、生态性与环保性,显示了广阔的应用前景。诚然,目前对生物膜在养殖系统中的作用机制尚未完全明了,诸如生物膜中各类微生物的功能定位,与环境的互作以及由群体感应引发的群落演替等问题仍有待于进一步揭示。此外,在水产养殖生产应用方面,人工基质-生物膜挂膜慢、菌群难以定向调控、抗逆性差等不足仍影响其功效发挥。由此,通过定向筛选或微生物基因工程技术构建高效、抗逆功能菌,并结合固定化微生物技术更好地发挥其作用;借鉴新型材料技术研发比表面积大、亲水性强、传质性高的生物膜载体材料,辅以营养定向平衡技术实现快速挂膜等解决方式将在今后不断呈现。相信,随着科学技术的不断进步与多学科创新融合,人工基质在对虾养殖中的应用会愈发成熟与广泛。
4. 结论
1) 人工基质的使用,使得基质组水体中细菌群落的丰富性稍低于对照组,但其水体中菌群结构的稳定性高于对照组。
2) 基质组水体优势菌群在整个养殖周期中均以厚壁菌门芽孢杆菌属细菌和未培养细菌为主,而对照组养殖水体前期优势菌主要为变形菌门和未培养细菌,在养殖后期则以厚壁菌门芽孢杆菌属细菌为主。
-
图 2 各处理组水体菌群随时间变化的变性梯度凝胶电泳分析示意图
S1~S5,C1~C5分别代表基质组、对照组不同采样时间样品;带边框为测序条带,编号自上向下依次为S2-1、S4-2、S5-4、C2-7、C5-8、S4-9、C1-10、S2-11、C3-13、C5-15、C2-16、C4-17、C3-18、S3-20、S5-21、C3-22、C5-23、S3-24
Figure 2. Sketch map of DGGE analysis of bacteria in water with time in different groups
S1~S5 represent the experimental groups and C1~C5 represent the control groups at different sampling time. The sequencing bands are framed, and cutting band No. from top to bottom is S2-1, S4-2, S5-4, C2-7, C5-8, S4-9, C1-10, S2-11, C3-13, C5-15, C2-16, C4-17, C3-18, S3-20, S5-21, C3-22, C5-23 and S3-24.
表 1 特异性引物序列
Table 1 Specific sequence of primers
目的基因片段
target gene fragment引物名称
primer name引物序列(5′→3′)
primer sequence片段大小/ bp
product size16S rDNA[20] 8f AGAG TTTG ATCC TGGC TAAG 1 500 1492r GGTT ACCT TGTT ACGA CTT 16S rDNA-V3区[21] 341f-GC CGCCCGCCGCGCGCGGCGGGCGGGGCGGGGGCACGGGGGGCCTACGGGAGGCAG CAG 230 341f CCTACGGGAGGCAG CAG 534r ATTACCGCGGCTGCTGG -
[1] 崔和. 我国对虾产销现状与国际对虾市场格局的变化[J]. 中国水产, 2014(8): 28-29. doi: 10.3969/j.issn.1002-6681.2014.08.014 [2] 周远扬, 曹俊明, 万忠, 等. 2013年广东对虾产业发展形势与对策建议[J]. 广东农业科学, 2014(8): 8-11. doi: 10.3969/j.issn.1004-874X.2014.08.003 [3] De SCHRYVER P, CRAB R, DEFOIRDT T, et al. The basics of bio-flocs technology: the added value for aquaculture[J]. Aquaculture, 2008, 277(3/4): 125-137. https://www.semanticscholar.org/paper/The-basics-of-bio-flocs-technology%3A-The-added-value-Schryver-Crab/77fe55196d4452612028f131c87dbe8fafcb174a
[4] 罗亮, 李卓佳, 张家松, 等. 对虾精养池塘碳、氮和异养细菌含量的变化及其相关性研究[J]. 南方水产科学, 2011, 7(5): 24-29. doi: 10.3969/j.issn.2095-0780.2011.05.004 [5] LIU H D, WANG L, LIU M, et al. The intestinal microbial diversity in Chinese shrimp (Fenneropenaeus chinensis) as determined by PCR-DGGE and clone library analyses[J]. Aquaculture, 2011, 317(1/2/3/4): 32-36. https://www.semanticscholar.org/paper/The-intestinal-microbial-diversity-in-Chinese-as-by-Liu-Wang/23054fffb0ae499e01911fe85fc8c0af13d39f65
[6] 吴伟, 陈家长, 胡庚东, 等. 利用人工基质构建固定化微生物膜对池塘养殖水体的原位修复[J]. 农业环境科学学报, 2008, 27(4): 1501-1507. doi: 10.3321/j.issn:1672-2043.2008.04.039 [7] ANAND P, KUMAR S, PANIGRAHI A, et al. Effects of C : N ratio and substrate integration on periphyton biomass, microbial dynamics and growth of Penaeus monodon juveniles[J]. Aquac Int, 2013, 21(2): 511-524. doi: 10.1007/s10499-012-9585-6
[8] THOMPSON F L, ABREU P C, WASIELESKY W. Importance of biofilm for water quality and nourishment in intensive shrimp culture[J]. Aquaculture, 2002, 203(3/4): 263-278. https://europepmc.org/article/AGR/IND23334984
[9] 陈亮亮, 董宏标, 张家松, 等. 6种改性剂对生物膜附着基质亲水性及其虾池挂膜效果的影响[J]. 南方水产科学, 2014, 10(2): 42-47. https://xueshu.baidu.com/usercenter/paper/show?paperid=d533da41798b09b38c3af4423ad14266&site=xueshu_se&hitarticle=1 [10] BRATVOLD D, BROWDY C L. Effects of sand sediment and vertical surfaces (AquaMatsTM) on production, water quality, and microbial ecology in an intensive Litopenaeus vannamei culture system[J]. Aquaculture, 2001, 195(1): 81-94. https://www.researchgate.net/publication/223661252_Effects_of_sand_sediment_and_vertical_surfaces_AquaMatsTM_on_production_water_quality_and_microbial_ecology_in_an_intensive_Litopenaeus_vannamei_culture_system
[11] OTOSHI C A, MONTGOMERY A D, MATSUDA E M, et al. Effects of artificial substrate and water source on growth of juvenile Pacific white shrimp, Litopenaeus vannamei[J]. J World Aquac Soc, 2006, 37(2): 210-213. doi: 10.1111/j.1749-7345.2006.00029.x
[12] 宋协法, 马真, 万荣. 纳米生态基在凡纳滨对虾养殖中的应用研究[J]. 中国海洋大学学报(自然科学版), 2010, 40(10): 24-28. https://xueshu.baidu.com/usercenter/paper/show?paperid=06931221c5f7354233d001b4313f5530&site=xueshu_se&hitarticle=1 [13] ZHANG B. Influence of the artificial substrates on the attachment behavior of Litopenaeus vannamei in the intensive culture condition[J]. Int J Anim Vet Adv, 2011, 3(1): 37-43. https://www.semanticscholar.org/paper/Influence-of-the-Artificial-Substrates-on-the-of-in-Zhang/b43d19eb558d482b050e09e33dd1f2e74a7647dd
[14] SCHVEITZER R, ARANTES R, BALOI M F, et al. Use of artificial substrates in the culture of Litopenaeus vannamei (Biofloc System) at different stocking densities: effects on microbial activity, water quality and production rates[J]. Aquac Eng, 2013, 54(3): 93-103. https://www.researchgate.net/publication/256974162_Use_of_artificial_substrates_in_the_culture_of_Litopenaeus_vannamei_Biofloc_System_at_different_stocking_densities_Effects_on_microbial_activity_water_quality_and_production_rates
[15] ZHANG J, CHEN L, DONG H, et al. Artificial substrates in zero-water-exchange culture system regulate the rearing performance of Pacific white shrimp Litopenaeus vannamei (Boone, 1931)under the winter indoor condition[J]. Aquac Res, 2014: 1-10. DOI: 10.1111/are.12473.
[16] PACE N R. A molecular view of microbial diversity and the biosphere[J]. Science, 1997, 276(5313): 734-740. doi: 10.1126/science.276.5313.734
[17] 罗鹏, 胡超群, 张吕平, 等. 凡纳滨对虾海水养殖系统内细菌群落的PCR-DGGE分析[J]. 中国水产科学, 2009, 16(1): 31-38. https://xueshu.baidu.com/usercenter/paper/show?paperid=073cb871c675aff240a054b145c665c6&site=xueshu_se&hitarticle=1 [18] 姚延丹, 李谷, 陶玲, 等. 复合池塘循环水养殖系统微生物群落结构分析[J]. 中国水产科学, 2011, 18(2): 407-415. https://xueshu.baidu.com/usercenter/paper/show?paperid=ffb8870503ceef6c39572b93c79fbdbc&site=xueshu_se&hitarticle=1 [19] 王亭芳, 金风杰, 马士禹, 等. DGGE技术对南美白对虾养殖水体中微生物多样性的研究[J]. 生物技术通报, 2012(10): 131-136. https://xueshu.baidu.com/usercenter/paper/show?paperid=db9b5e97433278bdfeadd56a0639f81f&site=xueshu_se&hitarticle=1 [20] PORTEOUS L A, ARMSTRONG J L. Recovery of bulk DNA from soil by a rapid, small-scale extraction method[J]. Curr Microbiol, 1991, 22(6): 345-348. doi: 10.1007/BF02092152
[21] MUYZER G. DGGE/TGGE a method for identifying genes from natural ecosystems[J]. Curr Opin Microbiol, 1999, 31(2): 7-22. https://www.semanticscholar.org/paper/DGGE-TGGE-a-method-for-identifying-genes-from-Muyzer/970566056daf1a863a5a515d63827a3440b7e374
[22] 张瑜, 韩玲, 谢露蓉. 基于移动窗口分析法的西安市热岛效应研究[J]. 测绘科学, 2012, 37(5): 148-150. [23] 李敬美, 金若菲, 周集体, 等. 活性污泥与生物膜复合工艺处理石化废水的研究[J]. 环境工程学报, 2011, 5(3): 523-527. http://hjhx.rcees.ac.cn/article/id/20110310 [24] FOUAD M, BHARGAVA R. Mathematical model for the bio-film-activated sludge reactor[J]. J Environ Eng, 2005, 131(4): 557-562. doi: 10.1061/(ASCE)0733-9372(2005)131:4(557)
[25] ZOKAEIFAR H, BABAEI N, SAAD C R, et al. Administration of Bacillus subtilis strains in the rearing water enhances the water quality, growth performance, immune response, and resistance against Vibrio harveyi infection in juvenile white shrimp, Litopenaeus vannamei[J]. Fish Shellfish Immunol, 2014, 36(1): 68-74. doi: 10.1016/j.fsi.2013.10.007
[26] DONG H B, SU Y Q, MAO Y, et al. Dietary supplementation with Bacillus can improve the growth and survival of the kuruma shrimp Marsupenaeus japonicus in high-temperature environments[J]. Aquac Int, 2014, 22(2): 607-617. doi: 10.1007/s10499-013-9688-8
[27] 李卓佳, 郭志勋, 冯娟, 等. 应用芽孢杆菌调控虾池微生态的初步研究[J]. 海洋科学, 2006, 30(11): 28-31. doi: 10.3969/j.issn.1000-3096.2006.11.007 [28] 陆家昌, 李活, 黄翔鹄. 枯草芽孢杆菌对水质及凡纳滨对虾幼体免疫指标影响的研究[J]. 南方水产, 2010, 6(1): 19-24. doi: 10.3969/j.issn.1673-2227.2010.01.004 -
期刊类型引用(3)
1. 胡晓娟,文国樑,李卓佳,孙志伟,杨铿,张家松,韩宁,曹煜成. 养殖中后期高位池对虾水体微生物群落结构及水体理化因子. 生态学杂志. 2018(01): 171-178 .  百度学术
百度学术
2. 张月,段亚飞,董宏标,张真真,张家松. 人工基质对凡纳滨对虾免疫和消化相关指标的影响. 水产科学. 2017(04): 462-466 .  百度学术
百度学术
3. 段亚飞,黄忠,林黑着,董宏标,张家松. 深水网箱和池塘养殖凡纳滨对虾肌肉营养成分的比较分析. 南方水产科学. 2017(02): 93-100 .  本站查看
本站查看
其他类型引用(2)




 下载:
下载:

 粤公网安备 44010502001741号
粤公网安备 44010502001741号
