Genetic structure of Priacanthus macracanthus population from the South China Sea
-
摘要:
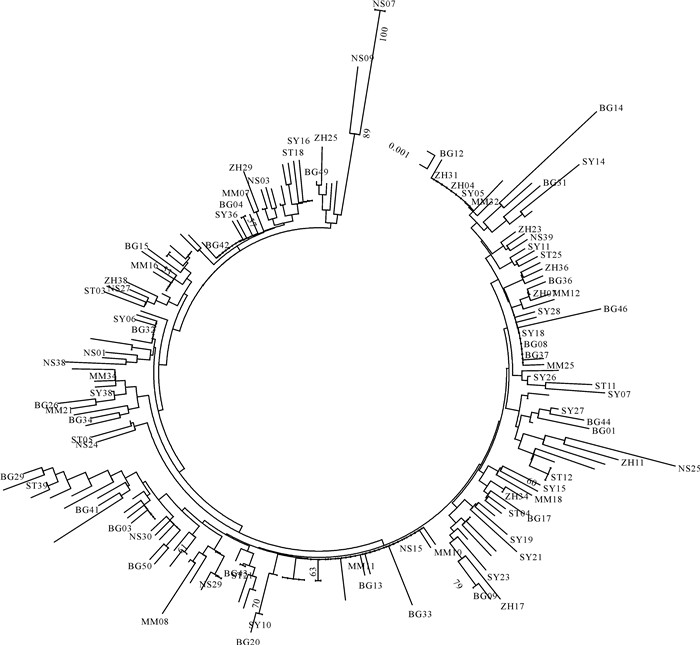
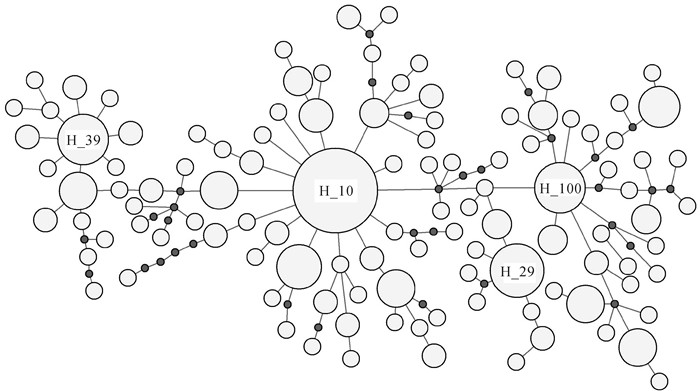
在南海北部陆架和南海南沙西南陆架6个地理位点采集224尾短尾大眼鲷(Priacanthus macracanthus)样品,采用线粒体控制区(mtDNA D-loop区)序列分析了其种群遗传结构,探讨了6个采样位点之间短尾大眼鲷种群归属关系。所得224条同源序列D-loop片段(729 bp)中检测到101个突变位点和94个核苷酸多态位点,定义了172种单倍型;遗传多样性表现出高单倍型多样性(0.980 5~0.997 1)和低核苷酸多样性(0.048 2~0.060 9)的特点;系统发育分析、分子方差分析和成对的Fst分析显示南海各群体之间有较高的遗传同质性,遗传分化不显著,也没有出现明显的地理分支或聚簇;中性检测和不对称分布分析发现南海的短尾大眼鲷群体发生过种群扩张。结果表明,南海海域的6个短尾大眼鲷群体属于同一种群,该结果为今后中国与南海周边国家渔业资源共享和争取捕捞配额提供了依据。
Abstract:We investigated the genetic structure of red bigeye (Priacanthus macracanthus) population in six sampling sites off the South China Sea including five coastal waters of Beibu Gulf, Sanya, Maoming, Zhuhai, Shantou and the Southwest Nansha. Sequences of the mitochondrial control region (D-loop) were amplified by polymerase chain reaction. Fragments of 729 bp of the mitochondrial D-loop were sequenced from 224 individuals, and 101 mutated sites, 94 nucleotide polymorphism sites as well as 172 haplotypes were identified. All the six groups were characterized with high haplotype diversity (0.980 5~ 0.997 1) and low nucleotide diversity (0.048 2~0.060 9). Analyses of phylogeny, molecular variances and pairwise Fst showed no significant genealogical clades or clusters of samples corresponding to sampling localities. Both neutrality tests and mismatch distribution analyses revealed a recent population expansion in P.macracanthus. Thus, the six P.macracanthus groups in the South China Sea belong to one population. The results provide references for the fishery resources sharing and quotas winning between China and the South China Sea surrounding countries.
-
几丁质由N-乙酰-D-氨基葡萄糖 (GlcNAc) 通过β-1,4-糖苷键连接而成[1],在自然界中广泛分布于虾和蟹的外壳[2]、真菌细胞壁、昆虫外骨骼以及一些绿藻中。仅水生生态系统中,几丁质每年的产量就超过1×1011 t[3]。天然几丁质由于分子内和分子间强大的氢键作用而性质稳定,难溶于水、稀酸、碱,仅溶于浓盐酸、硝酸等强酸以及一些离子液体 (如咪唑基离子液体等)[4],所以很难得到有效利用,造成了严重的资源浪费。然而,几丁质的水解产物N-乙酰壳寡糖却具有水溶性、细胞膜渗透性和易吸收性等理化特点;还具有抗菌、抗病毒、抗肿瘤、抗氧化、免疫调节、降血压和降低胆固醇等多种生物活性[5],广泛应用于生物化工、生物医药、农产品、化妆品、保健品等领域[6]。其中,N, N-二乙酰基壳二糖 [(GlcNAc)2] 具有抗肿瘤、抗菌和免疫刺激作用等生物活性[7]。(GlcNAc)2在医疗方面,能够防止术后组织黏连[8];在农业方面,能够促进植物生长和诱导植物先天免疫[9]。此外,(GlcNAc)2还能诱导几丁质酶在链霉菌、蘑菇等生物中的表达[10-11]。
降解几丁质的方法包括物理法、化学法和生物酶法。迄今为止,采用物理法制备N-乙酰壳寡糖的研究较少,在工业上尚未形成规模化生产。传统的化学法采用强酸处理几丁质,反应非常复杂,难以控制,并会产生各种二级化合物,难以去除。生物酶法作为绿色生产技术,可克服物理和化学法的缺点[12],其反应条件温和、产率高、对环境无污染,因而受到越来越多的关注[13]。
几丁质酶 (Chitinase, EC 3.2.1.14) 属于糖苷水解酶,通过水解几丁质的β-(1→4) 糖苷键将其降解为N-乙酰壳寡糖或GlcNAc[14]。在碳水化合物-活性酶数据库 (CAZy) 中,根据催化结构域氨基酸序列的保守性,被分为糖苷水解酶18家族 (GH18)、19家族 (GH19) 和20家族 (GH20)。GH18家族几丁质酶主要由细菌、真菌、昆虫、植物和动物产生,GH19家族几丁质酶主要来源于高等植物和某些细菌[15],而GH20家族几丁质酶来源于细菌和人类。不同来源的几丁质酶已在大肠杆菌 (Escherichia coli) 和枯草芽孢杆菌 (Bacillus subtilis) 等异源宿主系统中以重组蛋白的形式表达[16-17]。几丁质酶有许多潜在的应用,除了用于制备N-乙酰壳寡糖[18-19]外,还可以用作致病菌的抗真菌剂[20],利用和清理几丁质废弃物并将其转化为生物乙醇[21]。
海洋环境中含有丰富的几丁质,是筛选产几丁质酶微生物的重要来源地之一。在前期研究中,本课题组从红树林虾场土壤中筛选到一株能够利用几丁质的放线菌橙黄小单孢菌(Micromonospora aurantiaca)。迄今为止,已有诸多放线菌来源的几丁质酶被分离和鉴定[22-23],但是橙黄小单孢菌来源几丁酶的相关研究尚未见报道。本研究以橙黄小单孢菌的基因组DNA为模板,克隆到了一条几丁质酶基因Machi3,并成功在大肠杆菌中表达。之后,对重组酶进行纯化及酶学特性的研究,采用扫描电镜 (SEM) 进一步验证了该重组酶对几丁质底物的水解活性。最后,利用该重组酶水解胶体几丁质制备 (GlcNAc)2,为几丁质资源的开发和利用奠定了基础。
1. 材料与方法
1.1 菌株与质粒
橙黄小单孢菌:于海南省海口市东寨港红树林虾场土壤中筛选得到,保存于本实验室;pMD-19T载体购于宝生物工程 (大连) 有限公司;E. coli DH5α、E. coli BL21 (DE3) 和表达载体pET-28a购于Novagen公司。
1.2 主要材料与仪器
细菌基因组DNA提取试剂盒、质粒提取试剂盒 (上海捷瑞生物工程有限公司);PCR产物纯化试剂盒、DNA胶回收试剂盒、蛋白Marker [生工生物工程 (上海) 股份有限公司];T4 DNA连接酶、DNA Marker [宝生物工程 (大连) 有限公司];限制性内切酶 (赛默飞世尔科技公司);GlcNAc (上海源叶生物科技有限公司);(GlcNAc)2-6标准品 (山东青岛博智汇力有限公司);牛血清白蛋白 (武汉赛维尔生物科技有限公司);其他试剂均为国产分析纯。
DYCZ-24DN型双板垂直电泳槽、DYPC-31DN型水平电泳槽 (北京六一生物科技有限公司);A300型PCR扩增仪、LG2020D型凝胶成像系统 (杭州朗基科学仪器有限公司);高效液相色谱柱:Shodex Asahipak NH2P-50 4E (4.6 mm×250 mm, 5 μm) (北京迪科马科技有限公司);K6600B型全波长酶标仪 (北京凯奥科技发展有限公司);NRY-200型全温培养摇床 (上海南荣实验室设备有限公司);Phenom ProX型SEM (飞纳科学仪器有限公司)。
1.3 实验方法
1.3.1 目的基因的克隆和生物信息学分析
本研究根据美国国家生物信息中心 (NCBI) 已公布的橙黄小单孢菌ATCC 27029的全基因组DNA序列信息 (NCBI登陆号CP002162),从中挖掘出一条可能编码几丁质酶的基因序列,根据该基因序列采用Primer Premier 5.0软件设计特异性引物Primer-F (CATATGGTGGTCCTCCCGAACAACTTC,酶切位点为Nde I) 和Primer-R (AAGCTTTCAGGCCGGCAGCCGC,酶切位点为Hind III),委托生工生物工程 (上海) 股份有限公司合成。PCR反应体系 (50 µL):TaKaRa LA Taq 0.5 µL,Primer-F (20 µmol·L−1) 1 µL,Primer-R (20 µmol·L−1) 1 µL,基因组DNA 1 µL,dNTP Mixture 8 μL,2 × GC Buffer I 25 μL,ddH2O 13.5 µL。PCR扩增条件:94 ℃预变性 1 min;94 ℃变性 30 s,58 ℃退火 30 s,72 ℃ 延伸100 s,30个循环;72 ℃ 延伸 7 min。PCR产物经1% (质量分数) 琼脂糖凝胶电泳验证后,根据DNA胶回收试剂盒说明书回收目的产物片段。
生物信息学分析软件及网站:氨基酸序列分析采用NCBI的BLAST功能,信号肽序列分析用SignalP 5.0在线服务器 (http://www.cbs.dtu.dk/services/SignalP/),蛋白质同源序列比对采用DNAMAN软件,保守域的分析采用NCBI的CD-search工具 (https://www.ncbi.nlm.nih.gov/cdd/),理论分子量和等电点的预测采用ExPASy的ProtParam工具 (http://web.expasy.org/protparam/)。
1.3.2 重组表达载体的构建
将目的基因连接至pMD19-T载体,之后转化至E. coli DH5α感受态细胞中。根据菌落PCR鉴定结果,挑选阳性克隆子送至北京擎科生物科技有限公司测定目的基因序列。将测序正确的基因序列和pET-28a表达载体分别用Nde I和Hind III双酶切,回收目的基因片段和酶切质粒,用T4 DNA连接酶连接,构建重组表达质粒,并转化至E. coli BL21 (DE3) 感受态细胞中。
1.3.3 重组几丁质酶的诱导表达和分离纯化
将含有目的基因的重组菌接种到含50 μg·mL−1卡那霉素的Luria-Bertani (LB) 液体培基中,在37 ℃、180 r·min−1下振荡培养至菌液OD600 nm为0.8时,加入终浓度为0.2 mmol·L−1的异丙基-β-D-硫代半乳糖苷 (IPTG),在16 ℃、180 r·min−1下诱导培养16 h。诱导培养结束后,离心 (4 ℃, 6 000 r·min−1, 5 min) 收集菌体,用0.85% (质量分数) 的生理盐水清洗后用pH 7.0的磷酸盐缓冲液重悬,之后置于冰水浴中超声破碎 (功率350 W,工作 2 s,间隔5 s)。超声破碎结束后在4 ℃、6000 r·min−1下离心15 min,上清液即为粗酶液。将粗酶液上样于镍亲和层析色谱柱进行分离纯化,并利用SDS-PAGE分析蛋白的纯度和分子量。
1.3.4 胶体几丁质的制备
参照Pan等[24]的方法制备胶体几丁质,略有改动。称取10 g α-几丁质粉末,缓慢加入到200 mL预冷的浓盐酸中 (边加边搅拌),在4 ℃冰箱中静置至溶液呈黄色。之后,向上述混合物中加入2 L预冷的50% (体积分数) 乙醇,快速搅拌后于4 ℃下放置过夜。然后在4 ℃、4 000 r·min−1下离心15 min,并收集沉淀部分。用大量去离子水反复冲洗沉淀部分至中性,离心 (10 000 r·min−1, 10 min) 后获得的沉淀物即为胶体几丁质。
1.3.5 重组几丁质酶的酶活力测定
以胶体几丁质作为底物,采用3,5-二硝基水杨酸 (DNS) 法测定还原糖产量[25]。用0~5 μmol·L−1的GlcNAc制作还原糖标准曲线,得到标准曲线方程y=0.122 6x−0.030 9,R2=0.990 8。酶活测定方法如下:将胶体几丁质加入至pH 7.0的Tris-HCl缓冲液中,混合均匀后制成终质量浓度为50 mg·mL−1的胶体几丁质混悬液。取0.5 mL上述胶体几丁质混悬液与0.5 mL酶液混合,于50 ℃水浴中磁力搅拌反应30 min。反应结束后离心 (10 000 r·min−1, 2 min),获得上清液。取0.5 mL上清液,加入0.5 mL DNS溶液,摇匀后煮沸5 min进行显色反应,用酶标仪于540 nm处测定吸光值,根据还原糖标准曲线计算还原糖产量。同时,以加热灭活的酶液作为空白对照组。酶活力单位定义为:在上述反应条件下,每分钟催化胶体几丁质产生1 μmol GlcNAc所需要的酶量,用U表示。
1.3.6 重组几丁质酶的蛋白浓度测定
采用Bradford法[26]测定重组酶的蛋白浓度。以0~0.1 mg·mL−1的牛血清白蛋白制作蛋白浓度标准曲线,得到标准曲线方程y=4.579 1x−0.002,R2=0.990 7。根据吸光值和标准曲线方程计算待测样的蛋白浓度。
1.3.7 重组几丁质酶的酶学性质研究
1) 最适反应温度及热稳定性研究。取等量的酶液,在不同反应温度 (25~65 ℃) 下按照标准方法测定重组几丁质酶的酶活。将最适反应温度下的相对酶活定义为100%。为了测定重组几丁质酶的热稳定性,将酶液置于不同温度 (40~60 ℃) 下孵育1 h后取样,按标准方法,在最适反应温度下测定残余酶活。将孵育前几丁质酶的酶活定义为100%。
2) 最适反应pH及pH稳定性研究。取等量的酶液,在不同pH的缓冲液 (50 mmol·L−1,pH 3.0~5.0为柠檬酸-柠檬酸钠缓冲液;pH 5.0~8.0为磷酸盐缓冲液;pH 7.0~9.0为Tris-HCl缓冲液;pH 9.0~10.0为甘氨酸-NaOH缓冲液) 中按照标准方法测定重组几丁质酶的酶活。将最适反应pH下的相对酶活力定义为100%。为了测定重组几丁质酶的pH稳定性,将酶液加入到不同pH的缓冲液 (50 mmol·L−1,pH 3.0~4.0为柠檬酸-柠檬酸钠缓冲液;pH 5.0~6.0为磷酸盐缓冲液;pH 7.0~8.0为Tris-HCl缓冲液;pH 9.0~10.0为甘氨酸-NaOH缓冲液) 中,在4 ℃下孵育1 h。之后,在最适反应温度和pH条件下,按照标准方法测定残余酶活。孵育前重组几丁质酶的酶活被定义为100%。
3) 化学试剂和金属离子对酶活力的影响。为了研究化学试剂对重组几丁质酶酶活力的影响,向反应体系中分别加入不同的化学试剂,包括尿素 (终浓度200 mmol·L−1),十二烷基硫酸钠 (SDS,终浓度10 mmol·L−1),乙二胺四乙酸 (EDTA,终浓度10 mmol·L−1),吐温 (Tween 20、40、60、80,终体积分数1%) 和曲拉通X-100 (终体积分数1%),在最适反应温度和pH条件下,按照标准方法测定重组酶MaChi3的酶活力。为了研究金属离子对重组几丁质酶酶活力的影响,向反应体系中分别加入终浓度1 mmol·L−1的钾离子 (K+)、镁离子 (Mg2+)、亚铁离子 (Fe2+)、钙离子 (Ca2+)、钴离子 (Co2+)、铁离子 (Fe3+)、钡离子 (Ba2+)、锌离子 (Zn2+)、铜离子 (Cu2+) 和银离子 (Ag+),在最适反应温度和pH条件下,按照标准方法测定重组酶MaChi3的酶活力。将未添加任何化学试剂和金属离子时的相对酶活定义为100%。
4) 底物特异性及动力学参数测定。将酶液分别与终质量浓度为2 mg·mL−1的α-几丁质、胶体几丁质、虾壳粉、淀粉、纤维素、不同脱乙酰度的壳聚糖 (脱乙酰度50%、70%、80%、85%、90%、95%) 进行反应,在最适反应温度和pH条件下,测定重组酶MaChi3的酶活力,研究该重组酶对不同底物的特异性。
在含有不同质量浓度 (1~8 mg·mL−1) 胶体几丁质的反应体系中,在最适反应温度和pH条件下测定重组酶MaChi3的酶活力。采用双倒数作图法,利用Origin 8.5软件进行线性拟合,根据拟合公式计算MaChi3的动力学参数米氏常数Km和最大反应速率Vmax值。
1.3.8 几丁质的SEM表征
将10 mg α-几丁质和胶体几丁质分别加入到50 mmol·L−1 pH 7.0的Tris-HCl缓冲液中,均匀分散后加入2 mL酶液,于55 ℃下振荡孵育24 h。孵育结束后,在10 000 r·min−1下离心10 min,收集底物样品,在60 ℃下干燥24 h。采用SEM对孵育前后的α-几丁质和胶体几丁质的表面微观形态进行观察。观察前用导电双面胶带将样品固定在样品台上,在真空环境下进行镀金处理,物料表面被一层薄薄的金覆盖形成导电膜以获得导电性。使用SEM的颗粒表面分析系统观察样品的表面形态,加速电压为10 kV,放大倍数为2500倍[27]。
1.3.9 N, N-二乙酰基壳二糖的制备
将10 mg胶体几丁质加入到50 mmol·L−1 pH 7.0的Tris-HCl缓冲液中,均匀分散后加入5 mL酶液,于55 ℃下振荡反应。定时从反应瓶中取样 (0、2、4、6、8、10、12、24 h),在沸水浴中孵育10 min后离心 (10 000 r·min−1, 5 min),取上清液用0.22 μm滤头过滤,滤液用于HPLC检测分析。检测条件:安捷伦1260高效液相色谱仪,配备Asahipak NH2P-504E (250 mm×4.6 mm,Shodex,日本) 色谱柱。流动相为75% (体积分数) 乙腈,流速为0.5 mL·min−1,柱温箱温度设定为25 ℃,检测器温度为25 ℃,进样量为10 μL,检测波长为210 nm。(GlcNAc)1-6的保留时间依次为13.2、18.0、25.3、34.1、49.7和77.3 min。
1.3.10 数据处理
采用Origin Pro 2018软件对数据进行统计分析,每组实验做3次平行。
2. 结果
2.1 几丁质酶基因的克隆及生物信息学分析
以本实验室保存的橙黄小单孢菌的基因组DNA为模板,利用特异性引物进行PCR扩增,获得一条大约1 400 bp的基因条带 (图1-a)。将测序结果提交至NCBI数据库进行BLAST分析,发现其与查找到的几丁质酶基因序列一致性达到100% (NCBI登录号WP_013285221),表明已成功从M. aurantiaca的基因组DNA中克隆到几丁质酶基因,并将其记为Machi3。Machi3基因的全长为1 371 bp,编码456个氨基酸,预测的蛋白理论相对分子质量和pI分别为47.44 kD和7.55。信号肽预测结果表明,在MaChi3的N端含有信号肽,信号肽的切割位点在第33—第34个氨基酸之间。MaChi3含有一个Glyco_18结构域 (位于40—305位氨基酸残基) 和一个具有碳水化合物结合作用的RICIN结构域 (位于331—455位氨基酸残基,图1-b)。将MaChi3的氨基酸序列提交至NCBI数据库进行BLAST分析,发现其与来源于那拉提瓦小单孢菌 (M. narathiwatensis) (NCBI登录号WP_091195038)、邱氏疣孢菌 (Verrucosispora sioxanthis) (NCBI登录号WP_164447165) 和石渣球孢菌 (Allocatelliglobosispora scoriae) (NCBI登录号WP_184833644) 的几丁质酶序列一致性分别为87.3%、83.5%和78.3%。但是,上述几丁质酶均为未经验证的假定几丁质酶。因此,将MaChi3的氨基酸序列提交至PDB数据库,结果表明该酶与来源于约氏黄杆菌 (Flavobacterium johnsoniae) UW101的GH18家族几丁质酶具有32%的一致性。GH18家族几丁质酶具有保守序列DxDxE和SxGG,DxDxE基序为几丁质酶的催化中心,而SxGG基序负责酶与底物的结合。在催化过程中,SxGG基序中的丝氨酸对DxDxE基序中第一个天冬氨酸上产生的暂时过剩的负电荷起稳定作用[28-29]。如图1-c所示,在MaChi3的催化域中含有符合上述保守基序的145DMDWE149序列,但是SxGG基序中的丝氨酸残基被丙氨酸残基 (103AVGG106) 所代替,这一发现与Vaaje-Kolstad等[30]报道的来源于乳酸乳球菌乳酸亚种 (Lactococcus lactis ssp. Lactis)的LlChi18A相一致。事实上,除了SxGG基序中的丝氨酸外,酪氨酸也具有稳定电荷的作用。尽管MaChi3缺乏丝氨酸残基,但却含有酪氨酸残基 (Y45,相当于LlChi18A的Y48)。另外,Vaaje-Kolstad等[30]研究发现,在与LlChi18A催化域相似性较高的50个不同来源的GH18家族几丁质酶的催化域中,约有一半蛋白质的丝氨酸或酪氨酸残基被替代,但是这两个保守氨基酸残基不会被同时替代。总之,以上序列分析结果表明,MaChi3是一种新型的GH18家族几丁质酶。
![]() 图 1 MaChi3 基因 PCR 扩增产物 (a)、保守域分析 (b) 和部分催化域氨基酸多序列比对 (c)注:(a) M为DNA分子量标准;(c)图中列出的几丁质酶氨基酸序列来源于B. circulans WL-12 (PDB ID : 1ITX),C. shinanonensis (PDB ID : 6KXL),B. thuringiensis (PDB ID : 6BT9),Paenibacillus sp. FPU-7 (PDB ID : 5GZU) 和L. lactis ssp. Lactis (NCBI登录号AAK06048)。保守基序SxGG和DxDxE在底部用带下划线的红色字母标识,保守的酪氨酸残基在上部用※标识。Fig. 1 PCR products (a), conserved domain analysis (b) and sequence alignment of partial predicted catalytic domain (c) of MaChi3Note: (a) M is DNA molecular mass standard. (c) The listed sequences include the chitinases from B. circulans WL-12 (PDB ID: 1ITX), C. shinanonensis (PDB ID: 6KXL), B. thuringiensis (PDB ID: 6BT9), Paenibacillus sp. FPU-7 (PDB ID: 5GZU) and L. lactis ssp. Lactis (NCBI accession NO.: AAK06048). The conserved sequence motifs SxGG and DxDxE are showed at the bottom with red color underlined, and the conserved tyrosine residue is indicated by ※.
图 1 MaChi3 基因 PCR 扩增产物 (a)、保守域分析 (b) 和部分催化域氨基酸多序列比对 (c)注:(a) M为DNA分子量标准;(c)图中列出的几丁质酶氨基酸序列来源于B. circulans WL-12 (PDB ID : 1ITX),C. shinanonensis (PDB ID : 6KXL),B. thuringiensis (PDB ID : 6BT9),Paenibacillus sp. FPU-7 (PDB ID : 5GZU) 和L. lactis ssp. Lactis (NCBI登录号AAK06048)。保守基序SxGG和DxDxE在底部用带下划线的红色字母标识,保守的酪氨酸残基在上部用※标识。Fig. 1 PCR products (a), conserved domain analysis (b) and sequence alignment of partial predicted catalytic domain (c) of MaChi3Note: (a) M is DNA molecular mass standard. (c) The listed sequences include the chitinases from B. circulans WL-12 (PDB ID: 1ITX), C. shinanonensis (PDB ID: 6KXL), B. thuringiensis (PDB ID: 6BT9), Paenibacillus sp. FPU-7 (PDB ID: 5GZU) and L. lactis ssp. Lactis (NCBI accession NO.: AAK06048). The conserved sequence motifs SxGG and DxDxE are showed at the bottom with red color underlined, and the conserved tyrosine residue is indicated by ※.2.2 MaChi3的诱导表达
重组菌经诱导表达后,利用镍亲和层析色谱柱对粗酶液进行纯化,在大约47 kD处出现一条蛋白条带 (图2),与推测的蛋白相对分子质量 (47.44 kD) 基本吻合,表明重组酶MaChi3已成功在大肠杆菌中表达。
2.3 MaChi3的酶学性质研究
2.3.1 MaChi3的最适反应温度及热稳定性
为了测定重组几丁质酶MaChi3的最适反应温度,以胶体几丁质作为底物,在25~65 ℃下进行反应。结果显示,MaChi3的最适反应温度为55 ℃,并且在50~60 ℃范围内具有较高的相对酶活力 (>90.4%)。当反应温度提高至65 ℃时,相对酶活力急剧下降,仅为23.5% (图3-a)。为了测定MaChi3的温度稳定性,将其置于40~60 ℃水浴中孵育1 h,然后在最适反应温度下测定其残余酶活力。由图3-b可知,MaChi3的温度稳定性良好,在50 ℃下孵育1 h后残余酶活力超过70%,在其最适反应温度55 ℃下孵育1 h后残余酶活力保持在67.3%。
2.3.2 MaChi3的最适反应pH及pH稳定性
重组几丁质酶MaChi3的最适反应pH为7.0,并且在pH 6.0~9.0范围内表现出较好的酶活力 ,相对酶活力高于64% (图4-a)。为了测定MaChi3的pH稳定性,将该酶置于pH 3.0~10.0的缓冲液中孵育,之后在最适反应温度和pH条件下测定其残余酶活力。MaChi3在pH 6.0~9.0范围内孵育1 h后残余酶活力保持在80%以上,展现出了较好的稳定性 (图4-b)。此外,在其最适反应pH (pH 7.0) 下孵育1 h后对其酶活力基本无影响,残余酶活力为98%。
2.3.3 化学试剂和金属离子对MaChi3的影响
如表1所示,Triton X-100和EDTA对MaChi3的酶活力无显著影响。尿素、SDS、吐温 20和吐温60对MaChi3具有不同程度的抑制作用,相对酶活力为58.1%~90.4%。吐温40和吐温80对MaChi3的酶活力有轻微的促进作用,相对酶活力分别为104.1%和108.4%。不同的金属离子对MaChi3表现为不同程度的激活或抑制作用。Mg2+和Ca2+对其酶活力具有激活作用,相对酶活力分别为117.7%和107.3%。Zn2+和Ba2+对MaChi3的酶活力几乎无影响。而K+、Ag+、Co2+、Fe2+、Cu2+和Fe3+对MaChi3的酶活力呈现出不同程度的抑制作用,相对酶活力降低至43.3%~87.2% (表1)。
表 1 化学试剂和金属离子对重组酶 MaChi3 酶活力的影响Table 1 Effects of metal ions and chemical reagents on activity of recombinant chitinase MaChi3化学试剂
Chemical reagent终浓度/体积分数
Final concentration/
Volume fraction相对酶活力
Relative activity/
%对照 Control 100 尿素 Urea 200 mmol·L−1 70.9±0.8 乙二胺四乙酸 EDTA 10 mmol·L−1 94.2±1.6 十二烷基硫酸钠 SDS 10 mmol·L−1 58.1±0. 5 吐温20 Tween 20 1% 90.4±0.8 吐温40 Tween 40 1% 104.1±1.6 吐温60 Tween 60 1% 79.6±1.0 吐温80 Tween 80 1% 108.4±0.8 曲拉通 X-100 Triton X-100 1% 101.0±0.9 钾离子 K+ 1 mmol·L−1 86.4±0.7 银离子 Ag+ 1 mmol·L−1 43.3±0.4 镁离子 Mg2+ 1 mmol·L−1 117.7±0.7 亚铁离子 Fe2+ 1 mmol·L−1 65.4±0.7 钙离子 Ca2+ 1 mmol·L−1 107.3±0.4 钴离子 Co2+ 1 mmol·L−1 87.2±0.7 钡离子 Ba2+ 1 mmol·L−1 99.5±1.3 锌离子 Zn2+ 1 mmol·L−1 97.6±1.1 铜离子 Cu2+ 1 mmol·L−1 45.0±0.7 铁离子 Fe3+ 1 mmol·L−1 59.3±1.1 2.3.4 MaChi3的底物特异性和动力学参数
分别以α-几丁质、胶体几丁质、淀粉、纤维素、虾壳粉和不同脱乙酰度的壳聚糖作为底物,研究重组几丁质酶MaChi3的底物特异性。如图5所示,MaChi3在催化胶体几丁质水解时表现出了最高的酶活力 (2.24 U·mg−1)。但是,以α-几丁质粉末作为底物时,酶活力仅为0.59 U·mg−1,前者约是后者的3.8倍。当MaChi3以虾壳粉为底物时,酶活力为0.84 U·mg−1,与其催化α-几丁质水解的活性相当。以50%脱乙酰度的壳聚糖为底物时,MaChi3展现了较高的水解能力 (1.66 U·mg−1)。但是,随着脱乙酰度的增加水解效率逐渐降低。以淀粉和纤维素为底物时,酶活力均较低,分别为0.40和0.39 U·mg−1。此外,以胶体几丁质作为底物,MaChi3的Km和Vmax值分别为5.93 mg·mL−1和8.58 μmol·(min·mg)−1。
2.3.5 SEM分析
为了进一步验证重组几丁质酶MaChi3对几丁质底物的水解能力,对酶解前及酶解后的α-几丁质和胶体几丁质进行SEM观察。α-几丁质的纤维结构紧密、表面略光滑 (图6-a)。经MaChi3酶解后,表面微观结构被破坏,变得粗糙、不平整,并出现大小不均匀的碎片附着在表面 (图6-b)。α-几丁质被制成胶体几丁质后,纤维结构变得松散 (图6-c),暴露出更多的酶切位点,因而经MaChi3酶解后,表面变得更加粗糙和褶皱,有更多的小碎片附着在表面 (图6-d)。以上结果表明,重组几丁质酶MaChi3对几丁质具有水解活力,并对胶体几丁质的水解活力更高,该结论与底物特异性结果 (图5) 一致。
2.3.6 N, N-二乙酰基壳二糖的制备
如图7-a所示,在反应初始阶段,重组几丁质酶MaChi3快速地将胶体几丁质水解为 (GlcNAc)2,同时生成了少量的GlcNAc。反应24 h后,(GlcNAc)2的产率基本保持稳定,为285.5 mg·g−1几丁质,GlcNAc的产率为39.8 mg·g−1几丁质,考虑到MaChi3的热稳定性 (图3-b),此时几丁质酶可能已经失去了活性。
3. 讨论
虾、蟹等甲壳类动物在生产加工过程中会生成大量的废弃物,其中,外壳占废物干质量的20%~58%。这些外壳中富含蛋白质 (20%~40%)、矿物质 (30%~60%) 和几丁质 (20%~40%) 等成分。在工业上,虾、蟹的外壳是生产几丁质的主要原料。几丁质经水解后得到的N-乙酰壳寡糖和单糖具有多种生物活性,已被应用于多个领域。因此,从甲壳类动物的壳中提取几丁质并将其转化为更具有应用价值的水解产物 [ 如(GlcNAc)2] 是缓解废弃物污染问题的有效途径。
在原核生物中,放线菌是最好的几丁质分解者之一,其体内含有丰富的几丁质降解酶系[31],可以利用几丁质作为唯一的碳源和氮源。本研究从放线菌橙黄小单孢菌的基因组DNA中克隆到了一条几丁质酶基因Machi3,经过生物信息学分析得知,MaChi3属于GH18家族几丁质酶。为了简化酶的分离纯化过程,并提高酶的产量[32],本研究将几丁质酶基因Machi3在E. coli BL21 (DE3) 中重组表达,经过纯化获得了电泳纯的重组几丁质酶。
不同微生物来源的几丁质酶对温度表现出不同的敏感性,如来源于橙色假交替单胞菌(Pseudoalteromonas aurantia) 的几丁质酶最适反应温度为60 ℃[33],而来源于冷喜几丁质节杆菌(Chitiniphilus shinanonensis) 的几丁质酶最适反应温度为50 ℃[34]。Huo等[35]克隆表达了来源于维氏气单胞菌 (Aeromonas veronii) B565的几丁质酶,该重组酶的最适反应温度为40 ℃,在50 ℃下孵育1 h后残余酶活力仅为20%。相比之下,本研究克隆表达的重组几丁质酶MaChi3具有较高的最适反应温度和良好的热稳定性,为拓宽几丁质酶的应用奠定了良好的基础。同时,不同来源的几丁质酶对酸碱适应性差异较大,已报道的几丁质酶最佳pH值范围为3.0~10.0[36-37]。由于几丁质酶在不同的氢离子(H+)浓度下解离程度不同,其高级结构中S-S键、疏水键及氢键受到影响,从而影响几丁质酶构象的变化而表现出不同的活性。Wang等[38]克隆、表达了假交替单胞菌 (Pseudoalteromonas sp.) DL-6来源的几丁质酶ChiA,其最适反应pH为9.0,在碱性条件下 (pH 8.0~12.0) 较稳定。Chen等[39]研究发现铜绿假单胞菌 (Pseudomonas aeruginosa) 几丁质酶的最适pH为4.5,在酸性条件下 (pH 4.5~6.0) 较稳定。而本研究的重组几丁质酶MaChi3是一种中性酶,并且在中性pH附近 (pH 6.0~9.0) 具有较好的稳定性。由表1可知,金属离子螯合剂EDTA对MaChi3的酶活性无显著影响,说明MaChi3为非金属依赖性蛋白酶。除了SDS外,MaChi3对所测试的化学试剂表现出了较强的抵抗力。据报道,SDS能够破坏蛋白质分子间和分子内的氢键,从而破坏蛋白质的二级和三级结构[40]。MaChi3与大多数几丁质酶相似,表现出严格的底物特异性[41-42],对胶体几丁质的酶活力高于其他底物,这可能与底物的物理形态和脱乙酰度有关。此外,本研究对MaChi3降解胶体几丁质的产物进行了分析。在反应过程中 (GlcNAc)2为主要产物,GlcNAc为唯一副产物,产物类型简单,有利于后续的分离纯化。(GlcNAc)2已被广泛用作药用生物活性物质以及抗菌剂和螯合剂。因此,MaChi3在制备 (GlcNAc)2 方面具有潜在应用价值。
4. 结论
本研究从海洋微生物橙黄小单孢菌的基因组DNA中克隆到了一条几丁质酶基因Machi3,并成功在大肠杆菌中表达。MaChi3的相对分子质量为47 kD,为新型的GH18家族几丁质酶。MaChi3的最适反应温度为55 ℃,最适反应pH为7.0。在体外的稳定性较好,在55 ℃下孵育1 h后仍能保持大部分的酶活力 (67%),在pH 6.0~9.0范围内孵育1 h后残余酶活力超过80%。吐温40、吐温80、Ca2+和Mg2+对MaChi3的酶活力有轻微的促进作用,Triton X-100、EDTA、Zn2+和Ba2+对MaChi3无显著性影响,其他化学试剂和金属离子对MaChi3表现为不同程度的抑制作用。该酶对α-几丁质、胶体几丁质、虾壳粉、不同脱乙酰度 (50%~95%) 的壳聚糖、淀粉以及纤维素均有一定的水解活力,其中胶体几丁质是MaChi3的最适底物,酶活力为2.24 U·mg−1。α-几丁质被制备成胶体几丁质后,纤维结构变的更加松散,因而更有利于MaChi3的水解作用。以胶体几丁质为底物时,MaChi3的Km和Vmax值分别为5.93 mg·mL−1和8.58 μmol·(min·mg)−1。此外,胶体几丁质经MaChi3水解后主要产物为 (GlcNAc)2,反应24 h后产率为285.5 mg·g−1几丁质,同时生成了少量的GlcNAc。MaChi3优良的酶学特性使其在 (GlcNAc)2制备方面有一定的应用前景。
-
表 1 不同群体短尾大眼鲷的碱基频率、中性检验和不对称分布参数
Table 1 Statistics of Tajima′s D, Fu′s Fs and mismatch distribution parameter of P.macracanthus
群体group 编号ID T/% C/% A/% G/% Tajima′s D Fu′s Fs mismatch distribution D P Fs P τ θ0 θ1 南沙Nansha NS 34.2 19.1 31.8 15.0 -1.798 0 0.014 2 -25.346 8 0.000 0 3.675 7 1.453 7 99 999.000 0 北部湾Beibu Gulf BG 34.1 19.1 31.8 15.0 -1.696 4 0.022 7 -25.345 2 0.000 0 5.718 7 0.000 0 99 999.000 0 三亚Sanya SY 34.1 19.1 31.8 15.0 -1.801 9 0.015 0 -25.642 2 0.000 0 4.500 0 0.000 0 99 999.000 0 茂名Maoming MM 34.1 19.1 31.8 15.0 -1.641 3 0.031 0 -25.779 2 0.000 0 4.343 7 0.001 7 99 999.000 0 珠海Zhuhai ZH 34.1 19.1 31.8 15.0 -1.730 1 0.020 9 -25.597 2 0.000 0 4.679 6 0.014 0 143.359 3 汕头Shantou ST 34.1 19.1 31.7 15.0 -1.704 2 0.024 5 -25.104 1 0.000 0 4.187 5 1.221 4 260.625 0 平均average 34.1 19.1 31.8 15.0 -2.062 6 0.000 3 -25.101 2 0.000 9 4.931 6 0.008 7 99 999.000 0 表 2 短尾大眼鲷样本信息及遗传多样性参数
Table 2 Sampling information and descriptive statistics of genetic diversity of P.macracanthus
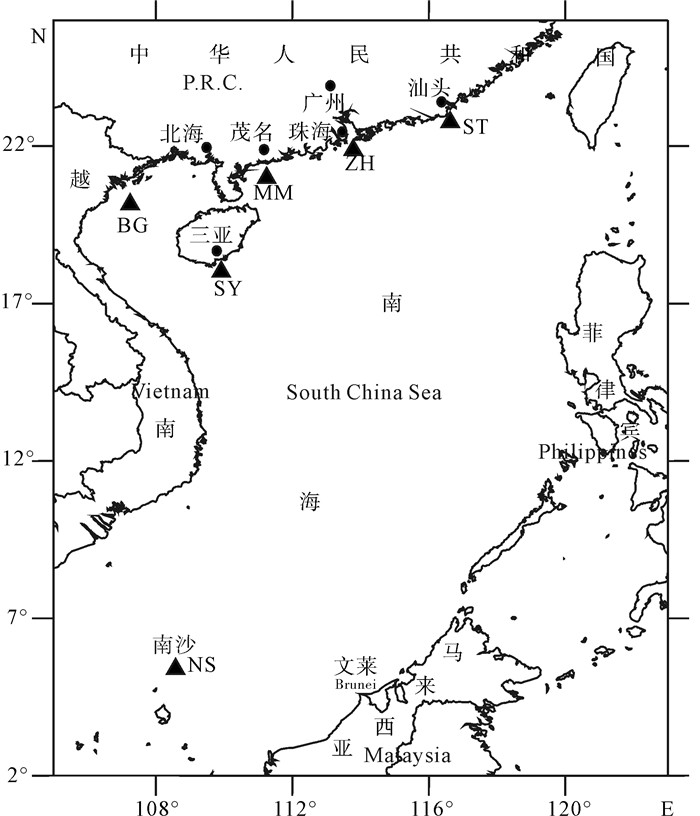
编号ID 群体group 经度longitude 纬度latitude 采样数量quantity of sample 单倍型数(私有单倍型数) No.of haplotypes (private) 多态位点(S) segregating site 单倍型多样性(h) haplotype diversity 核苷酸多样性(π) nucleotide diverity 平均核苷酸差异(k) mean number of nucleotide differences NS 南沙Nansha 109.650E 6.037N 40 37(28) 49 0.994 9±0.007 5 0.060 6±0.032 9 5.698 7±2.788 8 BG 北部湾Beibu Gulf 107.483E 20.427N 46 42(37) 49 0.997 1±0.005 7 0.060 9±0.032 9 5.725 6±2.792 8 SY 三亚Sanya 110.135E 18.075N 37 30(26) 39 0.989 5±0.009 2 0.048 9±0.027 3 4.603 6±2.311 9 MM 茂名Maoming 111.548E 21.132N 34 31(23) 32 0.992 9±0.009 9 0.044 7±0.025 3 4.210 3±2.142 9 ZH 珠海Zhuhai 113.774E 22.353N 37 29(23) 37 0.980 5±0.013 5 0.048 2±0.026 9 4.531 5±2.280 2 ST 汕头Shantou 116.932E 23.062N 30 28(21) 42 0.993 1±0.011 8 0.060 7±0.033 3 5.710 3±2.813 9 平均average 224 172(158) 94 0.990 3±0.002 3 0.094 2±0.048 1 9.140 7±4.218 3 表 3 短尾大眼鲷的遗传分化系数Fst(对角线下)和P值(对角线上)
Table 3 Pairwise Fst (below diagonal) and P value of population differentiation (above diagonal) of P.macracanthus
南沙NS 北部湾BG 三亚SY 茂名MM 珠海ZH 汕头ST 南沙NS 0.040 0 0.135 7 0.036 1 0.324 2 0.958 9 北部湾BG 0.013 2 0.607 4 0.669 9 0.652 3 0.044 9 三亚SY 0.008 1 -0.002 9 0.793 9 0.976 5 0.338 8 茂名MM 0.017 1 -0.003 4 -0.006 9 0.832 0 0.015 6 珠海ZH 0.002 4 -0.003 7 -0.011 4 -0.008 0 0.238 2 汕头ST -0.012 6 0.014 6 0.001 8 0.024 0 0.004 9 表 4 不同群体短尾大眼鲷遗传差异的AMOVA多样性分析
Table 4 Analysis of molecular variance (AMOVA) among different groups of P.macracanthus
变异来源source of variation 自由度degrees of freedom 平方和sum of squares 变异组成variance components 变异百分比percentage of variation F P 群体间among populations 5 14.087 0.007 1 0.28 群体内within populations 218 556.654 2.553 5 99.72 总计total 223 570.741 2.560 6 100 Fst=0.002 8 0.207 9 -
[1] SOKOLOVSKAYA T G, SOKOLOVSKII A S, SOBOLEVSKII E I. A list of fishes of Peter the Great Bay (the Sea of Japan)[J]. J Ichthyol, 1998, 38(1): 1-11. https://www.semanticscholar.org/paper/A-LIST-OF-FISHES-OF-PETER-THE-GREAT-BAY-(THE-SEA-OF-Sokolovskaya-Sokolovskii/c65b189aad74d9abcdf1cd8052fe67600840f0b1
[2] 卢占晖, 朱增军, 林显鹏. 东海区短尾大眼鲷食性分析[J]. 海洋渔业, 2010, 32(2): 199-203. doi: 10.3969/j.issn.1004-2490.2010.02.014 [3] 张仁斋, 陆穗芬. 短尾大眼鲷卵及仔、稚鱼的发育[J]. 水产学报, 1982(3): 243-251. http://scxuebao.ijournals.cn/scxuebao/article/abstract/19820306 [4] 何筱洁. 大眼鲷匹里虫病及其病原体的研究——包括匹里虫属一新种的描述[J]. 水产学报, 1982(2): 97-106. http://scxuebao.ijournals.cn/scxuebao/article/abstract/19820202 [5] 孙典荣, 邱永松. 北部湾长尾大眼鲷生长和死亡参数估计[J]. 海洋湖沼通报, 2004(3): 27-34. doi: 10.3969/j.issn.1003-6482.2004.03.005 [6] LIU K M, CHENG Y L. Virtual population analysis of the big eye Priacanthus macracanthus in the waters off northeastern Taiwan[J]. Fish Res, 1999, 41(3): 243-254. doi: 10.1016/S0165-7836(99)00025-9
[7] LIU K M, HUNG K Y, CHEN C T. Reproductive biology of the big eye Priacanthus macracanthus in the north-eastern waters off Taiwan[J]. Fish Sci, 2001, 67(6): 835-838. doi: 10.1046/j.1444-2906.2001.00355.x
[8] BENJAKUL S, OUNGBHO K, VISESSANGUAN W, et al. Characteristics of gelatin from the skins of bigeye snapper, Priacanthus tayenus and Priacanthus macracanthus[J]. Food Chem, 2009, 116(2): 307-325. doi: 10.1016/j.foodchem.2009.02.063
[9] 孙冬芳, 李永振, 董丽娜. AFLP技术在鱼类种群分析中的应用[J]. 南方水产, 2009, 5(6): 71-75. doi: 10.3969/j.issn.1673-2227.2009.06.013 [10] 宁平, 俞存根, 虞聪达, 等. 浙江南部外海渔场鱼类区系特征研究[J]. 浙江海洋学院学报, 2008, 27(3): 267-270. https://www.zhangqiaokeyan.com/academic-journal-cn_journal-zhejiang-ocean-university-natural-science_thesis/0201248287722.html [11] KOICHIRO T, GLEN S, DANIEL P, et. al. MEGA6: Molecular evolutionary genetics analysis version 6.0[J]. Mol Biol Evol, 2013, 30(12): 2725-2729. doi: 10.1093/molbev/mst197
[12] LIBRADO P, ROZAS J. DnaSP v5: a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 2009, 25(11): 1451-1452. doi: 10.1093/bioinformatics/btp187
[13] EXCOFFIER L, LAVAL G, SCHNEIDER S. Arlequin ver. 3.0: an integrated software package for population genetics data analysis[J]. Evol Bioinformatics, 2005, 1: 47-50. https://www.researchgate.net/publication/24238444_ARLEQUIN_Ver_30_An_integrated_software_package_for_population_genetic_data_analysis
[14] NEI M. Molecular evolutionary genetics[M]. New York: Columbia University Press, 1987: 153-187. doi: 10.7312/nei-92038
[15] TAJIMA F. Evolutionary relationship of DNA sequences in finite populations[J]. Genetics, 1983, 105(2): 437-467. doi: 10.1093/genetics/105.2.437
[16] WRIGHT S. The genetical structure of populations[J]. Annals of Eugenics, 1951, 15(1): 323-354. doi: 10.1111/j.1469-1809.1949.tb02451.x
[17] WRIGHT S. Evolution and the genetics of populations[M]//Variability within and among natural populations Vol. 4, Chicago: University of Chicago Press, 1978: 179-182. https://www.semanticscholar.org/paper/Evolution-and-the-Genetics-of-Populations%2C-Volume-Curnow-Wright/7ffa4dd7ecfb20e985c073866956be97764d86a1
[18] FREELAND J R. Molecular ecology[M]. Chichester: John Wiley & Sons Ltd, 2005: 112-116. https://onlinelibrary.wiley.com/toc/1365294x/2005/14/1
[19] BANDELT H J, FORSTER P, ROHL A. Median joining networks for inferring intraspecific phylogenics[J]. Mol Biol Evol, 1999, 16(1) : 37-48. doi: 10.1093/oxfordjournals.molbev.a026036
[20] ROGERS A R, HARPENDING H. Population growth makes waves in the distribution of pairwise genetic differences[J]. Mol Biol Evol, 1992, 9(3): 552-569. doi: 10.1093/oxfordjournals.molbev.a040727
[21] LI W H. Molecular evolution[M]. Sunderland, Massachusetts, USA: Sinauer Associates, Inc. Publishers, 1997: 59-63.
[22] HARPENDING H C. Signature of ancient population growth in a low-resolution mitochondrial DNA mismatch distribution[J]. Hum Biol, 1994, 66(4): 591-600. https://pubmed.ncbi.nlm.nih.gov/8088750/
[23] LEE W J, CONROY J, HOWELL W H, et al. Structure and evolution of teleost mitochondrial control region[J]. Mol Evol, 1995, 41(4): 54-66. doi: 10.1007/BF00174041
[24] RAMOS-ONSINS S E, ROZAS J. Statistical properties of new neutrality tests against population growth[J]. Mol Biol Evol, 2002, 19(12): 2092-2100. doi: 10.1093/oxfordjournals.molbev.a004034
[25] TAJIMA F. Statistical-method for testing the neutral mutation hypothesis by DNA polymorphism[J]. Genetics, 1989, 123(3): 585-595. doi: 10.1093/genetics/123.3.585
[26] FU Y X. Statistical tests of neutrality of mutations against population growth, hitchhiking and background selection[J]. Genetics, 1997, 147(2): 915-925. doi: 10.1093/genetics/147.2.915
[27] 牛素芳, 苏永全, 王军, 等. 福建近海蓝圆鲹群体遗传结构分析[J]. 厦门大学学报: 自然科学版, 2012, 51(4): 759-766. https://www.cnki.com.cn/Article/CJFDTotal-XDZK201204024.htm [28] 牛素芳, 苏永全, 王军, 等. 福建近海竹筴鱼线粒体DNA控制区和细胞色素b遗传多态性[J]. 中国水产科学, 2011, 19(1): 66-74. https://www.doc88.com/p-9734747774803.html [29] LIU J X, GAO T X, WU S F, et al. Pleistocene isolation in the Northwestern Pacific marginal seas and limited dispersal in a marine fish, Chelon haematocheilus (Temminck & Schlegel, 1845)[J]. Mol Ecol, 2007, 16(2): 275-288. doi: 10.1111/j.1365-294X.2006.03140.x
[30] 苏天凤, 江世贵, 马之明, 等. 二长棘犁齿鲷线粒体DNA控制区结构和进化[J]. 南方水产科学, 2012, 8(6): 1-8. doi: 10.3969/j.issn.2095-0780.2012.06.001 [31] 罗旭光, 齐景伟, 安晓萍, 等. 内蒙古达里湖及岗更湖东北雅罗鱼D-loop基因序列及多样性研究[J]. 南方水产科学, 2014, 10(2): 92-96. doi: 10.3969/j.issn.2095-0780.2014.02.014 [32] TUDELA S, GARCÍA-MARÍN J L, PLA C. Genetic structure of the European anchovy, Engraulis encrasicolus, in the north-west Mediterranean [J]. J Exp Mar Biol Ecol, 1999, 234(1): 95-109. doi: 10.1016/S0022-0981(98)00142-7
[33] 张东亚, 汪登强, 刘绍平, 等. 怒江濒危鱼类缺须盆唇鱼基于线粒体Cyt b序列的群体遗传结构分析[J]. 中国水产科学, 2009, 17(4): 477-486. doi: 10.3321/j.issn:1005-8737.2009.04.002 [34] 刘进贤, 高天翔, 吴世芳, 等. 梭鱼的分子系统地理学研究——晚更新世西北太平洋边缘海隔离分化及其有限的扩散能力[J]. 中国海洋大学学报: 自然科学版, 2007, 37(6): 931-938. doi: 10.16441/j.cnki.hdxb.2007.06.014 [35] AVISE J C. Phylogeography: the history and formation of species[M]. Cambridge, Massachusets: Harvard Unversity Press, 2000. https://www.nature.com/articles/6887654
[36] HEWITT G M. The genetic legacy of the Quaternary ice age[J]. Narture, 2000, 405(6789): 907-913. https://www.nature.com/articles/35016000
[37] DYNESIUS M, JANSSON R. Evolutionary consequences of changes in species′ geographical distribution driven by Milankovitch climate oscillations[J]. Proc Natl Acad Sci USA, 2000, 97(16): 9115-9120. doi: 10.1073/pnas.97.16.9115
[38] LAMBECK K, HONZA E. Global tectonics, and formation of marginal basins: role of the westerm Pacific[J]. Episodes, 1991, 14: 224-230. doi: 10.18814/epiiugs/1991/v14i3/005
[39] 杨海军, 刘秦玉. 南海海洋环流研究综述[J]. 地球科学进展, 1998(4): 47-51. doi: 10.3321/j.issn:1001-8166.1998.04.007 [40] 苏纪兰. 南海环流动力机制研究综述[J]. 海洋学报: 中文版, 2005, 27(6): 3-10. http://www.hyxb.org.cn/aos/ch/reader/view_abstract.aspx?file_no=20050601&st=alljournals [41] 管秉贤, 袁耀初. 中国近海及其附近海域若干涡旋研究综述Ⅰ. 南海和台湾以东海域[J]. 海洋学报: 中文版, 2006, 28(3): 1-16. https://qikan.cqvip.com/Qikan/Article/Detail?id=21970714 [42] 孙冬芳, 董丽娜, 李永振, 等. 南海北部海域多齿蛇鲻的种群分析[J]. 水产学报, 2010, 34(9): 1387-1394. doi: 10.3724/SP.J.1231.2010.06975 [43] 孙冬芳, 卢伟华, 艾红, 等. 中沙和南沙群岛密点胡椒鲷的种群分析[J]. 广东农业科学, 2010(5): 4-7. doi: 10.3969/j.issn.1004-874X.2010.05.002 [44] 董丽娜. 南海北部陆架区金线鱼(Nemipterus virgatus)种群分子分析[D]. 上海: 上海海洋大学, 2011. https://cdmd.cnki.com.cn/Article/CDMD-10264-1011304986.htm [45] 李玉芳, 李永振, 陈国宝, 等. 基于线粒体Cyt b基因的黄鳍马面鲀种群分析[J]. 水产学报, 2014, 38(1): 23-32. doi: 10.3724/SP.J.1231.2014.48864




 下载:
下载:






 粤公网安备 44010502001741号
粤公网安备 44010502001741号
