Sequence polymorphism of 5′-flanking regions of GHSR genes and genetic diversity of two populations of Nile tilapia (Oreochromis niloticus)
-
摘要:
该试验以快长尼罗罗非鱼(Oreochromis niloticus)和普通尼罗罗非鱼2个群体为研究对象,通过PCR扩增与测序,获得GHSR基因5′侧翼区序列77条,片段大小为1 217 bp。共检测出变异位点57个,包括12个插入/缺失位点和45个多态性位点。共发现16种单倍型,其中Hap2可能为原始单倍型。16种单倍型在进化树中聚为A和B 2支,A支中有11种单倍型,在普通群体和快长群体中均有分布,但快长群体中所占比例较高;B支中有5种单倍型,均分布于普通群体中。遗传多样性参数显示普通尼罗罗非鱼群体的核苷酸多样性(Pi)、单倍型多样性(Hd)和平均核苷酸差异数(K)都高于快长尼罗罗非鱼群体。AMOVA分析结果显示快长和普通尼罗罗非鱼2个群体之间的Fst为-0.200 0(P >0.1),表明2个群体间遗传分化不明显,且2个群体的遗传差异主要来自群体内(120%)。
Abstract:We obtained 77 sequences of 5′-flanking regions of GHSR genes from two populations (fast-growth group and normal-growth group) of Nile tilapia (Oreochromis niloticus) by PCR amplification and sequencing method, and detected 57 variation sites (12 inserting or deleting sites and 45 polymorphic sites) and 16 kinds of haplotypes. Hap2 might be the original haplotype. There were two branches (A and B) in the phylogenetic tree of 16 kinds of haplotypes. Branch A contained 11 kinds of haplotypes which could be found in both groups but showed higher-frequency distribution in fast-growth group; while Branch B contained five kinds of haplotypes which only distributed in normal-growth group. The nucleotide diversity (Pi), haplotype diversity (Hd) and average number of nucleotide differences (K) in fast-growth group were higher than those in normal-growth group. The Fst value (-0.200 0, P >0.1) and genetic difference (120%, mainly from intragroup) between the two groups suggest that no genetic differentiation exist between the two groups.
-
Keywords:
- Oreochromis niloticus /
- GHSR gene /
- 5′-flanking region /
- polymorphism /
- genetic diversity
-
尼罗罗非鱼(Oreochromis niloticus)俗称非洲鲫鱼,隶属于鲈形目、丽鱼科、罗非鱼属。原产于非洲,由于其具有生长快、繁殖力强、抗病力强、食性杂、耐低氧、群体产量高和适应高密度养殖等特点,1976年联合国粮农组织(FAO)向世界各国推广这一优良养殖品种,目前已经成为养殖区域分布最广的淡水养殖品种[1-3]。中国罗非鱼产量约占世界罗非鱼产量的45%,目前主要养殖品种为尼罗罗非鱼吉富品系。
生长是鱼类养殖的重要经济性状,也是反映生产水平和经济效益的重要指标。然而,随着罗非鱼的大规模养殖,制种过程中近亲繁殖、种质退化现象比较普遍,快长等优良性状丢失的现象逐渐显现。选育出生长速度快的罗非鱼新品种已成为广大养殖业者的迫切需求,也是罗非鱼品种改良的重要方向之一。罗非鱼新品种通常是通过传统的选择和杂交育种途径获得,但传统的育种方法存在选育周期长和优良性状遗传不稳定等缺点;而利用候选基因法,筛选与数量性状密切相关的DNA标记进行分子标记辅助育种,则可以显著缩短育种周期,增加选择强度,有利于定向选育,使优良性状稳定遗传,从而成为新兴的高效育种方法[4]。生长激素促分泌素受体(growth hormone secretagogue receptor,GHSR),是生长激素促分泌素(Ghrelin)的天然配体,Ghrelin是一种可促进机体摄食的脑肠肽[5]。GHSR基因是由2个外显子和1个内含子组成的G蛋白偶联蛋白受体[6]。它包括GHSR1a与GHSR1b 2个亚型,GHSR1a亚型含有366个氨基酸,有7个跨膜域,是主要的功能性受体[7]。GHSR1b亚型含有289个氨基酸,只有前5个跨膜域[8]。研究表明,GHSR与其配体Ghrelin结合后可激活磷脂酶C(phospholipase C,PLC),而PLC介导生成三磷酸肌醇(IP3)和甘油二脂(diacylglycerol,DAG);IP3促使钙离子(Ca2+)从IP3敏感的Ca2+储存池中释放出来,同时DAG激活PKC介导Ca2+从胞外进入,最后由Ca2+刺激生长激素(growth hormone,GH)的合成分泌,从而对机体的生长发育起到一定的促进作用[9]。GHSR与Ghrelin的结合还能促进摄食、调节能量代谢平衡、调节肠胃和心血管功能、参与细胞分化和免疫调节等[10-11]。GHSR基因作为筛选生长相关分子标记的重要候选基因,目前在畜禽类的研究中已有较多报道。张宝[12]在黄牛(Bos taurus)的GHSR基因中发现4个与生长相关的SNP位点,1个位于5′非翻译区,2个位于外显子1上,另一个位于3′非翻译区,而且都没有引起氨基酸的变化。JIN等[13]利用PCR单链构象多态性和DNA测序方法分析山羊(Capra hircus)的GHSR基因,发现2个位于GHSR基因外显子2上的生长相关SNP位点。方梅霞等[14]研究表明鸭(Cairina moschata)的GHSR基因中2个SNP位点T404C、G3427A与其生长及屠体相关性状显著相关,可以作为快长品种选育的重要分子标记。DARZI等[15]在肉鸡(Gallus domesticus)的GHSR基因上检测到1个与生长相关的SNP位点,其在调控肉鸡的生长性状中发挥着重要的作用。上述研究表明GHSR基因可作为筛选生长相关标记的重要候选基因。此外,俞菊华等[16]利用分段PCR的方法在建鲤(Cyprinus carpiovar Jian)的GHSR1a和GHSR1b基因上共找到了32个SNP位点,通过构建PCR-RFLP法对其中9个SNPs进行基因型检测,结果发现1a-C386T和1b-G159T与雌雄鱼鱼种和成鱼阶段增重分别呈极显著相关。但目前在尼罗罗非鱼中还没有GHSR生长相关分子遗传标记的报道。
真核生物基因表达调控所需的顺式作用元件(启动子、增强子、TATA盒和GC盒等)多位于5′侧翼非编码区,同时它也是反式作用因子的特异结合部位,因此,5′侧翼区的变异引起的生长性状的差异可能比编码区的变异引起的生长性状的差异更大[17]。该试验以快长和普通尼罗罗非鱼2个群体作为研究对象,PCR扩增、测序获得GHSR基因5′侧翼区序列,并分析GHSR基因序列多态性及2个群体的遗传多样性,为进一步研究GHSR基因多态性与生长之间的相关性提供基础材料,以期为快长罗非鱼分子辅助育种奠定理论基础。
1. 材料与方法
1.1 试验材料
快长尼罗罗非鱼群体采自广州番禺广东罗非鱼良种场,普通尼罗罗非鱼群体采自中国水产科学研究院珠江水产研究所高要水产良种基地。2个群体各取鳍条40份,100%的无水乙醇固定,-40 ℃保存备用。
1.2 试验方法
1.2.1 总DNA提取
采用TIANamp Genomic DNA Kit试剂盒(TIANGEN)从尼罗罗非鱼鳍条中提取总DNA,步骤参照试剂盒说明书。1%的琼脂糖凝胶电泳检测总DNA的提取效果,并用核酸蛋白定量仪(Bio-Rad Smartspic Plus)检测DNA纯度及浓度,-20 ℃保存备用。
1.2.2 PCR扩增及测序
以提取的总DNA为模板,根据笔者实验室克隆到的莫桑比克罗非鱼(O.mossambicus)GHSR基因序列(GenBank序列号EU910220),设计特异扩增引物扩增尼罗罗非鱼GHSR基因5′侧翼区序列。正向引物GHSR-335为5′-CCTAAGCCTGACTTTCCT-TGTC-3′;反向引物GHSR-SNP2为5′-GGAGGGCATTGTTGCCTCTG-3′,引物由上海立菲生物技术有限公司合成。50 μL PCR反应体系包括10×PCR Buffer(without Mg2+)5 μL,氯化镁(MgCl2)(25 mmol · L-1)5 μL,dNTP Mixture(10 mmol · L-1 each)1 μL,上下游引物(10 μmol · L-1)各1 μL,TaqDNA聚合酶(50 U · μL-1)0.5 μL(上海申能博彩),ddH2O 37.5 μL。扩增条件为94 ℃预变性3 min、94 ℃变性30 s、54 ℃退火30 s、72 ℃延伸90 s,30个循环,最后72 ℃再延伸7 min。扩增产物经1%的琼脂糖凝胶电泳检测,将目的片段大小的PCR产物直接送至上海立菲生物技术有限公司测序。
1.2.3 数据分析
用Vector NTI 8软件中的Conting Express程序对测序结果进行拼接并辅以手工校正[18]。应用Vector NTI 8软件中的Align X程序和Clustal X软件进行多重比对。用DnaSP 5.0软件计算序列的碱基组成,分析单倍型并输出变异位点及遗传多样性参数[19]。利用Arlequin 3.11软件中的分子方差分析(AMOVA)计算群体间的遗传分化[20]。用MEGA 5软件计算尼罗罗非鱼群体内与群体间的遗传距离,并利用拼接法(neighbor joining,NJ)构建单倍型的系统进化树[21]。利用Network 4.5软件构建尼罗罗非鱼GHSR基因5′侧翼区16种单倍型的网络进化图。
2. 结果
2.1 GHSR基因5′侧翼区片段的扩增
提取的尼罗罗非鱼鳍条总DNA,经核酸蛋白定量仪检测,所有样品的光密度OD260/OD280均为1.8~2.0,表明其纯度较高;经1.0%琼脂糖凝胶电泳检测,显示条带清晰明亮、DNA完整性较好,符合PCR反应要求,可用于下一步试验。通过特异引物扩增获得与预期大小一致的目的片段,大小为1 265 bp。
2.2 GHSR基因5′侧翼区序列的变异分析
测序结果通过Vector NTI8软件进行拼接,及手工校正,得到序列长度为1 217 bp的GHSR基因5′侧翼区部分序列。BLAST分析表明,所测得的序列与莫桑比克罗非鱼的GHSR基因5′侧翼区序列(Genbank序列号EU910220)有高度的相似性(99%),说明扩增获得的序列为尼罗罗非鱼GHSR基因5′侧翼区序列。由于80份PCR产物中有3份测序结果的峰图杂乱,对其进行删除后,对余下的77条GHSR基因5′侧翼区序列进行分析。MEGA 5.0软件分析表明,GHSR基因5′侧翼区基因片段中T、C、A、G碱基的平均含量分别为31.8%、20.5%、25.4%和22.3%,A+T的含量(57.2%)明显高于G+C的含量(42.8%)。
77条序列的1217个碱基中保守位点1 160个,变异位点57(包括12个插入/缺失位点和45个多态性位点),45个多态性位点中颠换多于转换,其中29个颠换位点(12个A/T颠换、5个G/T颠换、7个A/C颠换、5个G/C颠换),12个转换位点(8个A/G转换、4个T/C转换),4个位点既出现转换(A/G、T/C)又出现颠换(A/T、A/C)。45个多态性位点包括12个单核苷酸多态性位点(singleton variable sites),33个简约信息位点(parsimony informative sites)。77条序列共归为16种单倍型(Hap1~Hap16)。变异位点在不同单倍型中的分布情况见图 1。
![]() 图 1 尼罗罗非鱼GHSR基因5′侧翼区序列16种单倍型中变异位点分布Hap1~Hap16. 单倍型名称;上方数字表示变异位点在单倍型中的相应碱基位置,“.”表示与第一种单倍型Hap1有相同的碱基组成;“-”表示碱基缺失Figure 1. Location of mutative nucleotide acids in 16 haplotypes of 5′-flanking regions of GHSR genes from O.niloticusHap1~Hap16. names of 16 haplotypes; numbers on the top represrent locations of mutative nucleotide acids in the 16 haplotypes of O.niloticus; dots indicate the bases which are the same as haplotype 1(Hap1); transverse lines indicate the base deletion.
图 1 尼罗罗非鱼GHSR基因5′侧翼区序列16种单倍型中变异位点分布Hap1~Hap16. 单倍型名称;上方数字表示变异位点在单倍型中的相应碱基位置,“.”表示与第一种单倍型Hap1有相同的碱基组成;“-”表示碱基缺失Figure 1. Location of mutative nucleotide acids in 16 haplotypes of 5′-flanking regions of GHSR genes from O.niloticusHap1~Hap16. names of 16 haplotypes; numbers on the top represrent locations of mutative nucleotide acids in the 16 haplotypes of O.niloticus; dots indicate the bases which are the same as haplotype 1(Hap1); transverse lines indicate the base deletion.2.3 单倍型的分布及遗传距离
分析16种单倍型在2个群体中的分布,结果显示,共享单倍型比例较低,约为18.8%(3/16),其中Hap8和Hap2在2个群体中分布最为广泛。Hap5只存在于快长群体中,且占快长群体中个体总数的71.1%(27/38);Hap2在普通群体中占的比例最大,为33.3%(13/39)(表 1)。利用MEGA 5.0软件的Kimura-2-parameter模型计算尼罗罗非鱼群体中16种单倍型(Hap1~Hap16)的遗传距离(表 2),结果表明,尼罗罗非鱼16种单倍型之间的遗传距离为0~0.041 2,平均遗传距离为0.012 8;在16种单倍型中Hap5与Hap6之间及Hap4与Hap5、Hap6之间的遗传距离最小(0);Hap13与各单倍型之间的遗传距离较大,其中Hap5与Hap13之间的遗传距离最大(0.041 2)。
表 1 尼罗罗非鱼GHSR 5′侧翼区序列16种单倍型在2个群体中的分布Table 1. Distribution of 16 haplotypes of GHSR 5′-flanking regions from O.niloticus in fast- and normal-growth groups组别
group单倍型haplotype 合计
totalHap1 Hap2 Hap3 Hap4 Hap5 Hap6 Hap7 Hap8 Hap9 Hap10 Hap11 Hap12 Hap13 Hap14 Hap15 Hap16 快长群体fast-growth group 2 1 1 27 1 1 6 38 普通群体normal-growth group 6 13 2 9 1 1 1 1 1 1 1 1 39 合计total 6 15 3 1 27 1 1 15 1 1 1 1 1 1 1 1 77 表 2 基于尼罗罗非鱼GHSR 5′侧翼区序列单倍型的Kimura-2-parameter遗传距离Table 2. Kimura-2-parameter genetic distance based on haplotypes of GHSR gene fragments of O.niloticusHap1 Hap2 Hap3 Hap4 Hap5 Hap6 Hap7 Hap8 Hap9 Hap10 Hap11 Hap12 Hap13 Hap14 Hap15 Hap16 Hap1 - Hap2 0.000 8 - Hap3 0.001 7 0.000 8 - Hap4 0.003 3 0.001 4 0.003 3 - Hap5 0.003 3 0.001 4 0.003 3 0 - Hap6 0.003 3 0.001 4 0.003 3 0 0 - Hap7 0.011 6 0.012 4 0.011 6 0.008 3 0.008 3 0.008 3 - Hap8 0.002 5 0.003 3 0.002 5 0.000 8 0.000 8 0.000 8 0.009 1 - Hap9 0.000 8 0.001 7 0.002 5 0.002 5 0.002 5 0.002 5 0.010 8 0.001 7 - Hap10 0.000 8 0.001 7 0.002 5 0.002 5 0.002 5 0.002 5 0.010 8 0.003 3 0.001 7 - Hap11 0.001 7 0.002 5 0.003 3 0.005 0 0.005 0 0.005 0 0.010 8 0.004 1 0.002 5 0.002 5 - Hap12 0.021 8 0.020 9 0.021 8 0.025 3 0.025 2 0.025 2 0.028 6 0.024 3 0.028 6 0.022 6 0.021 8 - Hap13 0.027 8 0.026 9 0.027 8 0.030 5 0.041 2 0.031 2 0.032 9 0.030 3 0.028 6 0.028 6 0.029 5 0.029 5 - Hap14 0.019 2 0.020 1 0.020 9 0.022 8 0.022 6 0.022 6 0.025 2 0.021 8 0.020 1 0.020 1 0.019 3 0.019 2 0.012 5 - Hap15 0.024 3 0.025 2 0.025 2 0.027 9 0.027 8 0.027 8 0.029 5 0.026 9 0.025 2 0.025 2 0.024 3 0.024 3 0.014 2 0.006 6 - Hap16 0.018 4 0.019 2 0.019 2 0.021 9 0.021 8 0.021 8 0.023 5 0.020 9 0.019 2 0.019 2 0.018 4 0.018 4 0.015 0 0.007 5 0.007 5 - 2.4 单倍型的聚类分析
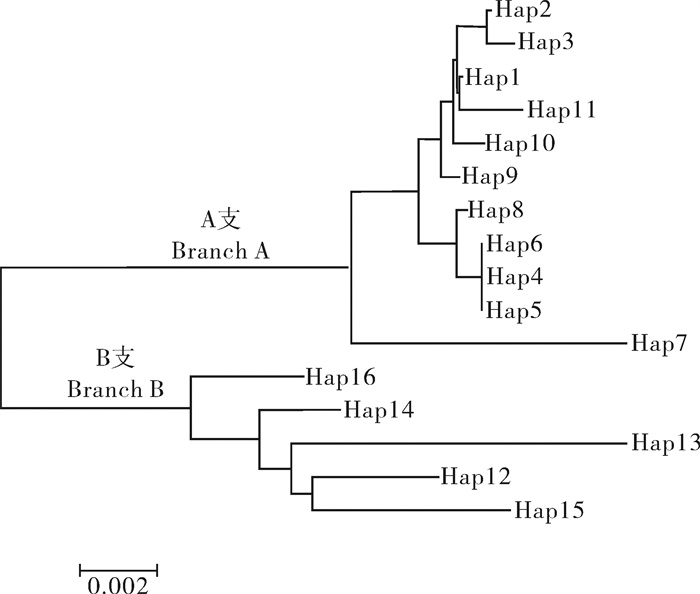
利用MEGA 5.0软件,根据Kimura-2-parameter进化参数模型,对上述所得到的16种单倍型(Hap1~Hap16)构建单倍型的NJ(Neighbor-Joining)分子系统进化树(图 2)。在此系统进化树中,16种单倍型可分为A支和B支,A支中有11种单倍型,而11种单倍型在普通群体和快长群体中都有分布,但快长群体中的分布比例较高。B支中有5种单倍型,均分布在普通群体中,在快长群体中没有分布;在A支中,Hap7与A支中的其他几个单倍型遗传距离相对较远,而B支中Hap13与其他几个单倍型的遗传距离较远。
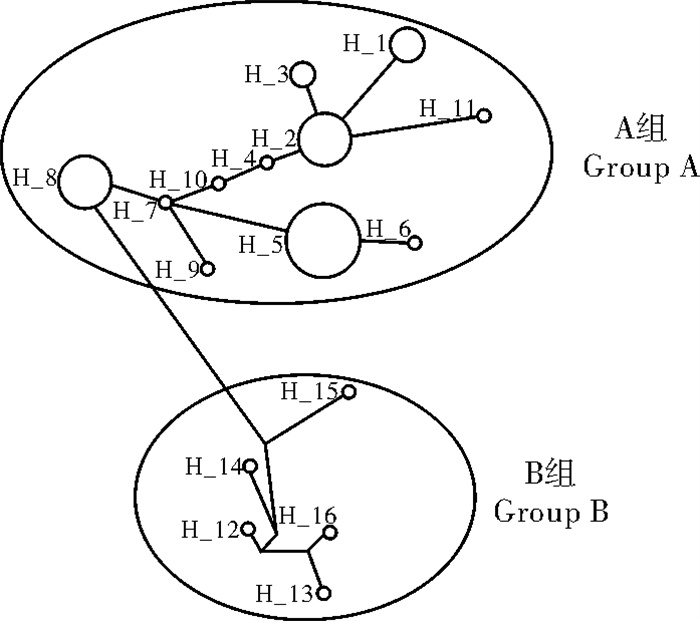
利用Network 4.5软件构建了尼罗罗非鱼GHSR基因16种单倍型的进化网络关系图(图 3)。16种单倍型可分为A和B 2组,B组包括了单倍型Hap12~16,A组包括了其他的单倍型,这与NJ分子系统进化树中16种单倍型的分支基本一致。此外,单倍型Hap2处于所有单倍型最为中心的位置,并且与其突变连接的单倍型数量最多;单倍型8是连接A、B 2组的唯一单倍型。
2.5 群体遗传多样性
利用Dnasp 5.0软件对快长与普通尼罗罗非鱼群体进行基于GHSR基因5′侧翼区序列的遗传多样性参数的分析,表明2个群体的遗传多样性存在差异(表 3)。从核苷酸多样性(Pi)看,普通群体的Pi较高,为0.016 6±0.005 4;而快长群体的Pi相对较低,为0.002 3±0.000 9。普通群体的单倍型多样性(Hd)和平均核苷酸差异数(K)高于快长群体。
表 3 尼罗罗非鱼群体内遗传多样性参数Table 3. Genetic diversity parameters in each population of O.niloticus群体
group样本数
number of samples单倍型数
number of haplotypes单倍型多样性
haplotype diversity (Hd±SD)平均核苷酸差异数
average number of nucleotide differences (K)核苷酸多样性
nucleotide diversity (Pi±SD)快长群体fast-growth group 39 7 0.862 0±0.239 0 3.091 2 0.002 3±0.000 9 普通群体normal-growth group 38 15 1.632 0±0.121 0 20.107 0 0.016 6±0.005 4 合计total 77 22 1.247 0±0.180 0 11.600 0 0.004 7±0.001 6 2.6 遗传距离和遗传分化
利用MEGA 5.0软件的Kimura-2-parameter模型计算尼罗罗非鱼群体内和群体间的遗传距离。结果显示快长群体和普通群体间的遗传距离为0.005 9,普通群体内的遗传距离大于快长群体内的遗传距离,分别为0.006 8和0.001 2。
AMOVA分析结果表明,快长和普通群体间的遗传差异主要来自群体内(120%),而群体间遗传差异为-20%,2个群体间的Fst为-0.200 0,P>0.1(表 4),表明这2个群体间差异不显著,遗传分化不明显。
表 4 基于尼罗罗非鱼GHSR基因5′侧翼区序列的AMOVA分析结果Table 4. AMOVA analysis based on GHSR genes 5′-flanking sequences of O.niloticus变异来源
source of variation自由度
d.f.平方和
sum of squares方差分量
variance components总变异百分比
percentage of variationFst P 群体间between populations 24 0 -0.088 9 Va -20 -0.200 0 >0.1 群体内within population 125 66.667 2 0.533 3 Vb 120 总数total 149 66.667 3 0.444 4 3. 讨论
3.1 尼罗罗非鱼GHSR基因5′侧翼区基因片段的多态性
鱼类生长受到脑(各种神经内分泌因子)-脑垂体(由生长激素细胞分泌生长激素)-肝脏(肝细胞产生类胰岛素生长因子)轴的调控,是多种生长相关基因共同作用的结果[7]。生长激素(GH)作为鱼类生长调节中最为重要的激素,其分泌主要受到生长激素释放激素(GHRH)和生长激素抑制素(somatostatin,SRIF)的调控,但最近研究表明GHSR参与组成GH分泌调节的另一个重要调节通路[22]。目前,在河豚(Spheroides nephelus)[23]、黑鲷(Acanthopagrus schlegeli)[24-25]、斜带石斑鱼(Epinephelus coioides)[26]、斑马鱼(Danio rerio)[27]、虹鳟(Oncorhynchus mykiss)[28]、荷那龙罗非鱼(Oreochromis hornorum)[29]等硬骨鱼类中已有GHSR基因克隆及其表达的相关研究报道,但有关鱼类GHSR基因多态性的研究却鲜有报道。关于家禽GHSR基因多态性的研究较多,NIE等[30]对4个地方品种鸭GHSR全基因(包括5′侧翼区)直接测序筛查SNP,共发现48个SNPs和5个插入缺失。FANG等[31]对全长为4 121 bp(包括侧翼区)的鸡GHSR基因进行SNP筛查,发现有1处6 bp的片段插入/缺失突变和37处SNPs。该研究采用PCR产物测序方法获得尼罗罗非鱼快长和普通群体1 217 bp的GHSR基因5′侧翼区序列片段,从中共检测出变异位点57个(包括12个插入/缺失位点和45个多态性位点),45个多态性位点包括12个单核苷酸多态性位点和33个简约信息位点。与以上家禽中GHSR全基因的多态性比较可发现,尼罗罗非鱼GHSR基因5′侧翼区序列具有更丰富的多态性。这可能是相对于家禽,罗非鱼的选育时间较短、选育强度较低的缘故。如对肉鸡个体增重性状的选育经历了近30年,且目前养殖的优良鸡品种一般至少经过6~8个世代的选育[32-34]。唐永凯等[35-36]通过对吉富罗非鱼生长相关基因——生长激素基因(GH)和肌细胞生长抑制素基因(MSTN)进行多态性分析,分别发现1个和3个SNPs位点,远少于该研究从尼罗罗非鱼GHSR基因中发现的SNPs位点,说明在尼罗罗非鱼生长相关基因中GHSR具有较丰富多态性,更适用于筛选生长相关分子标记。
3.2 单倍型的分布特点及进化分析
单倍型是在单核苷酸研究基础上发展起来的一种新方法,它是指一个DNA分子中具有统计学关联性的一类单核苷酸多态性位点,相对单个SNP可提供较多的多态信息[37]。该研究在2个尼罗罗非鱼群体中共发现16种单倍型(Hap1~Hap16),Hap5只存在于快长群体中,且占快长群体中个体总数的71.1%,Hap2在普通群体中占的比例最大(33.3%),从而可推测Hap5为快长尼罗罗非鱼群体中的主要单倍型,Hap2为普通群体中的主要单倍型。从表 2可知在16种单倍型中Hap5与Hap6之间及Hap4与Hap5、Hap6之间的遗传距离最小(都为0),Hap13与各单倍型之间的遗传距离都较大,这与单倍型NJ分子进化树(图 2)中结果一致。说明Hap4、Hap5及Hap6这3种单倍型可能具有相同的起源,且分化时间较短,而Hap13可能形成时间较早,由基因突变长期积累和组合产生。在进化树中,A支中有11种单倍型,且在普通群体和快长群体中均有分布,但快长群体中的分布比例较高,B支中有5种单倍型都只分布于普通群体中。这说明快长和普通尼罗罗非鱼群体之间GHSR基因5′侧翼区序列存在一定的差异,拥有各自特有的单倍型。虽然单凭GHSR基因5′侧翼区序列尚无法把2个尼罗罗非鱼群体完全分开,只能分成2个比较明显的分支,但在进一步的研究中,通过对GHSR全基因序列及对Ghrelin基因、IGF基因等生长相关基因SNP的联合分析,有可能筛选到尼罗罗非鱼快长分子标记并应用于进一步的品种改良中。
在物种系统进化的研究中,物种最为原始的单倍型是处于所有单倍型最为中心的位置,在物种的个体中出现频率也最高,并且与其突变连接的单倍型数量最多[38]。该研究发现,2个群体中出现频率最高的为Hap5、Hap8和Hap2,分别有27、15和15个体共享该单倍型(表 1)。进化网络关系图中,单倍型2处于所有单倍型最中心的位置,并且与其连接的单倍型数量最多,因此可认为单倍型2是最为原始的单倍型。另外,Hap8是A单倍型组中最靠近外群的单倍型,是连接A和B 2个单倍型组的唯一单倍型,且出现的频率较高(表 1),推测B组单倍型起源于Hap8。
3.3 群体遗传多样性及遗传分化
动物的遗传多样性是动物进化和适应环境的基础,群体的遗传多样性越丰富,适应环境变化的能力就越强,反之遗传多样性丧失对生存于不稳定环境中的动物群体则是极大的威胁[39]。该研究结果表明,普通尼罗罗非鱼群体的Pi、Hd和K都要高于快长尼罗罗非鱼群体,这说明选育在一定程度上导致了快长尼罗罗非鱼群体遗传多样性的下降,这不利于优良性状的稳定遗传,因此在今后的育种过程中应增加选育家系数量,避免近亲繁殖,保证选育群体足够的遗传多样性。群体内和群体间的遗传距离数据及AMOVA分析结果均表明,这2个群体之间遗传分化不明显,群体间遗传差异主要来自群体内(120%),这可能与快长群体是从吉富品系尼罗罗非鱼选育而来、选育代数仍较少有关。
综上所述,尼罗罗非鱼GHSR基因序列具有较丰富多态性,可作为生长相关分子遗传标记筛选的候选基因。快长尼罗罗非鱼和普通尼罗罗非鱼2个群体的遗传分化不明显,还未形成2个独立的地理种群;快长尼罗罗非鱼群体遗传多样性比普通群体的低,在进一步的选育中需采取必要的措施提高其遗传多样性。
-
图 1 尼罗罗非鱼GHSR基因5′侧翼区序列16种单倍型中变异位点分布
Hap1~Hap16. 单倍型名称;上方数字表示变异位点在单倍型中的相应碱基位置,“.”表示与第一种单倍型Hap1有相同的碱基组成;“-”表示碱基缺失
Figure 1. Location of mutative nucleotide acids in 16 haplotypes of 5′-flanking regions of GHSR genes from O.niloticus
Hap1~Hap16. names of 16 haplotypes; numbers on the top represrent locations of mutative nucleotide acids in the 16 haplotypes of O.niloticus; dots indicate the bases which are the same as haplotype 1(Hap1); transverse lines indicate the base deletion.
表 1 尼罗罗非鱼GHSR 5′侧翼区序列16种单倍型在2个群体中的分布
Table 1 Distribution of 16 haplotypes of GHSR 5′-flanking regions from O.niloticus in fast- and normal-growth groups
组别
group单倍型haplotype 合计
totalHap1 Hap2 Hap3 Hap4 Hap5 Hap6 Hap7 Hap8 Hap9 Hap10 Hap11 Hap12 Hap13 Hap14 Hap15 Hap16 快长群体fast-growth group 2 1 1 27 1 1 6 38 普通群体normal-growth group 6 13 2 9 1 1 1 1 1 1 1 1 39 合计total 6 15 3 1 27 1 1 15 1 1 1 1 1 1 1 1 77 表 2 基于尼罗罗非鱼GHSR 5′侧翼区序列单倍型的Kimura-2-parameter遗传距离
Table 2 Kimura-2-parameter genetic distance based on haplotypes of GHSR gene fragments of O.niloticus
Hap1 Hap2 Hap3 Hap4 Hap5 Hap6 Hap7 Hap8 Hap9 Hap10 Hap11 Hap12 Hap13 Hap14 Hap15 Hap16 Hap1 - Hap2 0.000 8 - Hap3 0.001 7 0.000 8 - Hap4 0.003 3 0.001 4 0.003 3 - Hap5 0.003 3 0.001 4 0.003 3 0 - Hap6 0.003 3 0.001 4 0.003 3 0 0 - Hap7 0.011 6 0.012 4 0.011 6 0.008 3 0.008 3 0.008 3 - Hap8 0.002 5 0.003 3 0.002 5 0.000 8 0.000 8 0.000 8 0.009 1 - Hap9 0.000 8 0.001 7 0.002 5 0.002 5 0.002 5 0.002 5 0.010 8 0.001 7 - Hap10 0.000 8 0.001 7 0.002 5 0.002 5 0.002 5 0.002 5 0.010 8 0.003 3 0.001 7 - Hap11 0.001 7 0.002 5 0.003 3 0.005 0 0.005 0 0.005 0 0.010 8 0.004 1 0.002 5 0.002 5 - Hap12 0.021 8 0.020 9 0.021 8 0.025 3 0.025 2 0.025 2 0.028 6 0.024 3 0.028 6 0.022 6 0.021 8 - Hap13 0.027 8 0.026 9 0.027 8 0.030 5 0.041 2 0.031 2 0.032 9 0.030 3 0.028 6 0.028 6 0.029 5 0.029 5 - Hap14 0.019 2 0.020 1 0.020 9 0.022 8 0.022 6 0.022 6 0.025 2 0.021 8 0.020 1 0.020 1 0.019 3 0.019 2 0.012 5 - Hap15 0.024 3 0.025 2 0.025 2 0.027 9 0.027 8 0.027 8 0.029 5 0.026 9 0.025 2 0.025 2 0.024 3 0.024 3 0.014 2 0.006 6 - Hap16 0.018 4 0.019 2 0.019 2 0.021 9 0.021 8 0.021 8 0.023 5 0.020 9 0.019 2 0.019 2 0.018 4 0.018 4 0.015 0 0.007 5 0.007 5 - 表 3 尼罗罗非鱼群体内遗传多样性参数
Table 3 Genetic diversity parameters in each population of O.niloticus
群体
group样本数
number of samples单倍型数
number of haplotypes单倍型多样性
haplotype diversity (Hd±SD)平均核苷酸差异数
average number of nucleotide differences (K)核苷酸多样性
nucleotide diversity (Pi±SD)快长群体fast-growth group 39 7 0.862 0±0.239 0 3.091 2 0.002 3±0.000 9 普通群体normal-growth group 38 15 1.632 0±0.121 0 20.107 0 0.016 6±0.005 4 合计total 77 22 1.247 0±0.180 0 11.600 0 0.004 7±0.001 6 表 4 基于尼罗罗非鱼GHSR基因5′侧翼区序列的AMOVA分析结果
Table 4 AMOVA analysis based on GHSR genes 5′-flanking sequences of O.niloticus
变异来源
source of variation自由度
d.f.平方和
sum of squares方差分量
variance components总变异百分比
percentage of variationFst P 群体间between populations 24 0 -0.088 9 Va -20 -0.200 0 >0.1 群体内within population 125 66.667 2 0.533 3 Vb 120 总数total 149 66.667 3 0.444 4 -
[1] KARAYUCEL I, TARIQ E, KARAYUCEL S, et al. Evidence for two unlinked "sex reversal" loci in the Nile tilapia, Oreochromis niloticus, and for linkage of one of these to the red body colour gene[J]. Aquaculture, 2004, 234(1/2/3/4): 51-63. https://www.semanticscholar.org/paper/Evidence-for-two-unlinked-sex-reversal-loci-in-the-Karay%C3%BCcel-Ezaz/c1703e4431bf1c4ba05071bcdd2257a594e24774
[2] 赵金良, 李思发. 尼罗罗非鱼育种学研究进展[J]. 水产科学, 2005, 24(26): 34-36. https://xueshu.baidu.com/usercenter/paper/show?paperid=1b2x02y0d55u02e0mw3g0ey0fx082834&site=xueshu_se&hitarticle=1 [3] 陈胜军, 李来好, 杨贤庆, 等. 中国罗非鱼产业现状分析及提高罗非鱼出口竞争的措施[J]. 南方水产, 2007, 3(1): 75-80. doi: 10.3969/j.issn.2095-0780.2007.01.013 [4] ALAIN V, DENIS M, MAGALI S C, et al. A review on SNP and other types of molecular marker and their use in animal genetics[J]. Genet Sel Evol, 2002, 34(18): 275-305. https://xueshu.baidu.com/usercenter/paper/show?paperid=e44a02bc9d25b26fa82254841ea1e604&site=xueshu_se&hitarticle=1
[5] ANDREA B, MICHAEL J, FISCHER M, et al. Genetic linkage and association of the growth hormone secretagogue receptor (Ghrelin Receptor) gene in human obesity[J]. Diabetes, 2005, 54(1): 259-267. doi: 10.2337/diabetes.54.1.259
[6] HOWARD A D, FEIGHNER S D, CULLY D F, et al. A receptor in pituitary and hypothalamus that functions in growth hormone release[J]. Science, 1996, 273(5277): 974-977. doi: 10.1126/science.273.5277.974
[7] ZHANG W L, YU G S, HUANG Y, et al. Expression of ghrelin and GHSR-1a in mammary glands of dairy goat during the lactation and the effects of ghrelin on regulation of mammary function in vitro[J]. Mol Cell Endocrinol, 2013, 370(1/2): 20-31. https://xueshu.baidu.com/usercenter/paper/show?paperid=48c1a4918049418d2908df50513c333e&site=xueshu_se&hitarticle=1
[8] KOJIMA M, HOSODA H, DATE Y, et al. Ghrelin is a growth-hormone-releasing acylated peptide from stomach[J]. Nature, 1999, 402(6762): 656-660. doi: 10.1038/45230
[9] SHUTO Y, SHIBASAKI T, OTAGRI A, et al. Hypothalamic growth hormone secretagogue receptor regulates growth hormone secretion, feeding, and adiposity[J]. J Clin Invest, 2002, 109(11): 1429-1436. doi: 10.1172/JCI0213300
[10] TOLLE V V, ZIZZARI P, TAMASETTO C, et al. In vivo and in vitro effects of ghrenlin/motilin-related peptide on growth hormone secretion in the rat[J]. Neuroendotinology, 2001, 73(35): 54-61. https://xueshu.baidu.com/usercenter/paper/show?paperid=92ecc090371c32b68171beb020393271&site=xueshu_se&hitarticle=1
[11] MCKEE K K, PALYHA O C, FEIGHNER S D, et al. Molecular analysis of rat pituitary and hypothalamic growth hormone secretagogue receptors[J]. Mol Endocrinol, 1997, 11(4): 415-423. doi: 10.1210/mend.11.4.9908
[12] 张宝. 6个黄牛品种ND5、GHSR基因遗传变异及其与生长性状关联分析[D]. 杨凌: 西北农林科技大学, 2008. https://xueshu.baidu.com/usercenter/paper/show?paperid=f9bbdff48e277758aad20583ed05b790&site=xueshu_se&hitarticle=1 [13] JIN Q J, FANG X T, LIU Y, et al. Novel SNPs of the caprine growth hormone secretagogue receptor (GHSR) gene and their association with growth traits in goats[J]. Biochem Genet, 2010, 11(5): 487-856. https://xueshu.baidu.com/usercenter/paper/show?paperid=3680beb6649116e1c6e6ef9dfc6b87c0&site=xueshu_se&hitarticle=1
[14] 方梅霞, 李莹, 徐海平, 等. GHRL及其受体GHSR基因多态性与鸭生长及屠体性状的关联性[J]. 畜牧兽医学报, 2011, 42(1): 18-24. https://xueshu.baidu.com/usercenter/paper/show?paperid=1s1e0je0kh6y0gd053120an0yj465065&site=xueshu_se&hitarticle=1 [15] NIARAMI M D, MASOUDI A A, TORSHIZI R V. Association of single nucleotide polymorphism of GHSR and TGFB2 genes with growth and body composition traits in sire and dam lines of a broiler chicken[J]. Anim Biotechnol, 2014, 25(1): 13-22. doi: 10.1080/10495398.2013.803478
[16] 俞菊华, 李红霞, 李建林. 建鲤生长激素促分泌素受体Ⅰ基因的特性及其与增重相关SNP位点的筛选[J]. 中国水产科学, 2012, 19(3): 390-398. http://qikan.cqvip.com/Qikan/Article/Detail?id=41910062 [17] LATCHMAN D S. Gene regulation: a eukaryotic perspective (Thirdedition)[M]. United Kingdom: Stanley Thornes Ltd, 1998: 23.
[18] 刘择隆, 于发科. Vector NTI Suite在分子生物学领域的应用[J]. 生物学杂志, 2006, 23(4): 46-49. doi: 10.3969/j.issn.2095-1736.2006.04.014 [19] LIBRADO P, ROZAS J. DnaSP v5: a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 2009, 25(11): 1451-1452. doi: 10.1093/bioinformatics/btp187
[20] GHANEM A, TORKHANI M, MAHJOUBI N, et al. Arlequin framework for multi-model, multi-time scale and heterogeneous time intergrators for structural transient dynamics[J]. Comput Method Appl M, 2013, 254(2): 292-308. https://xueshu.baidu.com/usercenter/paper/show?paperid=47fd78a66f9ddf3619b81a67ebcc6691&site=xueshu_se&hitarticle=1
[21] TAMURA K, DUDLEY J, NEI M, et al. MEGA 4: molecular evolutionary genetics analysis (MEGA) software version 4.0[J]. Mol Biol Evol, 2007, 24(8): 1596-1599. doi: 10.1093/molbev/msm092
[22] SMITH R G, LEONARD R, BAILEY A R T, et al. Growth hormone secretagogue receptor family members and ligands[J]. Endocrine, 2001, 14(1): 9-14. doi: 10.1385/ENDO:14:1:009
[23] PALYHA O C, FEIGHNER S D, TAN C P, et al. Ligand activation domain of human orphan growth hormone (GH) secretagogue receptor (GHS-R) conserved from pufferfish to humans[J]. Mol Endocrinol, 2000, 14(1): 160-169. doi: 10.1210/mend.14.1.0412
[24] CHAN C B, CHENG C H K. Identifcation and functional characterization of two alternatively spliced growth hormone secretagogue receptor transcripts from the pituitary of black seabream Acanthopagrus schlegeli[J]. Mol Cell Endocrinol, 2004, 214(2): 81-95. https://www.semanticscholar.org/paper/Identification-and-functional-characterization-of-Chan-Cheng/4bdfe547abd1de57c9324193773510bdc30d436c
[25] CHUNG M Y, CHI B C, CHRISTOPHER H K, et al. Isolation and characterization of the 5'-flanking region of the growth hormone secretagogue receptor gene from balck seabream Acanthopagrus schlegeli[J]. Mol Cell Endocrinol, 2004, 223(12): 5-15. https://europepmc.org/article/MED/15279906
[26] CHEN T, TANG Z, YAN A, et al. Molecular cloning and mRNA expression analysis of two GH secretagogue receptor transcripts in orange-spotted grouper (Epinephelus coioides)[J]. J Endocrinol, 2008, 199(2): 253-265. doi: 10.1677/JOE-08-0325
[27] OLSSON C, HOLBROOK J D, BOMPADRE G, et al. Identification of genes for the ghrelin and motilin receptors and a novel related gene in fish, and stimulation of intestinal motility in zebrafish (Danio rerio) by ghrelin and motilin[J]. Gen Comp Endocrinol, 2008, 155(1): 217-226. doi: 10.1016/j.ygcen.2007.05.016
[28] HIROYUKI K, TSUKASA M, MIKIYA M, et al. Ghrelin receptor (GHS-R)-like receptor and its genomic organisation in rainbow trout, Oncorhynchus mykiss[J]. Comp Biochem Physiol A, 2009, 153(4): 438-450. doi: 10.1016/j.cbpa.2009.04.612
[29] 高风英, 王欢, 叶星, 等. 荷那龙罗非鱼两种GHSR基因的克隆与序列分析[J]. 广东海洋大学学报, 2011, 31(4): 6-12. doi: 10.3969/j.issn.1673-9159.2011.04.002 [30] NIE Q, FANG M, XIE L, et al. Molecular Characterization of the ghrelin and ghrelin receptor genes and effects on fat deposition in chicken and duck[J]. J Biomed Biotechnol, 2009, 11(10): 1-12. https://xueshu.baidu.com/usercenter/paper/show?paperid=beb89381f87fd51a43a373226ae3dc3c&site=xueshu_se&hitarticle=1
[31] FANG M X, NIE Q H, LUO C, et al. Associations of GHSR gene polymorphisms with chicken growth and carcass traits[J]. Mol Biol Rep, 2010, 37(12): 423-428. https://xueshu.baidu.com/usercenter/paper/show?paperid=554e4cbb9512ca0bfcbf683d6871261a&site=xueshu_se&hitarticle=1
[32] HAVENSTEIN G B, FERKERT P R, GRIMES J L, et al. Comparison of the performance of 1966-versus 2003-type turkeys when fed representative 1966 and 2003 turkey diets: growth rate, livability, and feed conversion[J]. Poult Sci, 2007, 86(2): 232-240. doi: 10.1093/ps/86.2.232
[33] 王克华, 窦套存, 曲亮, 等. 优质鸡选育方案研究[J]. 中国家禽, 2012, 34(1): 7-10. doi: 10.3969/j.issn.1004-6364.2012.01.003 [34] 韦凤英, 陈宽维, 束婧婷, 等. 广西三黄鸡保护选育和利用研究[J]. 中国家禽, 2011, 33(13): 26-29. https://xueshu.baidu.com/usercenter/paper/show?paperid=a1d656675900e10834fefb750264f4d6&site=xueshu_se&hitarticle=1 [35] 唐永凯, 俞菊华, 徐跑, 等. 吉富罗非鱼GH基因的分离及其SNPs与增重的相关性[J]. 湖南农业大学学报: 自然科学版, 2012, 38(4): 422-425. https://xb.hunau.edu.cn/hnndzr/article/abstract/20120175?st=article_issue [36] 唐永凯, 李建林, 俞菊华, 等. 吉富罗非鱼MSTN基因结构及其多态性与生长性状的相关性[J]. 中国水产科学, 2010, 17(1): 44-51. https://www.zhangqiaokeyan.com/academic-journal-cn_journal-fishery-sciences-china_thesis/0201261550764.html [37] 方梅霞, 聂庆华, 何丹琳, 等. 四个鸡品种GHSR基因遗传多样性分析[C]//中国家禽科学研究进展: 第四次全国家禽科学学术讨论会论文集. 北京: 中国农业科学出版社, 2009, 312-317. https://xueshu.baidu.com/usercenter/paper/show?paperid=64ba28c5b20f8cb1df0376ec5d2c7e90&site=xueshu_se&hitarticle=1 [38] 周发林, 江世贵, 姜永杰, 等. 南海野生斑节对虾的线粒体16S rRNA基因序列单倍型分析[J]. 生态学杂志, 2008, 27(11): 1955-1959. https://xueshu.baidu.com/usercenter/paper/show?paperid=8075311d0e8ecae509586cd86e3b5ea6&site=xueshu_se&hitarticle=1 [39] 蒋宗良, 张明, 林勇, 等. 奥利亚罗非鱼线粒体基因组全序列测定与系统进化分析[J]. 中国水产科学, 2011, 18(4): 790-800. https://xueshu.baidu.com/usercenter/paper/show?paperid=6106f133afa4a5b8a90c0278fa367644&site=xueshu_se&hitarticle=1 -
期刊类型引用(8)
1. 马世豪,肖文富,解绶启,张健敏,董立学,文华,蒋明,郜卫华,田娟. 克氏原螯虾饲料亚麻酸与亚油酸的适宜比值. 水生生物学报. 2025(03): 106-118 .  百度学术
百度学术
2. 徐德峰,廖威龙,李彩虹,廖建萌,李欣. 冷藏凡纳滨对虾肌肉软化的生化特性及生物标记物. 广东海洋大学学报. 2024(01): 125-132 .  百度学术
百度学术
3. 邹宇凡,吴玮杰,白志毅,李典中,蒋军,程熙,李家乐. 克氏原螯虾三群体双列杂交组合生长性能和耐干露能力比较分析. 上海海洋大学学报. 2024(02): 361-370 .  百度学术
百度学术
4. 黄月,陈荔,钟传艳,陈果,龙晓文. 干露胁迫对中华绒螯蟹成体呼吸和能量代谢及抗氧化能力的影响. 水产学杂志. 2024(02): 61-69 .  百度学术
百度学术
5. 吴乐,李嘉尧,周文宗,成永旭. 氨氮短期胁迫与恢复对克氏原螯虾的影响. 水产科学. 2024(03): 390-399 .  百度学术
百度学术
6. 邹宇凡,吴玮杰,白志毅,冯建彬,吴敏,蒋军,李家乐. 5个群体克氏原螯虾生长和养殖生态适应性比较分析. 水产科学. 2024(03): 420-428 .  百度学术
百度学术
7. 苏禹,王力玄,孟泳岐,马源潮,鲁耀鹏,张泽龙,郑佩华,李军涛,冼健安,刘存歧,王冬梅. 环境因素对克氏原螯虾生长与繁育的影响. 中国饲料. 2024(21): 74-82 .  百度学术
百度学术
8. 郭子宇,鲁耀鹏. 克氏原螯虾活体运输技术研究进展. 河北渔业. 2022(09): 38-40+46 .  百度学术
百度学术
其他类型引用(7)




 下载:
下载:
 粤公网安备 44010502001741号
粤公网安备 44010502001741号
