Preliminary genetic analysis of conchocelis from 5 Porphyra species using microsatellite
-
摘要:
文章利用68对微卫星引物对坛紫菜(Porphyra haitanensis)、半叶紫菜(P.katadai)、条斑紫菜(P.yezoensis)、少精紫菜(P.oligospermatangia)和皱紫菜(P.crispata)5个紫菜属物种的丝状体进行初步的遗传分析,共筛选出20对高多态性适用引物,在10份紫菜丝状体样品扩增位点的多态性比例为95.24%,每对引物可扩增的等位基因位点数为1.22~4.05,平均为2.37;各样品之间的遗传多样性指数为0.254 6~0.899 4;不同物种的遗传距离为0.106 0~1.406 6,同一物种的丝状体材料优先聚类为一支,表明筛选出的微卫星引物具有良好的遗传分辨能力。
Abstract:We conducted a preliminary genetic analysis of conchocelis from 5 Porphyra species (P.haitanensis, P.katadai, P.yezoensis, P.oligospermatangia and P.crispate) by 68 microsatellite primer-pairs, and selected 20 microsatellite primer-pairs with high polymorphism. The polymorphism proportion among 10 Porphyra conchocelis reaches 95.24%, and the effective number of alleles for each primer is 1.22~4.05 (average 2.37). The genetic diversity index is 0.254 6~0.899 4 and the genetic distance between different species is 0.106 0~1.406 6. Conchocelis from the same species group into one cluster first, which reveals that the selected microsatellite primer-pairs have good genetic resolution power.
-
Keywords:
- Porphyra /
- microsatellites /
- allele loci /
- genetic variation
-
紫菜(Porphyra)属于红藻门、红毛菜科,已经报道有134个物种,广泛分布于从寒带到亚热带的潮间带海域。紫菜是一类重要的经济红藻,作为目前世界上人工养殖海藻中经济价值最高的种类,仅中、日、韩三国紫菜的初级加工品年产值就超过20×109美元[1]。中国是世界紫菜的主要生产国和出口国,主要栽培种类包括条斑紫菜(P.yezoensis)和坛紫菜(P.haitanensis)。长期以来紫菜的遗传育种研究以诱变育种、单性生殖、选择育种等经典育种技术为主,相对于蓬勃发展的动植物分子育种研究而言,分子标记技术尤其是共显性分子标记数量少、应用种类有限,制约了紫菜遗传改良工作的快速发展。微卫星又称为简单序列重复(simple sequence repeat,SSR),作为一种共显性遗传标记,具有多态性高、重复性好等优点[2],已被广泛应用到海带属(Laminaria; Saccharina)[3-4]、江蓠属(Gracilaria)[5]、龙须菜属(Gracilariopsis)[6]、紫菜属(Porphyra)[7-8]等重要大型海藻的遗传多样性、遗传图谱构建和QTL定位研究中。
皱紫菜(P.crispata)是中国南方沿海野生紫菜的主要种类之一,具有重要的经济价值和药用价值。研究表明,皱紫菜的高温耐受性较坛紫菜和条斑紫菜要强,对环境具有更强的适应能力[9]。目前中国已经进行皱紫菜人工栽培试验,增加紫菜栽培新种类对于解决坛紫菜栽培生产中因高温而出现“烂菜”等问题具有重要的意义。半叶紫菜(P.katadai)和少精紫菜(P.oligospermatangia)均为野生种类,主要分布于北方沿海地区,和条斑紫菜都属于雌雄同体的紫菜种类,也是紫菜属遗传学研究的良好试验材料。微卫星标记在紫菜属不同物种中的研究应用较少,关于皱紫菜、半叶紫菜和少精紫菜等紫菜属物种的微卫星标记尚未见报道。笔者利用微卫星DNA分子标记技术分析皱紫菜等5个紫菜属物种的遗传结构及种质,以便更有效地推动紫菜种质资源评价并辅助紫菜遗传改良,同时对微卫星标记在紫菜属物种遗传分析中的通用性进行探讨,以期为紫菜属的分子遗传学研究提供依据。
1. 材料与方法
1.1 试验材料
试验所用的紫菜属5个物种10个品系自由丝状体(表 1)由中国海洋大学汤晓荣老师惠赠,培养于中国海洋大学海洋生物遗传育种研究室。
表 1 此试验所用的10个品系紫菜丝状体信息表Table 1 Information of 10 lines of Porphyra conchocelis in this study序号
No.品系代码
line code物种名称
species name1 PH1 坛紫菜P.haitanensis 2 PH2 坛紫菜P.haitanensis 3 PH3 坛紫菜P.haitanensis 4 PH4 坛紫菜P.haitanensis 5 PK1 半叶紫菜P.katadai 6 PK2 半叶紫菜P.katadai 7 PY1 条斑紫菜P.yezoensis 8 PY2 条斑紫菜P.yezoensis 9 PO 少精紫菜P.oligospermatangia 10 PC 皱紫菜P.crispata 1.2 基因组DNA的提取
紫菜丝状体基因组DNA的提取均采用植物基因组DNA提取试剂盒(北京天根)法提取,用Nanodrop 1000分光光度计测定DNA的光密度(OD)和浓度,并在1.0%琼脂糖凝胶上电泳(100 V 30 min)检测,用JS-380A自动凝胶图像分析仪拍照并进一步检验DNA的质量和浓度。
1.3 微卫星引物合成及筛选
现有的文献报道紫菜微卫星引物共68对:25对引自SUN等[10]发布的微卫星引物,10对引自刘必谦等[11]发布的微卫星引物,11对引自ZUO[12]发布的微卫星引物,8对引自XIE等[13]发布的微卫星引物,14对引自KONG等[14]发布的微卫星引物,引物由华大基因公司合成。以5个不同物种的丝状体(PH2, PK2, PY2, PO和PC)DNA样品为模板,对所有引物进行PCR筛选,从中选择扩增稳定且条带清晰的微卫星引物(引物信息见表 2)。
表 2 20对微卫星引物序列和特异退火温度Table 2 Sequence of 20 pairs of microsatellite marker primers and specific annealing temperature for PCR amplification引物编号
primer No.引物序列(5′→3′)
primer sequence重复序列
repetitive sequence退火温度/℃
annealing temperature片段范围
fragment rangeGenBank登录号
GenBank accession No.参考文献
referenceSSR-ZC1 TTCGCTGCGTTTCACCTTACATTT (TGCG)6 60 205 AV434771 [10] ACAAGGCCAACCCGAACACA SSR-ZC2 GGCTGCGGCTGAGTCACAGA (AGC)8 60 235 AU194221 [10] GTCGCTCCAACTCCTCCTGCT SSR-ZC3 TGGTGCTGTCTTCCAACGAGTA (AGC)13 60 245 AU196122 [10] CGGCTGTCGCACCTCGTTATA SSR-ZC7 CGCTCAACCACTTCGTCAG (AAC)10+1 58 261 AU191538 [10] CATTGTTGGCGTTGTTGTCATA SSR-ZC9 CCGTCGTCAGCAGGAGCA (GGC)7 60 200 AV434560 [10] ATGTGAGAAGCCAGTAGGGAAAGT SSR-ZC10 ACTTCCATCGCTGTCTTCGCT (AGC)8 60 196 AU192094 [10] CTGAGCTGCGTGTTGTGGTT SSR-ZC12 TCAACCATCAGCCATACCGAC (ACC)11 58 192 AV435510 [10] GACATGTCCGCCACCTTGTA SSR-ZC15 TGACTTCCTCATCGACATTGT (AGC)8 55 319 AU187439 [10] GCCATAGTACATTTGTTGCTG SSR-ZC16 TACCAGGTCGACCAGGAGCA (AGC)11 60 291 AU193458 [10] TCCAACTCTGCAGTGTCCGTT SSR-ZC19 ACCTCCTCGGCTACTTCAGA (AGC)7 58 276 AV431181 [10] GGATACAACGCCTGCTCCAT SSR-ZC20 GGCAGCAGCCATGATGTA (AGC)8+8 55 339 AV433675 [10] CCGTCAGGCAGAAGAGAAT SSR-ZC23 CAAGGGCTACTGCTACTA CAA (AAC)17 55 249 AU195299 [10] TACAAAAAGACTCTCGTG GCA SSR-ZC25 CCGTGCTACTACGGCTA CAA (AAC)9+11 58 302 DN606134 [10] GTCCGGTGCAGGTTG TTCT SSR-ZC36 CCAACGATGGGTTTCTTCAA 55 300 [7] ACTTCATGCCCCTGCCGATG SSR-ZC38 GGACAAGGGGTAATGGCT (CA)17 50 212~226 DQ831155 [12] TGGAAAACTTCCTGGGTG SSR-ZC43 GGAATGCCTTGCACCTGG (GT)26 55 408~420 DQ831185 [12] CACTATTGACCGAATCCGCTAC SSR-ZC44 CGTGCGAGTCATAGTCTGCT (GTGA)26 55 191~200 DQ831194 [12] ACAGCCAGTGCAAGAACACC SSR-ZC47 CTAGACGGAGTGCGGCTGAC (CA)9 58 176~196 DQ831197 [12] GCTCCTCCACGAGCATCAGG SSR-ZC54 GGTGGCAGTGAAGCGAAACA (CG)7 60 229 DN607742 [13] ACCCAGCAAGTGCGTGAGC SSR-ZC61 AACAGAGATACGGAGAGC (GT)8..(TG)11 50 276 EU670707 [14] ATCAGATTGGACTTGCCT 1.4 PCR扩增及电泳
利用筛选出的微卫星引物对5个物种的10个丝状体品系进行PCR扩增。
PCR反应在PCR仪(BIOER)上进行。反应体系为20 μL,试剂终浓度分别为1.5 mmol·L-1,10×PCR buffer[含氯化镁(MgCl2)], 0.125 mmol·L-1dNTP, 20 ng模板DNA,0.05 U TaqDNA聚合酶,微卫星引物1.0 μL。
PCR反应程序为94 ℃变性5 min;接35个循环为94 ℃ 50 s,退火45 s(退火温度因引物而定),72 ℃ 1 min;72 ℃最后延伸10 min, 4 ℃保存,1.0%琼脂糖电泳,100 V 50 min,检测PCR产物。取部分选扩产物(3 μL)与等体积的上样缓冲液(3 μL)充分混匀,95 ℃变性5 min。点样6 μL,在6%变性聚丙烯酰胺凝胶上电泳,仪器为DYY-12C电泳仪(北京市六一仪器厂出品),电泳前进行30 min预电泳,最大功率恒功率电泳2 h。银染程序参照SANGUINETTI等[15]报道的方法。室温下自然干燥,用扫描仪扫描并保存图像。
1.5 数据统计分析
微卫星标记按共显性标记进行数据统计及分析。根据分子量大小对扩增结果读带,以二倍体形式记录,从大到小依次记为A,B,C……。利用POPGENE32(V 1.31) (ftp://ftp.microsoft.com/Softlib/MSLFILES/HPGL.EXE)软件计算各群体的观测有效等位基因数(Ne)、多态位点百分率(P)、基因多样性指数(Nei′s)、Shannon信息指数(I)、表观杂合度(Ho)和预期杂合度(He)、Nei′s标准遗传距离(D)、遗传相似度和遗传分化指数(Fst)。根据Nei′s标准遗传距离,利用MEGA 4.0 (http://www.megasoftware. net/)中的非加权平均算术法(UPGMA)对各种群进行聚类分析,自展检验1 000次。
2. 结果与分析
2.1 紫菜丝状体基因组DNA的提取
所提取的紫菜丝状体基因组DNA,OD260/OD280为1.7~1.9,质量浓度为20~40 ng·μL-1,没有DNA降解、RNA残留及蛋白质污染,可以满足微卫星遗传分析的需要(图 1)。
![]() 图 1 紫菜丝状体基因组DNA在1.0%的琼脂糖电泳检测结果M.DL 2 000分子量标准;1~4. PH1、PH2、PH3、PH4,坛紫菜; 5~6. PK1、PK2,半叶紫菜; 7~8. PY1、PY2, 条斑紫菜;9. PO,少精紫菜;10.PC, 皱紫菜Fig. 1 Electrophoresis in 1% agarosegel of genomic DNA obtained from conchocelis of PorphyraM. DL 2 000 DNA marker; 1~4. PH1, PH2, PH3, PH4, P.haitanensis; 5~6. PK1, PK2, P.katadai; 7~8. PY1, PY2, P.yezoensis; 9. PO, P.oligospermatangia; 10. PC, P.crispata
图 1 紫菜丝状体基因组DNA在1.0%的琼脂糖电泳检测结果M.DL 2 000分子量标准;1~4. PH1、PH2、PH3、PH4,坛紫菜; 5~6. PK1、PK2,半叶紫菜; 7~8. PY1、PY2, 条斑紫菜;9. PO,少精紫菜;10.PC, 皱紫菜Fig. 1 Electrophoresis in 1% agarosegel of genomic DNA obtained from conchocelis of PorphyraM. DL 2 000 DNA marker; 1~4. PH1, PH2, PH3, PH4, P.haitanensis; 5~6. PK1, PK2, P.katadai; 7~8. PY1, PY2, P.yezoensis; 9. PO, P.oligospermatangia; 10. PC, P.crispata2.2 引物筛选结果
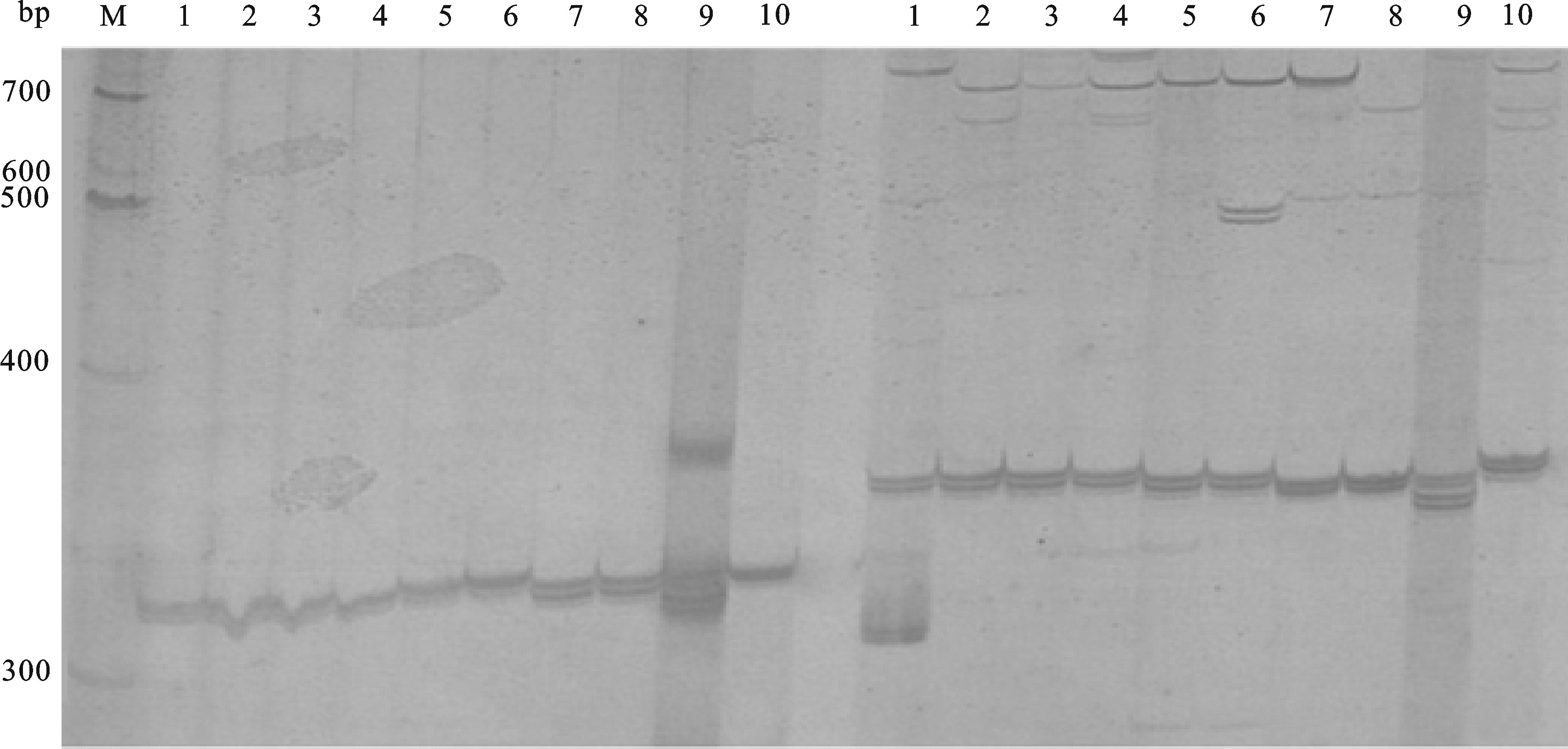
从68对微卫星引物中筛选出20对扩增稳定、多态性高的引物(表 2),其部分扩增结果见图 2。其中有14对引物在5个物种中均可扩增出稳定的条带,扩增片段大小为192~420 bp,且这14对引物中有10对可在10个丝状体中扩增出稳定的条带。
![]() 图 2 部分微卫星引物(左:SSR-ZC15;右:SSR-ZC20)在10个紫菜丝状体中的扩增结果M.DL100分子量标准;1~10. PH1,PH2,PH3,PH4,PK1,PK2,PY1,PY2,PO,PCFig. 2 Electrophoresis of part of microsatellite primer-pairs (L: SSR-ZC15; R: SSR-ZC20) in 10 lines of Porphyra conchocelisM. DNA maker DL100; 1~10. PH1, PH2, PH3, PH4, PK1, PK2, PY1, PY2, PO, PC
图 2 部分微卫星引物(左:SSR-ZC15;右:SSR-ZC20)在10个紫菜丝状体中的扩增结果M.DL100分子量标准;1~10. PH1,PH2,PH3,PH4,PK1,PK2,PY1,PY2,PO,PCFig. 2 Electrophoresis of part of microsatellite primer-pairs (L: SSR-ZC15; R: SSR-ZC20) in 10 lines of Porphyra conchocelisM. DNA maker DL100; 1~10. PH1, PH2, PH3, PH4, PK1, PK2, PY1, PY2, PO, PC2.3 有效等位基因数和遗传杂合度
10个紫菜丝状体个体间的平均Ne为2.37,平均He为0.550 9,I为0.899 9,Fst为0.400 0~1.000 0,平均为0.733 3。10个品系的紫菜丝状体具有良好的遗传多样性,且不同材料之间遗传分化较明显(表 3)。
表 3 20个微卫星的遗传多样性指数Table 3 Genetic diversity index of 20 microsatellite locilocus 表观杂合度
Ho预期杂合度
He有效等位基因数
NeShannon信息指数
I遗传分化指数
Fst基因流
NmSSR-ZC1 0.700 0 0.647 4 2.60 1.010 4 0.430 9 0.330 2 SSR-ZC2 1.000 0 0.733 3 2.57 1.011 4 0.844 6 0.046 0 SSR-ZC3 0.333 3 0.333 3 1.38 0.450 6 0.946 5 0.014 1 SSR-ZC7 0.000 0 0.484 8 1.80 0.636 5 1.000 0 0 SSR-ZC9 0.000 0 0.189 5 1.22 0.325 1 1.000 0 0 SSR-ZC10 0.100 0 0.721 1 3.17 1.565 4 0.927 0 0.019 7 SSR-ZC12 0.600 0 0.526 3 2.00 0.693 1 0.400 0 0.375 0 SSR-ZC15 0.100 0 0.415 8 1.65 0.687 4 0.873 4 0.036 2 SSR-ZC16 0.444 4 0.679 7 2.79 1.161 6 0.718 3 0.098 0 SSR-ZC19 0.666 7 0.797 4 4.05 1.565 4 0.625 0 0.150 0 SSR-ZC20 0.784 2 0.784 2 3.92 1.449 0 0.932 9 0.018 0 SSR-ZC23 0.500 0 0.575 0 2.17 0.921 5 0.716 3 0.099 0 SSR-ZC25 0.600 0 0.563 2 2.15 0.845 1 0.439 3 0.319 1 SSR-ZC36 0.111 1 0.385 6 1.57 0.654 7 0.896 9 0.028 7 SSR-ZC38 0.500 0 0.763 2 3.64 1.415 0 0.655 2 0.131 6 SSR-ZC43 0.666 7 0.732 0 3.24 1.239 5 0.600 0 0.166 7 SSR-ZC44 0.571 4 0.571 4 2.13 0.991 1 0.740 3 0.087 7 SSR-ZC47 0.285 7 0.439 6 1.69 0.598 3 0.859 2 0.041 0 SSR-ZC54 0.100 0 0.542 1 2.06 0.823 7 0.902 9 0.026 9 SSR-ZC61 0.500 0 0.684 2 2.86 1.161 1 0.615 4 0.156 3 平均mean 0.375 2 0.550 9 2.37 0.899 9 0.733 0 0.091 1 标准差
standard deviation0.288 5 0.205 5 0.872 7 0.392 6 2.4 遗传距离、遗传相似指数和聚类分析
10个品系紫菜丝状体的遗传相似指数为0.254 6~0.899 4,平均为0.474 9,最小值出现在皱紫菜(PC)和半叶紫菜(PK1)中,最大值出现在条斑紫菜的2个不同品系中;遗传距离为0.106 0~1.406 6,平均为0.792 3,最大值出现在坛紫菜(PH1)和少精紫菜(PO)中,最小值出现在条斑紫菜的2个不同品系中(表 4)。
表 4 10个紫菜品系遗传距离(左下方)和相似性系数(右上方)矩阵Table 4 Genetic distance (lower-trangular data matrix) and genetic similarity (upper-triangular data matrix) of 10 Porphyra linesPOP ID 坛紫菜1
PH1坛紫菜2
PH2坛紫菜3
PH3坛紫菜4
PH4半叶紫菜1
PK1半叶紫菜2
PK2条斑紫菜1
PY1条斑紫菜2
PY2少精紫菜
PO皱紫菜
PC坛紫菜1 PH1
P.haitanensis 10.445 4 0.471 9 0.506 0 0.307 7 0.302 2 0.296 9 0.373 1 0.245 0 0.423 1 坛紫菜2 PH2
P.haitanensis 20.808 9 0.753 8 0.619 8 0.335 0 0.296 1 0.348 5 0.377 2 0.384 7 0.477 0 坛紫菜3 PH3
P.haitanensis 30.751 1 0.282 7 0.860 8 0.555 6 0.527 4 0.485 8 0.465 1 0.391 2 0.436 4 坛紫菜4 PH4
P.haitanensis 40.681 2 0.478 4 0.149 8 0.474 3 0.414 0 0.444 9 0.380 4 0.338 9 0.379 5 半叶紫菜1 PK1
P.katadai 11.178 5 1.093 6 0.587 8 0.745 8 0.872 9 0.502 5 0.465 1 0.425 3 0.254 6 半叶紫菜2 PK2
P.katadai 21.196 7 1.217 1 0.639 9 0.881 8 0.136 0 0.526 4 0.488 2 0.467 7 0.357 1 条斑紫菜1 PY1
P.yezoensis 11.214 3 1.054 2 0.722 0 0.809 8 0.688 1 0.641 8 0.899 4 0.692 4 0.493 5 条斑紫菜2 PY2
P.yezoensis 20.985 9 0.975 1 0.765 5 0.966 6 0.765 5 0.717 0 0.106 0 0.721 8 0.519 7 少精紫菜PO
P.oligospermatangia1.406 6 0.955 4 0.938 4 1.082 1 0.855 1 0.759 9 0.367 6 0.326 0 0.567 9 皱紫菜PC
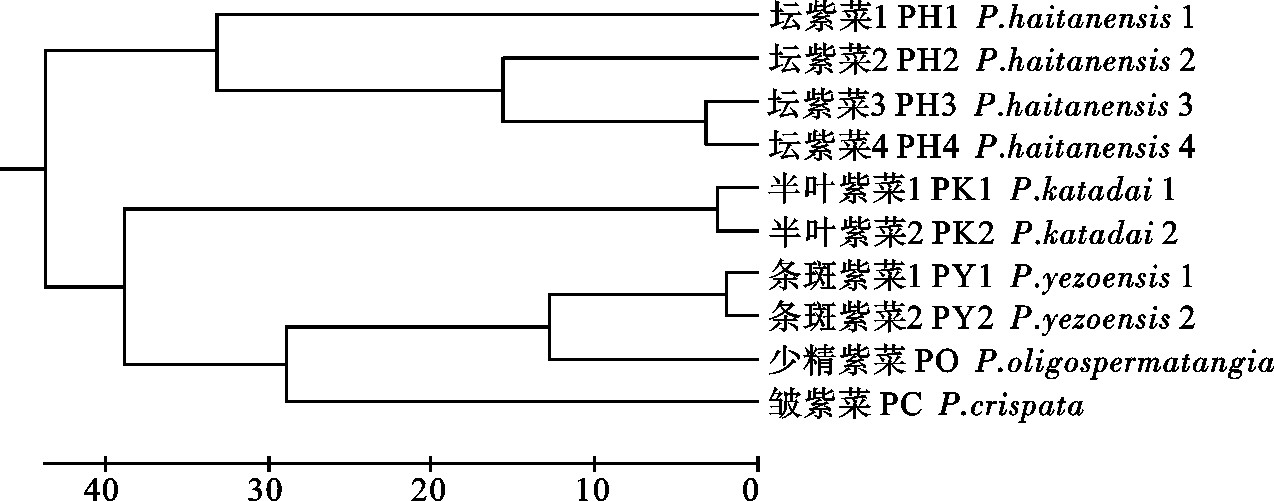
P.crispata0.860 2 0.740 2 0.829 1 0.968 8 1.368 1 1.029 6 0.706 3 0.654 5 0.565 8 根据各个体间的遗传距离进行UPGMA聚类分析(图 3),这5个物种的10个品系的紫菜丝状体可以分成2个亚群,坛紫菜单独聚为一支;半叶紫菜、条斑紫菜、少精紫菜和皱紫菜聚为另一支,其中条斑紫菜的2个品系首先聚在一块,再与少精紫菜聚在一起,接着与皱紫菜相聚,最后与半叶紫菜汇聚。
3. 讨论
3.1 引物通用性
微卫星标记的缺点在于微卫星引物具有种属特异性,在对一个物种首次进行微卫星分析时必须要进行微卫星引物的开发,较费时耗力[10]。有关研究认为微卫星引物可以在近缘物种间进行转移应用,但能否转移以及转移的成功率在不同的物种间表现较大的差异[16-17]。SUN等[10]、刘必谦等[11]、XIE等[13]和KONG等[14]通过对一个紫菜属物种设计的微卫星引物进行了引物种间转移扩增研究,证明微卫星引物在不同物种间具有高的通用性。笔者从68对微卫星引物中筛选出20对通用微卫星引物,筛选比例为29.4%,其中14对引物均能在5个物种中进行有效扩增,证实上述选扩的微卫星位点的侧翼序列在坛紫菜、条斑紫菜、少精紫菜、半叶紫菜及皱紫菜具有比较高的保守性。
所研究的68对微卫星引物中63.24%来自紫菜EST序列,36.76%来自基因组文库,而筛选出的20对引物中有15对来自GenBank中紫菜EST数据库,占34.88%,5对来自基因组,占20%。EST序列来自于有表达意义的mRNA反转录的cDNA,相对于含有内含子的基因组文库,在物种进化中更具保守性,来自于EST序列的微卫星引物可以在更大范围的紫菜属内扩增出多态性位点。因此,EST-SSR与基因组SSR相比更容易在不同物种中进行转移扩增。
3.2 紫菜属的遗传多样性
Ne作为衡量SSR引物多态性高低的一个标准,表明了物种或品系间的遗传多样性。Ne越高,物种或品系间的遗传多样性越高,在笔者的研究中每对引物可扩增的Ne平均为2.37。张鹏等[18]对坛紫菜9个品系的亲缘关系进行SSR标记分析,平均Ne为1.72。KONG等[14]用微卫星标记对条斑紫菜进行遗传分析,平均Ne为1.81。XIE等[13]用相同标记分析了坛紫菜15品系的遗传多样性,平均Ne为2.81,此研究获得的Ne结果与前人研究相似,同时显示种间的遗传多样性高于种内。
DAYANANDAN等[19]用4个微卫星引物对就可区分白杨(Populus tremuloides)的34个品系,只有2个品系没有被区分开;BECHER等[20]用3对引物就可以很好区分44个天竺葵(Pelargonium)品系。此研究从68对微卫星引物中筛选出20对引物,多态性比例高达95.25%,这可能与引物来源有关,笔者所用的引物63.24%来自EST序列。然而,刘必谦等[7]认为从基因组DNA筛选出的微卫星,其信息量要比来自EST的丰富,而此研究结果并不支持这一观点。崔灵英等[21]用ISSR标记对4种紫菜叶状体进行分析,获得多态性条带比例达95.5%。贾建航等[22]在对15个紫菜种(系)的丝状体进行RAPD分析时得到多态性比例达97.1%;杨锐等[23]用AFLP对坛紫菜8个品系的丝状体进行分析得到96.97%的多态性位点,这些结果与笔者的研究结果相似,进一步显示了紫菜物种的遗传变异相当丰富。
3.3 5种紫菜的种间遗传关系
5个紫菜属物种10个品系丝状体的遗传相似指数平均为0.474 9,说明个体间的亲缘关系比较远。曾呈奎等[1]曾根据叶片边缘的细胞排列方式将真紫菜亚属分为全缘紫菜组、刺缘紫菜组和边缘紫菜组,并且认为该特征具有进化上的意义。根据这一分类系统皱紫菜、坛紫菜均属于刺缘紫菜组,条斑紫菜、半叶紫菜和少精紫菜属于全缘紫菜组。此研究中5个物种中少精紫菜和条斑紫菜首先聚在一起,两者的遗传距离最为接近,这一结果与崔灵英等[21]用ISSR分子标记对紫菜叶状体分析的结果一致,也符合经典分类学结果;根据笔者的研究结果,皱紫菜聚类在条斑紫菜、半叶紫菜和少精紫菜的内部,而不是与坛紫菜聚类为一支,这与杨立恩等[24]对紫菜叶状体进行rbcL基因分类分析结果相矛盾,同时此研究结果与经典形态分类学认为皱紫菜和坛紫菜都属于刺缘紫菜组的结论也不一致。造成这些差异的原因可能是:1)试验材料不一样,笔者采用的试验材料为丝状体,而其他学者为叶状体;2)各种标记手段本身也存在着差异。
前人的研究表明,坛紫菜、条斑紫菜、皱紫菜、半叶紫菜和少精紫菜这5种紫菜之间具有重要生物学特征的差异,例如少精紫菜和条斑紫菜染色体n=3,皱紫菜、半叶紫菜和坛紫菜染色体n=5[1],这一研究结果与笔者的聚类分析结果有一定的相关性。通过聚类分析结果可以较真实地反映这10个品系紫菜丝状体间的亲缘关系,进一步显示微卫星分子标记在大型海藻分类学上具有一定的应用价值。
-
图 1 紫菜丝状体基因组DNA在1.0%的琼脂糖电泳检测结果
M.DL 2 000分子量标准;1~4. PH1、PH2、PH3、PH4,坛紫菜; 5~6. PK1、PK2,半叶紫菜; 7~8. PY1、PY2, 条斑紫菜;9. PO,少精紫菜;10.PC, 皱紫菜
Figure 1. Electrophoresis in 1% agarosegel of genomic DNA obtained from conchocelis of Porphyra
M. DL 2 000 DNA marker; 1~4. PH1, PH2, PH3, PH4, P.haitanensis; 5~6. PK1, PK2, P.katadai; 7~8. PY1, PY2, P.yezoensis; 9. PO, P.oligospermatangia; 10. PC, P.crispata
图 2 部分微卫星引物(左:SSR-ZC15;右:SSR-ZC20)在10个紫菜丝状体中的扩增结果
M.DL100分子量标准;1~10. PH1,PH2,PH3,PH4,PK1,PK2,PY1,PY2,PO,PC
Figure 2. Electrophoresis of part of microsatellite primer-pairs (L: SSR-ZC15; R: SSR-ZC20) in 10 lines of Porphyra conchocelis
M. DNA maker DL100; 1~10. PH1, PH2, PH3, PH4, PK1, PK2, PY1, PY2, PO, PC
表 1 此试验所用的10个品系紫菜丝状体信息表
Table 1 Information of 10 lines of Porphyra conchocelis in this study
序号
No.品系代码
line code物种名称
species name1 PH1 坛紫菜P.haitanensis 2 PH2 坛紫菜P.haitanensis 3 PH3 坛紫菜P.haitanensis 4 PH4 坛紫菜P.haitanensis 5 PK1 半叶紫菜P.katadai 6 PK2 半叶紫菜P.katadai 7 PY1 条斑紫菜P.yezoensis 8 PY2 条斑紫菜P.yezoensis 9 PO 少精紫菜P.oligospermatangia 10 PC 皱紫菜P.crispata 表 2 20对微卫星引物序列和特异退火温度
Table 2 Sequence of 20 pairs of microsatellite marker primers and specific annealing temperature for PCR amplification
引物编号
primer No.引物序列(5′→3′)
primer sequence重复序列
repetitive sequence退火温度/℃
annealing temperature片段范围
fragment rangeGenBank登录号
GenBank accession No.参考文献
referenceSSR-ZC1 TTCGCTGCGTTTCACCTTACATTT (TGCG)6 60 205 AV434771 [10] ACAAGGCCAACCCGAACACA SSR-ZC2 GGCTGCGGCTGAGTCACAGA (AGC)8 60 235 AU194221 [10] GTCGCTCCAACTCCTCCTGCT SSR-ZC3 TGGTGCTGTCTTCCAACGAGTA (AGC)13 60 245 AU196122 [10] CGGCTGTCGCACCTCGTTATA SSR-ZC7 CGCTCAACCACTTCGTCAG (AAC)10+1 58 261 AU191538 [10] CATTGTTGGCGTTGTTGTCATA SSR-ZC9 CCGTCGTCAGCAGGAGCA (GGC)7 60 200 AV434560 [10] ATGTGAGAAGCCAGTAGGGAAAGT SSR-ZC10 ACTTCCATCGCTGTCTTCGCT (AGC)8 60 196 AU192094 [10] CTGAGCTGCGTGTTGTGGTT SSR-ZC12 TCAACCATCAGCCATACCGAC (ACC)11 58 192 AV435510 [10] GACATGTCCGCCACCTTGTA SSR-ZC15 TGACTTCCTCATCGACATTGT (AGC)8 55 319 AU187439 [10] GCCATAGTACATTTGTTGCTG SSR-ZC16 TACCAGGTCGACCAGGAGCA (AGC)11 60 291 AU193458 [10] TCCAACTCTGCAGTGTCCGTT SSR-ZC19 ACCTCCTCGGCTACTTCAGA (AGC)7 58 276 AV431181 [10] GGATACAACGCCTGCTCCAT SSR-ZC20 GGCAGCAGCCATGATGTA (AGC)8+8 55 339 AV433675 [10] CCGTCAGGCAGAAGAGAAT SSR-ZC23 CAAGGGCTACTGCTACTA CAA (AAC)17 55 249 AU195299 [10] TACAAAAAGACTCTCGTG GCA SSR-ZC25 CCGTGCTACTACGGCTA CAA (AAC)9+11 58 302 DN606134 [10] GTCCGGTGCAGGTTG TTCT SSR-ZC36 CCAACGATGGGTTTCTTCAA 55 300 [7] ACTTCATGCCCCTGCCGATG SSR-ZC38 GGACAAGGGGTAATGGCT (CA)17 50 212~226 DQ831155 [12] TGGAAAACTTCCTGGGTG SSR-ZC43 GGAATGCCTTGCACCTGG (GT)26 55 408~420 DQ831185 [12] CACTATTGACCGAATCCGCTAC SSR-ZC44 CGTGCGAGTCATAGTCTGCT (GTGA)26 55 191~200 DQ831194 [12] ACAGCCAGTGCAAGAACACC SSR-ZC47 CTAGACGGAGTGCGGCTGAC (CA)9 58 176~196 DQ831197 [12] GCTCCTCCACGAGCATCAGG SSR-ZC54 GGTGGCAGTGAAGCGAAACA (CG)7 60 229 DN607742 [13] ACCCAGCAAGTGCGTGAGC SSR-ZC61 AACAGAGATACGGAGAGC (GT)8..(TG)11 50 276 EU670707 [14] ATCAGATTGGACTTGCCT 表 3 20个微卫星的遗传多样性指数
Table 3 Genetic diversity index of 20 microsatellite loci
locus 表观杂合度
Ho预期杂合度
He有效等位基因数
NeShannon信息指数
I遗传分化指数
Fst基因流
NmSSR-ZC1 0.700 0 0.647 4 2.60 1.010 4 0.430 9 0.330 2 SSR-ZC2 1.000 0 0.733 3 2.57 1.011 4 0.844 6 0.046 0 SSR-ZC3 0.333 3 0.333 3 1.38 0.450 6 0.946 5 0.014 1 SSR-ZC7 0.000 0 0.484 8 1.80 0.636 5 1.000 0 0 SSR-ZC9 0.000 0 0.189 5 1.22 0.325 1 1.000 0 0 SSR-ZC10 0.100 0 0.721 1 3.17 1.565 4 0.927 0 0.019 7 SSR-ZC12 0.600 0 0.526 3 2.00 0.693 1 0.400 0 0.375 0 SSR-ZC15 0.100 0 0.415 8 1.65 0.687 4 0.873 4 0.036 2 SSR-ZC16 0.444 4 0.679 7 2.79 1.161 6 0.718 3 0.098 0 SSR-ZC19 0.666 7 0.797 4 4.05 1.565 4 0.625 0 0.150 0 SSR-ZC20 0.784 2 0.784 2 3.92 1.449 0 0.932 9 0.018 0 SSR-ZC23 0.500 0 0.575 0 2.17 0.921 5 0.716 3 0.099 0 SSR-ZC25 0.600 0 0.563 2 2.15 0.845 1 0.439 3 0.319 1 SSR-ZC36 0.111 1 0.385 6 1.57 0.654 7 0.896 9 0.028 7 SSR-ZC38 0.500 0 0.763 2 3.64 1.415 0 0.655 2 0.131 6 SSR-ZC43 0.666 7 0.732 0 3.24 1.239 5 0.600 0 0.166 7 SSR-ZC44 0.571 4 0.571 4 2.13 0.991 1 0.740 3 0.087 7 SSR-ZC47 0.285 7 0.439 6 1.69 0.598 3 0.859 2 0.041 0 SSR-ZC54 0.100 0 0.542 1 2.06 0.823 7 0.902 9 0.026 9 SSR-ZC61 0.500 0 0.684 2 2.86 1.161 1 0.615 4 0.156 3 平均mean 0.375 2 0.550 9 2.37 0.899 9 0.733 0 0.091 1 标准差
standard deviation0.288 5 0.205 5 0.872 7 0.392 6 表 4 10个紫菜品系遗传距离(左下方)和相似性系数(右上方)矩阵
Table 4 Genetic distance (lower-trangular data matrix) and genetic similarity (upper-triangular data matrix) of 10 Porphyra lines
POP ID 坛紫菜1
PH1坛紫菜2
PH2坛紫菜3
PH3坛紫菜4
PH4半叶紫菜1
PK1半叶紫菜2
PK2条斑紫菜1
PY1条斑紫菜2
PY2少精紫菜
PO皱紫菜
PC坛紫菜1 PH1
P.haitanensis 10.445 4 0.471 9 0.506 0 0.307 7 0.302 2 0.296 9 0.373 1 0.245 0 0.423 1 坛紫菜2 PH2
P.haitanensis 20.808 9 0.753 8 0.619 8 0.335 0 0.296 1 0.348 5 0.377 2 0.384 7 0.477 0 坛紫菜3 PH3
P.haitanensis 30.751 1 0.282 7 0.860 8 0.555 6 0.527 4 0.485 8 0.465 1 0.391 2 0.436 4 坛紫菜4 PH4
P.haitanensis 40.681 2 0.478 4 0.149 8 0.474 3 0.414 0 0.444 9 0.380 4 0.338 9 0.379 5 半叶紫菜1 PK1
P.katadai 11.178 5 1.093 6 0.587 8 0.745 8 0.872 9 0.502 5 0.465 1 0.425 3 0.254 6 半叶紫菜2 PK2
P.katadai 21.196 7 1.217 1 0.639 9 0.881 8 0.136 0 0.526 4 0.488 2 0.467 7 0.357 1 条斑紫菜1 PY1
P.yezoensis 11.214 3 1.054 2 0.722 0 0.809 8 0.688 1 0.641 8 0.899 4 0.692 4 0.493 5 条斑紫菜2 PY2
P.yezoensis 20.985 9 0.975 1 0.765 5 0.966 6 0.765 5 0.717 0 0.106 0 0.721 8 0.519 7 少精紫菜PO
P.oligospermatangia1.406 6 0.955 4 0.938 4 1.082 1 0.855 1 0.759 9 0.367 6 0.326 0 0.567 9 皱紫菜PC
P.crispata0.860 2 0.740 2 0.829 1 0.968 8 1.368 1 1.029 6 0.706 3 0.654 5 0.565 8 -
[1] 张学成, 秦松, 马家海, 等. 海藻遗传学[M]. 北京: 中国农业出版社, 2005: 184-186. ZHANG Xuecheng, QIN Song, MA Jiahai, et al. Genetics of algae[M]. Beijing: Chinese Agricultrue Press, 2005: 184-186. (in Chinese)
[2] 周延清, 杨清香, 张改娜. 生物遗传标记与应用[M]. 北京: 化学工业出版社, 2008: 155-156. ZHOU Yanqing, YANG Qingxiang, ZHANG Gaina. Biological genetic markers and application[M]. Beijing: Chemical Industry Press, 2008: 155-156. (in Chinese)
[3] LIU Fuli, WANG Xueliang, YAO Jianting, et al. Development of expressed sequence tag-derived microsatellite markers for Saccharina(Laminaria) japonica[J]. J Appl Phycol, 2010, 22(2): 109-111. doi: 10.1007/s10811-009-9426-9
[4] ENRIQUE A M, LEYLA C, BILLOT C, et al. Microsatellites of Laminaria digitata tested in Lessonia nigrescens: evaluation and improvement of cross amplification between kelps of two different families[J]. J Appl Phycol, 2005, 17(3): 245-253. doi: 10.1007/s10811-005-3750-5
[5] LUO H, MÖRCHEN M, ENGEL C R, et al. Characterization of microsatellite markers in the red alga Gracilaria gracilis[J]. Mol Ecol, 1999, 8(4): 700-702. doi: 10.1046/j.1365-294x.1999.00879.x
[6] 张学成, 贺扬, 徐涤. 龙须菜微卫星DNA标记筛查及系统分析[J]. 中国海洋大学学报: 自然科学版, 2009, 39(2): 259-264. doi: 10.16441/j.cnki.hdxb.2009.02.016 ZHANG Xuecheng, HE Yang, XU Di. Screening microsatellite sequences from Gracilaria lemaneiformis and its phylogenetic analysis[J]. Periodic Ocean Univ China: Natural Science, 2009, 39(2): 259-264. (in Chinese) doi: 10.16441/j.cnki.hdxb.2009.02.016
[7] 刘必谦, 曾庆国, 骆其君, 等. 微卫星标记在坛紫菜丝状体品系DNA指纹构建中的应用[J]. 水产学报, 2005, 29(3): 324-336. doi: 10.3321/j.issn:1000-0615.2005.03.007 LIU Biqian, ZENG Qingguo, LUO Qijun, et al. Establishment of the DNA fingerprint of Porphyra haitanensis filament lines with microsatellite markers[J]. J Fish China, 2005, 29(3): 324-336. (in Chinese) doi: 10.3321/j.issn:1000-0615.2005.03.007
[8] 胡则辉, 周志刚. 微卫星DNA标记技术及其在海洋生物遗传学中的应用[J]. 海洋湖沼通报, 2006(1): 37-44. doi: 10.3969/j.issn.1003-6482.2006.01.005 HU Zehui, ZHOU Zhigang. Microsatellite DNA marker technique and its application to the genetic of marine organisms[J]. Trans Oceanol Limnol, 2006(1): 37-44. (in Chinese) doi: 10.3969/j.issn.1003-6482.2006.01.005
[9] 姚春燕, 姜红霞, 朱建一, 等. 温度胁迫对紫菜丝状体叶绿素荧光特性的影响[J]. 江苏农业科学, 2011(1): 277-282. doi: 10.3969/j.issn.1002-1302.2011.01.102 YAO Chunyan, JIANG Hongxia, ZHU Jianyi, et al. Temperature stress on chlorophyll fluorescence characteristics of Porphyra conchocelis[J]. Jiangsu Agric Sci, 2011(1): 277-282. (in Chinese) doi: 10.3969/j.issn.1002-1302.2011.01.102
[10] SUN Jianwei, LIU Tao, GUO Baotai, et al. Development of SSR primers from EST sequences and their application in germplasm identification of Porphyra lines(Rhodophyta)[J]. Eur J Phycol, 2006, 41(3): 324-336. doi: 10.1080/09670260600740906
[11] 刘必谦, 曾庆国, 骆其君, 等. 条斑紫菜(Porphyra yezoensis) dbEST中筛选微卫星位点及引物种间转移扩增[J]. 海洋与湖沼, 2005, 36(3): 248-254. doi: 10.3321/j.issn:0029-814X.2005.03.009 LIU Biqian, ZENG Qingguo, LUO Qijun, et al. Isolation of microsatellite loci from dbEST of algae Porphyra yezoensis and primer amplification of interspecies transfer[J]. Oceanologia et Limnologia Sinica, 2005, 36(3): 248-254. (in Chinese) doi: 10.3321/j.issn:0029-814X.2005.03.009
[12] ZUO Zhenghong, WANG Chonggang, CAO Xiaohua, et al. Isolation and characterization of microsatellite loci from a commercial cultivar of Porphyra haitanensis[J]. Mol Ecol Notes, 2007, 7(3): 522-524. doi: 10.1111/j.1471-8286.2006.01642.x
[13] XIE Chaotian, CHEN Changsheng, JI Dehua, et al. Characterization, development and exploitation of EST-derived microsatellites in Porphyra haitanensis Chang et Zheng (Bangiales, Rhodophyta)[J]. J Appl Phycol, 2009, 21(3): 367-374. doi: 10.1007/s10811-008-9379-4
[14] KONG Fanna, MAO Yunxiang, YANG Hui, et al. Genetic analysis of Porphyra yezoensis using microsatellite markers[J]. Plant Mol Biol Rep, 2009, 27(4): 496-502. doi: 10.1007/s11105-009-0101-8
[15] SANGUINETTI C J, DIAS N E, SIMPSON G. Rapid silver straining and recovery of PCR products separated on polyaerylamide gels[J]. Biotechniques, 1994, 17(5): 914-921. https://www.semanticscholar.org/paper/Rapid-silver-staining-and-recovery-of-PCR-products-Sanguinetti-Dias/9485fa8c6f2ee8747706ec5180c0ea9e567d4c9a
[16] PEAKALL R, GILMORE S, KEYS W, et al. Cross-species amplification of soybean (Glycine max) simple sequence repeats (SSRs) within the genus and other legume genera: implications for the transferability of SSRs in plants[J]. Mol Biol Evol, 1998, 15(10): 1275-1287. doi: 10.1093/oxfordjournals.molbev.a025856
[17] VARSHNEYA R K, SIGMUNDA R, BORNERA A, et al. Interspecific transferability and comparative mapping of barley EST-SSR markers in wheat, rye and rice[J]. Plant Sci, 2005, 168(1): 195-202. doi: 10.1016/j.plantsci.2004.08.001
[18] 张鹏, 张源, 王铁杆, 等. 坛紫菜不同品系亲缘关系的SSR标记分析[J]. 中国水产科学, 2009, 16(9): 842-849. https://kns.cnki.net/kcms2/article/abstract?v=At0rObma_qOzb9jtHSBwpOXyT3jOj_gAnoqho8-zsj174qbbxkMPzR7owaDA7ZCgOgBVmo9jnnoxbGg_vEkLtRTXeQLLVG_xCR8tN_xs3rak05Szao8R6dlBLbroTGBrtH0-IhaKZjWFOUjws0HmQ4QOv9e0MjhQkVzuI_jqfg9Rqa3TMYl5Sv65wgxLuydm&uniplatform=NZKPT&language=CHS ZHANG Peng, ZHANG Yuan, WANG Tiegan, et al. Phylogenetic relationship of the lines of Porphyra haitanensis (Rhodophyta, Bangials) determined by microsatellite DNA markers[J]. J Fish Sci China, 2009, 16(9): 842-849. (in Chinese) https://kns.cnki.net/kcms2/article/abstract?v=At0rObma_qOzb9jtHSBwpOXyT3jOj_gAnoqho8-zsj174qbbxkMPzR7owaDA7ZCgOgBVmo9jnnoxbGg_vEkLtRTXeQLLVG_xCR8tN_xs3rak05Szao8R6dlBLbroTGBrtH0-IhaKZjWFOUjws0HmQ4QOv9e0MjhQkVzuI_jqfg9Rqa3TMYl5Sv65wgxLuydm&uniplatform=NZKPT&language=CHS
[19] DAYANANDAN S, RAJORA O P, BAWA K S. Isolation and characterization of microsatellites in trembling aspen (Populus tremuloides)[J]. Theor Appl Genel, 1998, 96(6/7): 950-956. doi: 10.1007/s001220050825
[20] BECHER S A, STEINMETZ K, WEISING K, et al. Microsatellites for cultivar identification in Pelargonium[J]. Theor Appl Genel, 2000, 101(4): 643-651. doi: 10.1007/s001220051526
[21] 崔灵英, 许璞, 朱建一, 等. 4种紫菜叶状体的ISSR分子标记分析[J]. 中国水产科学, 2006, 13(3): 371-377. doi: 10.3321/j.issn:1005-8737.2006.03.006 CUI Lingyin, XU Pu, ZHU Jianyi, et al. Inter-simple sequence repeats(ISSR) applied in four species of Porphyra (Rhodophyta, Bangiales)[J]. J Fish Sci China, 2006, 13(3): 371-377. (in Chinese) doi: 10.3321/j.issn:1005-8737.2006.03.006
[22] 贾建航, 王萍, 金德敏, 等. RAPD标记在紫菜遗传多样性检测和种质鉴定中的应用[J]. 植物学报, 2000, 42(4): 403-407. doi: 10.3321/j.issn:1672-9072.2000.04.012 JIA Jianhang, WANG Ping, JIN Demin, et al. The application of RAPD markers in diversity detection and variety identification of Porphyra[J]. Acta Botanica Sinica, 2000, 42(4): 403-407. (in Chinese) doi: 10.3321/j.issn:1672-9072.2000.04.012
[23] 杨锐, 刘必谦, 骆其君, 等. 利用片段长度多态性(AFLP)研究坛紫菜的遗传变异[J]. 高技术通讯, 2002, 12(1): 83-86. doi: 10.3321/j.issn:1002-0470.2002.01.025 YANG Rui, LIU Biqian, LUO Qijun, et al. Genetic variation of Porphyra haitanensis by applying AFLP[J]. High Technol Lett, 2002, 12(1): 83-86. (in Chinese) doi: 10.3321/j.issn:1002-0470.2002.01.025
[24] 杨立恩, 金杰, 朱建一, 等. 紫菜(Porphyra)叶状体rbcL基因分类学分析[J]. 常熟理工学院学报: 自然科学, 2008, 23(8): 66-71. doi: 10.16101/j.cnki.cn32-1749/z.2009.08.020 YANG Lien, JIN Jie, ZHU Jianyi, et al. Taxonomy of Porphyra (Rhodophyta, Bangiales) based on rbcL sequences from the thallus[J]. J Changshu Inst Technol: Natural Science, 2008, 23(8): 66-71. (in Chinese) doi: 10.16101/j.cnki.cn32-1749/z.2009.08.020



 下载:
下载:

 粤公网安备 44010502001741号
粤公网安备 44010502001741号
