Optimization of SRAP-PCR system for Portunus trituberculatus based on orthogonal experimental design
-
摘要:
相关序列扩增多态性(sequence related amplified polymorphism,SRAP)是一种多态性高的新型分子标记,其扩增结果稳定,容易操作和引物通用性高,在遗传多样性分析、种质鉴定和遗传图谱构建等方面的广泛应用逐渐从植物转移到水产动物中。为了建立三疣梭子蟹(Portunus trituberculatus)SRAP技术体系,文章利用正交设计L16 (45)对三疣梭子蟹SRAP-PCR反应体系的5因素[TaqDNA聚合酶、镁离子(Mg2+)、模板、三磷酸脱氧核苷(dNTPs)和引物]在4个水平上进行优化试验,结果得出各因素水平变化对PCR反应的影响从大到小依次为模板(纯度1.75~1.90,质量浓度10~70 ng · μL-1),Mg2+(1.60~2.00 mmol · L-1),dNTPs(0.10~0.40 mmol · L-1),TaqDNA聚合酶(0.50~2.00 U)和引物(0.10~0.40 μmol · L-1);筛选出各反应因素的最佳水平,建立三疣梭子蟹SRAP-PCR反应的最佳体系(25 μL)为TaqDNA聚合酶0.50 U,Mg2+ 2.20 mmol · L-1,模板DNA 10 ng,dNTPs 0.20 mmol · L-1,引物0.20 μmol · L-1。这一优化体系可望在三疣梭子蟹遗传多样性和性别连锁标记等研究中应用。
Abstract:SRAP is a novel molecular marker with high polymorphism. It is reliable in amplification, easy to operate and has a high universality in primer pairs. SRAP technique has been widely used for studies from plants to aquatic animals in the fields of genetic diversity, germplasm identification, genetic map construction and so on. Based on an orthogonal experimental design, we optimized the five factors of SRAP-PCR amplification system for Portunus trituberculatus (TaqDNA polymerase, Mg2+, template, dNTPs and primer) from four levels. The results show that the effects of these factors on PCR reaction from strong to weak is as follows: template (purity 1.75~1.90, concentration 10~70 ng · μL-1), Mg2+ (1.60~2.00 mmol · L-1), dNTPs (0.10~0.40 mmol · L-1), TaqDNA polymerase (0.50~2.00 U) and primer (0.10~0.40 μmol · L-1).The optimal SRAP-PCR reaction system (25 μL) can be summarized as follows: 0.50 U TaqDNA polymerase, 2.20 mmol · L-1 Mg2+, 10 ng DNA template, 0.20 mmol · L-1 dNTPs and 0.20 μmol · L-1 primer. This optimized system may be helpful in the studies of genetic diversity and sex-linked markers for P.trituberculatus.
-
Keywords:
- Portunus trituberculatus /
- SRAP /
- orthogonal design /
- optimization
-
三疣梭子蟹(Portunus trituberculatus)广泛分布于中国沿海,作为一种重要的大型海产经济蟹类,其营养价值和经济价值较高,并且肉质细嫩,味道鲜美。过去几十年里,研究者在梭子蟹的苗种培育、人工养殖技术及疾病防治等方面做了大量工作。近年来,许多学者在生化和DNA水平上研究梭子蟹野生群体遗传结构及种质资源的鉴定方面,同工酶(isozyme)[1-6]、线粒体DNA(mitochondrial DNA,mtDNA)[7-11]、随机扩增多态性DNA(random amplified polymorphic DNA,RAPD)[12]、扩增片段长度多态性(amplified fragment length polymorphism,AFLP)[13]和简单重复序列间多态性(inter-simple sequence repeat,ISSR)[6]等先进分子标记的应用加速了梭子蟹遗传基础的研究进程,已经分离出能够区分不同种梭子蟹的分子标记[12]。
SRAP是由LI与QUIROS[14]发展而来的一种新型分子标记。SRAP分子标记在许多植物得到成功应用,如甘蓝(Brassica oleracea)[14]、棉花(Gossypium spp.)[15-16]、野牛草(Buchloe dactyloides)[17-18]和西葫芦(Cucurbita pepo)[19-20],足以证明SRAP标记能够有效地评价物种遗传多样性、品系鉴定和构建遗传图谱等。考虑到SRAP-PCR扩增结果易受镁离子(Mg2+)、三磷酸脱氧核苷(dNTPs)、引物、TaqDNA聚合酶、模板等因子及其协同作用的影响,因此,在利用SRAP标记进行梭子蟹遗传差异分析,寻找与性别相关的分子标记前,建立一个最佳的SRAP-PCR反应体系,以期获得稳定、可靠的试验结果。
1. 材料与方法
1.1 材料
2009年7月采自舟山普陀东河菜场,从活蟹的螯肢上取适量肌肉,保存于95%乙醇以备基因组DNA的提取。
从大连宝生物工程有限公司购置TaqDNA聚合酶和标准分子量(Marker)100 bp DNA ladder。
提交SRAP引物序列由上海生工合成,通过引物两两组合筛选,选取引物组合M8E9[14]进行正式扩增,重复3次。
1.2 梭子蟹基因组DNA模板的提取与检测
采用高盐法提取梭子蟹基因组DNA,利用U2100pro核酸蛋白检测仪检测其纯度和浓度,并进行0.8%琼脂糖凝胶检测,验证其是否降解,稀释基因组DNA为10 ng · μL-1以备扩增用。
1.3 反应因素水平的确定与正交表设计
根据王燕青和季孔庶[21]提供的方法,试验中采用正交设计L16 (45)进行试验。反应各因素水平见表 1。
表 1 PCR反应体系的因素水平Table 1 Factors and levels of PCR reaction因素
factors水平(体系终浓度)/levels(final concentration) 1 2 3 4 Mg2+/mmol·L-1 1.60 2.00 2.20 2.40 dNTPs/mmol·L-1 0.10 0.20 0.30 0.40 TaqDNA聚合酶/U TaqDNA polymerase 0.50 1.00 1.50 2.00 引物/μmol·L-1 primer 0.10 0.20 0.30 0.40 模板/ng template 10 30 50 70 在My CyclerTM thermal cycler仪(BIO-RAD,美国产)上进行SRAP-PCR扩增,反应程序参照王燕青和季孔庶[21]提供的程序。
PCR扩增产物用2%琼脂糖凝胶电泳(含EB)分离,电泳缓冲液为0.5×TBE,80 V稳压电泳40 min,电泳结束后在凝胶成像系统中拍照观察。
电泳结果评分标准是依据电泳条带的多少、清晰度及背景颜色进行打分[21-22]。此试验中,条带丰富记为6分;清晰度高记为6分;背景颜色浅记为4分,依此对各引物组合扩增结果进行打分与加和统计。
2. 结果与讨论
2.1 电泳结果评分
根据评分原则和标准,对3次重复试验[3次重复结果差异不大,仅附1次的电泳图谱(图 1)]分别单独进行统计,得分见表 2。从3次重复的得分来看,结果的一致性较好。其中以组合8、10和15效果最好,达到15分,而以组合4效果最差,仅为1.67分,这与图 1中的扩增结果表现大体一致。各因素的极差R与对反应结果的影响程度成正比,当某因素的极差(R)最大时,该因素对PCR反应的影响也最大。由此可知,试验中模板浓度对反应结果影响最大,引物浓度的影响最小,各因素水平变化对PCR结果的影响从大到小依次为模板、Mg2+、dNTPs、TaqDNA聚合酶和引物。
表 2 PCR反应的因素水平正交试验设计Table 2 Factors and levels of PCR reaction based on orthogonal experimental design编号
No.Mg2+
/mmol·L-1dNTPs
/mmol·L-1TaqDNA聚合酶/U
TaqDNA polymerase引物/μmol·L-1
primer模板/ng
template得分score 平均得分
average score1 2 3 1 1.60 0.10 0.50 0.10 10 11 10 10 10.33 2 1.60 0.20 1.00 0.20 30 13 12 11 12.00 3 1.60 0.30 1.50 0.30 50 14 11 10 11.67 4 1.60 0.40 2.00 0.40 70 2 1 2 1.67 5 2.00 0.10 1.00 0.30 70 10 9 9 9.33 6 2.00 0.20 0.50 0.40 50 10 7 14 10.33 7 2.00 0.30 2.00 0.10 30 11 14 10 11.67 8 2.00 0.40 1.50 0.20 10 16 15 14 15.00 9 2.20 0.10 1.50 0.40 30 13 15 13 13.67 10 2.20 0.20 2.00 0.30 10 15 16 14 15.00 11 2.20 0.30 0.50 0.20 70 15 12 13 13.33 12 2.20 0.40 1.00 0.10 50 12 11 12 11.67 13 2.40 0.10 2.00 0.20 50 11 12 11 11.33 14 2.40 0.20 1.50 0.10 70 10 12 12 11.33 15 2.40 0.30 1.00 0.40 10 13 16 16 15.00 16 2.40 0.40 0.50 0.30 30 11 10 10 10.33 T1 35.67 44.67 44.33 45.00 55.33 - - - - T2 46.33 48.67 48.00 51.67 47.67 - - - - T3 53.67 51.67 51.67 46.33 45.00 - - - - T4 48.00 38.67 39.67 40.67 35.67 - - - - t1 8.92 11.17 11.08 11.25 13.83 - - - - t2 11.58 12.17 12.00 12.92 11.92 - - - - t3 13.42 12.92 12.92 11.58 11.25 - - - - t4 12.00 9.67 9.92 10.17 8.92 - - - - R 4.50 3.25 3.00 2.75 4.91 - - - - 注:T1~T4为各水平的平均得分总和;t1~t4分别为T1,T2,T3和T4的平均值Note:T1~T4 represent the sum of average scores of each treatment; t1~t4 represent the mean of T1, T2, T3, T4. 2.2 模板质量浓度对PCR结果的影响
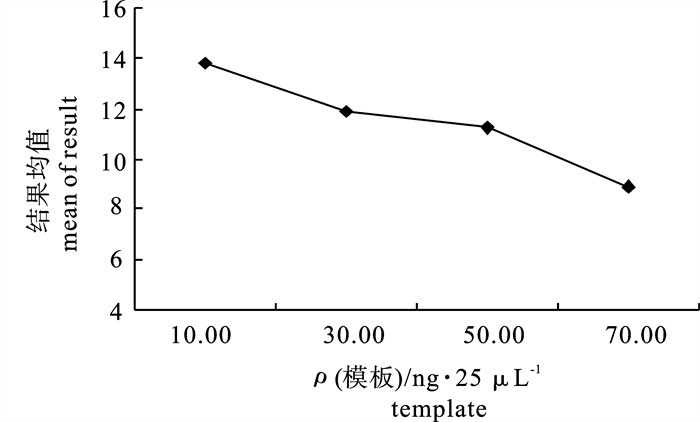
当ρ (模板)为30~50 ng · μL-1时,各水平间差异不明显,其余每2水平间均达到显著差异(图 2)。从图 2明显看出ρ (模板)10 ng · μL-1时,SRAP-PCR扩增效果好,达到了反应体系的最佳效果。在保证有效扩增效果的前提下应尽量降低模板的用量。因此,该试验最终选定10 ng ·μL-1作为最佳反应水平。
2.3 Mg2+对PCR结果的影响
Mg2+作为该试验中的第二关键因子,对PCR反应的影响是多方面的,包括对TaqDNA聚合酶的活性和引物退火温度的影响。当dNTPs和模板同时与Mg2+结合,降低了游离c (Mg2+)。在包含0.20 mmol · L-1 dNTPs的典型PCR反应中,c (Mg2+)以1.50~2.00 mmol · L-1为宜。然而在该试验中,尽管c (Mg2+)(2.00~2.40 mmol · L-1)明显高于常规的c (Mg2+),仍然得到较好的扩增结果。
较高的游离c (Mg2+)可以增加PCR产量,但也会增加非特异性扩增,降低忠实性。浓度过低会降低TaqDNA聚合酶的活性,使反应产物减少。当c (Mg2+)从1.60 mmol · L-1增加到2.40 mmol · L-1的过程中,发现在2.20 mmol · L-1时反应均值最高,随后下降与2.00 mmol · L-1相当。因此,最终确定c (Mg2+)2.20 mmol · L-1为最佳反应水平(图 3)。
2.4 dNTPs浓度对PCR结果的影响
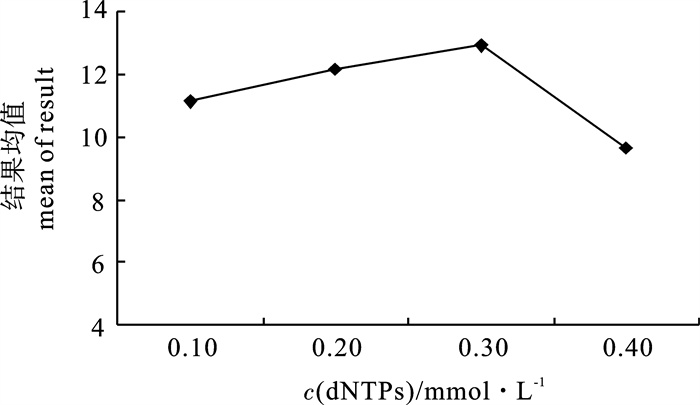
c (dNTPs)在0.10~0.30 mmol · L-1无明显差异,呈现上升的趋势,但与0.40 mmol · L-1差异显著。浓度过高时,错误掺入率大大增加,且dNTPs能与Mg2+结合,降低游离的c (Mg2+)。0.10~0.30 mmol · L-1结果差异不显著,均值几乎相同。为防止浓度过低所导致无扩增产物或扩增产物不稳定或扩增出来的条带模糊不清,再加上经济角度的考虑,选择0.20 mmol · L-1为反应的最佳水平(图 4)。
2.5 TaqDNA聚合酶浓度对PCR结果的影响
TaqDNA聚合酶在0.50~1.50 U水平间差异不显著,与2.00 U水平达到显著差异。当TaqDNA聚合酶用量为1.50 U时,SRAP-PCR扩增效率出现峰值,达到最佳(图 5)。一般来说,在100 μL PCR反应体系中,TaqDNA聚合酶用量约为2.50 U。浓度高可引起非特异性扩增,忠实性降低,浓度低则减少PCR产物,导致扩增模糊。考虑TaqDNA聚合酶在0.50~1.50 U水平间差异不显著的因素,该试验中25 μL反应体系选择0.50 U TaqDNA聚合酶作为最佳反应水平(图 5)。
2.6 引物浓度对PCR结果的影响
c (引物)0.10~0.40 μmol · L-1在水平间无显著差异(图 6)。较高的引物浓度能大量扩增产物,但同时诱发了错配、非特异性扩增和二聚体的生成,降低了PCR反应的忠实度。通常,最佳的引物浓度一般在0.10~0.50 μmol ·L-1,以最低引物量扩增出所需要的产物为好,因此该试验中应选择峰值0.20 μmol · L-1为反应的最佳水平。
3. 结论
SRAP标记是一种新型的分子标记技术,利用17碱基正向引物和18碱基反向引物进行扩增,正向引物的核心引物序列为14个碱基,后接CCGG碱基,反向引物的核心序列后接AATT碱基[23]。SRAP标记比RAPD结果稳定,而同时减少了AFLP繁琐的试验操作,并降低了试验成本。值得一提的是,SRAP的上游和下游引物可以通用,两两搭配组合甚至三引物组合,省却了SSR标记需要进行引物开发的前期工作。与RAPD、AFLP和微卫星DNA(simple sequence repeat,SSR)相比,SRAP标记的这种重复性好、容易操作、引物通用性好和多态性高等优点,在种群遗传多样性、种质鉴定和遗传图谱构建等方面发挥了日益重要的作用。
不同反应体系、扩增程序和物种的差别对SRAP-PCR的扩增效率均有一定影响。该试验结果证明,在扩增程序不变的前提下,不同反应体系对三疣梭子蟹的SRAP-PCR扩增结果影响较大。基于正交试验设计,该试验优化并建立了三疣梭子蟹SRAP-PCR最佳反应体系(25 μL)为TaqDNA聚合酶0.50 U,c (Mg2+)2.20 mmol · L-1,模板10 ng,c (dNTPs)0.20 mmol · L-1,c (引物)0.20 μmol · L-1。这与图 1中条带最优组合8和10(表 2)大体相似。
PCR反应是各因素相互作用的综合结果,一般来说,DNA的提取、扩增体系和扩增产物的检测都有可能影响其结果,特别是反应程序的影响较大。郭大龙和罗正荣[24]在对柿属植物(Diospyros spp.)进行SRAP体系优化时发现,较高的退火温度可以保证更加稳定和理想的结果。该研究采用SRAP-PCR的改进反应程序(即Tm为50 ℃)取得了较好的效果。SRAP一般采用PAGE凝胶检测所扩增的产物,然而在后续的雌、雄梭子蟹遗传差异分析(另文发表)中,利用该优化体系所扩增的产物用较高质量分数(2%)的琼脂糖凝胶电泳检测,同样能获得较丰富的多态性,而且结果清晰稳定,这与FERRIOL等[20]和郭大龙和罗正荣[24]的结论一致。
-
表 1 PCR反应体系的因素水平
Table 1 Factors and levels of PCR reaction
因素
factors水平(体系终浓度)/levels(final concentration) 1 2 3 4 Mg2+/mmol·L-1 1.60 2.00 2.20 2.40 dNTPs/mmol·L-1 0.10 0.20 0.30 0.40 TaqDNA聚合酶/U TaqDNA polymerase 0.50 1.00 1.50 2.00 引物/μmol·L-1 primer 0.10 0.20 0.30 0.40 模板/ng template 10 30 50 70 表 2 PCR反应的因素水平正交试验设计
Table 2 Factors and levels of PCR reaction based on orthogonal experimental design
编号
No.Mg2+
/mmol·L-1dNTPs
/mmol·L-1TaqDNA聚合酶/U
TaqDNA polymerase引物/μmol·L-1
primer模板/ng
template得分score 平均得分
average score1 2 3 1 1.60 0.10 0.50 0.10 10 11 10 10 10.33 2 1.60 0.20 1.00 0.20 30 13 12 11 12.00 3 1.60 0.30 1.50 0.30 50 14 11 10 11.67 4 1.60 0.40 2.00 0.40 70 2 1 2 1.67 5 2.00 0.10 1.00 0.30 70 10 9 9 9.33 6 2.00 0.20 0.50 0.40 50 10 7 14 10.33 7 2.00 0.30 2.00 0.10 30 11 14 10 11.67 8 2.00 0.40 1.50 0.20 10 16 15 14 15.00 9 2.20 0.10 1.50 0.40 30 13 15 13 13.67 10 2.20 0.20 2.00 0.30 10 15 16 14 15.00 11 2.20 0.30 0.50 0.20 70 15 12 13 13.33 12 2.20 0.40 1.00 0.10 50 12 11 12 11.67 13 2.40 0.10 2.00 0.20 50 11 12 11 11.33 14 2.40 0.20 1.50 0.10 70 10 12 12 11.33 15 2.40 0.30 1.00 0.40 10 13 16 16 15.00 16 2.40 0.40 0.50 0.30 30 11 10 10 10.33 T1 35.67 44.67 44.33 45.00 55.33 - - - - T2 46.33 48.67 48.00 51.67 47.67 - - - - T3 53.67 51.67 51.67 46.33 45.00 - - - - T4 48.00 38.67 39.67 40.67 35.67 - - - - t1 8.92 11.17 11.08 11.25 13.83 - - - - t2 11.58 12.17 12.00 12.92 11.92 - - - - t3 13.42 12.92 12.92 11.58 11.25 - - - - t4 12.00 9.67 9.92 10.17 8.92 - - - - R 4.50 3.25 3.00 2.75 4.91 - - - - 注:T1~T4为各水平的平均得分总和;t1~t4分别为T1,T2,T3和T4的平均值Note:T1~T4 represent the sum of average scores of each treatment; t1~t4 represent the mean of T1, T2, T3, T4. -
[1] 李鹏飞, 刘萍, 李健, 等. 莱州湾三疣梭子蟹的生化遗传分析[J]. 海洋水产研究, 2007, 28(2): 90-96. doi: 10.3969/j.issn.1000-7075.2007.02.014 [2] 余红卫, 朱冬发, 韩宝芹. 三疣梭子蟹不同组织同工酶的分析[J]. 动物学杂志, 2005, 40(1): 84-87. doi: 10.3969/j.issn.0250-3263.2005.01.014 [3] 朱冬发, 余红卫, 王春琳. 三疣梭子蟹个体发育早期的同工酶谱变化[J]. 水产学报, 2005, 29(6): 751-756. [4] 王国良, 金珊, 李政, 等. 三疣梭子蟹养殖群体同工酶的组织特异性及生化遗传分析[J]. 台湾海峡, 2005, 24(4): 474-480. doi: 10.3969/j.issn.1000-8160.2005.04.009 [5] 高保全, 刘萍, 李健, 等. 三疣梭子蟹野生群体同工酶的遗传多态性分析[J]. 水产学报, 2007, 31(1): 1-6. doi: 10.3321/j.issn:1000-0615.2007.01.001 [6] 陈淑吟, 吉红九, 丁亚平, 等. 吕泗渔场三疣梭子蟹自然群体同工酶与ISSR遗传多样性分析[J]. 上海水产大学学报, 2008, 17(4): 406-410. https://xueshu.baidu.com/usercenter/paper/show?paperid=96ae324a3f5db3bc37f4d01704b5e62a&site=xueshu_se [7] 王敏强, 崔志峰, 刘晓玲, 等. 2种三疣梭子蟹居群线粒体Cyt b和S-rRNA基因片段序列变异研究[J]. 烟台大学学报: 自然科学与工程版, 2008, 21(3): 191-197. doi: 10.3969/j.issn.1004-8820.2008.03.008 [8] 郭天慧, 孔晓瑜, 陈四清, 等. 三疣梭子蟹线粒体DNA 16S rRNA和COI基因片段序列的比较研究[J]. 中国海洋大学学报, 2004, 34(1): 22-28. doi: 10.3969/j.issn.1672-5174.2004.01.002 [9] 张姝, 李喜莲, 崔朝霞, 等. 线粒体基因片段在梭子蟹系统发育及物种鉴定中的应用[J]. 海洋科学, 2008, 32(4): 9-18. https://xueshu.baidu.com/usercenter/paper/show?paperid=d662fff511c2b719a004bd69a41f7a99&site=xueshu_se&hitarticle=1 [10] 冯冰冰, 李家乐, 牛东红, 等. 我国沿海三疣梭子蟹9个野生群体线粒体CR和COI片段比较分析[J]. 动物学杂志, 2008, 43(2): 28-36. doi: 10.3969/j.issn.0250-3263.2008.02.005 [11] 冯冰冰, 李家乐, 牛东红, 等. 我国四大海域三疣梭子蟹线粒体控制区基因片段序列比较分析[J]. 上海水产大学学报, 2008, 17(2): 134-139. [12] 金珊, 赵青松, 王春琳, 等. 梭子蟹科六种海产蟹的RAPD标记[J]. 动物学研究, 2004, 25(2): 172-176. doi: 10.3321/j.issn:0254-5853.2004.02.016 [13] 刘爽, 薛淑霞, 孙金生. 黄海和东海三疣梭子蟹(Portunus triuberbuculatus)的AFLP分析[J]. 海洋与湖沼, 2008, 39(2): 152-156. doi: 10.11693/hyhz20080224024 [14] LI G, QUIROS C F. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica[J]. Theor Appl Genet, 2001, 103(2): 455-461. doi: 10.1007/s001220100570
[15] LIN Zhongxu, ZHANG Xianlong, NIE Yichun. Construction of a genetic linkage map for cotton based on SRAP[J]. Chin Sci Bull, 2003, 48(19): 2063-2067. doi: 10.1360/03wc0193
[16] LIN Zhongxu, ZHANG Xianlong, NIE Yichun. Evaluation of application of a new molecular marker SRAP analysis of F2 segregation population and genetic diversity in cotton[J]. Acta Genet Sin, 2004, 31(6): 622-626. doi: 10.1088/1009-0630/6/5/011
[17] BUDAK H, SHEARMAN R C, PARMAKSIZ I. Molecular characterization of buffalograss germplasm using sequence-related amplified polymorphism markers[J]. Theor Appl Genet, 2004, 108(2): 328-334. doi: 10.1007/s00122-003-1428-4
[18] BUDAK H, SHEARMAN RC, PARMAKSIZ I, et al. Comparative analysis of seeded and vegetative biotype buffalograsses based on phylogenetic relationship using ISSRs, SSRs, RAPDs and SRAPs[J]. Theor Appl Genet, 2004, 109(2): 280-288. doi: 10.1007/s00122-004-1630-z
[19] FERRIOL M, PICO B, NUEZ F. Genetic diversity of a germplasm collection of Cucurbita pepo using SRAP and AFLP markers[J]. Theor Appl Genet, 2003, 107(2): 271-282. doi: 10.1007/s00122-003-1242-z
[20] FERRIOL M, PICO B, NUEZ F. Genetic diversity of some accessions of Cucurbita maxima from Spain using RAPD and SRAP markers[J]. Genet Resour Crop Evol, 2003, 50(3): 227-238. doi: 10.1023/A:1023502925766
[21] 王燕青, 季孔庶. 利用正交设计优化牡丹SRAP-PCR反应体系[J]. 分子植物育种, 2009, 7(1): 199-203. doi: 10.3969/j.issn.1672-416X.2009.01.033 [22] 谢运海, 夏德安, 姜静, 等. 利用正交设计优化水曲柳ISSR-PCR反应体系[J]. 分子植物育种, 2005, 3(3): 445-450. doi: 10.3969/j.issn.1672-416X.2005.03.022 [23] 柳李旺, 龚义勤, 黄浩, 等. 新型分子标记—SRAP与TRAP及其应用[J]. 遗传, 2004, 26(5): 777-778. doi: 10.3321/j.issn:0253-9772.2004.05.039 [24] 郭大龙, 罗正荣. 部分柿属植物SRAP-PCR反应体系的优化[J]. 果树学报, 2006, 23(10): 138-141. doi: 10.3969/j.issn.1009-9980.2006.01.034 -
期刊类型引用(3)
1. 刘明泰, 李杨, 刘小林, 许淑芬, 常亚青. 皱纹盘鲍SRAP反应体系的正交优化. 大连海洋大学学报. 2013(01): 12-16 .  百度学术
百度学术
2. 智艳平, 武喜艳, 安瑶瑶, 郜刚. 青枯菌SRAP-PCR体系的建立与优化. 山西师范大学学报(自然科学版). 2013(04): 69-72 .  百度学术
百度学术
3. 程长洪, 张敏莹, 刘凯, 徐东坡, 段金荣, 周彦锋, 施炜纲. 暗纹东方鲀SRAP-PCR体系的建立与优化. 浙江农业学报. 2011(06): 1084-1089 .  百度学术
百度学术
其他类型引用(0)



 下载:
下载:




 粤公网安备 44010502001741号
粤公网安备 44010502001741号
