Molecular phylogenetics of genus Mytilus based on COI and 16S rRNA sequences
-
摘要:
比较分析了贻贝属5个种的线粒体DNA COI及16S rRNA基因片段序列,确定了贻贝属的系统进化关系。以翡翠贻贝(Perna viridis)和条纹隔贻贝(Septifer virgatus)为外群,基于距离法(NJ)、最大简约法(MP)、最大似然法(ML)、贝叶斯演绎法(BI)及贝叶斯联合模型分析法构建分子系统树。系统发育分析结果表明,5种贻贝[Mytilus edulis,紫贻贝(M.galloprovincialis),盖勒贻贝(M.trossulus),厚壳贻贝(M.coruscus),加州贻贝(M.coruscus)]在系统树上聚为2支。加州贻贝最为原始,厚壳贻贝次之。其中,紫贻贝和M.edulis具有很近的亲缘关系,而厚壳贻贝和加州贻贝的亲缘关系近。研究结果为贻贝属物种进化、迁移及其育种研究提供了理论基础。
Abstract:In the present study, we compare the partial sequences of mitochondrial COI and 16S rRNA of five genus Mytilus to study their phylogenetic relationship. Taking Perna viridis and Septifer virgatus as outgroups, phylogenetic trees are constructed based on neighbor-joining, maximum parsimony, maximum likelihood, Bayesian inference and combined Bayesian analyses methods. The result demonstrates that Mytilus edulis, M.galloprovincialis, M.trossulus, M.coruscus and M.coruscus form into two clades. Among the five species of genus Mytilus, M.coruscus is the most primitive, then comes M.coruscus. M.galloprovincialis and M.edulis show very close genetic relationship, and M.coruscus is closer to M.coruscus than to M.edulis, M.galloprovincialis and M.trossulus. The molecular phylogeny analysis provides theoretical reference for future comparative study on species evolution, immigration and breeding strategies of Mytilus.
-
Keywords:
- genus Mytilus /
- cytochrome oxidase Ⅰ /
- 16S rRNA /
- phylogeny /
- combined sequences analyses /
- divergent time
-
贻贝属隶属于软体动物门(Mollusca)、双壳纲(Bivalvia)、异柱目(Anisomyaria)、贻贝科(Mytilidae),作为海洋无脊椎动物中分布最为广泛的属之一,其种类出现在高纬度的所有海洋以及世界主要海域,而且贻贝已成为研究海洋生物种群分化和物种形成的重要模型[1]。在中国和欧洲许多国家,贻贝已成为重要的养殖贝类。
贻贝属常见的有5个种,分别为Mytilus edulis、紫贻贝(M.gallprovincialis)、盖勒贻贝(M.trossulus)、加州贻贝(M.coruscus)和厚壳贻贝(M.coruscus)。M.edulis主要分布于西欧和北美洲东部,紫贻贝主要分布于地中海、西欧、加利福尼亚及东亚,而盖勒贻贝则分布于波罗的海、西伯利亚、加拿大东部及北美西部。几乎在这些种类分布重叠的区域均发现了杂交现象,引起了很多关于贻贝分类地位的讨论[2]。
有关厚壳贻贝的研究相对较少,SCARLATTO[3]首次报道了来自于亚洲太平洋沿海的厚壳贻贝。遗传学方面仅见SHEN等[4]采用线粒体DNA COI基因序列对厚壳贻贝2个养殖群体与2个野生群体遗传多样性的研究报道。加州贻贝主要分布于北美西部沿海及南美等地区,其主要在潮间带生活,个体相对比较大。中国沿海主要有紫贻贝和厚壳贻贝分布。黄渤海盛产紫贻贝,大连的紫贻贝产量居中国之首,人工南移后,东海和南海也有一定产量。厚壳贻贝分布于黄渤海和东海。迄今对贻贝属不同物种之间的进化关系尚不明确。
线粒体DNA(mtDNA)是一种共价闭环双链DNA分子,由于其具有遵循严格的母系遗传、几乎没有重组、结构简单、进化速度快以及在不同的区域进化速度存在差异等优点,逐渐成为种群遗传学和分子系统发育研究的重要标记[5]。细胞色素氧化酶Ⅰ亚基(cytochrome oxidase subunit I,COI)是线粒体氧化呼吸链的重要成员,由于COI基因变异较大[6],其序列已被广泛应用于贝类的种质鉴定[7]、种群遗传结构分析[8]和分子系统发育分析[9]等研究。线粒体16S rRNA基因是非编码蛋白质基因,由于未受密码子编码的选择压力影响,其大部分区域发生的变异为中性突变[10],加之16S rRNA基因进化速度适中,因此常被应用于不同阶元物种的系统进化和分类研究[11]。该研究运用线粒体COI与16S rRNA基因部分序列对不同贻贝物种进化关系进行了探讨,以期为进一步的遗传分析、种质鉴定和系统进化研究提供理论依据。
1. 材料与方法
1.1 试验材料
试验所用的2个野生紫贻贝样品于2008年10月采自青岛花石楼东侧中潮区,另3个于2009年2月采自大连金州; 2个厚壳贻贝于2009年4月采自浙江温州平阳南麂列岛海域,另3个于2009年7月采自福建福鼎。取样品闭壳肌组织置于95%乙醇溶液中固定,保存备用。贻贝属其他种类及外群[翡翠贻贝(Perna viridis)和条纹隔贻贝(Septifer virgatus)]的序列信息来自GenBank(表 1)。
表 1 该研究所用样本种类、采样地点、采样时间、分子标记和GenBank注册号Table 1 Sample species, sampling sites and dates, and their molecular markers and GenBank accession No.种类
species采样地点
sampling site采样时间
sampling date16S rRNA COI 缩写编号
abbreviation No.来源
source缩写编号
abbreviation No.来源
source紫贻贝
M.galloprovincialis青岛
Qingdao2008.10 M.galloprovincialis1~
M.galloprovincialis2该研究 M.galloprovincialis1~
M.galloprovincialis2该研究 大连Dalian 2009.02 M.galloprovincialis3~
M.galloprovincialis5该研究 M.galloprovincialis3~
M.galloprovincialis5该研究 - - M.galloprovincialis6 AY497292 M.galloprovincialis6 AY497292 厚壳贻贝
M.coruscus温州
Wenzhou2009.04 M.coruscus1~
M.coruscus2该研究 M.coruscus1~
M.coruscus2该研究 福鼎
Fuding2009.07 M.coruscus3~
M.coruscus5该研究 M.coruscus3~
M.coruscus5该研究 - - M.coruscus6~
M.coruscus10GQ472146~
GQ472149,
AF317545M.coruscus6~
M.coruscus10FJ495279,
FJ495282,
FJ495284~
FJ495286M.edulis - - M.edulis1~
M.edulis2AF317054,
AY484747M.edulis1~
M.edulis2AY484747,
NC_006161盖勒贻贝
M.trossulus- - M.trossulus1~
M.trossulus3MTU22879~
MTU22881M.trossulus1~
M.trossulus2AF242033,
AF242034加州贻贝
M.coruscus- - M.coruscus AF317057, U68770 M.coruscus MCU68776 翡翠贻贝
P.viridis- - P.viridis AB265680 P.viridis DQ343574 条纹隔贻贝
S.virgatus- - S.virgatus GQ472165 S.virgatus AB076941 1.2 试验方法
1.2.1 基因组DNA提取
取约100 mg贻贝闭壳肌组织,采用标准的酚-氯仿方法[12]提取基因组DNA,将乙醇沉淀后的基因组DNA加入100 μL TE溶液溶解,置于4 ℃下保存备用。
1.2.2 目的片段的PCR扩增、纯化
使用无脊椎动物通用COI引物COI-F(5’-GGTCAACAAAT CATAAAGATATTGG) 和COI-R(5’ -TAAACTTCAG-GGTGACCAAAAAATCA-3’)[13]扩增了贻贝类线粒体COI基因片段。用于扩增16S rRNA基因片段的引物序列为16S rRNA-RA:5’-CGCCTGTTTATCAAAAACAT-3’;16S rRNA-RB:5’-CCGGTCTGAACTCAGATCACGT-3’[14]。PCR反应体系总体积为50 μL,每个反应体系内含10×PCR缓冲液[200 mmol · L-1 Tris-HCl,pH 8.4;200 mmol ·L-1 KCl;100 mmol · L-1 (NH4)2SO4;15 mmol · L-1 MgCl2] 5 μL,dNTP 200 μmol · L-1,上、下游引物各0.2 μmol · L-1,Taq酶1.25 U(大连宝生物公司出品),DNA模板20 ng,加Milli-Q H2O至50 μL。应用Eppendorf热循环仪进行PCR反应,COI基因反应条件为94 ℃变性40 s,52 ℃退火30 s,72 ℃延伸60 s,共35个循环。反应前94 ℃预变性4 min,反应后72 ℃延伸7 min。16S rRNA的扩增条件为95 ℃变性45 s,50 ℃退火45 s,72 ℃延伸45 s,共35个循环。反应前95 ℃预变性3 min,反应后72 ℃延伸10 min。
为排除DNA污染,以上反应均设有阴性对照。取2 μL PCR扩增产物采用1%琼脂糖凝胶电泳对扩增效果加以检测(U=5 V · cm-1)。应用UNIQ-10柱式DNA胶回收试剂盒(上海华舜)对PCR扩增产物进行回收纯化。
1.2.3 目的片段序列测定
应用ABI公司3700型全自动DNA序列分析仪进行双向测序以确保结果的准确性,测序反应采用的引物与PCR反应一致。
1.3 数据分析
应用DNAStar软件(DNASTAR,Inc)中的SeqMan软件对正反序列进行组装,得到COI和16S rRNA基因片段的一致序列。用EditSeq对所得到的序列进行编辑和分析,使用无脊椎动物线粒体遗传密码从第1个碱基开始把扩增的COI基因片段翻译成氨基酸序列;用MegAlign对序列进行比对,并计算序列差异百分比。用MEGA 2.0[15]统计变异位点数和转换(Ts)颠换(Tv)位点数,计算Ts/Tv以及简约信息位点(parsim-informative site)数。
利用PAUP*4.0b10[16]计算COI序列碱基组成及对序列碱基组成的异质性进行卡方检验(chisquare test,χ2)。用PAUP*4.0b10和Modeltest 3.7[17]得到COI和16S rRNA基因片段核苷酸最佳替换模型。使用PAUP*4.0b10和MrBayes 3.0[18]软件,应用距离法(NJ)、最大简约法(MP)、最大似然法(ML)和贝叶斯演绎法(Bayesian Inference,BI)分别重建了系统发育树。前3种方法在PAUP*4.0b10中进行,邻接法分析采用K2P距离建树;在重建贝叶斯发育树时,同时建立4条马尔科夫链,其中3条热链和1条冷链;共预算10×104~20×104代,对每100代进行1次抽样。然后根据对数似然值老化曲线,忽略动态抽样(这里取前2 500次),对剩余的树在PAUP*4.0b10里计算合意树和各分支的后验概率。同时对2个片段进行贝叶斯联合模型分析,采用分划同质性检验(partition homogeneity test)[19]评价序列片段联合分析的可靠性。对COI片段的1、2和3密码子及16S rRNA基因片段分别运用MrModeltest 2.0[20]软件获得最适合该研究序列矩阵的核苷酸替代模型及相关参数,选择最佳的分区策略,联合各个片段构建系统发育树。该研究采用了4种不同的分区策略(表 2),所有的分区策略均用大写字母P表示,数字下标表示分区的数目。
表 2 该研究所采用的分区策略Table 2 Partitioning strategies used in this study分区策略
partitioning strategies分区方法
partitioning methodP1 1个分区 P2 COI;16S rRNA P3 COI密码子第1和第2位点;COI密码子第3位点;16S rRNA P4 COI按3个密码子位点分区;16S rRNA 2. 结果
2.1 DNA序列组成及变异
5种贻贝COI基因片段同源序列为432 bp。在432 bp比对位点中有121个变异位点,107个简约信息位点;A、T、C和G的含量分别为26.4%、35.0%、16.8%和21.7%,A+T含量61.4%,具有明显的AT偏倚。转换/颠换的平均值2.79,转换主要为TC转换,但在颠换中,TA颠换与CG颠换基本一致。异质性检验表明密码子3个位点的组成均是同质的:第1位点χ2=75.218,df=63,P=0.139;第2位点χ2=50.226,df=63,P=0.878;第3位点χ2=42.428,df=63,P=0.978。COI的同质性表明该研究所用的数据不会对系统树的构建产生负面影响。
5种贻贝16S rRNA基因总长为460 bp。在460 bp长度片段中有68个变异位点,包含了56个简约信息位点;A、T、C和G的含量分别为29.6%、30.6%、15.8%和24.0%,A+T含量60.2%,具有明显的AT含量偏倚。转换/颠换的平均值2.53,TC和AG转换基本一致,但在颠换中,主要发生的是TA颠换,而CG颠换很少发生。
2.2 遗传分化
贻贝属种间的COI和16S rRNA基因的遗传距离见表 3。基于COI和16S rRNA基因片段得到物种间遗传距离分别为0.014~0.219和0.006~0.078。紫贻贝与M.edulis之间的遗传距离最小,基于COI与16S rRNA片段分别为0.014和0.006,而M.edulis与厚壳贻贝之间的遗传距离最大,达到0.219(COI)和0.078(16S rRNA)。加州贻贝与厚壳贻贝遗传距离为0.127(COI)和0.023(16S rRNA),小于与其他贻贝间的遗传距离。
表 3 贻贝属不同种类间的平均K2-P遗传距离(左下角:16S rRNA,右上角:COI)Table 3 Average K2-P genetic distance based on 16S rRNA region (below diagonal) and COI region (above diagonal) among different species of genus Mytilus种类
species紫贻贝
M.galloprovincialis厚壳贻贝
M.coruscusM.edulis 盖勒贻贝
M.trossulus加州贻贝
M.californianus紫贻贝M.galloprovincialis - 0.203 0.014 0.139 0.178 厚壳贻贝M.coruscus 0.070 - 0.219 0.187 0.127 M.edulis 0.006 0.078 - 0.154 0.188 盖勒贻贝M.trossulus 0.028 0.072 0.033 - 0.146 加州贻贝M.coruscus 0.068 0.023 0.071 0.071 - 该研究结果显示,在COI蛋白质编码基因片段中,检测到的大部分突变都是同义突变(synonymous substitution),其中最普遍的核苷酸替代模式是发生在密码子第3位点上的转换,其次是密码子第3位点上的颠换和密码子第1位点上的无义转换。氨基酸水平上,在COI基因长度为143个氨基酸序列上,仅在M.edulis序列上检测到1处氨基酸替代(amino acid substitution)。
根据核苷酸替代速率公式R=K/2T,K为每个位点的核苷酸替代数,T为2基因的分歧时间,在分歧时间一定的条件下,核苷酸替代速率与K成正比。2个线粒体基因组片段的核苷酸替代数COI>16S rRNA,如紫贻贝与厚壳贻贝间COI(0.203)>16S rRNA(0.070)。贻贝类COI基因片段的替代速率快于16S rRNA(表 3)。
以上新世中期(大约350万年前)白令海峡首次开放作为盖勒贻贝与北大西洋M.edulis群体分化的分子钟标准[21],基于COI及16S rRNA基因片段计算推测,则M.edulis大约在498万~827万年前最先与厚壳贻贝分化,然后与加州贻贝约在427万~753万年前分化,最后与紫贻贝在31.8万~63.6万年前分化。分化事件主要发生在晚中新世(Late Miocene)、早上新世(Early Pliocene)及晚更新世(Late Pleistocene)。
2.3 系统发育分析
2.3.1 COI与16S rRNA基因片段分析
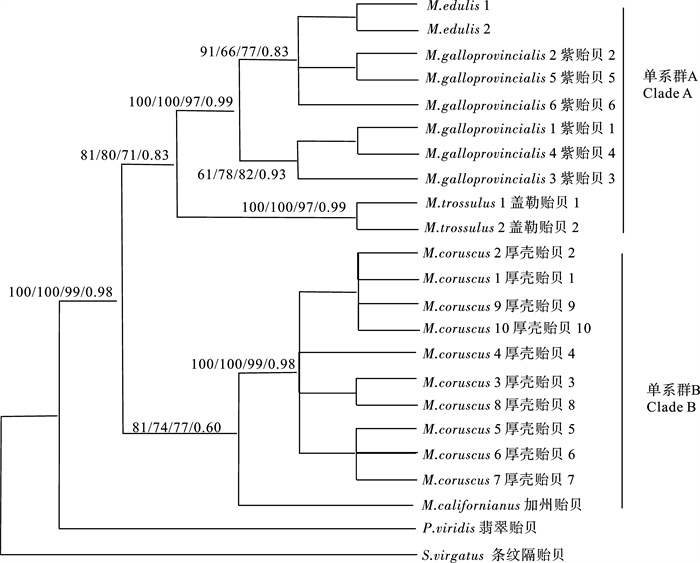
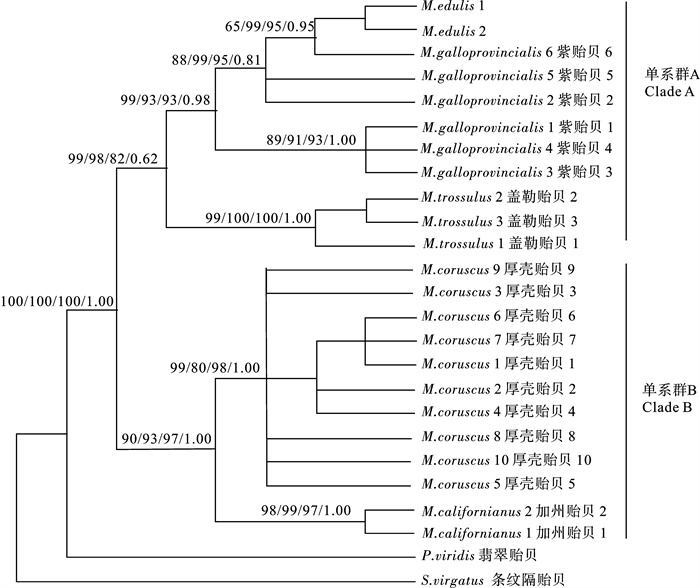
基于COI和16S rRNA基因序列,应用NJ、MP、ML和BI分别重建了系统发育树(图 1和图 2),得到的系统发育树拓扑结构基本一致,节点的支持率很高。5种贻贝属种类分为2个单系群(A和B), 单系群A包括贻贝属的M.edulis、紫贻贝和盖勒贻贝;单系群B包括厚壳贻贝和加州贻贝。其中加州贻贝与厚壳贻贝首先聚为1支,M.edulis与紫贻贝聚为1支再与盖勒贻贝聚类。M.edulis与紫贻贝的亲缘关系最近。
![]() 图 1 基于COI运用邻接法、MP、ML和BI构建的贻贝属种类系统树(后验概率及支持率标注在分支上, 后图同)Fig. 1 Phylogenetic tree of Mytilus species constructed with neighbor-joining, maximum parsimony, maximum likelihood and Bayes analyses based on COI (numbers on the tree represent posterior probability values and supporting values; the same case in the following figure)
图 1 基于COI运用邻接法、MP、ML和BI构建的贻贝属种类系统树(后验概率及支持率标注在分支上, 后图同)Fig. 1 Phylogenetic tree of Mytilus species constructed with neighbor-joining, maximum parsimony, maximum likelihood and Bayes analyses based on COI (numbers on the tree represent posterior probability values and supporting values; the same case in the following figure)2.3.2 COI与16S rRNA序列联合模型分析
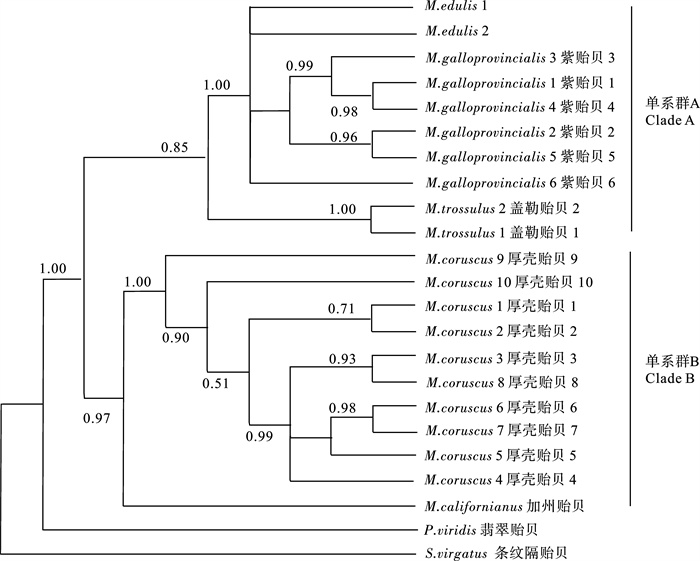
对COI和16S rRNA 2个基因片段进行贝叶斯联合模型分析,以得出正确的进化信息, 构建更为合理、正确的系统发育树。用PAUP软件对COI和16S rRNA 2个基因片段采用分划同质性检验(partition homogeneity test)[19],评价联合分析的可靠性,结果P值为0.07,说明可将2个基因片段进行联合分析。联合数据共包含901个碱基,不同分区的最适模型见表 4。由于每个贝叶斯联合模型分析所得到的系统树拓扑结构几乎完全一致,该研究以P3生成的贝叶斯树(图 3)来讨论贻贝属系统进化关系。其结果与运用2个基因片段独立分析的结果基本一致,并且具有较高的后验概率。即加州贻贝与厚壳贻贝先构成1支,再与M.edulis、紫贻贝和盖勒贻贝构成的分支聚类。
表 4 不同分区及其最适进化模型和所包含位点数Table 4 Partitions, optimal model of sequence evolution and total number of characters in each partition分区
partition进化模型
evolutionary model分区所包含位点数
number of characters in partitionCOI GTR+G 432 COI第1位点 HKY+G 144 COI第2位点 HKY 144 COI第3位点 HKY+G 144 COI第1和第2位点 HKY+G 288 16S rRNA GTR+G 469 联合数据 GTR+I+G 901 ![]() 图 3 基于COI与16S rRNA序列通过贝叶斯联合模型(P3)构建的50%一致树节点处的数字代表后验概率(小于50%的未显示)Fig. 3 50% consensus tree of Mytilus species constructed with Bayesian analysis (strategy P3) based on COI and 16S rRNA sequences.Bayesian posterior probabilities are indicated above the branch(bootstrap scores larger than 50% were shown)
图 3 基于COI与16S rRNA序列通过贝叶斯联合模型(P3)构建的50%一致树节点处的数字代表后验概率(小于50%的未显示)Fig. 3 50% consensus tree of Mytilus species constructed with Bayesian analysis (strategy P3) based on COI and 16S rRNA sequences.Bayesian posterior probabilities are indicated above the branch(bootstrap scores larger than 50% were shown)3. 讨论
一般认为,在线粒体基因组中,12S rRNA和16S rRNA基因进化速率比较低[22]且相对保守,而COI基因变异性较大,在不少无脊椎动物中检测到了较大的遗传变异[23]。从该试验的结果可以看出,在长度为460 bp的16S rRNA片段中,5种贻贝共检测到68个碱基变异,而在长度为432 bp的COI基因片段中,共检测到了121个碱基的变异。由此可见,在5个贻贝种类中COI基因的进化速率比16S rRNA基因快。贻贝类线粒体COI和16S rRNA基因在不同贻贝种间多态性明显差异,证实了COI和16S rRNA基因序列均普遍适用于贻贝类种以上阶元的系统发育分析。该研究结果显示贻贝2基因片段的A+T含量基本一致,分别为61.4%和60.2%,都明显高于G+C含量,这一结果与其他学者在头足类、甲壳类和双壳贝类等的16S rRNA、12S rRNA和COI基因研究结果一致[24-26],较高的AT含量应为无脊椎动物线粒体DNA中的普遍现象[27]。
该研究讨论了贻贝属种间的系统进化关系。贻贝属的广泛分布及生存环境对其贝壳外形的影响使该属的分类非常混乱[28]。在贻贝分类研究方面,国外做了大量的报道[29-32],特别是M.edulis、紫贻贝和盖勒贻贝的形态极其相似而难以区分。随着分子生物学的迅速发展,分子标记被广泛应用于贻贝类的分类研究。KOEHN等[33]通过5个同工酶基因位点对北美东部地区的贻贝进行了研究,认为M.edulis、紫贻贝和盖勒贻贝是不同的物种。McDONALD等[28]开展了贻贝的同工酶分析和形态特征研究,多元统计分析结果表明这3种贻贝构成了1个大的类群,但每种贻贝在遗传和形态表型上都各自独立。
该研究分析结果与McDONALD等[28]对3种贻贝进行的形态学和生物学研究结果一致。这3种贻贝在NJ树、MP树、ML树及BI树中形成1个单系群。3种贻贝进化关系分析表明M.edulis和紫贻贝最相似,而两者与盖勒贻贝距离稍远,盖勒贻贝形成了1个单系,且节点支持率较高(>80)(图 1~图 3)。M.edulis和紫贻贝间的遗传距离0.014(COI)和0.006(16S rRNA)小于紫贻贝种内个体间遗传距离0.021(COI)和0.007(16S rRNA),而且在聚类树中先聚为1个分支(图 1~图 3)。盖勒贻贝与M.edulis的净遗传距离为15.4%(COI),这与RIGINOS等[34]得出的紫贻贝与盖勒贻贝间遗传差异17.1%(4.89%/MY)相近。宋文涛等[35]基于线粒体基因组全序列、蛋白编码基因和rRNA基因构建的16种双壳贝类系统树中,M.edulis先与紫贻贝聚类再与盖勒贻贝聚在一起,这与该研究结果一致。HILBISH等[36]开展了贻贝类反热带分布起源研究,3种贻贝的聚类结果与该研究结果基本一致,盖勒贻贝单独1支,但M.edulis与紫贻贝个体有交叉。GERARD等[37]应用16S rRNA与COI序列对南北半球贻贝分化进行研究,也得到了类似的研究结果。
然而,该研究结果与沈玉帮等[38]运用16S rRNA及WENNE和SKIBINSKI[39]运用线粒体DNA限制性内切酶方法对这3种贻贝的进化关系的研究结果存在差异,2项研究均显示M.edulis与盖勒贻贝亲缘关系最近,遗传距离最小。与沈玉帮等[38]研究结果的差异可能是由于其用于分析的16S rRNA基因片段长度仅为282 bp,而该研究中用于分析的该基因片段长度为469 bp,且结合COI片段并进一步对2个基因片段进行贝叶斯联合模型分析,在一定程度上能更为全面、准确地反映其遗传信息。与WENNE和SKIBINSKI[39]研究结果的差异可能是由于在其研究中未将mtDNA 2种基因型(F型与M型)[40]加以区分,M.edulis与盖勒贻贝均有高频率的个体异质性导致两者相近。
VARVIO等[41]运用酶电泳法结合地质古生物(geological-palaeontological)认为紫贻贝是在更新世起源于地中海,从M.edulis的祖先进化而来,且2个种在形态上存在差异,从细胞遗传上亦可区分,推测盖勒贻贝可能是起源于北部的更为普遍古老种类的残余体(relict)。
分子钟假说认为,某一特定的大分子在所有的世系(lineage)中,核苷酸替代速率在时间上是稳定的。基于无脊椎动物的研究数据,以350万年前白令海峡开放作为分子钟标准[21],则M.edulis大约在498万~827万年前最先与厚壳贻贝分化,然后与加州贻贝在427万~753万年前分化,该结果与ORT和POGSON[42]关于M.edulis类与加州贻贝分化年代的结果相近(7.64~7.61 MYA),最后与紫贻贝在31.8万~63.6万年前分化。分化事件主要发生在晚中新世(Late Miocene)、早上新世(Early Pliocene)及晚更新世(Late Pleistocene)。
在线粒体中,COI和16S rRNA基因进化速率相对比较慢,为了更准确地确定贻贝属的系统发育关系,还需结合地理学、生态学及古生物学等方面的资料进行综合分析,其属内系统分化尚有待进一步研究。
致谢: 王茂林博士和张辉硕士帮忙采集紫贻贝样品,谨此致谢! -
图 1 基于COI运用邻接法、MP、ML和BI构建的贻贝属种类系统树(后验概率及支持率标注在分支上, 后图同)
Figure 1. Phylogenetic tree of Mytilus species constructed with neighbor-joining, maximum parsimony, maximum likelihood and Bayes analyses based on COI (numbers on the tree represent posterior probability values and supporting values; the same case in the following figure)
图 3 基于COI与16S rRNA序列通过贝叶斯联合模型(P3)构建的50%一致树节点处的数字代表后验概率(小于50%的未显示)
Figure 3. 50% consensus tree of Mytilus species constructed with Bayesian analysis (strategy P3) based on COI and 16S rRNA sequences.Bayesian posterior probabilities are indicated above the branch(bootstrap scores larger than 50% were shown)
表 1 该研究所用样本种类、采样地点、采样时间、分子标记和GenBank注册号
Table 1 Sample species, sampling sites and dates, and their molecular markers and GenBank accession No.
种类
species采样地点
sampling site采样时间
sampling date16S rRNA COI 缩写编号
abbreviation No.来源
source缩写编号
abbreviation No.来源
source紫贻贝
M.galloprovincialis青岛
Qingdao2008.10 M.galloprovincialis1~
M.galloprovincialis2该研究 M.galloprovincialis1~
M.galloprovincialis2该研究 大连Dalian 2009.02 M.galloprovincialis3~
M.galloprovincialis5该研究 M.galloprovincialis3~
M.galloprovincialis5该研究 - - M.galloprovincialis6 AY497292 M.galloprovincialis6 AY497292 厚壳贻贝
M.coruscus温州
Wenzhou2009.04 M.coruscus1~
M.coruscus2该研究 M.coruscus1~
M.coruscus2该研究 福鼎
Fuding2009.07 M.coruscus3~
M.coruscus5该研究 M.coruscus3~
M.coruscus5该研究 - - M.coruscus6~
M.coruscus10GQ472146~
GQ472149,
AF317545M.coruscus6~
M.coruscus10FJ495279,
FJ495282,
FJ495284~
FJ495286M.edulis - - M.edulis1~
M.edulis2AF317054,
AY484747M.edulis1~
M.edulis2AY484747,
NC_006161盖勒贻贝
M.trossulus- - M.trossulus1~
M.trossulus3MTU22879~
MTU22881M.trossulus1~
M.trossulus2AF242033,
AF242034加州贻贝
M.coruscus- - M.coruscus AF317057, U68770 M.coruscus MCU68776 翡翠贻贝
P.viridis- - P.viridis AB265680 P.viridis DQ343574 条纹隔贻贝
S.virgatus- - S.virgatus GQ472165 S.virgatus AB076941 表 2 该研究所采用的分区策略
Table 2 Partitioning strategies used in this study
分区策略
partitioning strategies分区方法
partitioning methodP1 1个分区 P2 COI;16S rRNA P3 COI密码子第1和第2位点;COI密码子第3位点;16S rRNA P4 COI按3个密码子位点分区;16S rRNA 表 3 贻贝属不同种类间的平均K2-P遗传距离(左下角:16S rRNA,右上角:COI)
Table 3 Average K2-P genetic distance based on 16S rRNA region (below diagonal) and COI region (above diagonal) among different species of genus Mytilus
种类
species紫贻贝
M.galloprovincialis厚壳贻贝
M.coruscusM.edulis 盖勒贻贝
M.trossulus加州贻贝
M.californianus紫贻贝M.galloprovincialis - 0.203 0.014 0.139 0.178 厚壳贻贝M.coruscus 0.070 - 0.219 0.187 0.127 M.edulis 0.006 0.078 - 0.154 0.188 盖勒贻贝M.trossulus 0.028 0.072 0.033 - 0.146 加州贻贝M.coruscus 0.068 0.023 0.071 0.071 - 表 4 不同分区及其最适进化模型和所包含位点数
Table 4 Partitions, optimal model of sequence evolution and total number of characters in each partition
分区
partition进化模型
evolutionary model分区所包含位点数
number of characters in partitionCOI GTR+G 432 COI第1位点 HKY+G 144 COI第2位点 HKY 144 COI第3位点 HKY+G 144 COI第1和第2位点 HKY+G 288 16S rRNA GTR+G 469 联合数据 GTR+I+G 901 -
[1] BEYNON C M, SKIBINSKI D O F. The evolutionary relationships between three species of mussel (Mytilus) based on anonymous DNA polymorphisms[J]. J Exp Mar Biol Ecol, 1996, 203(1): 1-10. doi: 10.1016/0022-0981(96)02565-8
[2] JOHANNESSON K, KAUTSKY N, TEDENGREN M. Genotypic and phenotypic differences between Baltic and North Sea populations of Mytilus edulis evaluated through reciprocal transplantations. Ⅱ Genetic variation[J]. Mar Ecol Prog Ser, 1990, 59(3): 211-219. doi: 10.3354/meps059221
[3] SCARLATTO O A. Bivalve mollusks of temperate latitudes of the western portion of the Pacific Ocean[M]. Moscow: Opred Faune SSSR, 1981, 126: 1-461. https://a.xueshu.baidu.com/usercenter/paper/show?paperid=36f12c41c240674962b05bf719855f99
[4] SHEN Yubang, LI Jiale, FENG Bingbing. Genetic analysis of cultured and wild populations of Mytilus coruscus based on mitochondrial DNA[J]. Zool Res, 2009, 30(3): 240-246. doi: 10.3724/SP.J.1141.2009.03240
[5] AVISE J C. Phylogeography[M]. London: Harvard University Press, 2000: 1-36. https://www.nature.com/articles/6887654
[6] HOEH W R, STEWART D T, SUTHERLAND B W, et al. Cytochrome c oxidase sequence comparisons suggest an unusually high rate of mitochondria DNA evolution in Mytilus (Mollusca: Bivalvia)[J]. Mol Biol Evol, 1996, 13(2): 418-421. doi: 10.1093/oxfordjournals.molbev.a025600
[7] 陈丽梅, 孔晓瑜, 喻子牛, 等. 3种蛏类线粒体16S rRNA和COI基因片段的序列比较及其系统学初步研究[J]. 海洋科学, 2005, 29(8): 27-32. doi: 10.3969/j.issn.1000-3096.2005.08.008 [8] KING T L, EACKLES M S, GJETVAJ B, et al. Intraspecific phylogeography of Lasmigona subviridis (Bivalvia: Unionidae): conservation implications of range discontinuity[J]. Mol Ecol, 1999, 8(S1): 65-78. doi: 10.1046/j.1365-294X.1999.00784.x
[9] AN H S, JEE Y J, MIN K S, et al. Phylogenetic analysis of six species of Pacific abalone (Haliotidae) based on DNA sequences of 16S rRNA and cytochrome c oxidase subunit I mitochondrial genes[J]. Mar Biotech, 2005, 7(4): 373-380. doi: 10.1007/s10126-004-4405-2
[10] 郑雷, 何明亮, 靳嘉瑞, 等. 蓖麻蚕核糖体大亚基RNA基因3'端序列分析及进化研究[J]. 遗传学报, 1997, 24 (4): 296-304. doi: 10.1007/BF02951625 [11] 蒙子宁, 庄志猛, 丁少雄, 等. 中国近海8种石首鱼类的线粒体16S rRNA基因序列变异及其分子系统进化[J]. 自然科学进展, 2004, 14(5): 514-521. doi: 10.3321/j.issn:1002-008X.2004.05.006 [12] SAMBROOK J, FRITSCH E F, MANIATIS T, et al. Molecular cloning: a laboratory manual[M]. New York: Cold Spring Harbor Laboratory Press, 1996: 464-468.
[13] FOLMER O, BLACK M, HOEH W, et al. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates[J]. Mol Mar Biol Biotech, 1994, 3(5): 294-299.
[14] PALUMBI S R, MARTIN A P, ROMANO S, et al. The simple fool's guide to PCR. version 2.0[M]. Honolulu: Department of Zoology, University of Hawaii, 1991. https://xueshu.baidu.com/usercenter/paper/show?paperid=3e4cec4de4499d3d3db63e0c20e8e3ef&site=xueshu_se
[15] KUMAR S, TAMURA K, JAKOBSEN I B, et al. MEGA: molecular evolutionary genetics analysis software[J]. Bioinformatics, 2001, 17(12): 1244-1245. doi: 10.1093/bioinformatics/17.12.1244
[16] SWOFFORD D L. PAUP*. Phylogenetic analysis using parsimony (* and other methods), version 4[M]. Sunderland, MA: Sinauer Assoc, 2002. doi: 10.1111/j.0014-3820.2002.tb00191.x
[17] POSADA D, CRANDALL K A. Modeltest: testing the model of DNA substitution[J]. Bioinformatics, 1998, 14(9): 817-818. doi: 10.1093/bioinformatics/14.9.817
[18] RONQUIST F, HUELSENBECK J P. Mrbayes 3: Bayesian phylogenetic inference under mixed models[J]. Bioinformatics, 2003, 19(12): 1572-1574. doi: 10.1093/bioinformatics/btg180
[19] FARRIS J, KALLERSJO M, KLUGE A, et al. Testing significance of incongruence[J]. Cladistics, 1995, 10: 315-319. doi: 10.1111/j.1096-0031.1994.tb00181.x
[20] NYLANDER J. MrModeltest 2.0. Program distributed by the author[M]. Sweden: Evolutionary Biology Centre, Uppsala University, 2004. https://www.researchgate.net/publication/285805344_MrModeltest_V2_Program_Distributed_by_the_Author
[21] RIGINOS C, HICKERSON M J, HENZLER C M, et al. Differential patterns of male and female mtDNA exchange across the Atlantic Ocean in the blue mussel, Mytilus edulis[J]. Evol, 2004, 58(11): 2438-2451. doi: 10.1111/j.0014-3820.2004.tb00873.x
[22] 毕潇潇, 高天翔, 肖永双, 等. 4种鳕鱼线粒体16S rRNA、COI和Cyt b基因片段序列的比较研究[J]. 南方水产, 2009, 5(3): 46-521. doi: 10.3969/j.issn.1673-2227.2009.03.008 [23] HOWLAND D E, HEWITT G M. Phylogeny of the coleoptera based on mitochondrial cytochrome oxidase I sequence data[J]. Insect Mol Biol, 1995, 4(3): 203-205. doi: 10.1111/j.1365-2583.1995.tb00026.x
[24] ANDERSON F E. Phylogeny and historical biogeography of the loliginid squids (Mollusca: Cephalopoda) based on mitochondrial DNA sequence data[J]. Mol Phylogenet Evol, 2000, 15(2): 191-214. doi: 10.1006/mpev.1999.0753
[25] 高天翔, 张秀梅, 吉崎悟朗, 等. 中华绒螯蟹和日本绒螯蟹线粒体12S rRNA基因序列研究[J]. 中国海洋大学学报, 2000, 30(1): 43-47. doi: 10.3321/j.issn:1000-0615.2000.05.005 [26] 孔晓瑜, 刘亚军, 喻子牛, 等. 栉孔扇贝和海湾扇贝线粒体DNA 16S rRNA基因片段序列研究[G]//中国贝类学会. 贝类学论文集第Ⅸ辑. 北京: 海洋出版社, 2001: 59-63. https://xueshu.baidu.com/usercenter/paper/show?paperid=4aefd57bc421ec93adf0ae0efa01aeae&site=xueshu_se [27] 黄桂菊, 喻达辉, 郭奕惠, 等. 珍珠贝基于16S rRNA基因序列的亲缘关系研究[J]. 南方水产, 2009, 5(6): 47-53. doi: 10.3969/j.issn.1673-2227.2009.06.009 [28] McDONALD J H, SEED R, AND KOEHN R K. Allozymes and morphometric characters of three species of Mytilus in the Northern and Southern Hemispheres[J]. Mar Biol, 1991, 111(3): 323-333. doi: 10.1007/BF01319403
[29] KOEHM R K. The genetics and taxonomy of species in the genus Mytilus[J]. Aquac, 1991, 94(2/3): 125-145. doi: 10.1016/0044-8486(91)90114-M
[30] MCDONALD J H, KOEHM R K. The mussels Mytilus galloprovincialis and M.trossulus on the Pacific of North America[J]. Mar Biol, 1988, 99(1): 111-118. doi: 10.1007/BF00644984
[31] EDWARDS C A, SKIBINSKI D O F. Genetic variation of mitochondrial DNA in mussel (Mytilus edulis and M. galloprovincialis) populations from South West England and South Wales[J]. Mar Biol, 1987, 94(4): 547-556. doi: 10.1007/BF00431401
[32] VARVIO S L, KOEHN R, VAINOLA R. Evolutionary genetics of the Mytilus edulis complex in the North Atlantic region[J]. Mar Biol, 1988, 98(1): 51-60. doi: 10.1007/BF00392658
[33] KOEHN R K, HALL J G, INNES D J, et al. Genetic differentiation in Mytilus edulis in eastern North America[J]. Mar Biol, 1984, 79(2): 117-126. doi: 10.1007/BF00951820
[34] RIGINOS C, HICKERSON M J, HENZLER C M, et al. Differential patterns of male and female mtDNA exchange across the Atlantic Ocean in the blue mussel, Mytilus edulis[J]. Evol, 2004, 58(11): 2438-2451. doi: 10.1111/j.0014-3820.2004.tb00873.x
[35] 宋文涛, 高祥刚, 李云峰, 等. 双壳贝类线粒体基因组结构的比较[J]. 遗传, 2009, 31(11): 1-11. doi: 10.3724/SP.J.1005.2009.01127 [36] HILBISH T J, MULLINAX A, DOLVEN S I, et al. Origin of the antitropical distribution pattern in marine mussels (Mytilus spp. ): routes and timing of transequatorial migration[J]. Mar Biol, 2000, 136(1): 69-77. doi: 10.1007/s002270050010
[37] GERARD K, BIERNE N, BORSA P, et al. Pleistocene separation of mitochondrial lineages of Mytilus spp. mussels from northern and southern hemispheres and strong genetic differentiation among southern populations[J]. Mol Phylogenet Evol, 2008, 49(1): 84-91. doi: 10.1016/j.ympev.2008.07.006
[38] 沈玉帮, 李家乐, 牟月军. 基于16S rRNA序列初步探讨贻贝属的系统发育[J]. 海洋科学, 2009, 33(12): 50-55. https://xueshu.baidu.com/usercenter/paper/show?paperid=6a91cff406d099a76e025bf5375c4520&site=xueshu_se&hitarticle=1 [39] WENNE R, SKIBINSKI D O F. Mitochondrial DNA heteroplasmy in European populations of the mussel, Mytilus trossulus[J]. Mar Biol, 1995, 122(4): 619-624. doi: 10.1007/BF00350683
[40] ZOUROS E, BALL A O, SAAVEDRA C, et al. A unusual type of mitochondrial DNA inheritance in the blue mussel Mytilus[J]. Proc Natl Acad Sci USA, 1994, 91(16): 7463-7467. doi: 10.1073/pnas.91.16.7463
[41] VARVIO S L, KOEHN R K, VAINOLA R. Evolutionary genetics of the Mytilus edulis complex in the North Atlantic region[J]. Mar Biol, 1988, 98(1): 51-60. doi: 10.1007/BF00392658
[42] ORT B S, POGSON G. H. Molecular population genetics of the male and female mitochondrial DNA molecules of the California Sea mussel, Mytilus californianus[J]. Genet, 2007, 177(2): 1087-1099. doi: 10.1534/genetics.107.072934
-
期刊类型引用(12)
1. 袁杭洋,周万铠,郑泽延,林国锡,徐永健. 基于CO I和16S rRNA基因序列分析与比较中国沿海7个脊尾白虾群体的遗传多样性. 中国水产科学. 2024(02): 165-176 .  百度学术
百度学术
2. 朱懿,马培振,李翠,左晨霞,王海艳,张振. 贻贝目软体动物分类学与系统演化研究进展. 海洋科学. 2024(08): 106-122 .  百度学术
百度学术
3. 顾忠旗,苗菁,马颜雯,黄继,叶莹莹. 基于线粒体COI及16S rRNA基因序列的笠贝科系统进化研究. 浙江海洋大学学报(自然科学版). 2023(03): 205-211+254 .  百度学术
百度学术
4. 吴晓雯,张华伟,余海,彭欣,张永普. 基于DNA条形码对浙南岛屿日本花棘石鳖的遗传特征分析. 海洋渔业. 2018(03): 265-278 .  百度学术
百度学术
5. 王朋云. 莆田南日岛泥东风螺的形态、DNA条形码和同工酶初步分析. 渔业研究. 2017(04): 249-263 .  百度学术
百度学术
6. 卫晓英,李苹苹,任秀娟,梁红霞,李娜. 贻贝油膏多酶协同水解工艺研究. 中国调味品. 2016(05): 76-80 .  百度学术
百度学术
7. 杨振雄,毛阳丽,宋娜,高天翔,蔡厚才,张朝晖. 浙江和福建沿海厚壳贻贝Mytilus coruscus群体的COI序列比较分析. 海洋湖沼通报. 2014(02): 82-88 .  百度学术
百度学术
8. 刘萍,段亚飞,毛智超,李吉涛,高保全,李健. 中华虎头蟹线粒体16S rRNA和COⅠ基因的序列比较及其系统进化分析. 水产学报. 2013(10): 1441-1451 .  百度学术
百度学术
9. 叶莹莹,徐梅英,郭宝英,吴常文. 我国东南沿海厚壳贻贝(Mytilus coruscus)4个群体线粒体16S rRNA序列及遗传结构分析. 海洋与湖沼. 2012(02): 376-381 .  百度学术
百度学术
10. 苏天凤,江世贵,张殿昌,周发林,黄建华,朱彩艳,杨丽诗. 粤东海域褐菖鲉种群线粒体DNA控制区序列变异及其在平鲉亚科中的分类地位. 渔业科学进展. 2012(02): 1-8 .  百度学术
百度学术
11. 曾晓起,张文峰,高天翔. 基于线粒体16S rRNA与COI基因序列的刻肋海胆属系统发育研究. 中国海洋大学学报(自然科学版). 2012(06): 47-51 .  百度学术
百度学术
12. 刘慧慧,薛超波,常抗美,杨刚. 厚壳贻贝M7lysin分子的克隆与表达. 水产学报. 2011(09): 1337-1342 .  百度学术
百度学术
其他类型引用(22)



 下载:
下载:
 粤公网安备 44010502001741号
粤公网安备 44010502001741号
