Molecular cloning and characterization analysis of glutathione peroxidase 1 from Nile tilapia (Oreochromis niloticus)
-
摘要:
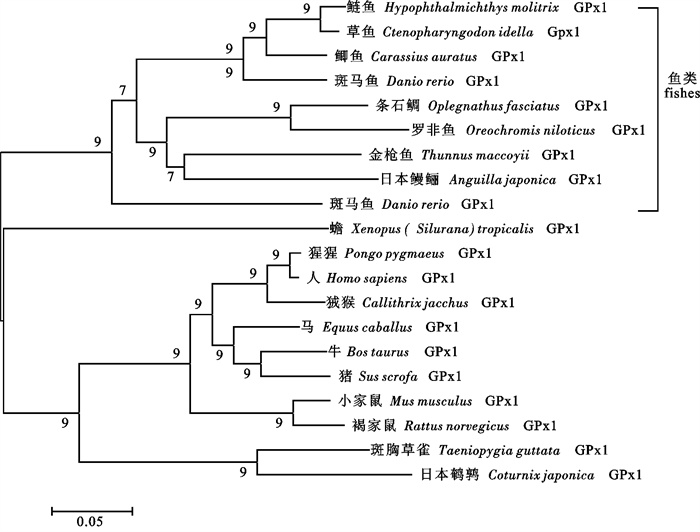
采用cDNA末端快速扩增(rapid amplification of cDNA ends,RACE)技术,克隆了罗非鱼(Oreochromis niloticus)谷胱甘肽过氧化物酶1(glutathione peroxidase1,GPx1)基因完整的编码序列(complete coding sequence,CDS)。GPx1基因全长984 bp,5′UTR 56 bp,CDS 576 bp,3′UTR 352 bp,PolyA 20 bp;第791~885位碱基(位于3′UTR)形成1个硒半胱氨酸插入序列(selenocysteine insertion sequence,SECIS),协助174~176位密码子TGA(UGA)编码1个硒半胱氨酸(Sec)。GPx1包含191个氨基酸,分子量21.8 kDa,等电点8.04,无信号肽和潜在的N-糖基化位点。蛋白结构分析表明该基因编码蛋白为非跨膜蛋白。序列比对显示,GPx1单体具有Sec、Trp、Gln和Asn构成的催化四联体。罗非鱼GPx1与其他脊椎动物GPx1相比较,核苷酸序列相似性为43.2%~58.2%,氨基酸序列相似性为58.1%~80.6%。进化分析显示,处在分类学上不同纲的脊椎动物GPx1分别占据了不同分支。利用Swiss-Model预测了罗非鱼GPx1的3D结构,序列分析显示,GPx1可以形成1个同源四聚体。
-
关键词:
- 罗非鱼 /
- 谷胱甘肽过氧化物酶1 /
- 基因克隆
Abstract:The complete coding sequence of glutathione peroxidase 1 (GPx1) of Nile tilapia (Oreochromis niloticus) was cloned by rapid amplification of cDNA ends (RACE).The GPx1 contains 984 bp, including a 56 bp 5′-untranslated region, a 576 bp coding sequence (CDS), a 352 bp 3′-untranslated region and a 20 bp PolyA tail. A selenocysteine (Sec) was encoded by the unusual stop codon, TGA, with the selenocysteine insertion sequence (SECIS) located at 3′UTR. The GPx1 was predicted to encode 191 amino acids, and its molecular weight was 21.8 kDa with a pI of 8.04; neither signal peptide nor N-Glycosylation site was found. The analysis showed that GPx1 was a non-transmembrane protein, and it possessed classic catalytic tetrad comprising selenocysteine, tryptophan, glutamine and asparagine. We compared GPx1 between Nile tilapia and other vertebrate animals, and found that the similarity of nucleotide acid sequence was 43.2%~58.2%, and that of amino acid sequence was 58.1%~80.6%. Phylogenic analysis indicated that GPx1s of different classes of vertebrates split into different clusters. The 3D structure of tilapia GPx1 was predicted with Swiss-Model software, and sequence analysis suggested that GPx1 was homotetrameric.
-
Keywords:
- Oreochromis niloticus /
- glutathione peroxidase1 /
- gene cloning
-
罗非鱼(Oreochromis spp.)属鲈形目,丽鱼科,是原产于非洲的热带性鱼类,得到联合国粮农组织的大力推荐,成为世界最重要的养殖鱼类之一[1-2]。2010年罗非鱼全球产量达到320×104 t,中国占其35%,居于世界首位[3-4]。当前实际生产中存在较多的罗非鱼种间杂交,从而使得种质发生退化和混杂,因此迫切需要寻找群体间的遗传标记[5]用于种质鉴定。近年来广泛应用的DNA分子标记在罗非鱼等水产动物的遗传多样性、近亲繁殖、品系鉴定等方面都有很大进展[6]。DNA分子标记由于不受环境、季节、发育阶段、组织的影响与限制,能够直接反应DNA的多态性,易于建立与目标性状的关联,其在鱼类种质鉴定、遗传多样性分析、遗传图谱构建、基因定位、亲缘关系分析、分子辅助育种中得到广泛的研究与应用[7-8]。其中基于保守序列、保守基序或保守调控核心序列或真核生物中基因序列的特点,以及表达序列标签(EST)的目标分子标记技术受到越来越多研究者的重视[9-10]。基于基因序列和EST的靶位区域扩增多态性(target region amplification polymorphism,TRAP)是HU和VICK[11]在向日葵(Helianthus annuus)上提出的新型目标分子标记,其利用EST或cDNA序列信息和生物信息工具,对目标区域序列进行扩增产生多态性分子标记。TRAP具有操作简单、重复性好、效率高和易测序等优点[12],随后在水稻(Oryza sativa)、莴苣(Lactuca sativa)、小麦(Triticum aestivum)、油菜(Brassica napus)等植物的遗传多样性分析、重要性状标记筛选、遗传图谱构建等得到应用[13-17]。同时TRAP在草鱼(Ctenopharyngodon idella)、鲤(Cyprinus carpio)、鲶(Silurus asotus)、鳜(Siniperca chuatsi)等水产动物研究中也得到运用[18-20]。但TRAP在罗非鱼上的应用以及应用过程中的PCR反应体系的建立未见报道。最早由何正文等[21]提出的正交设计法优化PCR反应体系方法,由于其试验规模小且能够考虑各因素间的交互作用,随后用于大麻哈鱼(Oncorhynchus keta)SRAR-PCR、葡萄(Vitis vinifera)SCoT-PCR等PCR反应体系优化中[22-23]。而水产动物TRAP反应体系优化都是以保持体系中其他参数不变针对一个影响因子浓度进行优化的单因素法,而未见正交设计法的使用[19, 24]。
笔者比较了正交试验设计和单因素分析法,建立了稳定的罗非鱼TRAP反应体系,以期为罗非鱼的遗传多样性分析、种质鉴定、分子辅助育种等提供新的研究手段。
1. 材料与方法
1.1 材料
试验所用4个罗非鱼群体均来自中国水产科学研究院淡水渔业研究中心宜兴养殖基地,分别是马来西亚红罗非鱼(O.niloticus)、奥尼罗非鱼(O.niloticus×O.aureus)、吉富罗非鱼(O.niloticus Gift strain)和新吉富罗非鱼(O.niloticus New Gift strain)。
1.2 DNA提取与检测
每个群体随机取20个个体,剪尾鳍于95%的酒精中固定并在-20 ℃冰箱保存。尾鳍在加入液氮的研钵中充分研磨后参照Takara的universal genomic DNA extraction kit Ver 3.0试剂盒中的动物组织提取基因组方法提取DNA。所得DNA用1%琼脂糖凝胶电泳检测质量并用分光光度计检测其浓度与纯度,稀释适当浓度后于-20 ℃保存。
1.3 TRAP-PCR扩增
固定引物序列参考HU和VICK[11]设计的4个固定引物,随机引物参考HU[25]的6个随机引物(表 1),引物由Invitrogen Biotechnology公司合成。DNA扩增程序参考HU和VICK[11]等,略改动为94 ℃ 2 min,94 ℃ 45 s,38 ℃ 45 s,72 ℃ 1 min,5个循环;94 ℃ 45 s,53 ℃ 45 s,72 ℃ 1 min,35个循环;72 ℃延伸8 min,最后于4 ℃保存。
表 1 试验采用的4个固定引物和6个随机引物序列Table 1. Sequences of 4 fixed primers and 6 arbitrary primers used in present study引物 primer 序列 sequence (5′→3′) 固定引物fixed primer E1 AGATTTCACCAAGGCTGT E2 TTATGAATTGCTGGCTTG E3 TGAATCCGTCTTCATTCA E4 ATCGATGCCTTTGATTTT 随机引物arbitrary primer Ga3-800 TCATCTCAAACCATCTACAC Odd26-700 CTATCTCTCGGGACCAAAC Sa12-700 TTCTAGGTAATCCAACAACA Trap03-700 CGTAGCGCGTCAATTATG Trap13-800 GCGCGATGATAAATTATC Em1 GGAACCAAACACATGAAGA 1.4 TRAP产物检测
PCR扩增结束后在300 V电压下用8%聚丙烯酰胺凝胶电泳3 h,用DNAGREEN绿如蓝低毒核酸染料(北京天恩泽基因科技有限公司出品)染色15 min,由美国UVP GelDoc-ItTM imaging system凝胶成像系统拍照保存。
1.5 试验设计
1.5.1 正交设计
以红罗非鱼DNA为模板筛选引物,选择一对较好的引物进行扩增,将影响罗非鱼TRAP反应体系的镁离子(Mg2+)浓度、dNTPs浓度、TaqDNA聚合酶浓度、模板DNA浓度和引物浓度5个因子设置4个水平,用L16(45)正交试验对罗非鱼TRAP反应体系进行分析、优化(表 2),3次重复。然后依据条带灵敏性、特异性及清晰度作1~16分打分[21],运用正交设计软件助手进行结果分析。
表 2 红罗非鱼TRAP反应体系L16(45)正交设计Table 2. L16(45) orthogonal design of red tilapia TRAP reaction system编号
No.DNA模板/ng
DNA template镁离子/mmol·L-1
Mg2+脱氧核苷酸/mmol·L-1
dNTPs随机引物/pmol·L-1
arbitrary primerTaqNDA聚合酶/U
Taq NDA polymerase1 30 1.0 0.20 0.5 0.3 2 30 1.5 0.25 2.0 0.5 3 30 2.5 0.30 7.0 0.8 4 30 3.0 0.35 10.0 1.1 5 60 1.0 0.25 7.0 1.1 6 60 1.5 0.20 10.0 0.8 7 60 2.5 0.35 0.5 0.5 8 60 3.0 0.30 2.0 0.3 9 90 1.0 0.30 10.0 0.5 10 90 1.5 0.35 7.0 0.3 11 90 2.5 0.20 2.0 1.1 12 90 3.0 0.25 0.5 0.8 13 120 1.0 0.35 2.0 0.8 14 120 1.5 0.30 0.5 1.1 15 120 2.5 0.25 10.0 0.3 16 120 3.0 0.20 7.0 0.5 1.5.2 单因素对比试验
参考HU和VICK[11]15 μL TRAP反应体系中的各参数,用DNA模板50 ng,Mg2+ 2.5 mmol·L-1、dNTPs 0.3 mmol·L-1、TaqDNA聚合酶1.5 U、固定引物10 pmol·L-1(随机引物浓度/固定引物浓度为1/1)和1×PCR buffer(Mg2+ Free)。针对某一因素设计不同浓度处理,而其他因素浓度不变。固定引物与buffer浓度不变,提高固定引物与随机引物的比例[26]。最后对正交设计和单因素法优化效果进行比较。
2. 结果与分析
2.1 正交设计
罗非鱼TRAP正交设计试验结果表明,Mg2+、dNTPs、TaqDNA聚合酶、模板DNA和引物等不同浓度混合进行PCR扩增,可产生显著差异的条带多态性以及条带清晰度(图 1)。其中9和13无扩增结果,泳道1杂带很多且难区分;3和7条带较亮且清晰,最佳处理组合为3。用正交设计软件助手对16个反应组合进行直观分析和方差分析,结果见图 2和表 3。
表 3 罗非鱼TRAP反应体系正交设计方差分析Table 3. Variance analysis of TRAP reaction system for tilapia optimized with orthogonal design变异来源
source极差
range方差
SSF 均方
MS标准差
X±SD模板 template 6.750 153.188 2.428 51.063 12.37 二价镁离子 Mg2+ 4.750 54.688 0.867 18.229 7.39 三磷脱氧核糖核酸 dNTPs 2.500 13.688 0.217 4.563 3.70 随机引物 arbitrary primer 4.750 50.688 0.803 16.896 7.12 TaqDNA聚合酶 TaqDNA 4.250 43.188 0.685 14.396 6.57 共计 total 315.440 均值大小代表各因素各水平间的差异,而极差大小代表该因素对试验的影响程度[22]。图 2中DNA不同浓度均值变化最大,影响最显著,其余依次为Mg2+、TaqDNA聚合酶、dNTPs和随机引物。表 3可看出模板影响最为显著。根据DNA浓度各水平均值, 显示条带并非浓度越高扩展扩增效果更好,60 ng时显著高于其他浓度水平,扩增条带较清晰丰富。dNTPs、随机引物和Mg2+浓度变化均值均出现了波峰,由此可见PCR反应各因子浓度均应控制在某一范围,扩增效果与各因子浓度不相关。综合各因素得到罗非鱼15 μL TRAP正交试验最优反应体系为DNA模板浓度为60 ng、Mg2+浓度为1.5 mmol·L-1、dNTPs浓度为0.3 mmol·L-1、TaqDNA聚合酶0.5 U和随机引物浓度为7 pmol·L-1。
2.2 单因素试验
2.2.1 dNTPs浓度、随机引物浓度、DNA浓度对罗非鱼TRAP扩增效果的影响
dNTPs浓度、随机引物浓度和DNA浓度对罗非鱼TRAP扩增效果的影响见图 3。结果显示,dNTPs浓度增加,特异性条带亮度增加;浓度较低时杂带较多,条带相对不够清晰。罗非鱼15 μL TRAP反应体系中,dNTPs浓度在0.3 mmol·L-1时条带亮度、清晰度以及特异性高于其他水平。当随机引物浓度增加时条带清晰且丰富;浓度较低时条带较少且不太清晰,但随机引物浓度为7 pmol·L-1时条带清晰度和丰富性最好。当DNA浓度较低时杂带较多且很模糊,罗非鱼15 μL TRAP反应体系中当DNA浓度达到60 ng以上,扩增效果没有明显差异。
![]() 图 3 dNTPs浓度、随机引物浓度和DNA浓度对罗非鱼TRAP的影响M. 100 bp DNA ladder;1~12. dNTPs浓度、随机引物浓度和DNA浓度各自1~4处理水平Figure 3. Effects of concentrations of dNTPs, arbitrary primers and template on TRAP reaction system for tilapiaM. 100 bp DNA ladder; 1~12. 1~4 levels of concentrations of dNTPs, random primers and template
图 3 dNTPs浓度、随机引物浓度和DNA浓度对罗非鱼TRAP的影响M. 100 bp DNA ladder;1~12. dNTPs浓度、随机引物浓度和DNA浓度各自1~4处理水平Figure 3. Effects of concentrations of dNTPs, arbitrary primers and template on TRAP reaction system for tilapiaM. 100 bp DNA ladder; 1~12. 1~4 levels of concentrations of dNTPs, random primers and template2.2.2 Mg2+浓度和TaqDNA聚合酶浓度对罗非鱼TRAP扩增效果的影响
Mg2+浓度较低时产生的条带较少且不清晰(图 4)。罗非鱼15 μL TRAP反应体系中,Mg2+浓度为2.5 mmol·L-1时其条带清晰度与亮度明显高于其他3个浓度水平。TaqDNA聚合酶在所选试验浓度范围内浓度较高时条带较清晰,但5和6条带质量差距不大,综合考虑成本和条带质量因素,罗非鱼15 μL TRAP反应体系TaqDNA聚合酶浓度在0.8 U时较好。运用单因素分析法得到罗非鱼15 μL TRAP最优反应体系为DNA模板浓度为60 ng、Mg2+ 浓度为2.5 mmol·L-1、dNTPs浓度为0.3 mmol·L-1、TaqDNA聚合酶浓度为0.8 U和随机引物浓度为7 pmol·L-1。
2.3 TRAP反应体系2种优化方法比较及验证
用正交设计和单因素方法分别优化罗非鱼TRAP反应体系的结果,对随机挑取的5个红罗非鱼个体PCR扩增对比,单因素优化罗非鱼TRAP-PCR产生的条带正交设计法优化结果几乎都能够产生,但正交设计法优化结果能扩增出较多单因素法优化结果无法扩增出的条带(图 5)。以4个罗非鱼群体材料对优化后体系进行验证,扩增条带清晰且特异性强,特异性条带较多,说明此反应体系适于罗非鱼TRAP-PCR分析(图 6)。
![]() 图 5 比较正交设计和单因素优化罗非鱼TRAP扩增图M. 100 bp DNA ladder;O. 正交设计;S. 单因素;1~5. 不同罗非鱼个体Figure 5. Comparison of amplification results of TRAP reaction system for tilapia between orthogonal design and single factor designM. 100 bp of DNA ladder; O. orthogonal design; S. single factor; 1~5. different tilapia individuals
图 5 比较正交设计和单因素优化罗非鱼TRAP扩增图M. 100 bp DNA ladder;O. 正交设计;S. 单因素;1~5. 不同罗非鱼个体Figure 5. Comparison of amplification results of TRAP reaction system for tilapia between orthogonal design and single factor designM. 100 bp of DNA ladder; O. orthogonal design; S. single factor; 1~5. different tilapia individuals![]() 图 6 4个不同罗非鱼群体的TRAP扩增图M. 100 bp DNA ladder;1~4. 马来西亚红罗非鱼;5~8. 新吉富罗非鱼;9~12. 吉富罗非鱼;13~16. 奥尼罗非鱼Figure 6. Amplification result of TRAP reaction system for 4 different tilapia populationsM. 100 bp DNA ladder; 1~4. red tilapia; 5~8. New GIFT strain Nile tilapia; 9~12. GIFT strain Nile tilapia; 13~16. O.niloticus×O.aureus
图 6 4个不同罗非鱼群体的TRAP扩增图M. 100 bp DNA ladder;1~4. 马来西亚红罗非鱼;5~8. 新吉富罗非鱼;9~12. 吉富罗非鱼;13~16. 奥尼罗非鱼Figure 6. Amplification result of TRAP reaction system for 4 different tilapia populationsM. 100 bp DNA ladder; 1~4. red tilapia; 5~8. New GIFT strain Nile tilapia; 9~12. GIFT strain Nile tilapia; 13~16. O.niloticus×O.aureus3. 讨论
在罗非鱼等水产动物中应用比较广的遗传标记主要是限制性片段长度多态性(restriction fragment length polymorphism,RFLP)、随机扩增多态性DNA(random amplified polymorphic RAPD)、扩增片段长度多态性(amplified fragment length polymorphism,AFLP)、单核苷酸的多态性(single nucleotide polymorphisms,SNP)、微卫星标记(simple sequence repeat,SSR)和表达序列标签(expressed sequence tag,EST)等[7, 27-28]。但随着大规模DNA测序技术的迅速发展,很多重要经济鱼类都获得了大量的DNA序列信息。分子标记技术也伴随着DNA测序技术发展逐步从传统的匿名性分子标记转向功能性及目标性分子标记[5]。TRAP标记与RAPD一样具有易于建立、操作、重复性强等特点,同时由于其特定引物中的固定引物可来自功能基因序列,所以具有能够偏向产生功能基因或者目标性区域条带的优势。TRAP能够把EST或cDNA序列与动物生长性状建立联系,能够快速筛选出生长相关的分子标记。TRAP标记既具有AFLP技术强大功能的优势,同时具有简单、多态性丰富、效率高、稳定性好的特点。其已经成功用于鲤、草鱼、鲶和鳜等水产动物的遗传多样分析以及分子标记辅助育种等[18-20, 29]。目前对于鲤、草鱼、棉花、水稻等[16, 19, 24, 26]TRAP反应体系的建立与优化,都是运用单因素分析法。单因素分析法是对每个反应影响因子进行逐项优化,不同因素、不同水平产生的反应结果对比鲜明,易于挑选单一因素最佳水平。但其不能够充分考虑单因素间的交互作用,不能保证各因素最佳浓度时组合为最佳反应组合。而运用统计学方法的正交设计法能够考察各因素间的交互作用,利用正交表安排试验方案,能够在最少的试验次数中获得最佳结果,一般各因素最好水平组合就是最优方案,但此方法具有一定局限性[22],如根据条带的清晰度和特异性对条带进行打分,具有一定的主观性。因此,该试验同时采用单因素分析法和正交设计法比较进行优化。
正交设计分析法和单因素分析法优化罗非鱼TRAP反应体系结果表明,2种分析方法结果为dNTPs浓度、随机引物浓度、DNA浓度相同,Mg2+浓度和TaqDNA聚合酶浓度不相同,两者在Mg2+浓度和TaqDNA聚合酶浓度对PCR扩增清晰度和多态性具有差异,在一定浓度范围内Mg2+浓度和TaqDNA聚合酶浓度与其他因子互作对PCR扩增具有显著性交互作用,可能是正交设计考虑了单因素间的交互作用。酶浓度超过一定范围对于PCR扩增效果没有显著作用,反而会引起非特异性扩增,因此,应比较分析2种方法中各因素使用浓度。该试验对正交设计与单因素法优化结果比较,正交设计法优化结果产生更多的多态性条带,因其能够综合考虑罗非鱼TRAP-PCR反应各因素之间的相互作用。该试验优化是建立在对其他鱼类TRAP反应各因子浓度有一定了解的基础上,如果对各因子浓度范围不清楚,可用正交设计试验探寻每个因素大概适宜浓度,然后再利用单因素分析法的快捷、对比鲜明等特点对PCR反应因子进行逐步优化[22]。为了获得稳定且重复性好的TRAP反应体系或是产生更加偏向靶位区域的条带,在草鱼、向日葵[24, 25]的TRAP反应体系都提高了起始退火温度到38 ℃和40 ℃,笔者试验亦采用38 ℃。罗非鱼TRAP反应体系中固定引物浓度与随机引物浓度之比与草鱼、鲶等[19, 23]均不相同而与鲤相似[19]。罗非鱼与草鱼、鲶染色体数量相似,但与鲤差距较大[30],可见不同鱼种的反应体系的差异与染色体的数量没有直接关系,可能与基因组核苷酸序列或空间结构相关,所以不同种鱼间的TRAP反应体系各有不同,不能简单套用。此外,该试验采用DNAGREEN对扩增产物染色,其具有EB相当的稳定性和灵敏度,但毒性较低。
综合单因素与正交设计试验能够获得更科学、完善的试验结果[23]。比较2种分析方法,正交设计方法比单因素法更简单、科学、合理。笔者试验建立反应稳定、重复性好的罗非鱼15μL TRAP最优反应体系是DNA模板浓度为60 ng、Mg2+浓度为1.5 mmol·L-1、dNTPs浓度为0.3 mmol·L-1、TaqDNA聚合酶0.5 U、随机引物浓度为7 pmol·L-1和固定引物浓度为10 pmol·L-1。该TRAP反应体系均能在马来西亚红罗非鱼、新吉富罗非鱼、吉富罗非鱼、奥尼罗非鱼群体间和群体中扩增出多态性较好的条带,大小主要在100~900 bp。该反应体系的建立为罗非鱼遗传多样性评价、种质鉴定、分子标记辅助育种等研究提供了新的手段。
-
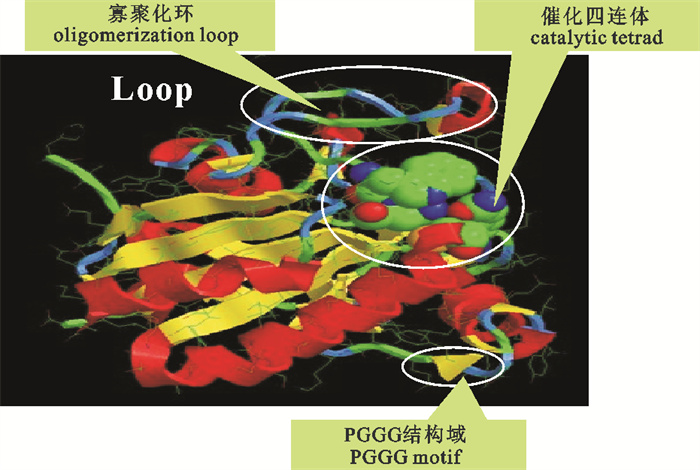
图 4 罗非鱼GPx1与其他脊椎动物GPx1氨基酸序列比对
*表示相同氨基酸,催化四联体用粗斜体字母标出,PGGG结构域和寡聚化环用灰色方、框标出,有助予稳定酶结构的3个环用粗下划线表示,传递底物至催化中心的重要氨基酸用黑体加粗字母标出
Figure 4. Comparison of Gpxl amino acid sequence between 0.niloticus and other vertebrate animals
* indicates the same amino acid; catalytic tetrad are shown in bold italic letters; PGGG motif and oligomerizxagion loop are boxed in grey three loops that stabiloize enzyme structure are undertlined; amino acid delivering the substrates towrd catalytic centers are shown in bold letters.
表 1 GPx1基因扩增引物序列表
Table 1 Primers used in GPx1 cloning
引物名称primer 序列(5′~3′) sequence(5′~3′) GPXS1 CAGTTYGGCCAYCARGA GPXR1 AGRAACTTYTCRAARTTCCA LGPXS2 AGAAGGTGGATGTGAATGGA LGPXS3 TGATGCCCTGGGTCTCAT LGPXR2 TATCTGCCTCGATGTCACTTGTC LGPXR3 AGTTCCAGGATACGTCATTC FAP GGCCACGCGTCGACTAGTAC17 RAP GGCCACGCGTCGACTAGTACT17 AP GGCCACGCGTCGACTAGTAC 注:Y. C/T;R. A/G 表 2 罗非鱼与其他脊椎动物GPx1基因核苷酸、氨基酸序列相似性比较
Table 2 Comparison of nucleotide and amino acid similarities between O.niloticus GPx1 and other vertebrate animals GPx1
% 种类
species核苷酸相似性
nucleotide similarity氨基酸相似性
amino acid similarity草鱼Ctenopharyngodon idella 58.2 80.6 斑马鱼Danio rerio 56.6 78.5 日本鳗鲡Anguilla japonica 51.5 78.5 金枪鱼Thunnus maccoyii 54.6 73.1 人Homo sapiens 45.2 63.8 马Equus caballus 43.9 64.3 鼠Mus musculus 44.1 63.2 蟾Xenopus (Silurana) tropicalis 44.0 58.4 斑胸草雀Taeniopygia guttata 43.2 58.1 -
[1] PASSARDI F, THEILER G, ZAMOCKY M, et al. PeroxiBase: the peroxidase database[J]. Phytochem, 2007, 68(12): 1605-1611. doi: 10.1016/j.phytochem.2007.04.005
[2] 王咏梅. 自由基与谷胱甘肽过氧化物酶[J]. 解放军药学学报, 2005, 21(5): 369-371. doi: 10.3969/j.issn.1008-9926.2005.05.017 [3] TOSATTO S C, BOSELLO V, FOGOLARI F, et al. The catalytic site of glutathione peroxidases[J]. Antioxid Redox Signal, 2008, 10(9): 1515-1525. doi: 10.1089/ars.2008.2055
[4] TOPPO S, FLOHÉ L, URSINI F, et al. Catalytic mechanisms and specificities of glutathione peroxidases: variations of a basic scheme[J]. Biochimica et Biophysica Acta (BBA) -General Subjects, 2009, 1790(11): 1486-1500. doi: 10.1016/j.bbagen.2009.04.007
[5] TOPPO S, VANIN S, BOSELLO V, et al. Evolutionary and structural insights into the multifaceted glutathione peroxidase (Gpx) superfamily[J]. Antioxid Redox Signal, 2008, 10(9): 1501-1513. doi: 10.1089/ars.2008.2057
[6] CONRAD M, SCHNEIDER M, SEILER A, et al. Physiological role of phospholipid hydroperoxide glutathione peroxidase in mammals[J]. Biol Chem, 2007, 388(10): 1019-1025. doi: 10.1515/BC.2007.130
[7] MARGIS R, DUNAND C, TEIXEIRA F K, et al. Glutathione peroxidase family: an evolutionary overview[J]. FEBS J, 2008, 275(15): 3959-3970. doi: 10.1111/j.1742-4658.2008.06542.x
[8] LIU C H, TSENG M C, CHENG W. Identification and cloning of the antioxidant enzyme, glutathione peroxidase, of white shrimp, Litopenaeus vannamei, and its expression following Vibrio alginolyticus infection[J]. Fish Shellfish Immuol, 2007, 23(1): 34-45. doi: 10.1016/j.fsi.2006.09.002
[9] LI G Z, LIANG X F, YAO W, et al. Molecular characterization of glutathione peroxidase gene from the liver of silver carp, bighead carp and grass carp[J]. BMB Rep, 2008, 41(3): 204-209. doi: 10.5483/BMBRep.2008.41.3.204
[10] MISRA S, NIYOGI S. Selenite causes cytotoxicity in rainbow trout (Oncorhynchus mykiss) hepatocytes by inducing oxidative stress[J]. Toxicol In Vitro, 2009, 23(7): 1249-1258. doi: 10.1016/j.tiv.2009.07.031
[11] SITJ-BOBADILLA A, CALDUCH-GINER J, SAERA-VILA A, et al. Chronic exposure to the parasite Enteromyxum leei (Myxozoa: Myxosporea) modulates the immune response and the expression of growth, redox and immune relevant genes in gilthead sea bream, Sparus aurata L[J]. Fish Shellfish Immunol, 2008, 24(5): 610-619. doi: 10.1016/j.fsi.2008.01.014
[12] CHOI C Y, AN K W, NELSON E R, et al. Cadmium affects the expression of metallothionein (MT) and glutathione peroxidase (GPX) mRNA in goldfish, Carassius auratus[J]. Comp Biochem Physiol Part C: Toxicol Pharmacol, 2007, 145(4): 595-600. doi: 10.1016/j.cbpc.2007.02.007
[13] WOO S, YUM S, KIM D W, et al. Transcripts level responses in a marine medaka (Oryzias javanicus) exposed to organophosphorus pesticide[J]. Comp Biochem Physiol C: Toxicol Pharmacol, 2009, 149(3): 427-432. doi: 10.1016/j.cbpc.2008.10.100
[14] KOROTKOV K V, NOVOSELOV S V, HATFIELD D L, et al. Mammalian selenoprotein in which selenocysteine (Sec) incorporation is supported by a new form of Sec insertion sequence element[J]. Mol Cell Biol, 2002, 22(5): 1402-1411. doi: 10.1128/MCB.22.5.1402-1411.2002
[15] 何珊, 梁旭方, 廖婉琴, 等. 鲢鱼、鳙鱼、草鱼谷胱甘肽过氧化物酶cDNA的克隆及肝组织表达[J]. 动物学杂志, 2007, 42(3): 40-47. doi: 10.3969/j.issn.0250-3263.2007.03.007 [16] FUKUHARA R, KAGEYAMA T. Structure, gene expression, and evolution of primate glutathione peroxidases[J]. Comp Biochem Physiol B: Biochem Mol Biol, 2005, 141(4): 428-436. doi: 10.1016/j.cbpc.2005.05.002
[17] EPP O, LADENSTEIN R, WENDEL A. The refined structure of the selenoenzyme glutathione peroxidase at 0.2-nm resolution[J]. Eur J Biochem, 1983, 133(1): 51-69. doi: 10.1111/j.1432-1033.1983.tb07429.x
[18] ARTHUR J R. The glutathione peroxidases[J]. Cell Mol Life Sci, 2000, 57(13/14): 1825-1835. doi: 10.1007/pl00000664
[19] GROSSMANN A, WENDEL A. Non-reactivity of the selenoenzyme glutathione peroxidase with enzymatically hydroperoxidized phospholipids[J]. Eur J Biochem, 1983, 135(3): 549-552. doi: 10.1111/j.1432-1033.1983.tb07687.x
[20] SEVANIAN A, MUAKKASSAH-KELLY S F, MONTESTRUQUE S. The influence of phospholipase A2 and glutathione peroxidase on the elimination of membrane lipid peroxides[J]. Arch Biochem Biophys, 1983, 223(2): 441-452. doi: 10.1016/0003-9861(83)90608-2
[21] HERBETTE S, ROECKEL-DREVET P, DREVET JR. Seleno-independent glutathione peroxidases. More than simple antioxidant scavengers[J]. FEBS J, 2007, 274(9): 2163-2180. doi: 10.1111/j.1742-4658.2007.05774.x
-
期刊类型引用(3)
1. 何福玲, 凌正宝, 肖俊, 郭忠宝, 钟欢, 杨弘, 陈文治, 唐瞻杨, 单丹, 梁军能, 甘西, 罗永巨. 两个人工选择奥利亚罗非鱼群体系统发育及其遗传多样性分析. 南方农业学报. 2017(02): 341-349 .  百度学术
百度学术
2. 颉晓勇, 钟金香, 赵金良. 遗传标记在罗非鱼良种选育研究中的应用与展望. 湖南农业科学. 2017(02): 123-126 .  百度学术
百度学术
3. 颉晓勇, 钟金香, 李思发. 3种分子标记分析罗非鱼选育群体的多态性效率比较. 广东农业科学. 2014(24): 114-117 .  百度学术
百度学术
其他类型引用(2)



 下载:
下载:






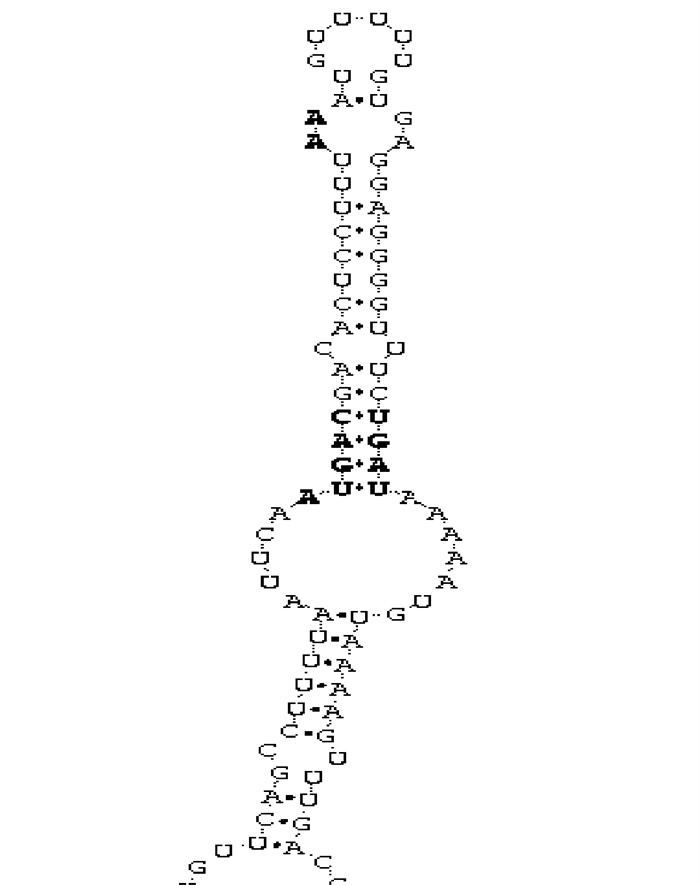

 粤公网安备 44010502001741号
粤公网安备 44010502001741号
