Cloning and sequence analysis of Cyt b gene in four Sparidae fishes in Zhoushan sea area
-
摘要:
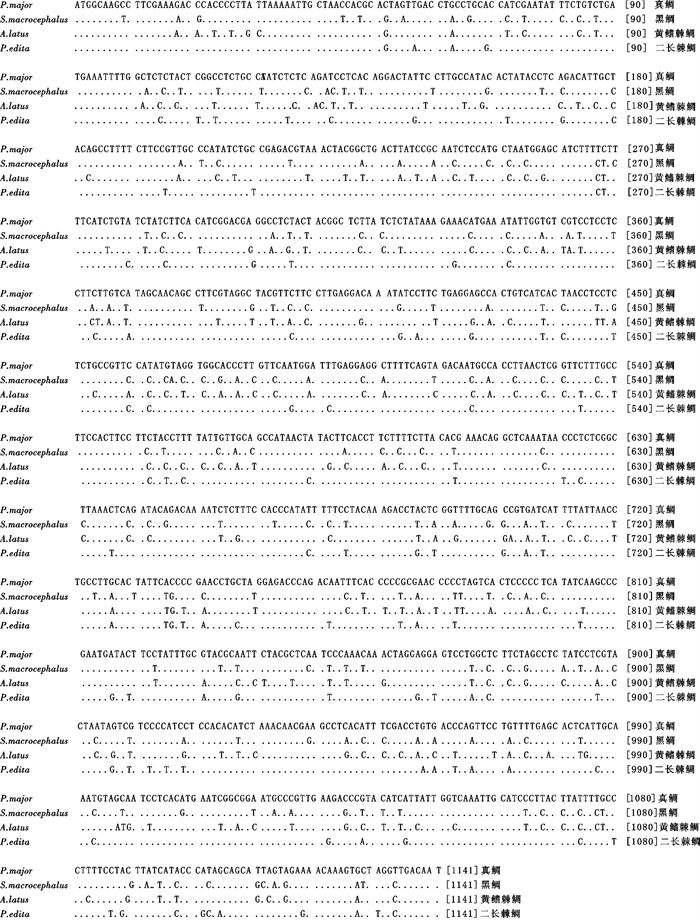
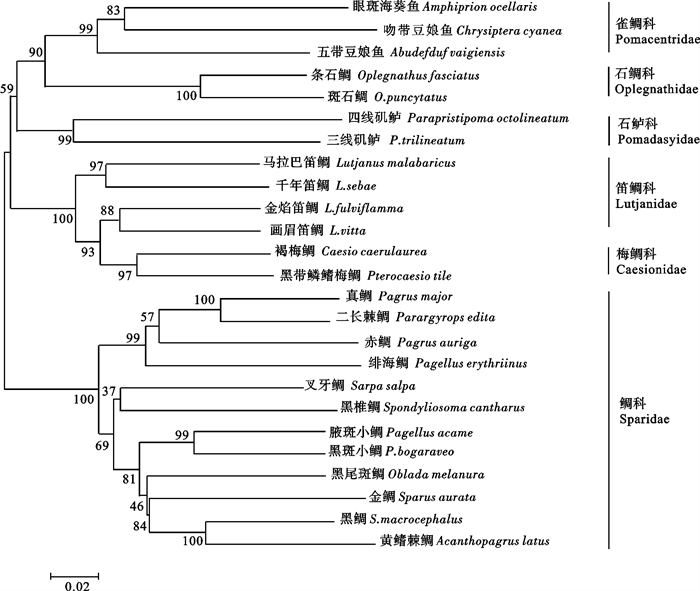
克隆测序了分布于舟山海域的真鲷(Pagrus pagrus)、黑鲷(Sparus macrocephalus)、黄鳍棘鲷(Acanthopagrus latus)和二长棘鲷(Parargyrops edita)4种鲷科鱼类的线粒体Cyt b基因1 141 bp的全序列。通过对4种鲷科鱼类Cyt b基因序列的比对分析,发现了292个核苷酸变异位点,其中存在186个信息位点,序列中的转换大于颠换,碱基替换多发生于密码子第3位。遗传距离分析表明,4种鲷科鱼类的遗传差异在0.096 7~0.227 5之间,其中真鲷与二长棘鲷之间遗传距离最小,为0.096 7,黄鳍棘鲷与二长棘鲷的遗传距离最大,为0.227 5;序列变异的转换/颠换比值在2.293 0~7.358 7之间。以25种鲈总科鱼类构建的系统树表明鲷科鱼类与其他科的鱼类存在较远的遗传亲缘关系。
Abstract:The complete sequences of mitochondrial Cyt b gene in Pagrus pagrus, Sparus macrocephalus, Acanthopagrus latus and Parargyrops edita in Zhoushan sea area were amplified, cloned and sequenced. The 1 141 bp Cyt b gene sequences of these four species were obtained. Compared with the same DNA fragment recorded in the GeneBank, the sequences of 1 141 bp DNA had 292 nucleotide variation sites, among which 186 parsimony informative sites were observed.Transitions were discovered to be more than transversions, and most of the base substitutions occurred at the third locus of the codon. The genetic distance among 4 Sparidae species ranged from 0.096 7 to 0.227 5. The shortest distance occurred between P.pagrus and P.edita, being 0.096 7, while the longest one between S.macrocephalus and A.latus, being 0.227 5. Moreover, the transition-transversion ratio of the sequence differentiation of 4 species ranged from 2.293 0 to 7.358 7. The NJ phylogenetic tree revealed that 25 species were divided into two clades, and obvious genetic divergence existed between fishes of Sparidae and the other fishes of Percoidea.
-
Keywords:
- Spridae /
- Cyt b /
- sequence analysis /
- genetic evolution
-
光合细菌是一大类能利用光能作为能源进行不放氧光合作用的原核生物的总称。光合细菌由于其显著的净化水质作用[1],其中的某些菌株能够拮抗病原菌[2]、提高水产动物免疫力[3]和成活率[4],且其丰富的菌体蛋白也可以为水产动物提供营养[5-7],在水产动物的育苗[8]及生产[9]中发挥了重要的作用。筴膜红假单胞菌(Rhodopseudomonas capsulate)PS2菌株可适应的温度、盐度、pH和光照度范围分别为(20~40) ℃、0~40.0、6.0~9.0、(500~5 000)lx;该菌株具有良好的水质净化能力,生长范围广泛,是一种潜在的益生菌[1]。赵卫红等[10]研究了筴膜红假单胞菌对异育银鲫(Allogynogenetie cruciaan)鱼种非特异性免疫机能的影响。笔者将此株光合细菌添加到饲料中研究了其对尖吻鲈(Lates calcarifer)的生长、消化酶及血清非特异性免疫酶的影响,以为光合细菌在鱼类饲料中的应用提供参考。
1. 材料与方法
1.1 试验菌株
试验所用的光合细菌由中国水产科学研究院南海水产研究所分离保存,并培养成液体制剂,该光合细菌被鉴定为筴膜红假单胞菌,其菌含量为8×108 cfu·mL-1。
1.2 试验饲料
投喂前将光合细菌液体制剂分别按照0.0%、0.5%、1.0%和1.5%的比例均匀喷洒在基础饲料表面,稍晾干后,在0.5 h内投喂完毕。基础饲料是恒兴牌海水鱼膨化饲料。
1.3 试验用鱼及饲料管理
试验在中国水产科学研究院南海水产研究所热带水产研究开发中心(海南三亚)进行。试验尖吻鲈购于当地养殖场,购回后在室内进行为期2周的驯养。挑选健康均匀的个体随机分为4个组,初始体质量为(10.95±0.25)g,每组3个平行,每个平行20尾鱼,在容量为0.5 m3的圆形玻璃纤维桶内养殖50 d。采用流水养殖,海水流速为2~3 L·min-1。试验用水是沙滤海水。每天饱食投喂2次,分别于8: 00和16: 00进行。溶解氧、氨氮、盐度和温度分别为(6.84±0.90)mg·L-1,(0.03±0.02)mg·L-1,29~31和26~31 ℃。
1.4 取样及计算指标
试验结束前经24 h禁食,称总质量,并统计每组鱼的数量。每桶随机取3尾鱼作为消化酶的样品。冰盘解剖活鱼,分别取出肝脏、胃、幽门垂和肠道,剔除脏器的脂肪和结缔组织并称重。样品放置在-20 ℃保存备用。每桶取5尾鱼断尾取血,用以测定血清免疫酶活性。增重率(weight gain,WG)、特定生长率(specific growth rate,SGR)、饲料系数(feed conversion ratio,FCR)和成活率(survival rate,SR)的计算公式如下:
增重率(%)=100×[终末均质量(g)-初始均质量(g)]/初始均质量(g)
特定生长率(%)=100×[ln终末均质量(g)-ln初始均质量(g)]/试验天数(d)
成活率(%) =100×试验结束鱼数/试验开始鱼数
饲料系数=投喂饲料干质量(g)/鱼增质量(g)
1.5 消化酶的测定
1.5.1 样品制备
样品制备参照WANG和XU[11]。将各个消化器官的组织在预冷的磷酸缓冲液(0.02 mol·L-1,pH 7.5)[1 g·(5 mL)-1]中用玻璃匀浆器冰浴匀浆,4 ℃、10 000 r·min-1冷冻离心30 min,上清液作为消化酶分析样品,4 ℃保存,24 h内分析完毕。酶液中可溶性蛋白浓度用BRADFORD[12]的方法测定,用牛血清蛋白做标准曲线。酶的活力单位定义为在37 ℃,相应的pH的条件下,每分钟催化底物释放1 μg的产物所需要的酶量。酶比活力定义为单位每毫克蛋白的酶活力(U·mg-1)。其中胃组织的测定pH 3.0,其他组织酶的活性均在pH 7.5时测定。
1.5.2 消化酶活力的测定
蛋白酶的测定参照ANSON[13]的方法,用酪氨酸做标准曲线,以福林酚试剂作为显色剂,分别用1.5%的酪蛋白(Sigma)和1.5%的牛血红蛋白(Sigma)作为碱性蛋白酶和酸性蛋白酶的底物,在680 nm测定吸光度。淀粉酶的测定参照BERNFELD[14]的方法,以DNS试剂为显色剂,用麦芽糖做标准曲线,以1%的可溶性淀粉做底物,在520 nm测定吸光度。
1.6 非特异性免疫酶活力的测定
试验结束时,血液样品在4 ℃过夜,5 000 r·min-1冷冻离心10 min取上清液分别测定碱性磷酸酶(AKP)、过氧化物酶(POD)和超氧化物歧化酶(SOD)。均使用南京建成试剂盒测定。
1.7 数据的统计分析
采用Excel和SPSS 13.0软件对数据进行统计分析,所有数值用平均数±标准误差表示,先对数据作单因素方差分(ANOVA),处理若有显著差异,再作Duncan′s多重比较,P < 0.05表示差异显著。
2. 结果与分析
2.1 对尖吻鲈生长的影响
在养殖过程中,部分鱼出现寄生虫病,影响了成活率和增重率,导致标准偏差较很大。尖吻鲈的增重率、特定生长率、成活率和饲料系数在组间均没有显著性差异(P>0.05),试验组鱼的增重率和特定生长率比对照组略微降低(表 1)。
表 1 饲料中不同添加量的光合细菌对尖吻鲈生长的影响Table 1. Growth performance of sea bass fed with different doses of photosynthetic bacterium in feed指标 index 饲料中光合细菌添加量 dietary R.capsulate levels 0.0% 0.5% 1.0% 1.5% 增重率 weight gain(WG) 427.05±26.02 391.75±116.52 404.28±115.25 337.07±41.05 特定生长率 specific growth rate(SGR) 3.32±0.10 3.15±0.46 3.20±0.46 2.94±0.19 成活率 survival rate(SR) 80.00±13.23 81.67±7.64 66.67±15.28 68.33±34.03 饲料系数 feed conversion ratio(FCR) 1.31±0.14 1.33±0.21 1.26±0.06 1.36±0.11 注:数据以3个重复的平均值±标准差表示,上标字母不同者为存在显著差异(P < 0.05)。后表同此
Note:Values are showed by Means ± SD of 3 replicates. Values within the same row with different superscript letters are significantly different(P < 0.05).The same as below.2.2 对尖吻鲈消化酶活性的影响
尖吻鲈的肠和肝蛋白酶均在1.0%组达到最大值,其中1.0%组鱼的肝蛋白酶显著高于对照组,但1.0%组肠蛋白酶与对照组没有显著差异。幽门垂蛋白酶在1.5%组达到最大值, 并显著高于其他各组(P < 0.05)。胃蛋白酶活性在组间没有显著性差异(P>0.05)(表 2)。
表 2 饲料中不同添加量的光合细菌对尖吻鲈各个器官蛋白酶活性的影响Table 2. Protease of organs of sea bass fed with different doses of photosynthetic bacterium in feedw(蛋白酶)/U·mg-1蛋白
protease饲料中光合细菌添加量 dietary R.capsulate levels 0.0% 0.5% 1.0% 1.5% 肠 intestinal 9.567±5.098ab 9.478±4.370ab 11.959±2.380b 5.516±1.214a 幽门垂 pyloric caeca 4.798±1.616a 5.568±1.355a 4.992±1.639a 14.199±2.377b 肝 liver 0.408±0.046ab 0.359±0.040a 0.595±0.125c 0.509±0.045bc 胃 stomach 3.656±0.731 3.521±0.902 4.275±0.770 3.777±0.587 尖吻鲈的肠及胃淀粉酶均在1.0%组显著高于对照组(P < 0.05);幽门垂淀粉酶在1.5%组显著高于其他各组(P < 0.05);肝淀粉酶在组间没有显著性差异(P>0.05)(表 3)。
表 3 饲料中不同添加量的光合细菌对尖吻鲈各个器官淀粉酶活性的影响Table 3. Amylase of organs of sea bass fed with different doses of photosynthetic bacterium in feedw(淀粉酶)/U·mg-1蛋白
amylase饲料中光合细菌添加量
dietary R.capsulate levels0.0% 0.5% 1.0% 1.5% 肠 intestinal 26.728±3.693a 33.433±6.292ab 38.633±13.095b 26.793±5.679a 幽门垂 pyloric caeca 43.497±11.147a 58.597±24.210a 51.357±6.590a 94.501±18.328b 肝 liver 137.029±31.855 239.006±79.602 230.824±60.875 231.406±22.863 胃 stomach 11.834±3.767a 13.002±1.991ab 15.628±3.468b 13.601±1.740ab 2.3 对尖吻鲈血清非特异性免疫酶活性的影响
尖吻鲈血清AKP、POD及SOD在组间均没有显著性差异(P>0.05)。但试验组的AKP均高于对照组,随着饲料中光合细菌添加量的增加,POD先减小后增大,SOD先增大后减小(表 4)。
表 4 饲料中不同添加量的光合细菌对尖吻鲈血清非特异性免疫酶活性的影响Table 4. Serum nonspecific immune enzymes of sea bass fed with different doses of photosynthetic bacterium in feedw(免疫酶)/U·mg-1蛋白
immune enzymes饲料中光合细菌添加量 dietary R.capsulate levels 0.0% 0.5% 1.0% 1.5% 碱性磷酸酶(AKP) 2.287±0.654 2.677±0.846 2.750±0.567 2.604±0.548 过氧化物酶(POD) 34.741±10.266 32.815±4.491 28.815±12.581 38.000±2.189 超氧化物歧化酶(SOD) 147.956±18.588 159.358±9.622 151.849±13.350 147.121±17.105 3. 讨论
在体外试验中,沼泽红假单胞菌(Rhodopseudomonas palustris)和混球红细菌(Rhodobacter sphaeroides)对人工模仿的胃酸及肠液具有一定的抵抗力,且对罗非鱼的小肠上皮细胞无害[15]。说明光合细菌的某些种类具有在鱼类消化道中生存的能力。在该试验中,饲料中添加光合细菌对尖吻鲈的生长及饲料系数均没有显著性影响。对草鱼(Ctenopharyngodon idellus)[16]、西伯利亚鲟鱼(Acipenser baeri)[17]、罗非鱼(Tilapia sp.)[18]和月鳢(Channa asiatica)[19]等的研究则表明,光合细菌以适宜的比例添加到饲料中可以显著地促进鱼类的生长。这可能与光合细菌的种类、鱼的食性以及养殖环境有关。不同种类的光合细菌在对酸及胆盐的耐受能力、对pH及盐度的适宜生长范围、对鱼类消化道的粘附能力等特性的影响会有很大差别。鱼类肠道菌群多样性与鱼类食性相关[20],尖吻鲈属于肉食性海水鱼类,可能与上述草食性、杂食性或肉食性淡水鱼类的微生态区系的菌群组成存在差别,因而也会影响光合细菌的作用。
此试验中,光合细菌在1.0%组促进肠及胃蛋白酶和淀粉酶活性,其中肠及胃的淀粉酶活性显著高于对照组;在1.5%组幽门垂的蛋白酶及淀粉酶都显著升高(P < 0.05),而此时肠及胃的消化酶却受到抑制。说明不同光合细菌添加量对各个消化器官的消化酶活性的影响不同。该研究的结果与陈鹏飞等[17]的研究结果有所不同,其研究显示,随着饲料中光合细菌添加量的增加,西伯利亚鲟鱼的胃、肠、肝及盲囊的消化酶变化趋势几乎一致。另外,该研究还显示消化酶的升高并没有促进尖吻鲈的生长,甚至有略微降低的趋势,这可能是由于该株光合细菌对尖吻鲈的生长没有促进作用,也可能养殖期间寄生虫病的发生对试验结果产生了影响。
张梁等[16]将菌浓度为1×108 cfu·mL-1的球形红假单胞菌(Rhodopseudomonas sphaeroides)分别按照0.0%、0.5%、1.5%、2.5%和3.5%的比例添加到草鱼饲料中,发现其血清及肝脏的SOD、ACP和AKP在2.5%组达到最大值,并显著高于对照组,而在3.5%组则出现降低的趋势。鲤鱼(Cyprinus carpio)血清溶菌酶及白细胞吞噬活性随着养殖水体中沼泽红假单胞菌(R.palustris)的添加量增加及添加时间的延长而升高[3]。赵卫红等[10]报道添加到水体中(5×108、5×109、5×1010 cfu·m-3)的筴膜红假单胞菌可提高异育银鲫白细胞的免疫性能,且其作用效果均随着添加量的增加而增加,但并未增加其血清溶菌酶的活力。而在此研究中光合细菌对尖吻鲈的血清AKP、POD及SOD均没有显著性差异。
尽管文献中报道光合细菌可以抑制病原菌的生长[2, 21],提高水产动物的成活率[22],显著地促进鱼类的生长[16-19],但该研究结果中多项指标却无显著性差异。这可能与光合细菌的种类、菌株特性具有专一性以及添加量有关,此外,养殖期间寄生虫病的发生对试验的结果也有一定的影响。对于尖吻鲈饲料中此株光合细菌的添加量和肠道微生态区系的菌群组成,以及两者之间的关系均有待于进一步研究。
-
表 1 25种鲈总科鱼类的Cyt b数据信息
Table 1 Accession No.of Cyt b of 25 Percoidea species
科
family物种
speciesCyt b基因GenBank登录号
accession No.鲷科 Sparidae 真鲷 Pagrus major GU131149;NC003196 黑鲷 Sparus macrocephalus GU131150;AB458395 黄鳍棘鲷 Acanthopagrus latus GU131151;AB458394 二长棘鲷 Parargyrops edita GU131152;EF107158 黑斑小鲷 Pagellus bogaraveo EF439560 赤鲷 Pagrus auriga NC005146 绯海鲷 Pagellus erythrinus EF439562 叉牙鲷 Sarpa salpa EF439572 黑椎鲷 Spondyliosoma cantharus EF439602 腋斑小鲷 Pagellus acarne EF439558 黑尾斑鲷 Oblada melanura EF439556 金鲷 Sparus aurata EF439595 梅鲷科 Caesionidae 黑带鳞鳍梅鲷 Pterocaesio tile AP000447 褐梅鲷 Caesio caerulaurea AF381273 雀鲷科 Pomacentridae 五带豆娘鱼 Abudefduf vaigiensis NC009064 眼斑海葵鱼 Amphiprion ocellaris NC009065 吻带豆娘鱼 Chrysiptera cyanea AB018992 石鲷科 Oplegnathidae 斑石鲷 Oplegnathus puncytatus NC013143 条石鲷 O.fasciatus AP006010 石鲈科 Pomadasyidae 四线矶鲈 Parapristipoma octolineatum DQ197977 三线矶鲈 P.trilineatum NC009857 笛鲷科 Lutjanidae 画眉笛鲷 Lutjanus vitta EF376182 金焰笛鲷 L.fulviflamma EF376177 千年笛鲷 L.sebae EF376168 马拉巴笛鲷 L.malabaricus EF376171 表 2 4种鲷科鱼类细胞色素b基因的碱基分布
Table 2 Nucleotide compositions of Cyt b gene of four Sparidae species
物种 species T C A G A+T G+C C-3 G-3 总长/bp total length 真鲷 P.major 30.0 30.1 24.9 15.1 54.9 45.1 37.6 5.3 1 141 黑鲷 S.macrocephalus 29.6 29.8 25.7 14.9 55.3 44.7 37.6 4.7 1 141 黄鳍棘鲷 A.latus 30.3 29.4 24.8 15.5 55.1 44.9 36.8 6.1 1 141 二长棘鲷 P.edita 30.9 29.2 24.2 15.7 55.1 44.9 35.5 6.3 1 141 平均值 average 30.2 29.6 24.9 15.3 55.1 44.9 36.9 5.6 1 141 注:表中数字3表示密码子的第3位
Note:The number 3 indicates the 3rd codon position.表 3 鲷科鱼类的相对遗传距离(对角线以下是转换加颠换,对角线以上是转换比颠换)
Table 3 Pair-wise genetic distance among Sparidae species (below the diagonal is transition+transversion; above the diagonal is transition/transversion)
物种
species真鲷 黑鲷 黄鳍棘鲷 二长棘鲷 黑尾斑鲷 腋斑小鲷 黑斑小鲷 绯海鲷 赤鲷 叉牙鲷 金鲷 黑椎鲷 真鲷 P.major 2.2930 2.5493 7.3587 2.3066 2.7524 2.4505 5.4167 5.4650 2.5751 2.4608 2.1017 黑鲷 S.macrocephalus 0.1999 5.1064 2.3704 4.1398 4.1897 3.8931 2.2294 2.1849 2.7756 3.0084 2.1049 黄鳍棘鲷 A.latus 0.2261 0.1261 2.6289 3.9624 4.0734 4.0647 2.0797 2.1247 3.2934 2.8186 2.1539 二长棘鲷 P.edita 0.0967 0.2013 0.2275 2.4808 2.5751 2.1049 4.9218 4.8678 2.4852 2.2636 2.0797 黑尾斑鲷 O.melanura 0.1812 0.1489 0.1763 0.1873 5.4304 4.7578 2.9703 2.3911 3.3673 4.6025 2.7345 腋斑小鲷 P.acarne 0.2167 0.1649 0.1659 0.2029 0.1564 7.2577 2.5642 2.7963 3.3551 4.6641 2.3129 黑斑小鲷 P.bogaraveo 0.2026 0.1600 0.1800 0.1854 0.1561 0.1104 2.7976 2.2580 2.7954 2.4658 2.4309 绯海鲷 P.erythrinus 0.1739 0.2154 0.2272 0.1550 0.2097 0.2199 0.2155 7.3169 2.3244 2.3791 2.2945 赤鲷 P.auriga 0.1692 0.2092 0.2274 0.1482 0.1825 0.2229 0.2010 0.1870 2.2468 2.7545 2.1911 叉牙鲷 S.salpa 0.2029 0.1738 0.2018 0.1944 0.1676 0.1630 0.1601 0.1952 0.1939 3.2594 3.6007 金鲷 S.aurata 0.2135 0.1729 0.1832 0.2046 0.1675 0.1641 0.1839 0.2085 0.2354 0.1838 2.7311 黑椎鲷 S.cantharus 0.2007 0.1854 0.2104 0.1900 0.1936 0.1685 0.1779 0.2131 0.2033 0.1678 0.2044 -
[1] 赵盛龙, 钟俊生. 舟山海域鱼类名录新考[J]. 浙江海洋学报: 自然科学版, 2005, 24(4): 364-379. doi: 10.3969/j.issn.1008-830X.2005.04.012 [2] MCVEIGH H P, DAVIDSON W S. A salmonid phylogeny inferred from mitochondrial cytochrome b gene sequence[J]. Fish Biol, 1991, 39(Sup A): 277-282. doi: 10.1111/j.1095-8649.1991.tb05090.x
[3] CANTATORE P, ROBERTI M, PESOLE G, et al. Evolutionary analysis of cytochrome b sequence in some Perciformes: evidence for a slower rate of evolution than in mammals[J]. Mol Evol, 1994, 39(6): 589-597. doi: 10.1007/BF00160404
[4] 李鹏飞, 周永东, 徐汉祥. 大黄鱼、鱼及美国红鱼线粒体DNA的Cyt b基因序列比较[J]. 南方水产, 2008, 4(3): 43-47. doi: 10.3969/j.issn.2095-0780.2008.03.008 [5] 朱世华, 郑文娟, 邹记兴, 等. 5种石鲈科鱼类细胞色素b基因序列及分子系统分析[J]. 热带海洋学报, 2006, 25(4): 42-45. doi: 10.3969/j.issn.1009-5470.2006.04.007 [6] 邵爱华, 朱江, 陈葵, 等. 暗纹东方鲀细胞色素b及其侧翼tRNA基因的克隆与序列分析[J]. 中国水产科学, 2005, 12(6): 675-681. doi: 10.3321/j.issn:1005-8737.2005.06.002 [7] 周发林, 江世贵, 苏天凤, 等. 6种笛鲷属鱼类Cyt b基因片段序列的比较[J]. 热带海洋学报, 2004, 23(4): 87-92. doi: 10.3969/j.issn.1009-5470.2004.04.012 [8] 高天翔, 张秀梅, 朱杰, 等. 养殖褐牙鲆细胞色素b基因序列的初步研究[J]. 中国水产科学, 2003, 10(2): 93-96. doi: 10.3321/j.issn:1005-8737.2003.02.002 [9] SWINDELL S R, PLASTERER T N, SEQMAN. Contig assembly[J]. Methods Mol Biol, 1997, 70: 75-89.
[10] TATUSOVA T A, MADDEN T L. BLAST 2 Sequences, a new tool for comparing protein and nucleotide sequences[J]. FEMS Microbiol Lett, 1999, 174: 247-250. doi: 10.1111/j.1574-6968.1999.tb13575.x
[11] TAMURA K, DUDLEY J, NEI M, et al. MEGA 4: molecular evolutionary genetics analysis (MEGA) software version 4.0[J]. Mol Biol Evol, 2007, 24(8): 1596-1599. doi: 10.1093/molbev/msm092
[12] IRWIN D M, KOCHER T D, WILSON A C. Evolution of the cytochrome b of mammals[J]. Mol Evol, 1991, 32(2): 128-144. doi: 10.1007/BF02515385
[13] NICOLAS V, GUILLAUME L. Weightion and congruence: a case study based on three mitochondrial genes in pitvipers[J]. Mol Phylogenet Evol, 1998, 9(3): 366-374. doi: 10.1006/mpev.1998.0509
[14] 周继亮, 张亚平, 黄美华, 等. 蝮亚科蛇线粒体Cyt b基因序列分析及其系统发育[J]. 动物学报, 2001, 47(4): 361-366. doi: 10.3969/j.issn.1674-5507.2001.04.001 [15] EDWARDS S V, ARCTANDER P, WILSON A C. Mitochondrial resolution of a deep branch in the genealogical tree for perching birds[J]. Proceedings: Biol Sci, 1991, B243: 99-107. doi: 10.1098/rspb.1991.0017
[16] KNIGHT A, MINDELL D P. Substitions bias, weighting of DNA sequence evolution, and the phylogenetic positions of fea′s viper[J]. Syst Biol, 1993, 42(1): 18-31. https://academic.oup.com/sysbio/article-abstract/42/1/18/1631945?login=false
[17] SALEMI M. Practice: the PHYL IP and TREE-PUZZLE software packages[M]//SALEMI M, VANDAMMME A M. The phylogenetic handbook: A practical approach to DNA and protein phylogeny. Cambridge, UK: Cambridge University Press, 2003. https://pubmed.ncbi.nlm.nih.gov/11934758/
-
期刊类型引用(8)
1. 张秀霞,汪蕾,李军涛,张泽龙,王冬梅,冼健安,孙敬锋. 饲料添加光合细菌对红螯螯虾幼虾生长、消化酶活力与免疫力的影响. 饲料研究. 2021(06): 62-65 .  百度学术
百度学术
2. 王一福,周胜杰,杨蕊,于刚,王志,马振华. 甘草对尖吻鲈生长特性、消化酶及免疫酶的影响. 南方农业学报. 2020(12): 3116-3125 .  百度学术
百度学术
3. 王一福,韩明洋,周胜杰,陈旭,付正祎,杨蕊,孟祥君,马振华. 饲料大蒜素水平对尖吻鲈消化酶、肝脏免疫酶活性的影响. 天津农学院学报. 2019(04): 71-76 .  百度学术
百度学术
4. 周胜杰,马婷,胡静,马振华,杨其彬,陈旭,杨蕊,刘亚娟,孟祥君. 尖吻鲈仔鱼骨骼发育观察. 南方农业学报. 2018(03): 592-598 .  百度学术
百度学术
5. 黄灵,李小梅,舒琥,张海发,石和荣. 饲料中添加不同益生菌对虎龙斑生长、免疫力及抗氧化能力的影响. 饲料工业. 2016(22): 15-20 .  百度学术
百度学术
6. 李小义,孔杰,赵凤,杨明举. 常用益生菌在鲟鱼饲料添加剂中的应用及研究进展. 贵州畜牧兽医. 2016(06): 61-63 .  百度学术
百度学术
7. 何伟聪,董晓慧,谭北平,杨奇慧,迟淑艳,刘泓宇,章双. 益生菌对军曹鱼幼鱼生长性能、消化酶和免疫酶活性的影响. 动物营养学报. 2015(12): 3821-3830 .  百度学术
百度学术
8. 文琳,雷燕,戚瑞荣,肖洋,王雪鹏. 尖吻鲈Lates calcarifer虹彩病毒病的诊断. 水产学杂志. 2015(04): 28-32 .  百度学术
百度学术
其他类型引用(10)



 下载:
下载:
 粤公网安备 44010502001741号
粤公网安备 44010502001741号
