Phylogenetic relationship of pearl oysters in genus Pinctada based on partial mitochondrial 16S rRNA sequence
-
摘要:
利用PCR技术分别扩增合浦珠母贝(Pinctada fucata)、长耳珠母贝(P.chemnitzi)和企鹅珍珠贝(Pteria penguin)的16S rRNA基因片段,PCR产物直接测序,删去引物及部分端部序列后,得到439 bp可供分析的核苷酸片段,用MEGA 3.1软件分析了核苷酸差异。结果表明,合浦珠母贝种内个体间序列完全相同,长耳珠母贝种内个体间有2个颠换(Transversion)突变位点,而企鹅珍珠贝种内个体间有1个转换(Transision)突变位点,5个颠换(Transversion)突变位点。与GenBank数据库中其他9种珍珠贝的序列进行比较,得到464个同源比对位点,包括51个插入/缺失位点和231个变异位点(173个简约信息位点和53个单突变子)。合浦珠母贝与长耳珠母贝的同源性为83.2%,合浦珠母贝、长耳珠母贝与企鹅珍珠贝的同源性分别为55.4%和59%。NJ系统进化树表明合浦珠母贝和长耳珠母贝与珠母贝属的种类聚在一起,而企鹅珍珠贝与珍珠贝属的种类聚在一起,与形态学分类一致。
-
关键词:
- 珍珠贝 /
- 16S rRNA基因 /
- 序列分析
Abstract:Mitochondrial 16S rRNA gene fragments were amplified from Pinctada fucata, P.chemnitzi and Pteria penguin using PCR technique. The PCR products were directly sequenced, and 439 bp nucleotide sequences were obtained after excluding the primers and truncating partial sequences at the end. Sequence variation was analyzed using MEGA 3.1 software. The results indicated that base substitution was not found among P.fucata, but two transversions were found among P.chemnitzi, one transition and five transversions among P.penguin. Alignment with nine homologous sequences from Pteridae species in GenBank revealed 51 insertion/deletions and 231 variable sites (173 parsimony-informative sites and 53 singletons) among 464 truncated alignment sites. The percentage of the identity of the 16S rRNA gene fragments was 83.2% between P.fucata and P.chemnitzi, 55.4% between P.fucata and Pteria penguin, and 59% between P.chemnitzi and P.penguin. The NJ tree indicated that P.fucata and P.chemnitzi were assembled together with Pinctada species, yet P.penguin assembled together with the Pteria species, which was consistent with the morphological classification.
-
Keywords:
- pearl oyster /
- 16S rRNA gene /
- sequence analysis
-
中国的珍珠贝主要包括珠母贝属(Pinctada)的合浦珠母贝(P.fucata)、大珠母贝(P.maxima)、白珠母贝(P.albina)、珠母贝(P.margaritifera)、黑珠母贝(P.nigra)、长耳珠母贝(P.chemnitzi)和珍珠贝属的企鹅珍珠贝(Pteria penguin)等[1-2]。这些珍珠贝是生产海水珍珠的主要母贝,具有重要的经济价值。但近年来在中国的海水珍珠生产中频频发生母贝大量死亡现象,以及珍珠质量的严重下降。除环境条件日益恶化的因素外[3],种质退化和遗传多样性下降可能有重要影响。对此,非常有必要研究和认识珍珠贝各自然群体的遗传结构和遗传变异水平,为有效管理、利用和保护珍珠贝种质资源和优良种质培育提供遗传学依据。
近年来分子系统学及DNA分子技术的快速发展,使得从分子水平研究物种间的亲缘关系及进行物种鉴定成为可能。动物线粒体基因组是一个小分子量环状分子(约为15.7~19.5 kb),一般认为它具有比核DNA高几倍的进化率[4]。在绝大多数动物中线粒体是单(母)系遗传,因此没有像核基因存在的重组现象[5]。线粒体DNA(mtDNA)作为遗传标记被广泛用于群体遗传和系统进化的研究。李刚等[6]运用同工酶谱对合浦珠母贝、长耳珠母贝和大珠母贝种间人工杂交进行了研究,证实这3个种的同工酶谱具有种的特异性,可作为鉴定种的一种分子手段。阎冰等[7]对马氏珠母贝和长耳珠母贝进行了RAPD分析,发现两者扩增带谱不同,具有各自的群体特征带。王小玉等[8]利用RAPD标记可将7种珍珠贝区分开来,而喻达辉等[9]对珠母贝6个种的ITS2序列进行了分析,发现不同种间序列差异较大,但种内差异非常小,认为ITS2序列可以作为种间鉴别标记。
文章拟通过分析合浦珠母贝、长耳珠母贝和企鹅珍珠贝的16S rRNA基因的核苷酸序列变异,并利用GenBank数据库其他珍珠贝种类的16S rRNA序列资料,探讨珍珠贝类的遗传变异与亲缘关系,为其遗传资源保护与利用提供理论依据。
1. 材料与方法
1.1 材料
样品的种类和采集地点见表 1。合浦珠母贝取3个个体,而长耳珠母贝和企鹅珍珠贝各取2个个体,分别取少许闭壳肌保存于95%的酒精中待用。
表 1 样品的种类和采集地点Table 1. Species and sampling location种类
species采样地点
sampling locationGenBank序列号
GenBank accession No.合浦珠母贝 Pinctada fucata 大亚湾、三亚和北海 长耳珠母贝 P.chemnitzi 大亚湾、香港 EF056475EF056476 企鹅珍珠贝 Pteria penguin 三亚 EF031242 1.2 方法
1.2.1 基因组DNA的提取
DNA的提取参照郭奕惠等[10]的方法并略作改进。取约20 mg珍珠贝肌肉组织,用纯水洗涤2次后,放入1.5 mL离心管内,先加入100 μL TEN9细胞裂解缓冲液,剪碎,再加入TEN9至600 L,加入SDS至终浓度为2%,混匀,56 ℃温浴15 min,加入蛋白酶K(20 mg · mL-1)10 μL,于56 ℃消化至溶液澄清。加入15 μL RNaseA,37 ℃反应约15 min,冷却后分别用等体积的饱和酚;酚: 氯仿: 异戊醇(25 : 24 : 1);氯仿: 异戊醇(24 : 1)各抽提1次,加入2倍体积无水乙醇和1/10体积NaAc(3 mol · L-1,pH 5.2),于-20 ℃沉淀2 h以上,离心,75%乙醇洗涤2次,干燥后加入200 L去离子超纯水溶解,4 ℃存放。提取的基因组样品用1%琼脂糖凝胶电泳,EB染色检测,紫外分光光度计测定OD260和OD280 nm,检测抽提的DNA质量和计算DNA浓度,然后配成浓度为20 ng · μL-1备用。
1.2.2 引物设计与PCR扩增
根据已知双壳贝类16S rRNA基因序列设计合成一对引物,16S-F:5′-CGCCTGGTTGATTAAAAACATTGCTGC-3′,16S-R:5′-CCGGTTTGAACTCAGATCACGTA-3′。PCR反应体积为20 μL,包含1×PCR缓冲液[10 mmol · L-1 Tris-HCl(pH 9.0,25 ℃),50 mmol · L-1 KCl,0.1% Triton X-100],3 mmol · L-1 MgCl2,0.1 mmol · L-1 dNTP,0.15 μmol · L-1引物,1 U TaqDNA聚合酶(上海申能博彩),DNA模板30~50 ng。反应开始为93 ℃变性4 min,之后35个循环:93 ℃ 30 s,55 ℃ 30 s,72 ℃ 30 s,最后为72 ℃ 5 min。扩增产物全部上样到1.0%的琼脂糖凝胶进行电泳分离,然后割胶,用试剂盒(V-gene)进行PCR产物纯化和回收。PCR产物经纯化回收后送往上海英骏公司测序。
1.2.3 数据分析
所测序列经手工校正后,用Clustal 1.81[11]软件与其他已知序列进行比对。利用MEGA3.1软件,依KIMURA[12]双参数法(two-parameter method)对核苷酸位点的替换数进行统计,Gap处理为缺失,用最大简约法(maximum parsimony method,MP)进行聚类分析构建进化树,并用Bootstrap重复1 000次计算各分支的置信度。
2. 结果
2.1 16S rRNA基因的碱基组成与同源比对分析
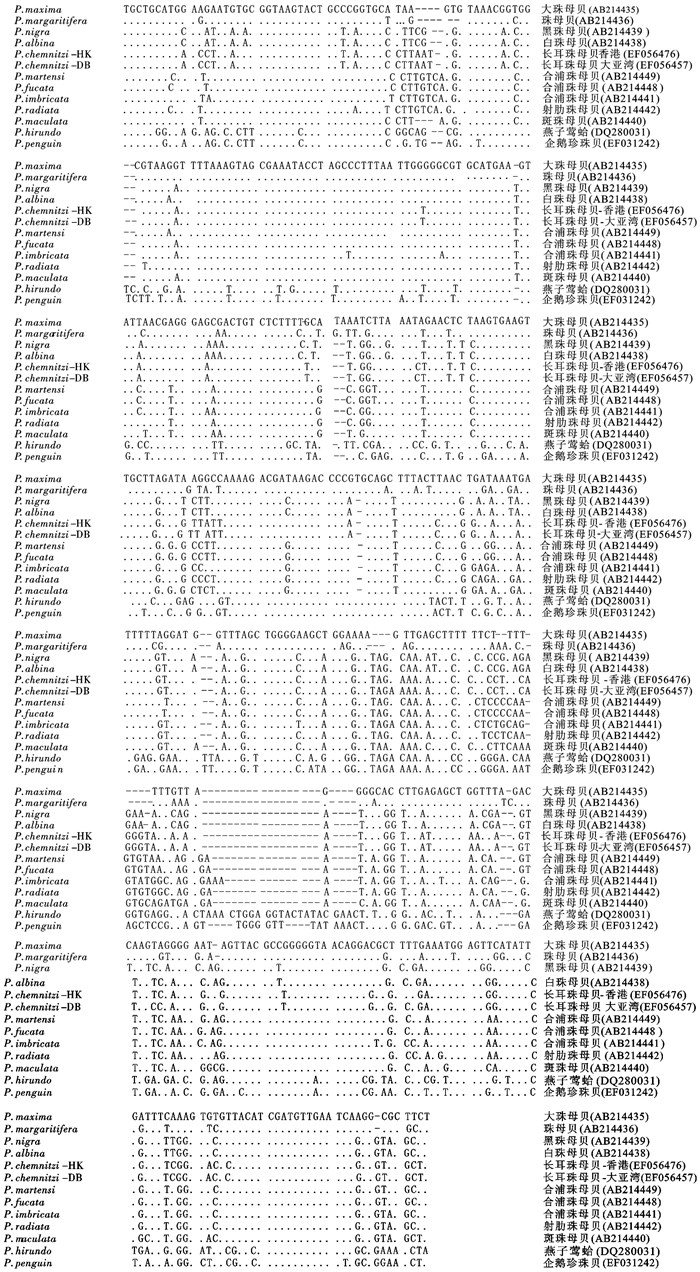
从所有个体的总DNA中均成功扩增出了大约500 bp的16S rRNA基因片段,直接把PCR产物送往上海英骏有限公司进行测序,将所测的3种核苷酸序列经手工校正后用Clustal 1.81软件对齐,删去引物及部分端部序列后,得到439 bp可供分析的核苷酸片段。同源比对结果显示,合浦珠母贝(3个个体)种内个体间序列完全相同,与网上已注册的合浦珠母贝序列(注册号:AB214441、AB214448和AB214449)完全一致;长耳珠母贝(2个个体)种内个体间有2个颠换(Transversion)突变位点,与其他珠母贝属的珍珠贝同源性都比较高;而企鹅珍珠贝(2个个体)种内个体间有1个转换(Transision)突变位点和5个颠换(Transversion)突变位点,与珍珠贝属的珍珠贝同源性较高,与其他珠母贝属的珍珠贝同源性较低。与其他珍珠贝一样,3种贝的16S rRNA基因中碱基A和T比G和C含量丰富(表 2)。同Genbank中其他9种珍珠贝的序列进行比较,得到464个同源比对位点,包括51个插入/缺失位点和231个变异位点(173个简约信息位点,53个单突变子,图 1)。合浦珠母贝与长耳珠母贝的同源性为83.2%,合浦珠母贝、长耳珠母贝与企鹅珍珠贝的同源性分别为55.4%和59.0%。表明同属珍珠贝同源性较高,不同属珍珠贝同源性较低。
表 2 16S rRNA基因片段的碱基组成Table 2. Base composition of 16S rRNA gene fragments in pearl oysters% 物种
species碱基组成base composition GC含量
GC contentA T C G 合浦珠母贝 Pinctada fucata 28.0 25.1 17.1 29.8 46.9 射肋珠母贝 P.radiata 27.5 25.3 17.6 29.7 47.3 长耳珠母贝 P.chemnitzi 29.0 27.9 15.8 27.2 43.0 黑珠母贝 P.nigra 29.7 26.8 16.2 27.3 43.5 斑珠母贝 P.maculata 28.0 25.6 17.4 28.9 46.3 大珠母贝 P.maxima 28.0 29.8 15.2 27.0 42.2 白珠母贝 P.albina 29.7 26.8 16.2 27.3 43.5 珠母贝 P.margaritifera 28.3 28.3 15.4 28.1 43.5 企鹅珍珠贝 Pteria penguin 27.9 30.2 14.8 27.1 41.9 燕子莺蛤 P.hirundo 26.0 26.9 17.3 29.8 47.1 2.2 遗传距离分析
根据Kimura双参数模型,得到12种珍珠贝类的遗传距离为0~0.490。珠母贝属的9种珍珠贝遗传距离为0~0.288,珍珠贝属的2种珍珠贝(P.penguin和P.hirundo)遗传距离为0.163 (表 3)。同属珍珠贝之间的遗传距离较小,不同属珍珠贝之间的遗传距离较大。合浦珠母贝(P.fucata)与长耳珠母贝(P.chemnitz)的遗传距离为0.168,合浦珠母贝(P.fucata)与企鹅珍珠贝(P.penguin)的遗传距离为0.446,长耳珠母贝(P.chemnitz)与企鹅珍珠贝(P.penguin)的遗传距离为0.410。合浦珠母贝(P.martensi)与(P.fucata)的遗传距离为0,白珠母贝(P.albina)与黑珠母贝(P.nigra)的遗传距离也为0。
表 3 12种珍珠贝类用Kimura双参数法计算的遗传距离Table 3. Pairwise genetic distances among 12 Pteriidae on the basis of Kimura two-parameter mode种类 species 1 2 3 4 5 6 7 8 9 10 11 12 13 1 大珠母贝
P.maxima2 珠母贝
P.margaritifera0.146 3 黑珠母贝
P.nigra0.288 0.279 4 白珠母贝
P.albina0.288 0.279 0.000 5 长耳珠母贝-香港
P.chemnitzi-HK0.279 0.276 0.122 0.122 6 长耳珠母贝-大亚湾
P.chemnitzi-DB0.286 0.283 0.125 0.125 0.007 7 合浦珠母贝
P.martensi0.245 0.244 0.144 0.144 0.168 0.171 8 合浦珠母贝
P.fucata0.245 0.244 0.144 0.144 0.168 0.171 0.000 9 合浦珠母贝
P.imbricata0.274 0.269 0.147 0.147 0.195 0.191 0.070 0.070 10 射肋珠母贝
P.radiata0.256 0.248 0.157 0.157 0.188 0.191 0.057 0.057 0.056 11 斑珠母贝
P.maculata0.270 0.253 0.155 0.155 0.161 0.164 0.103 0.103 0.120 0.097 12 燕子莺蛤
P.hirundo0.457 0.433 0.490 0.422 0.413 0.436 0.436 0.436 0.474 0.470 0.459 13 企鹅珍珠贝
P.penguin0.418 0.483 0.468 0.468 0.410 0.401 0.446 0.446 0.464 0.442 0.441 0.163 2.3 聚类分析
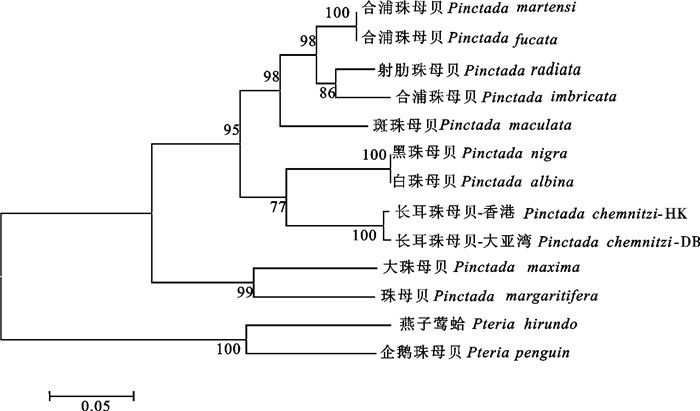
根据12种双壳贝类的16S rRNA基因序列用MEGA 3.1软件构建了它们的NJ系统树(图 2)。结果显示,系统进化树可分为2支,一支由珠母贝属的种类组成,包括合浦珠母贝、射肋珠母贝、斑珠母贝、黑珠母贝、白珠母贝、长耳珠母贝、大珠母贝和珠母贝等;另一支由珍珠贝属的种类组成,包括企鹅珍珠贝和燕子莺蛤(P.hirundo)。
3. 讨论
线粒体16S rRNA结构既具有保守性,又具有高变性。保守性能够反映生物物种的亲缘关系,为系统发育提供重要线索;高变性则能揭示出生物物种的特征核苷酸序列,是属种鉴定的分子基础,因此,一般认为16S rRNA适用于种以上水平的变异分析[13]。目前未见国内有关珍珠贝16S rRNA序列的报道,文章初步分析了合浦珠母贝、长耳珠母贝和企鹅珍珠贝的线粒体DNA 16S rRNA基因部分序列及其与珍珠贝科其他种类的亲缘关系。合浦珠母贝、长耳珠母贝和企鹅珍珠贝的16S rRNA基因部分序列的G、C含量分别为46.9%、43.0%和41.9%,和其他珍珠贝一样,碱基A、T含量比G、C丰富。
根据16S rRNA基因序列得到的遗传距离,合浦珠母贝与长耳珠母贝的遗传距离为0.168,合浦珠母贝、长耳珠母贝与企鹅珍珠贝的遗传距离分别为0.446和0.410,说明合浦珠母贝和长耳珠母贝亲缘关系较近,而与企鹅珍珠贝的亲缘关系较远。珠母贝属的9种珍珠贝遗传距离为0~0.288,珍珠贝属的2种珍珠贝(企鹅珍珠贝和燕子莺蛤)遗传距离为0.163。珠母贝属与珍珠贝属之间的遗传距离为0.410~0.490,表明珍珠贝科珠母贝属与珍珠贝属在遗传上存在较大的差异。
NJ系统进化树可分为2支,一支由珠母贝属的种类组成,另一支由珍珠贝属的种类组成。在珠母贝属的分支中,2种大型珠母贝(珠母贝和大珠母贝)最早分支出来,表明与小型珠母贝之间亲缘关系最远,这一结果与ITS[14-15]和RAPD[16]的结果一致。小型珠母贝之间又形成2个大的分支,其中P.fucata和P.martensi聚在一起,遗传距离为0,表明它们有可能是同种,射肋珠母贝(P.radiata)与P.imbricata聚在一起,遗传距离为0.057,表明两者之间的亲缘关系很近,此结果与BEAUMONT和KHAMDAN[17]的等位酶(allozyme)分析结果一致。BEAUMONT和KHAMDAN认为海湾地区的常见种射肋珠母贝(P.radiata)与澳大利亚的P.imbricata和日本的P.fucata为同种,而且在形态上很难区分射肋珠母贝与合浦珠母贝[2]。因此,射肋珠母贝与合浦珠母贝是同物异名的可能性很大。喻达辉和朱嘉濠[18]用ITS序列和AFLP标记对P.fucata、P.martensi和P.imbricata这3个种的遗传关系进行了分析,3个种群的ITS1和ITS2的种间遗传距离非常小,与种内遗传距离相互重叠,而系统发育分析也表明不同种的个体相互混聚在一起。此外,种间的遗传分化也很低。认为这3个种实际上是同一物种。P.fucata和P.martensi以及射肋珠母贝与P.imbricata汇合后与斑珠母贝聚合在一起形成一个大的分支,表明斑珠母贝与射肋珠母贝和合浦珠母贝的亲缘关系较近。
另一大分支由黑珠母贝、白珠母贝和长耳珠母贝组成。其中黑珠母贝和白珠母贝聚在一起,遗传距离为0,表明它们亲缘关系很近或有可能是同种。黑珠母贝和白珠母聚合后与长耳珠母贝汇合。王祯瑞[2]认为黑珠母贝和白珠母贝的形态差别主要是颜色方面的差异。喻达辉等[14]利用ITS1序列构建的系统树,黑珠母贝和白珠母贝聚为一支成为类群Ⅱ,遗传距离为0.013,认为可能为2个亚种。HE等[19]对中国珠母贝属5个种的ITS 2序列进行了分析,发现大珠母贝和珠母贝、黑珠母贝的遗传距离较近。长耳珠母贝与黑珠母贝首先聚在一起,再与马氏珠母贝聚在一起。王嫣等[16]利用RAPD标记分析了马氏珠母贝、大珠母贝、珠母贝、解氏珠母贝(也叫长耳珠母贝)以及企鹅珍珠贝的系统发育。发现大珠母贝与珠母贝的遗传距离最小,与解氏珠母贝的遗传距离最大。马氏珠母贝与大珠母贝的遗传距离较与珠母贝的大。进化程度由低到高的等级顺序为解氏珠母贝-马氏珠母贝-珠母贝-大珠母贝。此研究利用PCR技术扩增获得合浦珠母贝、长耳珠母贝和企鹅珍珠贝16S rRNA基因长约439 bp可供分析的核苷酸片段,了解了这3个种群的遗传差异及与珍珠贝科其他珍珠贝的分子遗传关系,可为今后珍珠贝的遗传选育以及养殖生产提供分子遗传学基础。
-
表 1 样品的种类和采集地点
Table 1 Species and sampling location
种类
species采样地点
sampling locationGenBank序列号
GenBank accession No.合浦珠母贝 Pinctada fucata 大亚湾、三亚和北海 长耳珠母贝 P.chemnitzi 大亚湾、香港 EF056475EF056476 企鹅珍珠贝 Pteria penguin 三亚 EF031242 表 2 16S rRNA基因片段的碱基组成
Table 2 Base composition of 16S rRNA gene fragments in pearl oysters
% 物种
species碱基组成base composition GC含量
GC contentA T C G 合浦珠母贝 Pinctada fucata 28.0 25.1 17.1 29.8 46.9 射肋珠母贝 P.radiata 27.5 25.3 17.6 29.7 47.3 长耳珠母贝 P.chemnitzi 29.0 27.9 15.8 27.2 43.0 黑珠母贝 P.nigra 29.7 26.8 16.2 27.3 43.5 斑珠母贝 P.maculata 28.0 25.6 17.4 28.9 46.3 大珠母贝 P.maxima 28.0 29.8 15.2 27.0 42.2 白珠母贝 P.albina 29.7 26.8 16.2 27.3 43.5 珠母贝 P.margaritifera 28.3 28.3 15.4 28.1 43.5 企鹅珍珠贝 Pteria penguin 27.9 30.2 14.8 27.1 41.9 燕子莺蛤 P.hirundo 26.0 26.9 17.3 29.8 47.1 表 3 12种珍珠贝类用Kimura双参数法计算的遗传距离
Table 3 Pairwise genetic distances among 12 Pteriidae on the basis of Kimura two-parameter mode
种类 species 1 2 3 4 5 6 7 8 9 10 11 12 13 1 大珠母贝
P.maxima2 珠母贝
P.margaritifera0.146 3 黑珠母贝
P.nigra0.288 0.279 4 白珠母贝
P.albina0.288 0.279 0.000 5 长耳珠母贝-香港
P.chemnitzi-HK0.279 0.276 0.122 0.122 6 长耳珠母贝-大亚湾
P.chemnitzi-DB0.286 0.283 0.125 0.125 0.007 7 合浦珠母贝
P.martensi0.245 0.244 0.144 0.144 0.168 0.171 8 合浦珠母贝
P.fucata0.245 0.244 0.144 0.144 0.168 0.171 0.000 9 合浦珠母贝
P.imbricata0.274 0.269 0.147 0.147 0.195 0.191 0.070 0.070 10 射肋珠母贝
P.radiata0.256 0.248 0.157 0.157 0.188 0.191 0.057 0.057 0.056 11 斑珠母贝
P.maculata0.270 0.253 0.155 0.155 0.161 0.164 0.103 0.103 0.120 0.097 12 燕子莺蛤
P.hirundo0.457 0.433 0.490 0.422 0.413 0.436 0.436 0.436 0.474 0.470 0.459 13 企鹅珍珠贝
P.penguin0.418 0.483 0.468 0.468 0.410 0.401 0.446 0.446 0.464 0.442 0.441 0.163 -
[1] 王祯瑞. 中国近海珍珠贝科的研究[M]//中国科学院海洋研究所. 海洋科学集刊. 北京: 科学出版社, 1978, 14: 101-117. http://qdhys.cnjournals.com/hykxjk/ch/reader/view_abstract.aspx?file_no=19781407&flag=1 [2] 王祯瑞. 软体动物门双壳纲珍珠贝亚目[M]//中国动物志无脊椎动物31卷. 北京: 科学出版社, 2002: 68-98. [3] 艾红, 李永振, 丁彦文. 海洋珍珠贝病害研究综述[J]. 上海水产大学学报, 2003, 12(1): 61-64. doi: 10.3969/j.issn.1004-7271.2003.01.012 [4] BROWN W M, GEORGE M J, WILSON A C. Rapid evolution of animal mitochondrial DNA[J]. PNAS, 1979, 76(4): 1 967-1 971. doi: 10.1073/pnas.76.4.1967
[5] WHITMORE D H, THAI T H, CRAFT C M. The large mouth bass ctyochrome b gene[J]. J Fish Biol, 1994, 44(4): 637-645. doi: 10.1111/j.1095-8649.1994.tb01240.x
[6] 李刚, 姜卫国, 魏贻尧. 合浦珠母贝、长耳珠母贝和大珠母贝种间人工杂交的研究: Ⅲ同工酶谱的比较研究[J]. 热带海洋, 1983, 2 (4): 321-328. https://kns.cnki.net/kcms2/article/abstract?v=QnUIp1GSfy66myH3-KMTFn4tiXRN77m2m_8slizx4XZew2KPSHOtk1uuHGgx7rVclTtJD6lYgY2mLS7oWUrRfKjnZHbUcg11nUBFqkKzJZiwo8cMD6QxEv4ZYA5jLDGr31kQC5_BJ2Ovs6wtEXUscOREQS_UFiXEMkkN3Mwo_sOeraZ-ZdxVlMyJcRSt9T-y&uniplatform=NZKPT&language=CHS [7] 阎冰, 叶力, 邓凤娇, 等. 马氏珠母贝与解氏珠母贝的随机扩增多态DNA分析[J]. 广西科学, 2001, 8(4): 287-290. doi: 10.3969/j.issn.1005-9164.2001.04.013 [8] 王小玉, 喻达辉, 郭奕惠, 等. 7种珍珠贝RAPD鉴别标记的初步研究[J]. 南方水产, 2006, 2(1): 18-22. doi: 10.3969/j.issn.2095-0780.2006.01.004 [9] 喻达辉, 朱嘉濠. 珠母贝属6个种的ITS2分子标记研究[J]. 南方水产, 2005, 1(4): 6-12. doi: 10.3969/j.issn.2095-0780.2005.04.002 [10] 郭奕惠, 黄桂菊, 喻达辉. 合浦珠母贝DNA的抽提和RAPD反应体系的优化[J]. 南方水产, 2006, 2(4): 59-64. doi: 10.3969/j.issn.2095-0780.2006.04.010 [11] THOMPSON J D, GIBSON T J, PLEWNIAK F J, et al. The Clustal X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Res, 1997, 25(24): 4 876-4 882. doi: 10.1093/nar/25.24.4876
[12] KUMAR M. A simple method for estimating evolutionary rate of base substitution through comparative studies of nucleotide sequence [J]. Mol Evol, 1980, 16(2): 111-120. doi: 10.1007/BF01731581
[13] 童金苟, 吴清江. 3个鲤品种线粒体基因片段序列保守性[J]. 水生生物学报, 2001, 25(1): 54-60. doi: 10.3321/j.issn:1000-3207.2001.01.008 [14] 喻达辉, 朱嘉濠, 贾晓平. 我国珠母贝属(Pinctada)主要种类亲缘关系的初步分析[J]. 海洋与湖沼, 2006, 37(3): 211-217. doi: 10.3321/j.issn:0029-814X.2006.03.004 [15] 喻达辉, 朱嘉濠. 珠母贝属的系统发育: 核rDNA ITS序列证据[J]. 生物多样性, 2005, 13(4): 315-323. doi: 10.3321/j.issn:1005-0094.2005.04.004 [16] 王嫣, 石耀华, 顾志峰, 等. 5种常见珍珠贝的系统发育和遗传多样性分析[J]. 基因组学与应用生物学, 2009, 28(2): 293-300. doi: 10.3969/gab.028.000293 [17] BEAUMONT A R, KHAMDAN S A A. Electrophoretic and morphometric characters in population differentiation of the pearl oyster, Pinctada radiata (Leach), from around Bahrain[J]. J Molluscan Study, 1991, 57(4): 433-441. doi: 10.1093/mollus/57.4.433
[18] 喻达辉, 朱嘉濠. 利用ITS和AFLP标记探讨中国、日本和澳大利亚3种珠母贝的亲缘关系[J]. 南方水产, 2006, 2(5): 36-44. doi: 10.3969/j.issn.2095-0780.2006.05.007 [19] HE M, HUANG L, SHI J, et al. Variability of ribosomal DNA ITS-2 and its utility in detecting genetic relatedness of pearl oyster[J]. Mar Biotechnol, 2005, 7(1): 40-45. doi: 10.1007/s10126-004-0003-6



 下载:
下载:
 粤公网安备 44010502001741号
粤公网安备 44010502001741号
