Sequence analysis of eukaryotic translation initiation factor 3 subunit G (eIF3g) of black tiger shrimp (Penaeus monodon) and its mRNA tissue expression
-
摘要:
通过mRNA差异显示(differential display,DD)技术筛选的差异片段,与斑节对虾(Penaeus monodon)混合组织cDNA文库中的EST进行拼接,获得斑节对虾真核生物翻译起始因子3亚基4(btseIF3g)的全序列(GenBank登录号:FJ829364)。btseIF3g全长1 041 bp,由72 bp 5′非编码区、90 bp 3′非编码区以及879 bp的开放阅读框组成,推测编码一个长度为292个氨基酸的蛋白。经BLAST分析,btseIF3g C端含有一个RNA识别基序(RNA recognition motif,RRM)的保守结构域(E值为2.72e-25)。利用氨基酸序列进行系统发育分析表明,btseIF3g与eIF3g属于同一分支。采用荧光定量PCR(Real-time PCR)方法对250日龄的雌、雄斑节对虾不同组织表达特性研究发现,btseIF3g在眼柄神经节、脑神经、肝胰腺、性腺和肌肉组织中均有表达,在卵巢中的相对表达量极显著(P < 0.01)高于其他组织,这一结果提示eIF3g可能与斑节对虾卵巢发育相关。
Abstract:In order to analyze the function of a differential expression cDNA fragment of gonads of domesticated broodstock black tiger shrimp (Penaeus monodon), the black tiger shrimp eukaryotic translation initiation factor 3 subunit G (btseIF3g) had been found by spliced with the EST, after screening in the P.monodon mix cDNA library. The full length cDNA of btseIF3g (GenBank accession no. FJ829364) contained a 5′ untranslated region (UTR) of 72 bp, a 3′UTR of 90 bp and an ORF of 879 bp encoding a polypeptide of 292 amino acids with an estimated molecular mass of 32.97 kDa. RNA recognition motif (RRM) was found in the btseIF3g deduced amino acid sequence. Phylogenetic analysis of the eIF3 gene family indicated that the btseIF3g was homologous to the eIF3g. mRNA expression of btseIF3g could be detected in 250-day-old P.monodon′s eyestalk, brain, hepatopancreas, gonad and muscle by fluorescent quantitative real-time PCR analysis. The highest expression level of btseIF3g in the ovary indicated that btseIF3g played an important role in oogenesis.
-
Keywords:
- Penaeus monodon /
- eIF3g /
- molecular cloning /
- tissue expression
-
真核生物蛋白质合成包括起始、延伸和终止3个阶段,每一阶段都涉及一组不同的蛋白质因子,分别称为起始因子、延长因子和终止因子,该过程主要在细胞质内的核糖体并在一系列真核生物翻译起始因子(eukaryotic translation initiation factor,eIFs)的参与下完成。目前在真核生物中已经鉴定了13个翻译起始因子,其中eIF3蛋白质分子量最大(~650 kDa),由8~13个多肽组成。eIF3在Met-tRNA与mRNA结合到40S核糖体亚基这一翻译起始过程中发挥核心作用,eIF3在缺失其他翻译元件的情况下与40S核糖体亚基结合从而保持40S和60S亚基的解聚状态,并可稳定和促进eIF2-GTP-Met-tRNAi三元复合物与mRNA的结合。此外,eIF3与40S亚基的结合参与mRNA结合的起始阶段。eIF3是迄今为止了解最少而又最为复杂的一种起始因子,它是一个多亚基复合物,组成亚基的分子量由28~180 kDa不等。目前已在小麦胚、Hela细胞、兔网织红细胞和酵母中分离纯化得到eIF3蛋白复合物。低等真核生物(如酵母)的eIF3包括8个不同的亚基,其中5个亚基(a、b、c、g和i,分别对应哺乳动物eIF3的p170、p116、p110、p44和p36亚基)组成核心复合物[1];高等植物(无论是单子叶还是双子叶植物)与哺乳动物的eIF3在亚基组成上则有很大的相似性,均包括11个亚基,其中10个亚基一致。最近研究发现eIF3g和eIF3i并非哺乳动物eIF3蛋白核心复合物所必需的,哺乳动物eIF3的功能核心由3个进化保守亚基(a、b和c)和3个不保守亚基(e、f和h)组成[2]。迄今为止,已在拟南芥、哺乳动物和酵母中克隆得到eIF3全部亚基的cDNA编码序列,对于各亚基生物学功能的研究主要集中于这几类生物。
此文以差异显示PCR技术筛选的斑节对虾(Penaeus monodon)性腺差异表达阳性克隆片段,与笔者实验室已测序完成的混合组织cDNA文库中的ESTs进行比对查找,首次在甲壳动物中分离得到编码斑节对虾eIF3g基因的全长cDNA。通过对其序列和组织表达分析探讨其在斑节对虾中可能的生物学功能,为进一步认识对虾生长发育与繁殖的调控机制奠定基础。
1. 材料与方法
1.1 试验虾
试验所用的斑节对虾为广东阳江地区全人工培育的同一批次苗种,全人工池塘养殖250日龄的斑节对虾。充氧后运回实验室,暂养3 d待其稳定后,随机选取健康无伤、活力好的雌、雄斑节对虾各6尾。雌虾纳精囊发育时期经组织切片,加以鉴定[4-5]。所取对虾性腺均发育成熟,其中雌虾卵巢发育处于Ⅰ期,体长为15.72±0.22 cm,体重为51.8±4.17 g;雄虾精荚饱满,体长为14.12±0.19 cm,体重为38.95±1.82 g。活体解剖取眼柄神经节、脑组织、肝胰腺、性腺和肌肉等组织,液氮保存备用。
1.2 方法
1.2.1 cDNA全长的获取
分别取50 mg雌、雄斑节对虾性腺组织各3套,液氮研磨。按照Trizol(Invitrogen)试剂操作说明书提取总RNA。无RNase的DNase-Ⅰ消化总RNA,去除DNA污染,酚-氯仿抽提除去总RNA中的蛋白质,获得纯净的总RNA。1.0%琼脂糖凝胶电泳检测总RNA完整性,紫外分光光度仪测定其浓度。分别将电泳条带边界清晰、OD260/OD280比值在1.8~2.0之间的卵巢和精巢总RNA等量混匀,构建总RNA混合池。
卵巢和精巢总RNA各取1 μg与3′端锚定引物H-T11G按照AMV试剂(Promega)说明书进行反转录,合成cDNA第一链。以反转录产物为模板,3′端锚定引物和5′端随机引物H-AP8配对进行cDNA扩增。PCR产物用6%尿素变性聚丙烯酰胺凝胶90 W恒定功率电泳2 h,硝酸银染色,对差异片段进行聚丙烯凝胶切胶回收并进行2次扩增,按照Wizard SV Gel and PCR Clean-Up System凝胶回收试剂盒(Promega)说明书对阳性的二扩PCR产物进行纯化回收。回收产物与pMD-18T载体(TaKaRa)连接后转化到大肠杆菌DH5α感受态细胞,经蓝白斑筛选挑选白色阳性克隆,提取质粒DNA,Pst I单酶切鉴定。将含有阳性克隆的菌液送往上海生工测序。此试验所用引物序列见表 1。
表 1 此研究所用引物信息Table 1. Primers information in this study引物名称 primer names 引物序列(5′to 3′) primer sequences 作用 function H-T11G AAGCTTTTTTTTTTTTTG 3′端锚定引物 H-AP8 AAGCTTTTACCGC 5′端随机引物 BtseIF3g-F GAGCAACAAAGATGAGGAGCAGAATG 荧光定量 BtseIF3g-R TCCCAGTAGGGATGACCGCAGA β-actin-F TACTCCTACACTCATAAACCAACGA 扩增内参 β-actin-R TTTCGGAATTTTATTATCAACTGTA 将获得的斑节对虾性腺差异表达片段与笔者实验室已测序完成的混合组织cDNA文库中的ESTs进行比对查找,利用DNAstar软件将差异片段序列与文库查找所得序列进行拼接获得全长cDNA序列。
1.2.2 序列分析
将拼接后的序列用Vector NTI 10.0软件及NCBI (http://www.ncbi.nlm.nih.gov/)在线工具预测确定开放阅读框(open reading frame,ORF)及其所编码的氨基酸序列。将预测的氨基酸序列BLAST比对进行同源性分析。用DNAStar、ClustalX 1.83和MEGA 4.0等软件分析拼接后的序列,并构建它与其他物种的系统发生树。用SMART在线工具预测蛋白质结构。
1.2.3 组织表达分析
根据GenBank中报道的斑节对虾β-actin mRNA序列(GenBank accession No:AF100987)和拼接后cDNA序列用Primer 5.0软件设计引物(表 1)。用Trizol试剂提取雌、雄斑节对虾各3尾的眼柄神经节、大脑、肝胰腺、性腺及肌肉组织的总RNA,紫外分光光度计定量后,DNase-Ⅰ消化除去DNA污染。按照ReverTRA ace-α-TM(TOYOBO)反转录试剂盒使用说明书进行反转录,反转录产物于-20℃保存备用。参照SYBR Green Real-time PCR Master Mix说明书,在Eppendorf realplex2上对目的基因进行Real-time RT-PCR组织表达分析。20 μL SYBR定量PCR体系为:灭菌水7.2 μL,10 μL 2×SYBR Green Realtime PCR Master Mix,10 μmol·L-1上、下游引物各0.4 μL,10倍稀释的反转录产物2 μL。反应条件为:95℃ 1 min;95℃ 15 s,58℃ 15 s,72℃ 30 s共进行40个循环;在反应结束后执行熔解曲线分析以确定得到的产物为目的产物。每个反应设置3个重复,以β-actin为内参,对cDNA模板的上样量进行调整,同时以不加模板的体系和以RNA为模板的体系做对照,以排除试剂污染和基因组DNA污染引起的假阳性的可能性。将获得的目的基因和其对应的β-actin的Ct值,参照LIVAK和SCHMITTGEN[6]的处理方法,根据公式2-△△Ct求出目的基因在各组织中的相对表达量,其中△Ct=Ct目标基因-Ctβ-actin。用SAS V8.02统计软件对目的基因在各组织及眼柄神经节、大脑、肝胰腺、性腺及肌肉这5类组织中的表达差异情况进行单因素方差分析。
2. 结果
2.1 斑节对虾eIF3g全长cDNA克隆及序列分析
通过差异显示方法筛选到一条编号为OG8-1,长度为787 bp的差异片段,同时在cDNA文库中发现1条长度为820 bp的EST(EST No. contig381),经BLAST发现两者均与真核细胞翻译起始因子3亚基4有高度同源性,将2条EST有效拼接得到长度为1 041 bp的cDNA序列(图 1)。用Vector NTI 10.0软件及NCBI在线预测,发现该cDNA序列由72 bp的5′端非编码区(Untranslated region,UTR),90 bp的3′端UTR和879 bp的ORF组成,推测编码一个长度为292个氨基酸的蛋白。预测该蛋白分子量(MW)为32.97 kDa,等电点(PI)为7.29。SMART在线预测蛋白质结构发现其C端存在一个RNA识别基序(RNA recognition motif,RRM)结构域(E值2.72e-25),这与eIF3g特征相似,将其命名为btseIF3g(GenBank登录号:FJ829364)。
![]() 图 1 斑节对虾eIF3g cDNA序列及其推导的氨基酸序列编码区用大写字母表示,非编码去用小写字母表示,氨基酸用单个大写字母表示,排在对应核苷酸序列下面,核苷酸阴影区分别为起始密码子和终止子;RNA识别模体用下划线标识Figure 1. The cDNA sequence of eIF3g and deduced amino acid sequence of the black tiger shrimpCoding region was shown in capital letters. Non-coding region was shown in small letters. The deduced amino acids were shown by single letter code of amino acids below the coding region. Initial codon and terminal codon were shaded. RBA was underlined.
图 1 斑节对虾eIF3g cDNA序列及其推导的氨基酸序列编码区用大写字母表示,非编码去用小写字母表示,氨基酸用单个大写字母表示,排在对应核苷酸序列下面,核苷酸阴影区分别为起始密码子和终止子;RNA识别模体用下划线标识Figure 1. The cDNA sequence of eIF3g and deduced amino acid sequence of the black tiger shrimpCoding region was shown in capital letters. Non-coding region was shown in small letters. The deduced amino acids were shown by single letter code of amino acids below the coding region. Initial codon and terminal codon were shaded. RBA was underlined.2.2 序列比对分析及系统进化树构建
利用MEGA 4.0软件中的邻位相联法(Neighbor-joining)构建btseIF3g推导的氨基酸序列与NCBI上已公布的eIF3氨基酸序列(表 2)的系统进化树(图 2)。结果表明eIF3各亚基很好的聚集于各自所属的类群中,btseIF3g被分在eIF3g分支中,其与斑马鱼(Danio rerio,NP_ 957293.1)的eIF3g同源性最高,为58%。同时与人(Homo sapiens,NP_ 003746.2)、小鼠(Mus musculus,NP_058572.2)、粟酒裂殖酵母(Schizosaccharomyces pombe,NP_ 595727.1)和拟南芥(Arabidopsis thaliana,NP_ 187747.1)等的eIF3g同源性较高,分别为53%、54%、40%和39%。这与最初的SMART结果一致。
表 2 NCBI上已公布的部分eIF3信息Table 2. eIF3 information published in NCBI序列名称
nameGenBank登录号
accession number物种
specieseIF3各亚基类型
eIF3 subunitP.monodon* FJ829364 斑节对虾 Penaeus monodon subunit G H.sapiens A/10 NP_003741.1 人 Homo sapiens subunit 10 theta, 150/170 kDa P.troglodytes A/10 XP_001153575.1 黑猩猩 Pan troglodytes subunit 10 theta, 150/170 kDa isoform 1 C.familiaris A/10 XP_852131.1 狗 Canis familiaris subunit 10 theta, isoform 2 B.taurus A/10 XP_884395.2 西藏黄牛 Bos taurus subunit A isoform 4 M.musculus A/10 NP_034253.2 小鼠 Mus musculus subunit 10 (theta) R.norvegicus A/10 XP_238649.4 褐家鼠 Rattus norvegicus similar to subunit 10 theta, 150/170 kDa D.rerio A/10 NP_956114.2 斑马鱼 Danio rerio subunit 10 (theta) D.melanogaster A/10 NP_649470.2 黑腹果蝇 Drosophila melanogaster S10, isoform A S.pombe A/10 NP_596379.1 粟酒裂殖酵母 Schizosaccharomyces pombe eIF3a A.thaliana A/10 NP_192881.1 拟南芥 Arabidopsis thaliana eIF3A H.sapiens B/9 NP_001032360.1 人 H.sapiens subunit 9 eta, 116 kDa C.familiaris B/9 XP_536894.2 狗 C.familiaris similar to subunit 9 M.musculus B/9 NP_598677.1 小鼠 M.musculus subunit 9 D.rerio B/9 XP_001335480.1 斑马鱼 D.rerio subunit B D.melanogaster B/9 NP_611228.1 黑腹果蝇 D.melanogaster S9, isoform B C.elegans B/9 NP_001022469.1 秀丽隐杆线虫 Caenorhabditis elegans eif-3.B A.thaliana B/9 NP_001031954.1 拟南芥 A.thaliana 3B1 H.sapiens C/8 NP_003743.1 人 H.sapiens subunit C C.familiaris C/8 XP_536923.2 狗 C.familiaris similar to subunit 8, 110 kDa B.taurus C/8 NP_001029790.1 西藏黄牛 B.taurus subunit C M.musculus C/8 NP_666312.1 小鼠 M.musculus subunit C R.norvegicus C/8 XP_215080.4 褐家鼠 R.norvegicus similar to subunit 8 D.rerio C/8 NP_998628.1 斑马鱼 D.rerio subunit C D.melanogaster C/8 NP_611242.1 黑腹果蝇 D.melanogaster S8 C.elegans C/8 NP_492638.1 秀丽隐杆线虫 C.elegans eif-3.C S.pombe C/8 NP_593828.2 粟酒裂殖酵母 S.pombe eIF3c A.thaliana C/8 NP_191174.1 拟南芥 A.thaliana EIF3C H.sapiens D/7 NP_003744.1 人 H.sapiens subunit D P.troglodytes D/7 XP_001158696.1 黑猩猩 P.troglodytes subunit 7 isoform 2 C.familiaris D/7 XP_861890.1 狗 C.familiaris similar to subunit 7 isoform 3 B.taurus D/7 NP_001029656.1 西藏黄牛 B.taurus subunit D M.musculus D/7 NP_061219.2 小鼠 M.musculus subunit 7 (zeta) R.norvegicus D/7 NP_001004283.1 褐家鼠 R.norvegicus subunit D G.gallus D/7 XP_001234643.1 红原鸡 Gallus gallus similar to subunit 7 zeta, 66/67 kDa D.rerio D/7 NP_956310.1 斑马鱼 D.rerio subunit D D.melanogaster D/7 NP_524463.2 黑腹果蝇 D.melanogaster p66 subunit C.elegans D/7 NP_498984.1 秀丽隐杆线虫 C.elegans eif-3.D H.sapiens E/6 NP_001559.1 人 H.sapiens subunit 6 48 kDa C.familiaris E/6 XP_532304.2 狗 C.familiaris similar to subunit 6 isoform 2 B.taurus E/6 NP_001029775.1 西藏黄牛 B.taurus subunit 6 48 kDa M.musculus E/6 NP_032414.1 小鼠 M.musculus subunit 6 R.norvegicus E/6 NP_001011990.1 褐家鼠 R.norvegicus subunit E G.gallus E/6 NP_001006349.1 红原鸡 G.gallus subunit E D.rerio E/6 NP_957133.1 斑马鱼 D.rerio subunit E, a C.elegans E/6 NP_492785.1 秀丽隐杆线虫 C.elegans eif-3.E A.thaliana E/6 NP_567047.1 拟南芥 A.thaliana eif3E H.sapiens F/5 NP_003745.1 人 H.sapiens subunit 5 epsilon, 47 kDa B.taurus F/5 XP_591540.1 西藏黄牛 B.taurus similar to 47 kDa subunit isoform 1 M.musculus F/5 NP_079620.1 小鼠 M.musculus subunit F R.norvegicus F/5 XP_215037.3 褐家鼠 R.norvegicus similar to subunit 5 epsilon, 47 kDa G.gallus F/5 XP_421624.1 红原鸡 G.gallus similar to 47 kDa subunit H.sapiens G/4 NP_003746.2 人 H.sapiens subunit 4 delta, 44 kDa P.troglodytes G/4 XP_001162705.1 黑猩猩 P.troglodytes subunit 4 delta, 44 kDa isoform 3 C.familiaris G/4 XP_868239.1 狗 C.familiaris similar to subunit 4(eIF3 p44) B.taurus G/4 XP_001251526.1 西藏黄牛 B.taurus subunit 4 delta M.musculus G/4 NP_058572.2 小鼠 M.musculus subunit G R.norvegicus G/4 NP_001013113.1 褐家鼠 R.norvegicus subunit G D.rerio G/4 NP_957293.1 斑马鱼 D.rerio subunit G S.pombe G/4 NP_595727.1 粟酒裂殖酵母 S.pombe eIF3g A.thaliana G/4 NP_187747.1 拟南芥 A.thaliana 3G1; RNA binding/translation initiation factor H.sapiens H/3 NP_003747.1 人 H.sapiens subunit 3 gamma, 40 kDa P.troglodytes H/3 XP_519914.2 黑猩猩 P.troglodytes subunit 3 gamma, 40 kDa isoform 6 C.familiaris H/3 XP_532315.1 狗 C.familiaris similar to subunit 3 gamma, 40 kDa isoform 1 B.taurus H/3 NP_001020493.1 西藏黄牛 B.taurus subunit 3 gamma, 40 kDa M.musculus H/3 NP_542366.1 小鼠 M.musculus subunit H R.norvegicus H/3 NP_942046.1 褐家鼠 R.norvegicus subunit H G.gallus H/3 NP_001026122.1 红原鸡 G.gallus subunit H D.rerio H/3 NP_001003763.1 斑马鱼 D.rerio subunit H D.melanogaster H/3 NP_524834.2 黑腹果蝇 D.melanogaster p40 subunit, isoform A A.thaliana H/3 NP_563880.1 拟南芥 A.thaliana subunit H1 H.sapiens I/2 NP_003748.1 人 H.sapiens subunit 2 beta, 36 kDa C.familiaris I/2 XP_535328.1 狗 C.familiaris similar to subunit 2 B.taurus I/2 NP_001015628.1 西藏黄牛 B.taurus subunit 2 beta, 36 kDa M.musculus I/2 NP_061269.1 小鼠 M.musculus subunit I D.rerio I/2 NP_998155.1 斑马鱼 D.rerio subunit 2 beta C.elegans I/2 NP_490988.2 秀丽隐杆线虫 C.elegans eif-3.I H.sapiens J/1 NP_003749.2 人 H.sapiens subunit 1 alpha, 35 kDa C.familiaris J/1 XP_544656.2 狗 C.familiaris similar to subunit 1(eif-3 alpha) B.taurus J/1 NP_001069611.1 西藏黄牛 B.taurus subunit J M.musculus J/1 NP_001069611.1 小鼠 M.musculus subunit J R.norvegicus J/1 XP_001080279.1 褐家鼠 R.norvegicus similar to subunit 1 (eif-3 alpha) G.gallus J/1 NP_001012789.1 红原鸡 G.gallus subunit J H.sapiens K/12 NP_037366.1 人 H.sapiens subunit 12 P.troglodytes K/12 XP_512639.2 黑猩猩 P.troglodytes subunit 12 C.familiaris K/12 XP_533679.1 狗 C.familiaris similar to subunit 12 isoform 1 B.taurus K/12 NP_001029661.1 西藏黄牛 B.taurus subunit K M.musculus K/12 NP_082935.1 小鼠 M.musculus subunit K R.norvegicus K/12 XP_214886.1 褐家鼠 R.norvegicus similar to subunit 12 C.elegans K/12 NP_506241.1 秀丽隐杆线虫 C.elegans eif-3.K H.sapiens L NP_057175 人 H.sapiens subunit 6 interacting protein C.familiaris L XP_531741 狗 C.familiaris similar to subunit 6 interacting protein isoform 1 D.rerio L NP_998293 斑马鱼 D.rerio subunit 6 interacting protein C.intestinalis L XP_002120603 玻璃海鞘 Ciona intestinalis similar to subunit 6 interacting protein H.sapiens M NP_006351.2 人 H.sapiens subunit M M.musculus M NP_663355.1 小鼠 M.musculus subunit M D.rerio M NP_001019906.1 斑马鱼 D.rerio subunit M 2.3 btseIF3g的组织表达
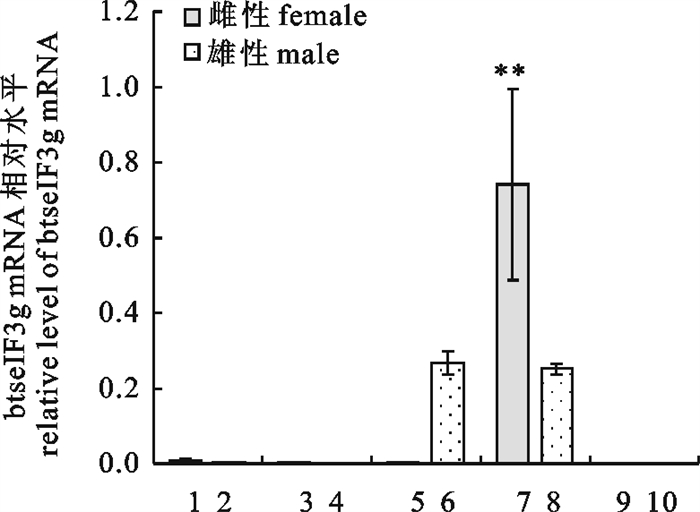
Real-time RT-PCR结果显示,试验重复性良好,Tm值与预期一致,表明反应体系和条件都得到很好的优化,引物的特异性高,real-time RT-PCR的验证结果可信。eIF3g在斑节对虾各组织中的表达丰度见图 3,btseIF3g在雌、雄个体的眼柄神经节、脑神经、肝胰腺、性腺和肌肉组织中均有表达,其中卵巢表达丰度最高,其次为雄性肝胰腺和精巢,而在雌性肝胰腺和雌、雄个体的眼柄神经节、脑神经和肌肉中的相对表达量较低。btseIF3g在雌、雄个体间的眼柄神经节、脑神经和性腺组织中的表达情况为雌性个体极显著(P < 0.01)高于雄性个体,在肌肉组织中没有表现出性别差异(P>0.05),而在肝胰腺组织中则表现为雄性极显著(P < 0.01)高于雌性。
![]() 图 3 各组织中btseIF3g mRNA表达1、3、5、7和9分别为雌性个体的眼柄神经节、脑神经、肝胰腺、性腺和肌肉;2、4、6、8和10分别为雄性个体的眼柄神经节、脑神经、肝胰腺、性腺和肌肉;n=3;X±SE;* *. 差异极显著(P<0.01)Figure 3. mRNA expression of btseIF3g in different tissues of Penaeus monodon1, 3, 5, 7 and 9 denoted female eyestalk, brain, hepatopancreas, ovary and muscle, respectively; 2, 4, 6, 8 and 10 denoted male eyestalk, brain, hepatopancreas, testis and muscle, respectively; n=3;X±SE; * *. extremely significant difference (P < 0.01)
图 3 各组织中btseIF3g mRNA表达1、3、5、7和9分别为雌性个体的眼柄神经节、脑神经、肝胰腺、性腺和肌肉;2、4、6、8和10分别为雄性个体的眼柄神经节、脑神经、肝胰腺、性腺和肌肉;n=3;X±SE;* *. 差异极显著(P<0.01)Figure 3. mRNA expression of btseIF3g in different tissues of Penaeus monodon1, 3, 5, 7 and 9 denoted female eyestalk, brain, hepatopancreas, ovary and muscle, respectively; 2, 4, 6, 8 and 10 denoted male eyestalk, brain, hepatopancreas, testis and muscle, respectively; n=3;X±SE; * *. extremely significant difference (P < 0.01)3. 讨论
由于斑节对虾的全人工繁育技术取得成功,其性腺发育的分子机制研究成为当前的研究热点,国内外学者先后建立了斑节对虾性腺差减cDNA文库[7]、卵巢cDNA文库[8]和精巢cDNA文库[9],并筛选到一大批ESTs并克隆了其中少量与性腺发育相关的重要基因。采用差异筛选的方法如抑制消减法、差异显示等可获取差异表达信息,但这些方法很难直接获得全长cDNA。因此,通过克隆或文库筛选等方法获取全长cDNA序列并对其进行序列和功能分析是研究基因结构、基因功能和基因表达的重要手段之一。尽管先前有很多关于eIF3亚基cDNA片段的报道,如Eukaryotic translation initiation factor 3 subunit 2(GenBank Acc: FD664040)[10],X-linked eukaryotic translation initiation factor isoform 3[8]等,但由于eIF3亚基数量多、各亚基间变异大,这些cDNA片段不能真正代表基因全长,这不仅限制了通过GenBank查找同源序列,而且难以实现对这些片段的准确定位。
此研究经DD-RT PCR筛选得到斑节对虾性腺差异表达片段,通过与斑节对虾cDNA文库中所获得的片段进行拼接,最终获得斑节对虾翻译起始因子3亚基4(btseIF3g)的全长cDNA序列。推测btseIF3g的氨基酸序列用SMART在线预测发现其C端存在一个RNA识别基序(RRM),RRM是eIF3g所具有的典型的保守结构域,也存在于eIF3b[11]。RRM是迄今研究得最多的一类RNA结合结构域,在真核生物基因表达调控中发挥着重要的功能。酵母在RRM缺失情况下导致生长变慢,表明RRM对eIF3g的功能不是必须的,但对其功能发挥的最佳化则是必须的[12]。eIF3g与eIF4b的相互作用则要求RRM的存在[13]。此研究并没有发现btseIF3g中存在可信的(E值< 1.0E-04)锌指结构域(zinc finger)。关于eIF3g的功能报道较少,eIF3g被认为是斑马鱼早期发育所必需的315个基因之一[14],研究表明eIF3g能结合到18S rRNA和β-螺旋(β-globin)mRNA上发挥其功能[15]。此研究通过对btseIF3g的组织表达分析发现其250日龄的斑节对虾卵巢中呈极显著的高表达现象,初步推测该基因与斑节对虾性腺发育相关,而eIF3g在斑节对虾中的表达调控模式还有待于进一步探讨。
-
图 1 斑节对虾eIF3g cDNA序列及其推导的氨基酸序列
编码区用大写字母表示,非编码去用小写字母表示,氨基酸用单个大写字母表示,排在对应核苷酸序列下面,核苷酸阴影区分别为起始密码子和终止子;RNA识别模体用下划线标识
Figure 1. The cDNA sequence of eIF3g and deduced amino acid sequence of the black tiger shrimp
Coding region was shown in capital letters. Non-coding region was shown in small letters. The deduced amino acids were shown by single letter code of amino acids below the coding region. Initial codon and terminal codon were shaded. RBA was underlined.
图 3 各组织中btseIF3g mRNA表达
1、3、5、7和9分别为雌性个体的眼柄神经节、脑神经、肝胰腺、性腺和肌肉;2、4、6、8和10分别为雄性个体的眼柄神经节、脑神经、肝胰腺、性腺和肌肉;n=3;X±SE;* *. 差异极显著(P<0.01)
Figure 3. mRNA expression of btseIF3g in different tissues of Penaeus monodon
1, 3, 5, 7 and 9 denoted female eyestalk, brain, hepatopancreas, ovary and muscle, respectively; 2, 4, 6, 8 and 10 denoted male eyestalk, brain, hepatopancreas, testis and muscle, respectively; n=3;X±SE; * *. extremely significant difference (P < 0.01)
表 1 此研究所用引物信息
Table 1 Primers information in this study
引物名称 primer names 引物序列(5′to 3′) primer sequences 作用 function H-T11G AAGCTTTTTTTTTTTTTG 3′端锚定引物 H-AP8 AAGCTTTTACCGC 5′端随机引物 BtseIF3g-F GAGCAACAAAGATGAGGAGCAGAATG 荧光定量 BtseIF3g-R TCCCAGTAGGGATGACCGCAGA β-actin-F TACTCCTACACTCATAAACCAACGA 扩增内参 β-actin-R TTTCGGAATTTTATTATCAACTGTA 表 2 NCBI上已公布的部分eIF3信息
Table 2 eIF3 information published in NCBI
序列名称
nameGenBank登录号
accession number物种
specieseIF3各亚基类型
eIF3 subunitP.monodon* FJ829364 斑节对虾 Penaeus monodon subunit G H.sapiens A/10 NP_003741.1 人 Homo sapiens subunit 10 theta, 150/170 kDa P.troglodytes A/10 XP_001153575.1 黑猩猩 Pan troglodytes subunit 10 theta, 150/170 kDa isoform 1 C.familiaris A/10 XP_852131.1 狗 Canis familiaris subunit 10 theta, isoform 2 B.taurus A/10 XP_884395.2 西藏黄牛 Bos taurus subunit A isoform 4 M.musculus A/10 NP_034253.2 小鼠 Mus musculus subunit 10 (theta) R.norvegicus A/10 XP_238649.4 褐家鼠 Rattus norvegicus similar to subunit 10 theta, 150/170 kDa D.rerio A/10 NP_956114.2 斑马鱼 Danio rerio subunit 10 (theta) D.melanogaster A/10 NP_649470.2 黑腹果蝇 Drosophila melanogaster S10, isoform A S.pombe A/10 NP_596379.1 粟酒裂殖酵母 Schizosaccharomyces pombe eIF3a A.thaliana A/10 NP_192881.1 拟南芥 Arabidopsis thaliana eIF3A H.sapiens B/9 NP_001032360.1 人 H.sapiens subunit 9 eta, 116 kDa C.familiaris B/9 XP_536894.2 狗 C.familiaris similar to subunit 9 M.musculus B/9 NP_598677.1 小鼠 M.musculus subunit 9 D.rerio B/9 XP_001335480.1 斑马鱼 D.rerio subunit B D.melanogaster B/9 NP_611228.1 黑腹果蝇 D.melanogaster S9, isoform B C.elegans B/9 NP_001022469.1 秀丽隐杆线虫 Caenorhabditis elegans eif-3.B A.thaliana B/9 NP_001031954.1 拟南芥 A.thaliana 3B1 H.sapiens C/8 NP_003743.1 人 H.sapiens subunit C C.familiaris C/8 XP_536923.2 狗 C.familiaris similar to subunit 8, 110 kDa B.taurus C/8 NP_001029790.1 西藏黄牛 B.taurus subunit C M.musculus C/8 NP_666312.1 小鼠 M.musculus subunit C R.norvegicus C/8 XP_215080.4 褐家鼠 R.norvegicus similar to subunit 8 D.rerio C/8 NP_998628.1 斑马鱼 D.rerio subunit C D.melanogaster C/8 NP_611242.1 黑腹果蝇 D.melanogaster S8 C.elegans C/8 NP_492638.1 秀丽隐杆线虫 C.elegans eif-3.C S.pombe C/8 NP_593828.2 粟酒裂殖酵母 S.pombe eIF3c A.thaliana C/8 NP_191174.1 拟南芥 A.thaliana EIF3C H.sapiens D/7 NP_003744.1 人 H.sapiens subunit D P.troglodytes D/7 XP_001158696.1 黑猩猩 P.troglodytes subunit 7 isoform 2 C.familiaris D/7 XP_861890.1 狗 C.familiaris similar to subunit 7 isoform 3 B.taurus D/7 NP_001029656.1 西藏黄牛 B.taurus subunit D M.musculus D/7 NP_061219.2 小鼠 M.musculus subunit 7 (zeta) R.norvegicus D/7 NP_001004283.1 褐家鼠 R.norvegicus subunit D G.gallus D/7 XP_001234643.1 红原鸡 Gallus gallus similar to subunit 7 zeta, 66/67 kDa D.rerio D/7 NP_956310.1 斑马鱼 D.rerio subunit D D.melanogaster D/7 NP_524463.2 黑腹果蝇 D.melanogaster p66 subunit C.elegans D/7 NP_498984.1 秀丽隐杆线虫 C.elegans eif-3.D H.sapiens E/6 NP_001559.1 人 H.sapiens subunit 6 48 kDa C.familiaris E/6 XP_532304.2 狗 C.familiaris similar to subunit 6 isoform 2 B.taurus E/6 NP_001029775.1 西藏黄牛 B.taurus subunit 6 48 kDa M.musculus E/6 NP_032414.1 小鼠 M.musculus subunit 6 R.norvegicus E/6 NP_001011990.1 褐家鼠 R.norvegicus subunit E G.gallus E/6 NP_001006349.1 红原鸡 G.gallus subunit E D.rerio E/6 NP_957133.1 斑马鱼 D.rerio subunit E, a C.elegans E/6 NP_492785.1 秀丽隐杆线虫 C.elegans eif-3.E A.thaliana E/6 NP_567047.1 拟南芥 A.thaliana eif3E H.sapiens F/5 NP_003745.1 人 H.sapiens subunit 5 epsilon, 47 kDa B.taurus F/5 XP_591540.1 西藏黄牛 B.taurus similar to 47 kDa subunit isoform 1 M.musculus F/5 NP_079620.1 小鼠 M.musculus subunit F R.norvegicus F/5 XP_215037.3 褐家鼠 R.norvegicus similar to subunit 5 epsilon, 47 kDa G.gallus F/5 XP_421624.1 红原鸡 G.gallus similar to 47 kDa subunit H.sapiens G/4 NP_003746.2 人 H.sapiens subunit 4 delta, 44 kDa P.troglodytes G/4 XP_001162705.1 黑猩猩 P.troglodytes subunit 4 delta, 44 kDa isoform 3 C.familiaris G/4 XP_868239.1 狗 C.familiaris similar to subunit 4(eIF3 p44) B.taurus G/4 XP_001251526.1 西藏黄牛 B.taurus subunit 4 delta M.musculus G/4 NP_058572.2 小鼠 M.musculus subunit G R.norvegicus G/4 NP_001013113.1 褐家鼠 R.norvegicus subunit G D.rerio G/4 NP_957293.1 斑马鱼 D.rerio subunit G S.pombe G/4 NP_595727.1 粟酒裂殖酵母 S.pombe eIF3g A.thaliana G/4 NP_187747.1 拟南芥 A.thaliana 3G1; RNA binding/translation initiation factor H.sapiens H/3 NP_003747.1 人 H.sapiens subunit 3 gamma, 40 kDa P.troglodytes H/3 XP_519914.2 黑猩猩 P.troglodytes subunit 3 gamma, 40 kDa isoform 6 C.familiaris H/3 XP_532315.1 狗 C.familiaris similar to subunit 3 gamma, 40 kDa isoform 1 B.taurus H/3 NP_001020493.1 西藏黄牛 B.taurus subunit 3 gamma, 40 kDa M.musculus H/3 NP_542366.1 小鼠 M.musculus subunit H R.norvegicus H/3 NP_942046.1 褐家鼠 R.norvegicus subunit H G.gallus H/3 NP_001026122.1 红原鸡 G.gallus subunit H D.rerio H/3 NP_001003763.1 斑马鱼 D.rerio subunit H D.melanogaster H/3 NP_524834.2 黑腹果蝇 D.melanogaster p40 subunit, isoform A A.thaliana H/3 NP_563880.1 拟南芥 A.thaliana subunit H1 H.sapiens I/2 NP_003748.1 人 H.sapiens subunit 2 beta, 36 kDa C.familiaris I/2 XP_535328.1 狗 C.familiaris similar to subunit 2 B.taurus I/2 NP_001015628.1 西藏黄牛 B.taurus subunit 2 beta, 36 kDa M.musculus I/2 NP_061269.1 小鼠 M.musculus subunit I D.rerio I/2 NP_998155.1 斑马鱼 D.rerio subunit 2 beta C.elegans I/2 NP_490988.2 秀丽隐杆线虫 C.elegans eif-3.I H.sapiens J/1 NP_003749.2 人 H.sapiens subunit 1 alpha, 35 kDa C.familiaris J/1 XP_544656.2 狗 C.familiaris similar to subunit 1(eif-3 alpha) B.taurus J/1 NP_001069611.1 西藏黄牛 B.taurus subunit J M.musculus J/1 NP_001069611.1 小鼠 M.musculus subunit J R.norvegicus J/1 XP_001080279.1 褐家鼠 R.norvegicus similar to subunit 1 (eif-3 alpha) G.gallus J/1 NP_001012789.1 红原鸡 G.gallus subunit J H.sapiens K/12 NP_037366.1 人 H.sapiens subunit 12 P.troglodytes K/12 XP_512639.2 黑猩猩 P.troglodytes subunit 12 C.familiaris K/12 XP_533679.1 狗 C.familiaris similar to subunit 12 isoform 1 B.taurus K/12 NP_001029661.1 西藏黄牛 B.taurus subunit K M.musculus K/12 NP_082935.1 小鼠 M.musculus subunit K R.norvegicus K/12 XP_214886.1 褐家鼠 R.norvegicus similar to subunit 12 C.elegans K/12 NP_506241.1 秀丽隐杆线虫 C.elegans eif-3.K H.sapiens L NP_057175 人 H.sapiens subunit 6 interacting protein C.familiaris L XP_531741 狗 C.familiaris similar to subunit 6 interacting protein isoform 1 D.rerio L NP_998293 斑马鱼 D.rerio subunit 6 interacting protein C.intestinalis L XP_002120603 玻璃海鞘 Ciona intestinalis similar to subunit 6 interacting protein H.sapiens M NP_006351.2 人 H.sapiens subunit M M.musculus M NP_663355.1 小鼠 M.musculus subunit M D.rerio M NP_001019906.1 斑马鱼 D.rerio subunit M -
[1] ASANO K, VORNLOCHER H P, RICHTER-COOK N J, et al. Structure of cDNAs encoding human eukaryotic initiation factor 3 subunits. Possible roles in RNA binding and macromolecular assembly[J]. J Biol Chem, 1997, 272(43): 27 042-27 052. doi: 10.1074/jbc.272.43.27042
[2] MASUTANI M, SONENBERG N, YOKOYAMA S, et al. Reconstitution reveals the functional core of mammalian eIF3[J]. EMBO J, 2007, 26(14): 3 373-3 383. doi: 10.1038/sj.emboj.7601765
[3] 李竑, 沈思师, 许智宏, 等. 水稻eIF3大亚基(eIF3a)编码基因的克隆及其表达模式分析[J]. 实验生物学报, 2003, 36(1): 54-60. doi: 10.3321/j.issn:1673-520X.2003.01.010 [4] 黄建华, 马之明, 周发林, 等. 南海北部野生斑节对虾卵巢解剖结构及组织学的研究[J]. 南方水产, 2005, 1(3): 49-52. doi: 10.3969/j.issn.2095-0780.2005.03.008 [5] 黄建华, 周发林, 马之明, 等. 南海北部斑节对虾卵巢发育的形态及组织学观察[J]. 热带海洋学报, 2006, 25(3): 47-52. doi: 10.3969/j.issn.1009-5470.2006.03.009 [6] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) [J]. Methods, 2001, 25(4): 402-408. doi: 10.1006/meth.2001.1262
[7] LEELATANAWIT R, KLINBUNGA S, PUANGLARP N, et al. Isolation and characterization of differentially expressed genes in ovaries and testes of the giant tiger shrimp (Penaeus monodon)[J]. Mar Biotechnol, 2004, 6: 506-510.
[8] PREECHAPHOL R, LEELATANAWIT R, SITTIKANKEAW K, et al. Expressed sequence tag analysis for identification and characterization of sex-related genes in the giant tiger shrimp Penaeus monodon[J]. J Biochem Mol, 2007, 40(4): 501-510. https://www.sciencedirect.com/science/article/pii/S1744117X09000471
[9] LEELATANAWIT R, SITTIKANKEAWB K, YOCAWIBUN P, et al. Identification, characterization and expression of sex-related genes in testes of the giant tiger shrimp Penaeus monodon[J]. Comp Biochem Phys B, 2009, 152(1): 66-76. doi: 10.1016/j.cbpa.2008.09.004
[10] LEELATANAWIT R, KLINBUNGA S, AOKI T, et al. Suppression subtractive hybridization (SSH) for isolation and characterization of genes related to testicular development in the giant tiger shrimp Penaeus monodon[J]. BMB Rep, 2008, 41(11): 796-802. doi: 10.5483/BMBRep.2008.41.11.796
[11] BURKS E A, BEZERRA P P, LE H, et al. Plant initiation factor 3 subunit composition resembles mammalian initiation factor 3 and has a novel subunit[J]. J Biol Chem, 2001, 276(3): 2 122-2 131. doi: 10.1074/jbc.M007236200
[12] HANACHI P, HERSHEY J W B, VORNLOCHER H P. Characterization of the p33 subunit of eukaryotic translation initiation factor-3 from Saccharomyces cerevisiae[J]. J Biol Chem, 1999, 274(13): 8 546-8 553. doi: 10.1074/jbc.274.13.8546
[13] VORNLOCHER H P, HANACHI P, RIBEIRO S, et al. A 110-kilodalton subunit of translation initiation factor eIF3 and an associated 135-kilodalton protein are encoded by the Saccharomyces cerevisiae TIF32 and TIF31 genes[J] J Biol Chem, 1999, 274(24): 16 802-16 812. doi: 10.1074/jbc.274.24.16802
[14] AMSTERDAM A, NISSEN R M, SUN Z, et al. Identification of 315 genes essential for early zebrafish development[J]. Proc Natl Acad Sci USA, 2004, 101(35): 12 792-12 797. doi: 10.1073/pnas.0403929101
[15] BLOCK K L, VORNLOCHER H P, HERSHEY J W B. Characterization of cDNAs encoding the p44 and p35 subunits of human translation initiation factor eIF3[J]. J Biol Chem, 1998, 273(48): 31 901-31 908. doi: 10.1074/jbc.273.48.31901



 下载:
下载:


 粤公网安备 44010502001741号
粤公网安备 44010502001741号
