Preliminary investigation of fish diversity in middle and lower reaches of Pearl River based on environmental DNA technology
-
摘要:
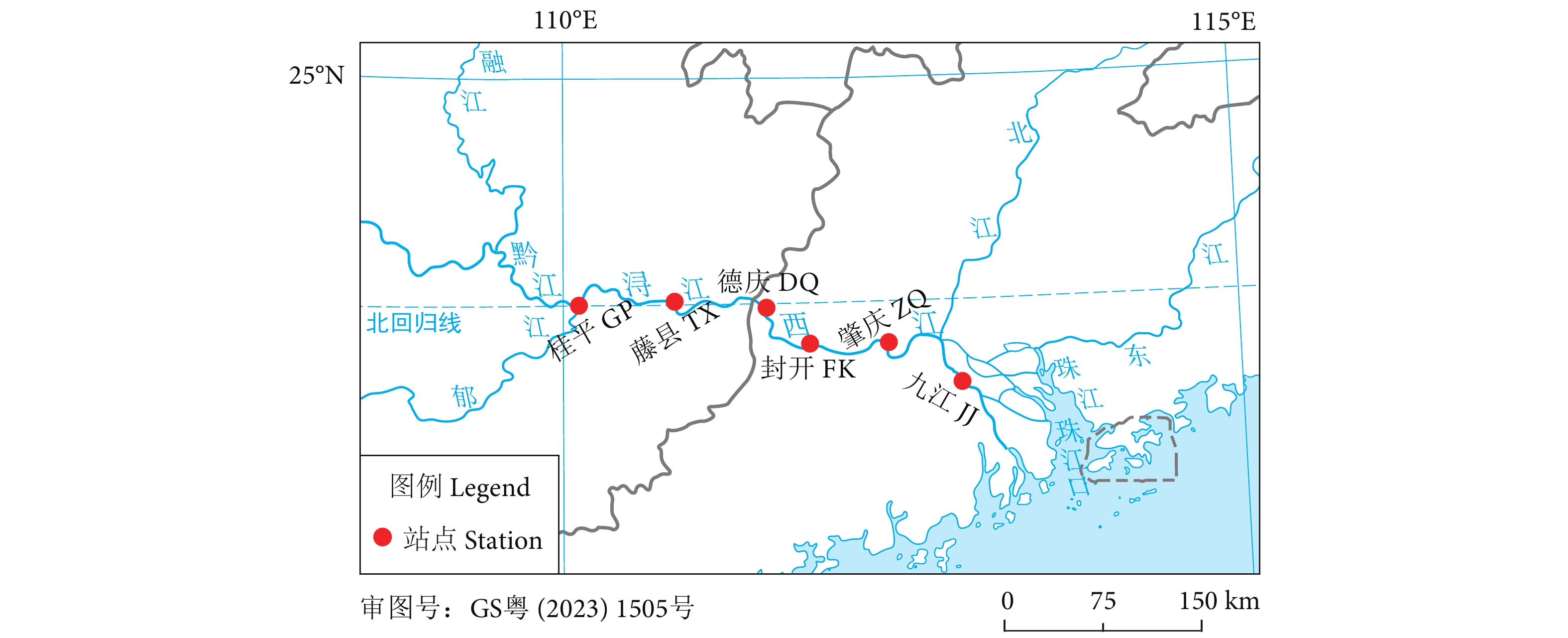
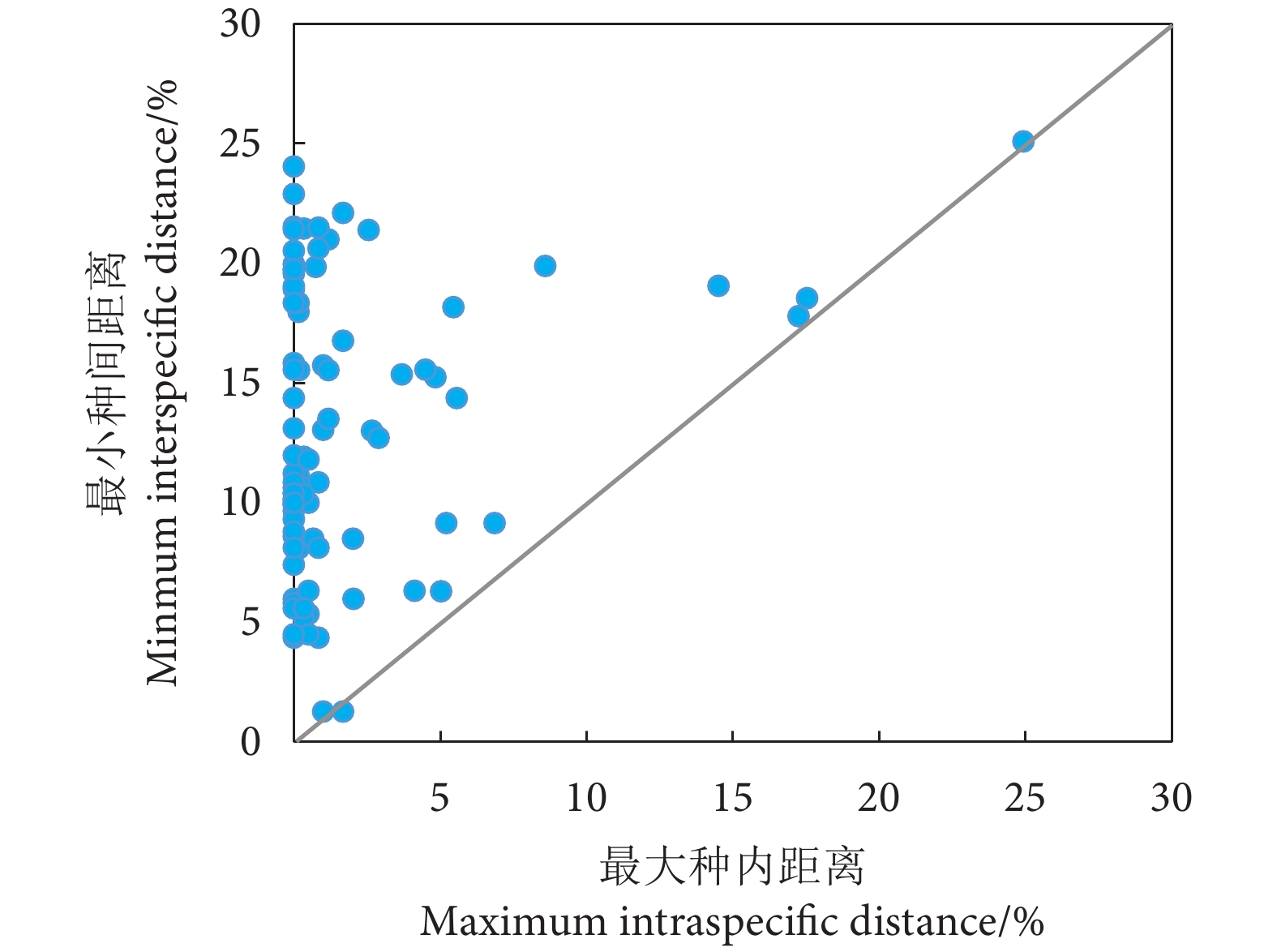
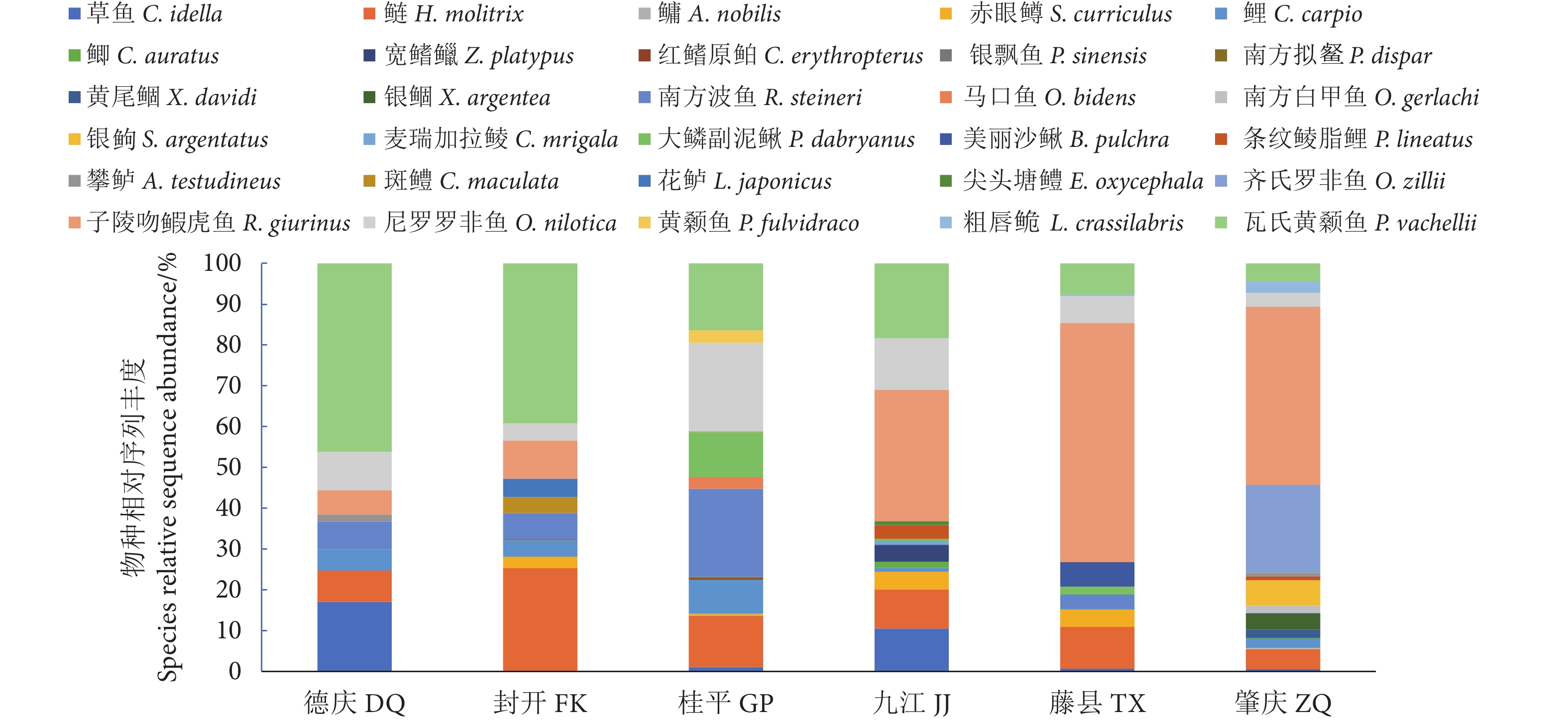
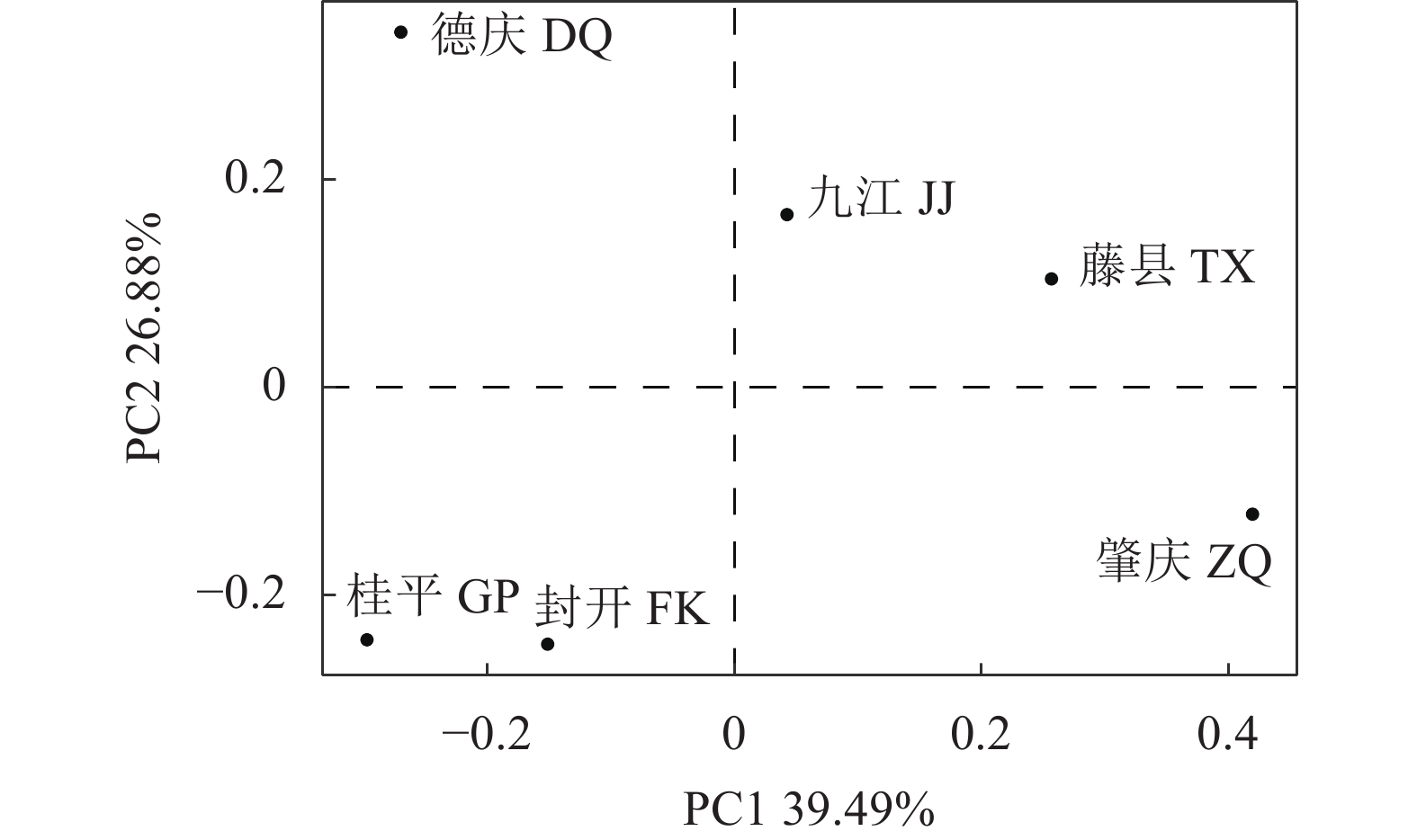
通过环境DNA技术 (Environmental DNA, eDNA) 检测珠江中下游鱼类生物多样性,探索珠江中下鱼类多样性监测和保护的新途径。2023年2月在珠江中下游设置了桂平、藤县、封开、德庆、肇庆和九江共6个采样点,通过水样采集及过滤、eDNA提取、遗传标记扩增及测序和数据库比对分析等流程检测鱼类多样性。结果表明,6个采样点共检测出30种鱼类,隶属于4目10科27属,其中土著鱼类26种,外来种4种。较已有传统调查数据新检出2种鱼类:美丽沙鳅 (Botia pulchra) 和齐氏罗非鱼 (Oceochromis zillii)。鱼类优势种为子陵吻鰕虎鱼 (Rhinogobius giurinus)、瓦氏黄颡鱼 (Pelteobagrus vachellii)、鲢 (Hypophthalmichthys molitrix)、尼罗罗非鱼 (O. nilotica)、齐氏罗非鱼、南方波鱼 (Rasbora steineri) 和鲤 (Cyprinus carpio)。根据Shannon指数和Simpson指数显示,eDNA检测九江和桂平站点的鱼类多样性最高,藤县的最低。作为一种新的检测方法,eDNA技术可用于快速检测珠江中下游鱼类的多样性及分布,在实际应用中可将eDNA技术与传统的监测方法相结合,以提供更全面的鱼类生物多样性数据信息。
-
关键词:
- 环境DNA (eDNA) /
- 鱼类多样性 /
- 珠江中下游
Abstract:To explore new ways for observing and protecting fish species diversity in the middle and lower reaches of the Pearl River, we used eDNA technology to detect fish species diversity in that river section. Six sampling localities, namely Guiping, Tengxian, Fengkai, Deqing, Zhaoqing and Jiujiang, were set up in February 2023. The fish diversity was detected by eDNA metabarcoding analysis that includes water collection, water filtration, eDNA extraction, genetic marker amplification, sequencing and bioinformatic analyses. The results show that 30 fish species had been detected in 6 sampling sites, belonging to 4 orders, 10 families and 27 genera, in which 26 were native species and 4 were non-native species. Compared with traditional survey reports, two new species (Botia pulchra and Oceochromis zillii) were detected. Rhinogobius giurinus, Pelteobagrus vachellii, Hypophthalmichthys molitrix, O. nilotica, O. zillii, Rasbora steineri and Cyprinus carpio were found at each sampling site, indicating that these species are the dominant species in the survey area. According to Shannon index and Simpson index, the fish diversity was highest in Jiujiang and Guiping, while lowest in Tengxian. As a new detection method, eDNA technology can efficiently investigate the fish biodiversity and distribution in the middle and lower reaches of the Pearl River. To sum up, eDNA technology can be combined with traditional fish resources monitoring methods to provide more comprehensive data information on fish species diversity.
-
三疣梭子蟹 (Portunus trituberculatus),俗称梭子蟹,属于甲壳纲、十足目、梭子蟹科、梭子蟹属,其肉质鲜美、营养丰富,广受消费者青睐,是中国沿海重要的经济蟹类[1]。梭子蟹生长速度快、养殖成本低、经济效益高,已成为中国沿海地区的重要养殖品种。由于雌、雄梭子蟹的营养成分不同,在市场销售前需要对其性别进行分类。目前,梭子蟹的性别分类主要靠人眼识别和手工挑选,效率低下。因此,开发一种计算机辅助自动分类系统来区分梭子蟹的性别非常必要。
早期的分类任务主要是手工设计提取特征,包括传统机器学习[2-4]和利用上下文信息[5-6],但是该类方法的表达能力较弱,所以分类模型的泛化能力不强。例如,Lecun等[7]提出的一种多层人工神经网络LeNet是为手写数字分类而设计的卷积神经网络 (Convolutional neural network, CNN)。2006年,深度学习 (Deep learning) [8]的概念被提出,在深度学习技术的推动下,图像分类及识别的相关研究迅速发展[9-13]。
基于深度学习的图像分类中,Krizhevsky等[9]构建了一种较深层数的网络AlexNet,并首次引入了Relu激活函数,同时在全连接层中使用Dropout,解决了模型的过拟合问题。牛津大学的几何视觉组 (Visual geometry group[10])设计的VGG模型在网络结构上并没有太大的创新,但是通过实验对比发现,增加网络的层数确实能够在一定程度上提高网络的训练效果。此外,VGG模型始终使用的是非常小的卷积核,通过串联很多小的卷积核后,其感受野和一个大卷积核相同,因此能很大程度地减少模型训练所需的参数。He等[12]开发出一种具有深度残差结构的卷积神经网络ResNet,很好地解决了网络深度达到一定层数时,模型的性能逐渐会趋向于饱和的问题,以及在网络达到某一深层时会使模型的性能急剧下降的问题。
在过去的几年里,图像的成熟分类技术主要得益于两个关键因素:一个是卷积神经网络,另一个是大量的可用图像数据集,如CIFAR数据集[14]和ImageNet 数据集[15]。当前,一些研究者已经将图像分类方法应用到实际场景中。例如,在医学图像分类领域中,杜丽君等[16]提出了一种基于注意力机制和多任务学习的阿尔兹海默症分类方法。该方法是一种不需要人工提取特征的网络,通过引入注意力机制,能够在不丢失重要特征信息的前提下,将分类任务所需要的关注重点放在目标区域中;随后通过不同的全连接层来实现多任务学习,从而提高了网络的泛化能力。实验结果表明,该方法能够对阿尔兹海默症进行准确分类。在遥感图像分类领域中,王宁等[17]在随机森林、支持向量机和BP神经网络的基础上设计出一种集成分类模型,并将其应用于水产养殖的水体资源遥感动态监测任务中。该模型很好地弥补了单个分类模型分类精度较低以及鲁棒性较差等缺点,在很大程度上避免了山体与建筑阴影等外在因素对水体特征提取的干扰。最终实验结果显示该分类模型拥有较高的分类精度,表明该模型能够成为水体资源遥感动态监测的分析工具之一。此外,朱明等[18]基于轻量级神经网络构建出一种鲈鱼捕食状态分类网络,满足了现代智能水产养殖的自动投喂需求。
然而,尚未见图像分类的相关技术应用于梭子蟹性别识别的研究报道。为此,本文提出一种应用于梭子蟹性别识别任务的分类方法:首先构建一个用于梭子蟹性别分类的数据集PGCD;然后搭建一种多组卷积神经网络 (Multi-group convolutional neural network, MGCNN),并使用注意力机制更专注地找出输入数据的有用信息;最后通过一系列的调整参数工作提高MGCNN的分类精度。
1. 材料与方法
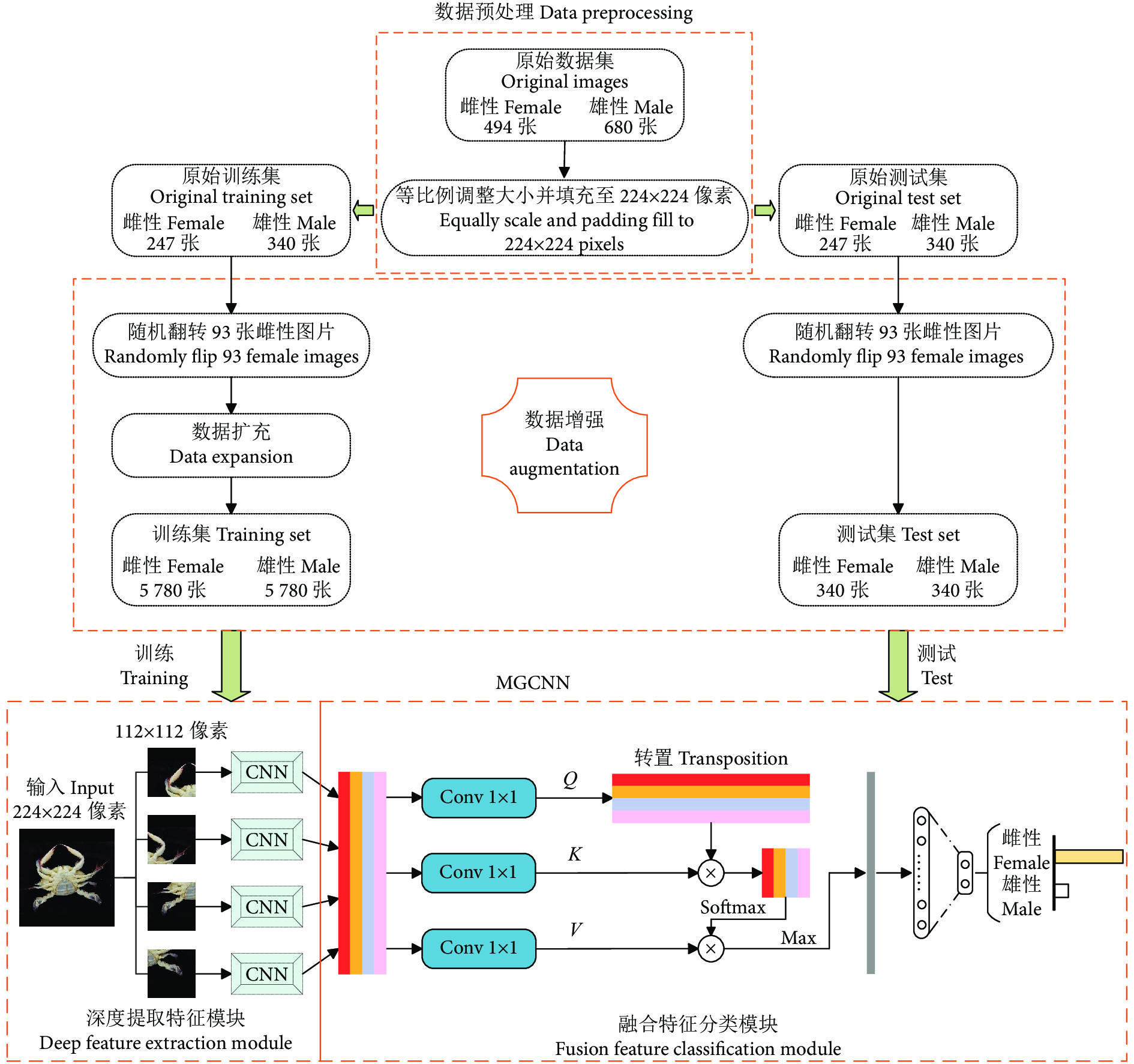
图1展示了本文所提方法的整体框架,包括数据预处理、数据增强以及MGCNN。 其中,MGCNN包含了深度提取特征模块和融合特征分类模块。数据预处理主要是将采集到的梭子蟹图像进行降低像素处理,从而解决后续处理速度过慢的问题;数据增强主要是对经过预处理后的梭子蟹图像进行数据扩充,解决了少量样本可能给网络带来的过度拟合问题,或者样本不均衡导致模型预测时对多数类的侧重问题;深度提取特征模块使用几组成熟的CNN 来提取图像的视觉特征,通过实验对比,最终的CNN本文选取ResNet50;融合特征分类模块主要是先将CNN提取到的特征进行信息融合,然后利用注意力机制代替传统池化层,在降低融合特征图维度的同时,去除了特征图的冗余信息,保留特征图的重要信息,最后经过输出分类层获取分类结果。
1.1 数据预处理
在公开数据库中,并没有梭子蟹性别分类的数据集,因此首先需要构建相关的数据集。本文的原始梭子蟹数据集主要由课题组在舟山水产品加工公司采集,采集所用设备的配置为10 800万像素的主摄+1 300万像素的超广角镜头+500万像素的长焦微距镜头。采集到的原始数据集共有1 174 张图像,其中雌、雄性图像各494和680 张,图像像素约为3 024×4 032。图2为部分采集的梭子蟹样品。
由于所采集的图像像素较大,导致后续操作中处理速度较慢,因此需要对图像进行像素降低处理,调整后的图像大小统一为224×224像素。传统的降低像素操作通常是利用opencv库的resize( ), 然而这种操作对于尺寸非N×N大小转化为N×N大小的图像来说,会改变图像原有的特征,影响网络的最终分类精度[19-21]。因此,本文采用一种等比例调整图像大小的方法,即在不改变宽高比的情况下进行图片调整,并填充至实验所需要的224×224像素的图像。resize ( ) 和等比例调整大小并填充的图像对比如图3所示。
1.2 数据增强
经过预处理后,将数据集随机分成原始训练集和原始测试集,均包含587 张梭子蟹图像 (雌性 247 张,雄性 340 张)。为了解决少量样本可能给网络带来的过度拟合问题,或者样本不均衡导致模型预测时对多数类的侧重问题,需要对原始数据集进行数据增强,以扩充出更多数据来提高数据集的数量和质量,提高网络模型的学习效果[22-24]。数据增强的过程如下所述:
(I) 随机翻转:为了解决样本不均衡问题,本文随机从原始训练集和原始测试集中各选取93 张雌性图像,进行随机翻转,从而使得原始训练集和原始测试集中雌性和雄性图像均有340 张。随机翻转是指从3种翻转方式中 (水平翻转、垂直翻转和水平-垂直翻转) 随机选取一种进行翻转。
(II) 随机旋转:对 (I) 中的原始训练集图像进行随机角度旋转,旋转角度在0°~90°、90°~180°、180°~270°和270°~360° 4个范围内进行随机选取。
(III) 随机平移:对 (I) 中的原始训练集图像进行随机像素平移,像素平移在−20至20之间进行随机选取,包括8个方向。
(IV) 随机明亮度:对 (I) 中的原始训练集图像进行随机明亮度调整,调整系数在0.8~1.0和1.0~1.2两个范围内随机选取。
(V) 随机噪声:对 (I) 中的原始训练集图像随机添加高斯噪声和椒盐噪声,添加噪声的系数均介于0.009~0.011随机选取。
值得注意的是,除了(I) 对训练集和测试集进行数据增强,(II)—(V) 均仅对训练集进行数据增强。其中,(IV) 和 (V) 主要是针对模型识别过程中图像出现的明亮度变化和噪声影响。通过 (IV) 和 (V) 两种数据增强技术可以让模型在现实环境中具有更好的应对能力。
5种不同类型的数据增强技术示例如图4所示。因此,经过数据增强后最终的数据集共有12 312 张梭子蟹图像,将其命名为PGCD。其中,训练集和测试集分别各有11 560 张 (雌性 5 780 张,雄性 5 780 张) 和680张 (雌性 340 张,雄性 340 张) 图像。
1.3 深度提取特征模块
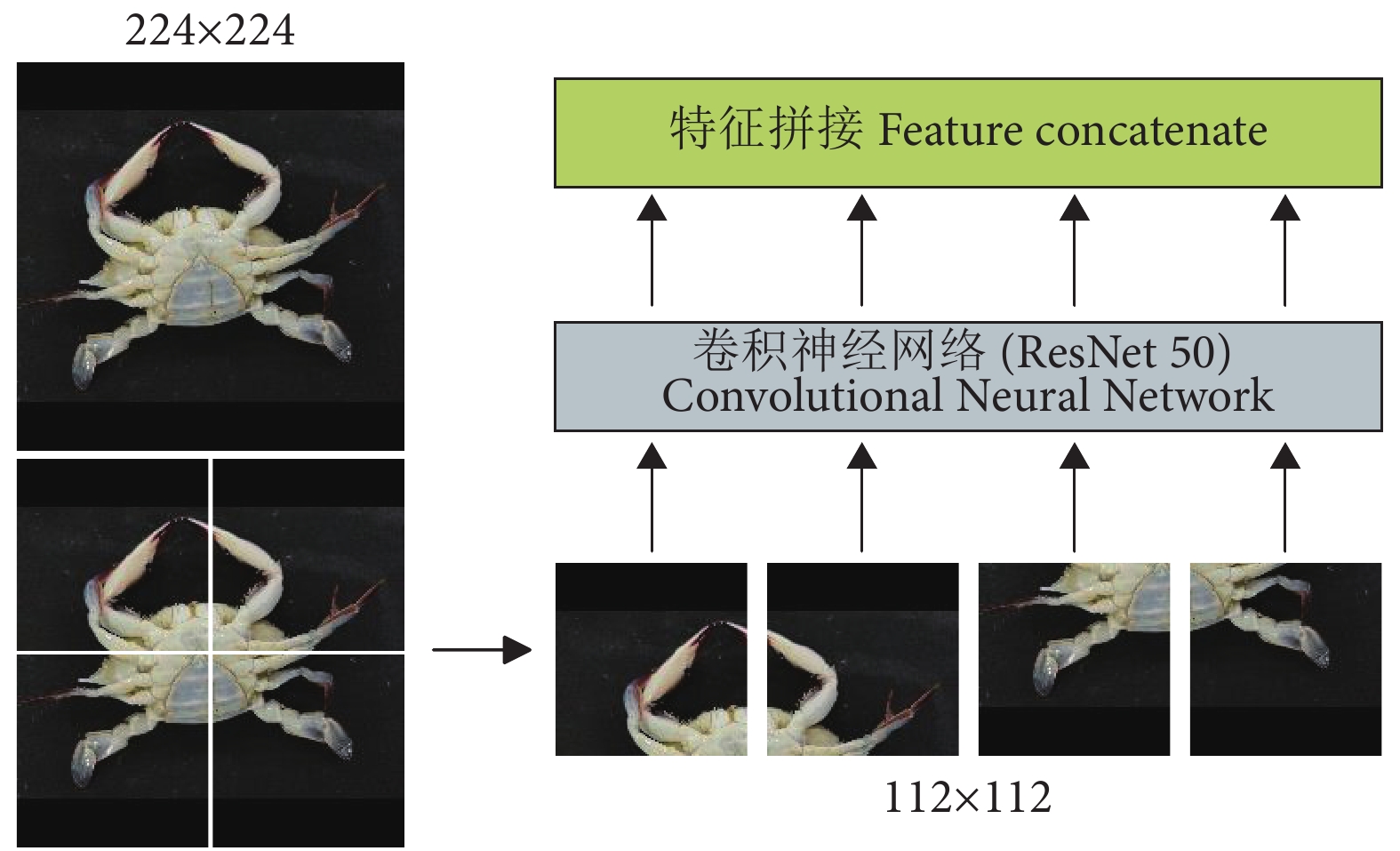
虽然Transformer[25]已经成为自然语言处理任务上的重要体系结构之一,但它在计算机视觉 (Computer Vision, CV) 领域的应用却非常有限。为了解决该问题,Dosovitskiy等[26]开发出一种Vision Transformer (ViT) 模型,可以直接应用于图像块序列 (Sequences of image patches),并且能够很好地执行图像分类任务。受这项工作的启发,本研究在提取特征之前设置了图像块来渲染每张图 (图5)。
具体操作是将输入图像切分成多个大小一致的图像块。在此假设输入图像的尺寸大小为N×N,需要切分的图像块数为n2,那么图像块的尺寸大小NP×NP用公式可表示为:
$$ {N_P} \times {N_P} = \frac{N}{n} \times \frac{N}{n} $$ (1) 式中:n为正整数。本文所提的MGCNN的输入图像尺寸为224×224 像素,图像块的个数n2=4,所以图像块的尺寸为112×112 像素。
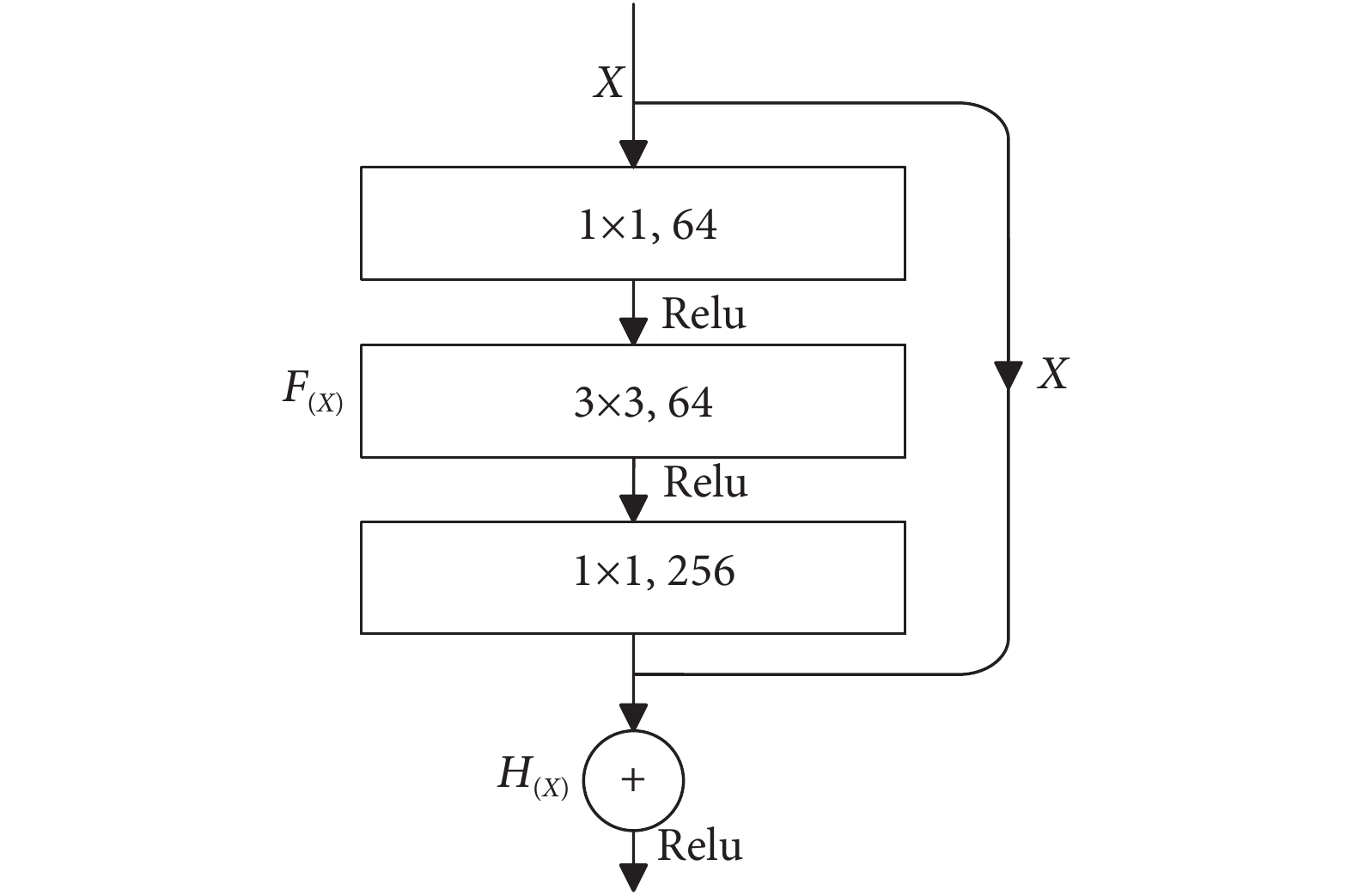
在4组图像块渲染图像之后,需要通过CNN来对每个图像块进行特征学习。为了减少特征提取过程的信息丢失,使得特征提取更有力,本文使用具有残差块的ResNet50作为深度提取特征模块中的主干CNN。ResNet50的残差块示意图见图6。
可以看出,残差块具有两条路径,一条是进行跳跃连接的输入特征X,另一条是经过3次卷积操作后得到的映射函数F(X),然后将这两条路径连接后就能得到残差模块的输出H(X),其过程可用公式 (2) 来表示:
$$ {H_{(X)}} = X + {F_{(X)}} $$ (2) 1.4 融合特征分类模块
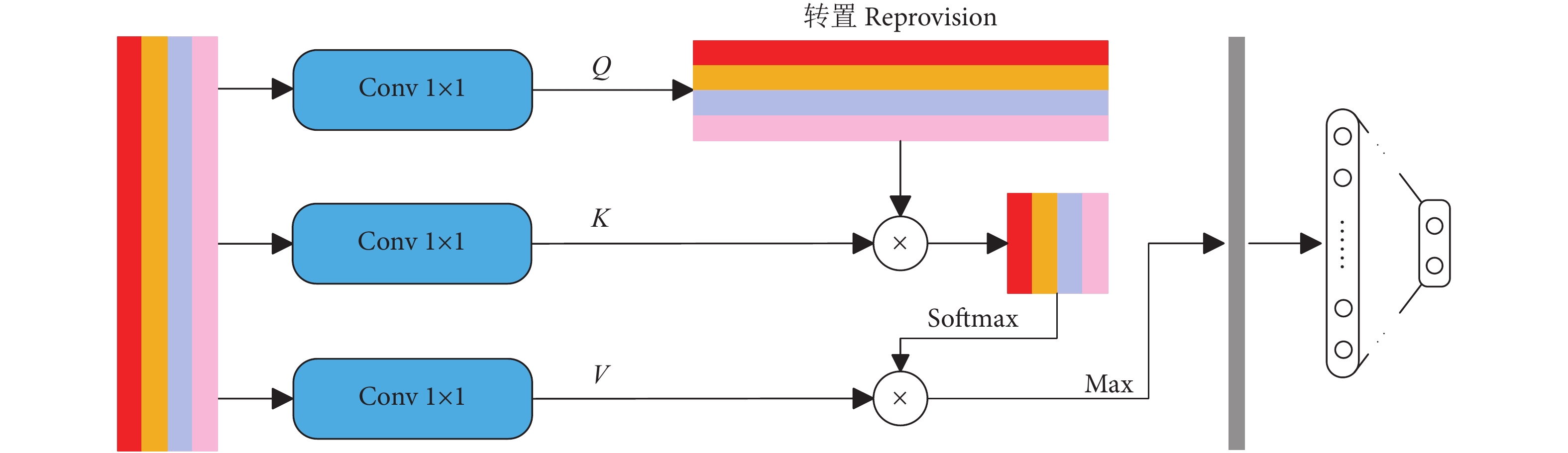
融合特征分类模块如图7所示。通过CNN学习不同图像块的视觉特征后,需要一个独特的全局描述符来表示图像。本文将不同图像块的视觉特征拼接 (Concatenate) 成一个全局特征图。最近一些研究表明,适当增加一些注意力机制可以更专注地找出输入数据的有用信息[27-29]。 为此,在分类之前本研究引入了一个注意力机制,来强调全局特征图中的细节重要性。首先,通过3个1×1的卷积层从全局特征图中获得Q、K、V 3个特征层;其次,将Q转置后与K做乘积运算形成一个新的特征图,再经过softmax函数后形成注意力权重;然后,将V与注意力权重做积运算,并使用最大池化对其结果进行降低维度处理;最后,通过全连接层获取待分类类别的概率分布。
2. 实验
2.1 实验设置
本实验均在同一环境配置的计算机上完成,训练集和测试集均来自于构建的PGCD数据集。用于实验的计算机配置为Windows10、NVidia GeForce GTX 1080 Ti GPU、16 GB 内存,实验基于Pytorch的方法[30]来实现,损失函数使用交叉熵损失 (Cross entropy loss) ,最大epoch值设置为100。通过实验对比,本研究提出方法的backbone model为ResNet50,学习率值设置为 0.000 1,batch size设置为32。此外,为了优化整体模型架构,本文使用 Adam[31]作为训练阶段优化器。
2.2 评估指标
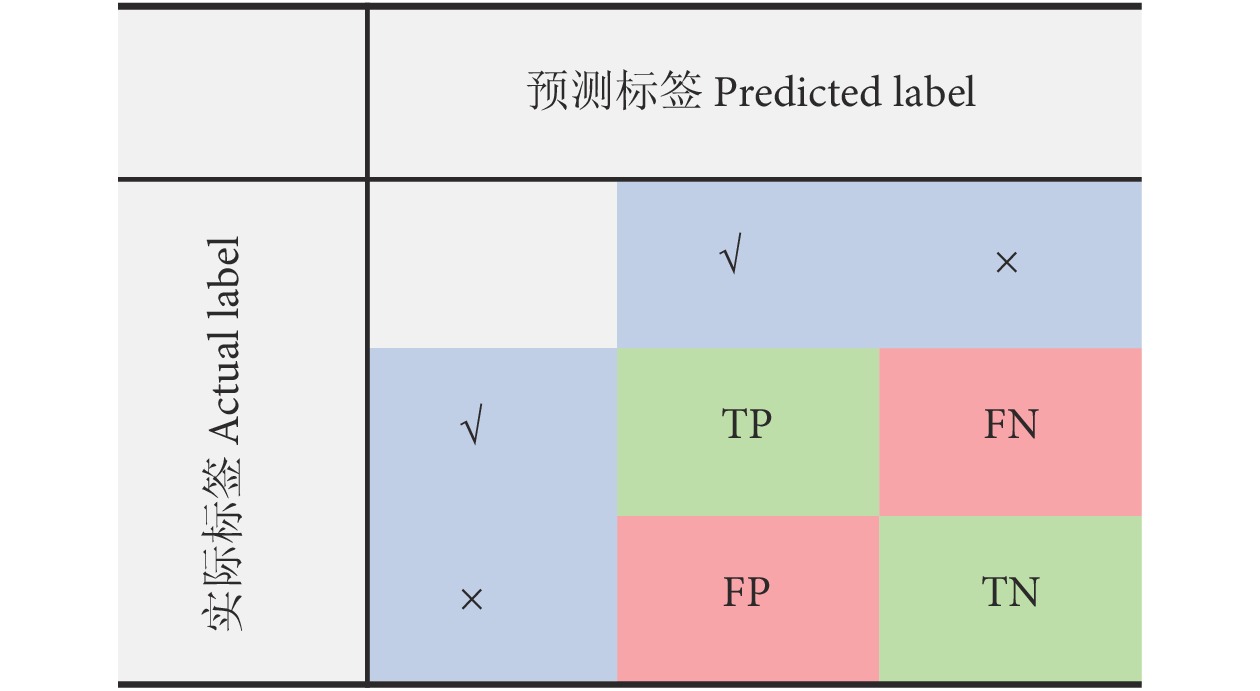
衡量模型分类性能的评估指标主要包括分类准确率 (又称分类精度)、召回率与查准率等[32-35]。本文主要采用这3个指标对所提方法进行评价。图8为混淆矩阵图。
为此,分类准确率可用公式 (3) 进行表征:
$$ A_{\rm{cc}} = \frac{{{{\rm{TP}}} + {{\rm{TN}}}}}{{{{\rm{TP}}} + {{\rm{FP}}} + {{\rm{TN}}} + {{\rm{FN}}}}} $$ (3) 召回率可用公式 (4) 进行表征:
$$ R = \frac{{{{\rm{TP}}}}}{{{{\rm{TP}}} + {{\rm{FN}}}}} $$ (4) 查准率可用公式 (5) 进行表征:
$$ P = \frac{{{{\rm{TP}}}}}{{{{\rm{TP}}} + {{\rm{FP}}}}} $$ (5) 式中:Acc代表分类准确率 (Accuracy);R代表召回率 (Recall);P代表查准率 (Precision)。
2.3 实验分析
2.3.1 不同骨干模型对MGCNN性能的影响
成熟的卷积神经网络能够很好地进行特征学习和特征提取[36-37]。为此,本文主要选取VGG模型[10]和ResNet模型[12]作为MGCNN的深度提取特征模块的主干模型。不考虑DenseNet模型[23]最主要的原因是该模型属于较大的网络,需要训练更多的参数,并且可能由于过拟合导致分类精度提高不明显。而VGG和ResNet系列属于较小的网络,可以节省额外的计算开销以及训练时间。为了公平比较,在这里统一将学习率值设置为 0.001,批大小设置为32;此外,本文选取了 SGD[38]作为不同骨干模型的对比实验训练阶段的优化器。结果如表1所示,可以看出,在相同的学习率、批大小以及相同优化器的情况下,ResNet 系列普遍优于VGG系列。值得注意的是,ResNet152的精度未超过90%,再次验证了较大网络的过拟合现象会影响分类精度。由于ResNet50以92.79%的分类精度位居第一,因此本研究选取ResNet50作为MGCNN的骨干模型。
表 1 不同骨干模型对 MGCNN 性能的影响Table 1. Effects of different backbone models on MGCNN performance骨干模型
Backbone model准确率
Accuracy/%VGG VGG11 76.88 VGG13 82.59 VGG16 89.76 VGG19 86.09 ResNet ResNet18 90.44 ResNet34 91.15 ResNet50 92.79 ResNet101 92.21 ResNet152 88.24 2.3.2 不同优化器对MGCNN性能的影响
深度学习的过程中会产生一定的损失[39],所以应尽可能地减少损失来优化所提出的MGCNN模型,使其拥有更好的分类性能。事实上,深度神经网络的每一层都有各自的权重参数,这些权重参数决定着神经网络的输出。因此需要利用优化器 (Optimizer) 来降低损失,从而更新模型的可学习权重参数来优化网络模型。本研究以SGD[38]、AdaGrad[40]、RMSprop[41]、Adam[31]、Adamax[42]、ASGD[43] 6种优化器作为探讨优化器选取的对比实验。在这里学习率仍为 0.001,批大小为32,结果如表2所示。可以看出,在相同骨干模型的情况下,上述6种优化器中Adam最为突出,分类精度达到95.29%,均领先于其他优化器。因此,本文选取Adam作为MGCNN的优化器。
表 2 不同优化器对MGCNN性能的影响Table 2. Effects of different optimizers on MGCNN performance骨干模型
Backbone model优化器
Optimizer准确率
Accuracy/%ResNet50 SGD 92.79 AdaGrad 89.56 RMSprop 95.15 Adam 95.29 Adamax 93.82 ASGD 92.65 2.3.3 不同参数对MGCNN性能的影响
通过调整学习率以及批大小可以提高模型的分类性能[28,44-45],在MGCNN的骨干模型和优化器均确定的情况下 (ResNet50+Adam),本研究进一步探讨了不同参数对其分类性能的影响,以实现最佳精度。对于模型训练而言,学习率是控制权重更新的重要参数[46]。一方面,使用过大的学习率可能会忽略最优值的位置,导致模型不收敛;另一方面,使用过小的学习率容易出现过拟合,导致模型收敛缓慢。因此,首先应确定最优学习率。在本实验中,将最开始的学习率设置为0.000 1,批大小设置为32。本次实验遵循从较小的速率开始,并依次增加直到发现最佳学习率。当最佳学习率确定后,需要进行实验来确定最佳批大小,以优化网络训练的收敛速度和稳定性[45]。本研究依次将批大小设置为64、32和16来训练网络,最终的实验结果如表3所示。结果表明,当学习率和批大小分别为0.001 5和32时,MGCNN的分类效果最高,分类精度达到95.59%。因此,本文将最佳学习率设置为0.001 5,最佳批大小设置为32。
表 3 不同参数对 MGCNN 性能的影响Table 3. Effects of different parameters on MGCNN performance学习率
Learning rate批大小
Batch size准确率
Accuracy/%0.000 1 32 92.94 0.000 5 95.00 0.001 0 95.29 0.001 5 95.59 0.002 0 92.65 0.001 5 64 94.56 32 95.59 16 95.15 2.4 实验结果及可视化
为了评估所提方法MGCNN的分类性能,本文在已构建的PGCD数据集上进行了相关的分类任务实验,并将所提方法与其他先进的模型[9-10,12-13,46]进行比较,包括AlexNet、VGG16、ResNet152、InceptionV3和DenseNet121。这些模型之前均在用于图像分类的ImageNet数据集[9]上训练过,但并没有训练过PGCD。为了适应PGCD数据集,实验过程中将上述网络模型的输出分类层替换为具有两个类别 (雌性和雄性) 的输出分类层。
表4给出了MGCNN与一些先进方法的实验对比结果。可以看出,AlexNet显示了最差的性能;ResNet152和DenseNet121网络较深,召回率和查准率相对平衡,但分类精度未超过95%;InceptionV3分类精度达到95%,却因其查准率高导致召回率低;而本文所提的网络相比InceptionV3而言,分类精度有所提升,并且在查准率仅降低0.54%的情况下,将召回率提升1.76%。网络分类错误率最低,仅占4.41%,分类性能均领先于其他方法。
表 4 MGCNN 与先进方法的比较Table 4. Comparison between MGCNN and state-of-the-art methods方法
Method准确率
Accuracy/%召回率
Recall/%查准率
Precision/%错误率
Error/%AlexNet 54.71 59.36 67.93 45.29 VGG16 89.85 88.24 91.19 10.15 ResNet152 94.56 94.71 94.43 5.44 DenseNet121 94.41 94.41 94.41 5.59 InceptionV3 95.00 92.65 97.22 5.00 MGCNN
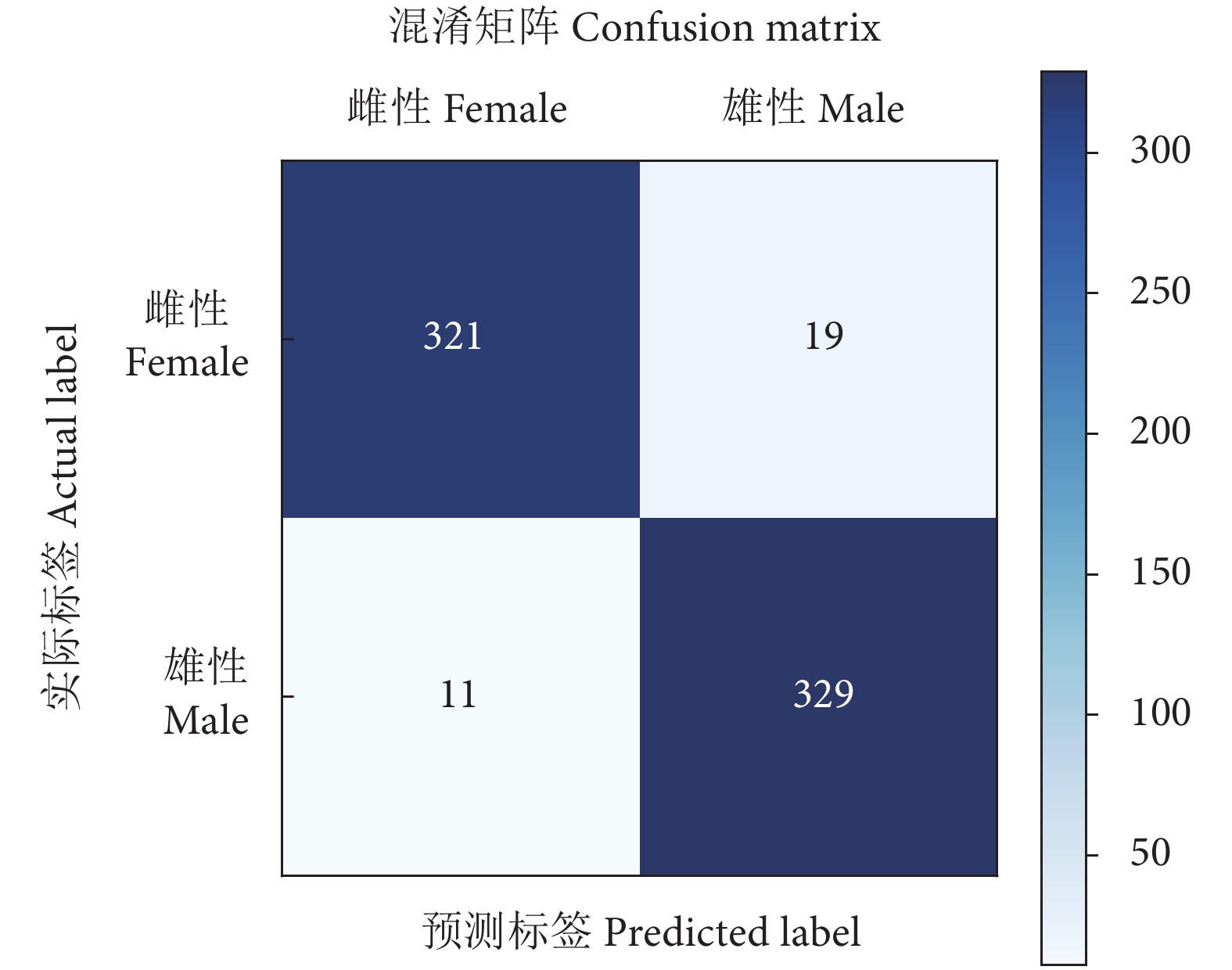
(本研究方法 Our method)95.59 94.41 96.68 4.41 为了更直观地显示所提方法的优越性,本文给出了PGCD测试集上梭子蟹性别分类的混淆矩阵 (Confusion matrix) ,如图9所示。图中主对角线显示的是识别正确的数目,副对角线显示的是识别错误的数目。可以看出,梭子蟹共有680 只,仅30 只被错误归类。其中,雌、雄蟹各有321、329 只被正确归类,仅11只雄蟹错归为雌性,19 只雌蟹错归为雄性。表明所提方法具有很好的分类效果。
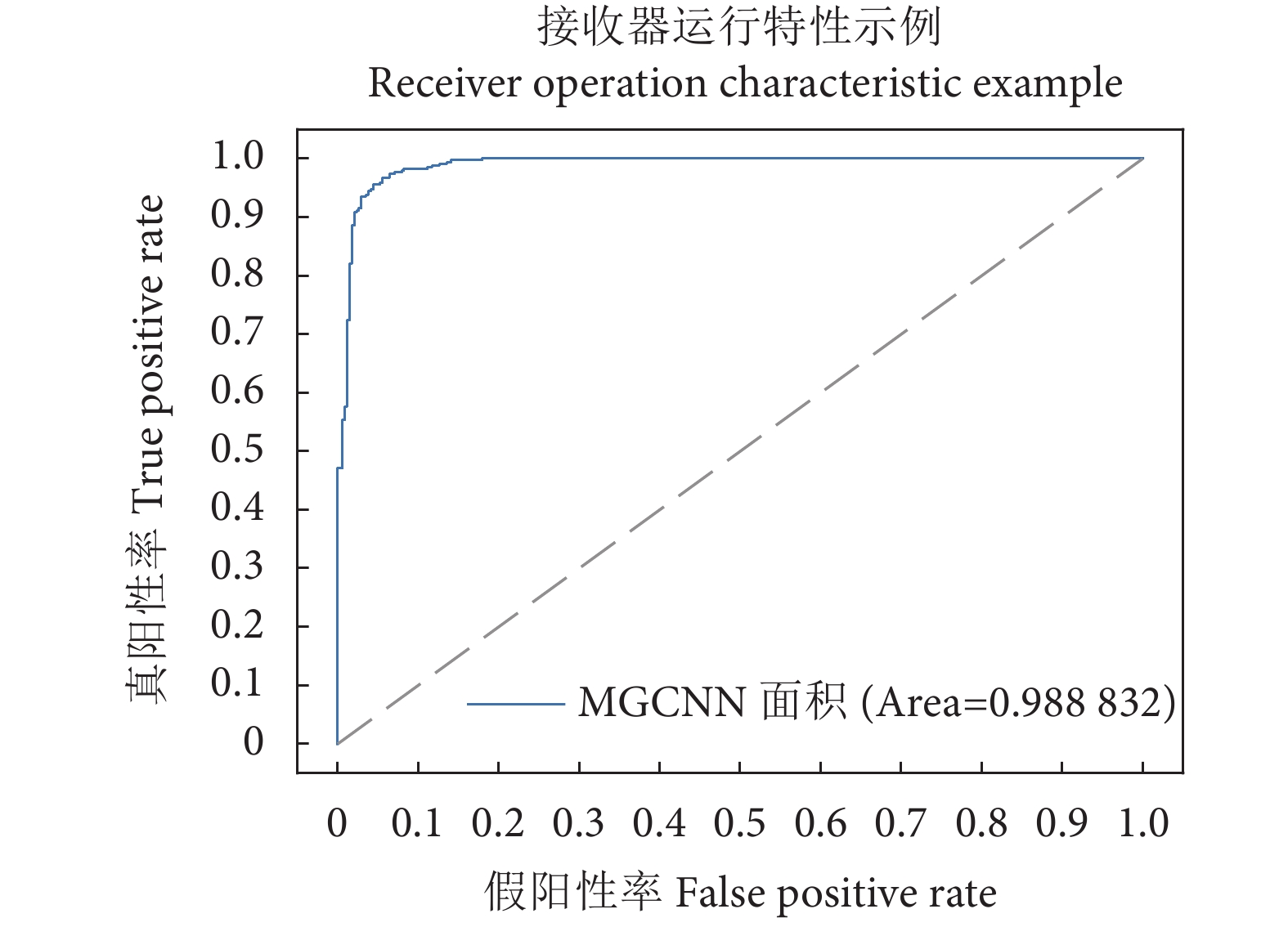
此外,受试者工作特征 (Receiver operating characteristic, ROC) 曲线和ROC曲线下的面积 (Area under the ROC curve, AUC) 也可以用于度量分类模型的好坏。MGCNN的ROC曲线和AUC如图10所示。可以看出,所提方法的AUC达到98.88%,在梭子蟹性别分类任务中取得优异的性能。
2.5 单幅图像预测结果
为了验证模型的实际应用效果,采购雌、雄梭子蟹各20只,在实验室对其性别进行自动识别验证。验证图像采用等比例调整大小并填充的预处理方式。图11展示了单幅图像预测的示例,可以看出预测均正确,且预测概率均超过95%。此外,经统计分析,识别一张图像的时间不超过1 s。因此,本研究所提的算法可以很好地应用在梭子蟹性别自动分类及识别系统。
3. 小结
为了实现梭子蟹性别的智能化识别,促进现代渔业分拣装备由半机械化、机械化走向智能化,本文构建了梭子蟹性别分类数据集 (PGCD),提出了一种用于梭子蟹性别分类的多组卷积神经网络框架。该网络首先通过引入ResNet对图像块提取特征,减少特征提取过程的信息丢失,使得特征提取更有力;然后提出一种注意力机制代替传统的池化层,从而更专注地找出输入数据的有用信息;最后进行了一系列的参数调整,使得所提的MGCNN拥有最优分类性能。实验结果表明,所提方法在PGCD数据集上具有分类优越性,分类准确率高达95.59%。未来,将围绕梭子蟹的尺寸、肥瘦、蟹钳完整程度等特征进行分类,进一步完善梭子蟹智能识别系统。
-
表 1 各采样点检测物种序列数
Table 1 Number of reads detected for each species at each sampling station
种类 Species 调查站位 Sampling station 德庆 DQ 封开 FK 桂平 GP 九江 JJ 藤县 TX 肇庆 ZQ 1 鲤形目 Cypriniformes 43 207 266 105 81 198 1.1鲤科 Cyprinidae 43 207 216 103 57 198 1.1.1草鱼 Ctenopharyngodon idella 20 0 5 34 2 5 1.1.2鲢 Hypophthalmichthys molitrix 9 135 57 31 31 43 1.1.3鳙 Aristichthys nobilis 0 0 0 0 0 2 1.1.4赤眼鳟 Squaliobarbus curriculus 0 15 2 14 13 1 1.1.5鲤 Cyprinus carpio 6 21 38 3 1 20 1.1.6鲫 Carassius auratus 0 0 0 5 0 2 1.1.7宽鳍鱲 Zacco platypus 0 0 0 13 0 0 1.1.8红鳍原鲌 Cultrichthys erythropterus 0 0 1 0 0 0 1.1.9银飘鱼 Pseudolaubuca sinensis 0 2 0 0 0 0 1.1.10南方拟䱗 Pseudohemiculter dispar 0 0 2 0 0 0 1.1.11黄尾鲴 Xenocypris davidi 0 0 0 0 0 18 1.1.12银鲴 Xenocypris argentea 0 0 0 0 0 35 1.1.13南方波鱼 Rasbora steineri 8 34 98 1 10 1 1.1.14马口鱼 Opsariichthys bidens 0 0 13 0 0 0 1.1.15南方白甲鱼 Onychostoma gerlachi 0 0 0 0 0 16 1.1.16银鮈 Squalidus argentatus 0 0 0 0 0 55 1.1.17麦瑞加拉鲮▲ Cirrhinus mrigala 0 0 0 2 0 0 1.2花鳅科 Cobitidae 0 0 50 2 24 0 1.2.1大鳞副泥鳅 Paramisgurnus dabryanus 0 0 50 2 6 0 1.2.2美丽沙鳅 Botia pulchra 0 0 0 0 18 0 2脂鲤目 Characiformes 0 0 0 11 0 8 2.1鲮脂鲤科 Prochilodontidae 0 0 0 11 0 8 2.1.1条纹鲮脂鲤▲ Prochilodus lineatus 0 0 0 11 0 8 3鲈形目 Perciformes 20 117 99 148 197 615 3.1攀鲈科 Anabantidae 2 0 0 0 0 9 3.1.1攀鲈 Anabas testudineus 2 0 0 0 0 9 3.2鳢科 Channidae 0 21 0 0 0 0 3.2.1斑鳢 Channa maculata 0 21 0 0 0 0 3.3狼鲈科 Moronidae 0 24 0 0 0 0 3.3.1花鲈 Lateolabrax japonicus 0 24 0 0 0 0 3.4塘鳢科 Eleotridae 0 0 0 3 0 0 3.4.1尖头塘鳢 Eleotris oxycephala 0 0 0 3 0 0 3.5鰕虎科 Gobiidae 7 50 1 104 177 386 3.5.1子陵吻鰕虎鱼 Rhinogobius giurinus 7 50 1 104 177 386 3.6丽鱼科 Cichlidae 11 22 98 41 20 220 3.6.1齐氏罗非鱼▲ Oceochromis zillii 0 0 0 0 0 190 3.6.2尼罗罗非鱼▲ Oceochromis nilotica 11 22 98 41 20 30 4鲇形目 Siluriformes 54 209 88 59 24 64 4.1鲿科 Bagridae 54 209 88 59 24 64 4.1.1黄颡鱼 Pelteobagrus fulvidraco 0 0 14 0 0 0 4.1.2粗唇鮠 Leiocassis crassilabris 0 0 0 0 1 25 4.1.3瓦氏黄颡鱼 Pelteobagrus vachellii 54 209 74 59 23 39 注:▲. 外来物种。 Note: ▲. Alien species. 表 2 各样本Alpha多样性指数
Table 2 Alpha diversity index of each sample
样本
SampleChao1指数
Chao1香农指数
Shannon辛普森指数
Simpson覆盖度
Coverage德庆 DQ 8.00 2.38 0.73 1.000 0 封开 FK 10.00 2.54 0.76 1.000 0 桂平 GP 13.33 2.89 0.84 0.995 6 九江 JJ 14.00 2.90 0.82 0.996 9 藤县 TX 11.50 2.15 0.63 0.993 4 肇庆 ZQ 18.33 2.73 0.75 0.997 7 -
[1] CHEN Y S, QU X, XIONG F Y, et al. Challenges to saving China's freshwater biodiversity: fishery exploitation and landscape pressures[J]. Ambio, 2020, 49(4): 926-938. doi: 10.1007/s13280-019-01246-2
[2] HE Y F, WANG J W, LEK S, et al. Structure of endemic fish assemblages in the upper Yangtze River Basin[J]. River Res Appl, 2011, 27(1): 59-75. doi: 10.1002/rra.1339
[3] REID A J, CARLSON A K, CREED I F, et al. Emerging threats and persistent conservation challenges for freshwater biodiversity[J]. Biol Rev, 2019, 94(3): 849-873. doi: 10.1111/brv.12480
[4] COULTHARD S, JOHNSON D, MCGREGOR J A. Poverty, sustainability and human wellbeing: a social wellbeing approach to the global fisheries crisis[J]. Global Environ Chang, 2011, 21(2): 453-463. doi: 10.1016/j.gloenvcha.2011.01.003
[5] DUDGEON D. Multiple threats imperil freshwater biodiversity in the Anthropocene[J]. Curr Biol, 2019, 29(19): 960-967. doi: 10.1016/j.cub.2019.08.002
[6] BONAR S A, MERCADO-SILVA N, HUBERT W A, et al. Standard methods for sampling freshwater fishes: opportunities for international collaboration[J]. Fisheries, 2017, 42(3): 150-156. doi: 10.1080/03632415.2017.1276352
[7] YAO M, ZHANG S, LU Q, et al. Fishing for fish environmental DNA: ecological applications, methodological considerations, surveying designs, and ways forward[J]. Mol Ecol, 2022, 31(20): 5132-5164. doi: 10.1111/mec.16659
[8] 秦传新, 左涛, 于刚, 等. 环境DNA在水生生态系统生物量评估中的研究进展[J]. 南方水产科学, 2020, 16(5): 123-128. doi: 10.12131/20190256 [9] 舒璐, 林佳艳, 徐源, 等. 基于环境DNA宏条形码的洱海鱼类多样性研究[J]. 水生生物学报, 2020, 44(5): 1080-1086. [10] DEINER K, BIK H M, MACHLER E, et al. Environmental DNA metabarcoding: transforming how we survey animal and plant communities[J]. Mol Ecol, 2017, 26(21): 5872-5895. doi: 10.1111/mec.14350
[11] ALICE V, PIERRE T, CLAUDE M, et al. Next-generation monitoring of aquatic biodiversity using environmental DNA metabarcoding[J]. Mol Ecol, 2015, 25(4): 929-942.
[12] QUENTIN M, LYNSEY R H, MICHAEL S, et al. The multiple states of environmental DNA and what is known about their persistence in aquatic environments[J]. Environ Sci Technol, 2022, 56(9): 5322-5333. doi: 10.1021/acs.est.1c07638
[13] PAWLOWSKI J, KELLY-QUINN M, ALTERMATT F, et al. The future of biotic indices in the ecogenomic era: integrating (e)DNA metabarcoding in biological assessment of aquatic ecosystems[J]. Sci Total Environ, 2018, 637/638(1): 1295-1310.
[14] TABERLET P, COISSAC E, HAJIBABAEI M, et al. Environmental DNA[J]. Mol Ecol, 2012, 21(8): 1789-1793. doi: 10.1111/j.1365-294X.2012.05542.x
[15] SHU L, LUDWIG A, PENG Z G. Standards for methods utilizing environmental DNA for detection of fish species[J]. Genes-Basel, 2020, 11(3): 296. doi: 10.3390/genes11030296
[16] BOHMANN K, EVANS A, GILBERT M T P, et al. Environmental DNA for wildlife biology and biodiversity monitoring[J]. Trends Ecol Evol, 2014, 29(6): 358-367. doi: 10.1016/j.tree.2014.04.003
[17] TAKAHARA T, MINAMOTO T, YAMANAKA H, et al. Estimation of fish biomass using environmental DNA[J]. PLoS One, 2012, 7(4): e35868. doi: 10.1371/journal.pone.0035868
[18] THOMSEN P F, KIELGAST J, IVERSEN L L, et al. Detection of a diverse marine fish fauna using environmental DNA from seawater samples[J]. PLoS One, 2012, 7(8): e41732. doi: 10.1371/journal.pone.0041732
[19] BELLE C C, STOECKLE B C, GEIST J. Taxonomic and geographical representation of freshwater environmental DNA research in aquatic conservation[J]. Aquat Conserv, 2019, 29(11): 1996-2009. doi: 10.1002/aqc.3208
[20] 赵明, 赵梦迪, 马春艳, 等. 环境DNA在水域生态中的研究进展[J]. 中国水产科学, 2018, 25(4): 714-720. [21] TSUJI S, TAKAHARA T, DOI H, et al. The detection of aquatic macroorganisms using environmental DNA analysis: a review of methods for collection, extraction, and detection[J]. Environ DNA, 2019, 1(2): 99-108. doi: 10.1002/edn3.21
[22] 《中国河湖大典》编纂委员会. 中国河湖大典 珠江卷[M]. 北京: 中国水利水电出版社, 2013: 1-10. [23] 张迎秋, 黄稻田, 李新辉, 等. 西江鱼类群落结构和环境影响分析[J]. 南方水产科学, 2020, 16(1): 42-52. [24] XING Y C, ZHANG C G, FAN E Y, et al. Freshwater fishes of China: species richness, endemism, threatened species and conservation[J]. Divers Distrib, 2016, 22(3): 358-370. doi: 10.1111/ddi.12399
[25] 李捷, 李新辉, 贾晓平, 等. 西江鱼类群落多样性及其演变[J]. 中国水产科学, 2010, 17(2): 298-311. [26] 李跃飞, 李新辉, 杨计平, 等. 珠江干流长洲水利枢纽蓄水后珠江鳡鱼 (Elopichthys bambusa) 早期资源现状[J]. 湖泊科学, 2015, 27(5): 917-924. [27] 帅方敏, 李新辉, 刘乾甫, 等. 珠江水系鱼类群落多样性空间分布格局[J]. 生态学报, 2017, 37(9): 3182-3192. [28] 盛强, 茹辉军, 李云峰, 等. 中国国家级水产种质资源保护区分布格局现状与分析[J]. 水产学报, 2019, 43(1): 62-80. [29] 杨君兴, 潘晓赋, 陈小勇, 等. 中国淡水鱼类人工增殖放流现状[J]. 动物学研究, 2013, 34(4): 267-280. [30] OWEN S W, CREU P, MAGDALENA G, et al. DNA metabarcoding of littoral hard-bottom communities: high diversity and database gaps revealed by two molecular markers[J]. PeerJ, 2018, 6: e4705. doi: 10.7717/peerj.4705
[31] CHEN W T, LI C, YANG J P, et al. Temporal species-level composition of larvae resources in the lower Pearl River drainage and implications for species' reproductive cycles[J]. Gene, 2021, 776: 145351. doi: 10.1016/j.gene.2020.145351
[32] 王梦, 杨鑫, 王维, 等. 基于eDNA技术的长江上游珍稀特有鱼类国家级自然保护区重庆段鱼类多样性研究[J]. 水生生物学报, 2022, 46(1): 2-16. [33] 郑慈英. 珠江鱼类志[M]. 北京: 科学出版社, 1989: 77-367. [34] 张春光, 赵亚辉. 中国内陆鱼类物种与分布[M]. 北京: 科学出版社, 2016: 210-213. [35] DIXON P. VEGAN, a package of R functions for community ecology[J]. J Veg Sci, 2003, 14(6): 927-930. doi: 10.1111/j.1654-1103.2003.tb02228.x
[36] 李捷, 李新辉, 谭细畅, 等. 广东肇庆西江珍稀鱼类省级自然保护区鱼类多样性[J]. 湖泊科学, 2009, 21(4): 556-562. doi: 10.3321/j.issn:1003-5427.2009.04.015 [37] 李跃飞, 李新辉, 谭细畅, 等. 西江肇庆江段渔业资源现状及其变化[J]. 水利渔业, 2008, 28(2): 80-83. [38] HEBERT P D, CYWINSKA A, BALL S L, et al. Biological identifications through DNA barcodes[J]. Proc R Soc B, 2003, 270(1512): 313-321. doi: 10.1098/rspb.2002.2218
[39] 蒋佩文, 李敏, 张帅, 等. 基于线粒体COI和12S rDNA基因构建珠江河口鱼类DNA宏条形码数据库[J]. 南方水产科学, 2022, 18(3): 13-21. [40] 郜星晨, 姜伟. 三峡库区常见鱼类DNA条形码本地BLAST数据库的构建和应用[J]. 基因组学与应用生物学, 2021, 40(5/6): 1952-1960. [41] 乐佩琦. 中国动物志. 硬骨鱼纲 鲤形目 中卷[M]. 北京: 科学出版社, 2000: 1-16. [42] 刘亚秋, 李新辉, 李跃飞, 等. 西江广东鲂 (Megalobrama terminalis) 繁殖生物学及繁殖策略[J]. 湖泊科学, 2021, 33(1): 232-241. [43] GOUTTE A, MOLBERT N, GUÉRIN S, et al. Monitoring freshwater fish communities in large rivers using environmental DNA metabarcoding and a long-term electrofishing survey[J]. J Fish Biol, 2020, 97(2): 444-452. doi: 10.1111/jfb.14383
[44] LAMY T, PITZ K J, CHAVEZ F P, et al. Environmental DNA reveals the fine-grained and hierarchical spatial structure of kelp forest fish communities[J]. Sci Rep-UK, 2021, 11(1): 14439. doi: 10.1038/s41598-021-93859-5
[45] CIVADE R, DEJEAN T, VALENTINI A, et al. Spatial representativeness of environmental DNA metabarcoding signal for fish biodiversity assessment in a natural freshwater system[J]. PLoS One, 2016, 11(6): e0157366.
[46] RUPERT A C, JUDITH B, OWEN S W, et al. Non-specific amplification compromises environmental DNA metabarcoding with COI[J]. Methods Ecol Evol, 2019, 10(11): 1985-2001. doi: 10.1111/2041-210X.13276
[47] 刘军, 赵良杰, 凡迎春, 等. 鱼类环境DNA研究中通用引物的筛选验证[J]. 淡水渔业, 2016, 46(1): 9-17. [48] MACKENZIE D I, NICHOLS J D, LACHMAN G B, et al. Estimating site occupancy rates when detection probabilities are less than one[J]. Ecology, 2002, 83(8): 2248-2255. doi: 10.1890/0012-9658(2002)083[2248:ESORWD]2.0.CO;2
[49] GU W D, SWIHART R K. Absent or undetected? Effects of non-detection of species occurrence on wildlife–habitat models[J]. Biol Conserv, 2004, 116(2): 195-203. doi: 10.1016/S0006-3207(03)00190-3
[50] GOTELLI N J, COLWELL R K. Estimating species richness[M]//MAGURRAN A E, McGILL B J. Frontiers in measuring biodiversity. New York: Oxford University Press, 2011: 39-54.
[51] CRISTESCU M E, HEBERT P D N. Uses and misuses of environmental DNA in biodiversity science and conservation[J]. Annu Rev Ecol Evol S, 2018, 49(1): 209-230. doi: 10.1146/annurev-ecolsys-110617-062306
[52] ZOU K S, CHEN J W, RUAN H T, et al. eDNA metabarcoding as a promising conservation tool for monitoring fish diversity in a coastal wetland of the Pearl River Estuary compared to bottom trawling[J]. Sci Total Environ, 2020, 702: 134704. doi: 10.1016/j.scitotenv.2019.134704
[53] ZHANG S N, ZHAO J D, YAO M. A comprehensive and comparative evaluation of primers for metabarcoding eDNA from fish[J]. Methods Ecol Evol, 2020, 11(12): 1609-1625. doi: 10.1111/2041-210X.13485
[54] SHIN-ICHIRO O, HIDEYUKI D, KEI M, et al. Environmental DNA metabarcoding for biodiversity monitoring of a highly diverse tropical fish community in a coral reef lagoon: estimation of species richness and detection of habitat segregation[J]. Environ DNA, 2021, 3(1): 55-69. doi: 10.1002/edn3.132
[55] 熊美华, 杨志, 胡兴坤, 等. 长江中游监利江段鱼类群落结构研究[J]. 长江流域资源与环境, 2019, 28(9): 2109-2118. [56] 武智, 李跃飞, 朱书礼, 等. 基于渔业声学调查的珠江东塔产卵场鱼类栖息地适宜性研究[J]. 南方水产科学, 2023, 19(3): 11-18. [57] 蒋佩文, 李敏, 张帅, 等. 基于环境DNA宏条码和底拖网的珠江河口鱼类多样性[J]. 水生生物学报, 2022, 46(11): 1701-1711. [58] 谭细畅, 李新辉, 李跃飞, 等. 尼罗罗非鱼早期发育形态及其在珠江水系的空间分布[J]. 生物安全学报, 2012, 21(4): 295-299. [59] 顾党恩, 牟希东, 罗渡, 等. 广东省主要水系罗非鱼的建群状况[J]. 生物安全学报, 2012, 21(4): 277-282. [60] TONY D, ALICE V, ANTOINE D, et al. Persistence of environmental DNA in freshwater ecosystems[J]. PLoS One, 2012, 6(8): e23398.
[61] FICETOLA G F, MIAUD C, POMPANON F, et al. Species detection using environmental DNA from water samples[J]. Biol Lett, 2008, 4(4): 423-425. doi: 10.1098/rsbl.2008.0118
[62] HOWALD G, DONLAN C J, GALVAN J P, et al. Invasive rodent eradication on islands[J]. Conserv Biol, 2007, 21(5): 1258-1268. doi: 10.1111/j.1523-1739.2007.00755.x
[63] BOOTHROYD M, MANDRAK N E, FOX M, et al. Environmental DNA (eDNA) detection and habitat occupancy of threatened spotted gar (Lepisosteus oculatus)[J]. Aquat Conserv, 2016, 26(6): 1107-1119. doi: 10.1002/aqc.2617
[64] JANOSIK A M, JOHNSTON C E. Environmental DNA as an effective tool for detection of imperiled fishes[J]. Environ Biol Fish, 2015, 98(8): 1889-1893. doi: 10.1007/s10641-015-0405-5
[65] 甘西, 蓝家湖, 吴铁军, 等. 中国南方淡水鱼类原色图鉴[M]. 郑州: 河南科学技术出版社, 2017: 75. [66] VALDIVIA-CARRILLO T, ROCHA-OLIVARES A, REYES-BONILLA H, et al. Integrating eDNA metabarcoding and simultaneous underwater visual surveys to describe complex fish communities in a marine biodiversity hotspot[J]. Mol Ecol Resour, 2021, 21(5): 1558-1574. doi: 10.1111/1755-0998.13375
[67] CILLEROS K, VALENTINI A, ALLARD L, et al. Unlocking biodiversity and conservation studies in high-diversity environments using environmental DNA (eDNA): a test with Guianese freshwater fishes[J]. Mol Ecol Resour, 2019, 19(1): 27-46. doi: 10.1111/1755-0998.12900
-
其他相关附件
-
PDF格式
20230111附录A 点击下载(649KB)
-



 下载:
下载:



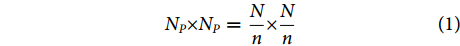
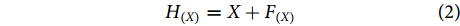
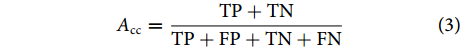
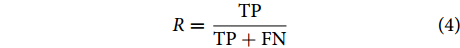
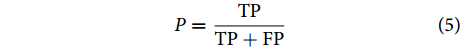


 粤公网安备 44010502001741号
粤公网安备 44010502001741号
